中国科学院微生物研究所、中国微生物学会主办
文章信息
- 霍亚楠, 吴凤礼, 宋国田, 涂然, 陈五九, 花尔并, 王钦宏
- Huo Yanan, Wu Fengli, Song Guotian, Tu Ran, Chen Wujiu, Hua Erbing, Wang Qinhong
- 定向进化改造酪氨酸解氨酶提高大肠杆菌合成对香豆酸产量
- Directed evolution of tyrosine ammonia-lyase to improve the production of p-coumaric acid in Escherichia coli
- 生物工程学报, 2020, 36(11): 2367-2376
- Chinese Journal of Biotechnology, 2020, 36(11): 2367-2376
- 10.13345/j.cjb.200122
-
文章历史
- Received: March 14, 2020
- Accepted: April 21, 2020
2. 中国科学院天津工业生物技术研究所 中国科学院系统微生物工程重点实验室, 天津 300308
2. CAS Key Laboratory of Systems Microbial Biotechnology, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
对香豆酸(p-coumaric acid),又名对羟基肉桂酸,在植物中广泛存在,是一种天然酚类化合物,具有多种药理活性,包括预防心血管疾病、抗氧化、抗菌消炎、保护心脏、利肝利胆、及抗病毒等功效[1-4]。因此,对香豆酸广泛应用于食品、化妆品、医药等领域。此外,对香豆酸也是咖啡酸、阿魏酸、柚皮素、香豆素、白藜芦醇等天然产物的合成前体[5-6],是一种重要的有机化工原料。目前,对香豆酸的生产方式有植物提取、化学合成和生物合成。植物提取法具有生长周期长、收益率低、受环境影响大、提取成本高等缺点[7]。化学合成生产对香豆酸具有能耗高、副产物多、产率低、高污染等问题。生物合成法具有低成本、高产率、可持续、绿色环保等优势,所以利用微生物合成技术生产对香豆酸近年来受到广泛关注[8]。
生物体中对香豆酸有两条不同的生物合成途径(图 1)。其中一条途径是由苯丙氨酸解氨酶(Phenylalanine ammonia-lyase,PAL)催化苯丙氨酸(L-phenylalanine)脱氨基生成肉桂酸(trans- cinnamic acid),再经肉桂酸4-羟化酶(Cinnamate 4-hydroxylase,C4H)催化肉桂酸生成对香豆酸。C4H仅通过与内质网膜结合才能发挥作用,在大肠杆菌等原核生物中很难实现活性表达[9]。另一条途径是以酪氨酸(L-tyrosine)为底物,在酪氨酸解氨酶(Tyrosine ammonia-lyase,TAL)作用下催化酪氨酸直接生成对香豆酸。该合成途径具有反应路径短、催化效率高等优点,因此通常采用这条路径在微生物中合成对香豆酸[10-12]。Cui等[13]比较糖丝菌Saccharothrix sp. NRRL B-16348和链霉菌Streptomyces sp. NRRL F-4489来源的酪氨酸解氨酶Sas-TAL和Sts-TAL发现,Sts-TAL的催化活力更高;利用表达Sts-TAL的大肠杆菌进行全细胞催化转化酪氨酸,对香豆酸的合成速率达到2.88 g/(L·h)。梁景龙等[14]对粘红酵母Rhodotorula glutinis RgTAL与RgPAL的蛋白序列进行比对发现,两者同源性为74%;经过同源建模得到RgTAL的蛋白结构为同源四聚体,二级结构主要是α螺旋,催化位点有4个,分别为Ala217、Ser218、Gly219、His143;Ala217-Ser218-Gly219所形成的MIO结构与L-酪氨酸上的氨基进行对接反应,确定His143是决定酪氨酸解氨酶具有活性的关键位点[15]。
|
| 图 1 对香豆酸生物合成途径示意图 Fig. 1 The biosynthetic pathway of p-coumaric acid.PPP: pentose phosphate pathway; GalP-Glk: galactose permease-glucokinase system; PTS–: inactivated phosphotransferase system; AroF/AroG/AroH: 3-deoxy-D-arabino-heptulosonate-7-phosphate synthase; TAL: tyrosine ammonia-lyase; PAL: phenylalanine ammonia-lyase; C4H: cinnamate 4-hydroxylase; G6P: glucose-6-phosphate; E4P: D-erythrose 4-phosphate; PEP: phosphoenolpyruvate; DAHP: 3-deoxy-D-arabino-heptulosonate-7-phosphate; CHA: chorismite. |
| |
通过微生物从头合成对香豆酸已有报道,但是对香豆酸产量较低。Vannelli等[16]将来源于粘红酵母的tal基因以质粒的形式在产苯丙氨酸的大肠杆菌Escherichia coli ATCC 31884菌株中用IPTG进行诱导表达,实现了以葡萄糖为原料,从头合成大约150 μmol/L对香豆酸(约24.6 mg/L)。随后该研究组又在大肠杆菌中诱导表达了丝孢酵母菌Trichosporon cutaneum的tal基因,在培养液上清中检测到了71–79 mg/L的对香豆酸[17]。对香豆酸产量较低,一方面是可能由于对香豆酸具有细胞毒性,抑制细胞生长;另一方面可能是由于TAL酶催化活性较低,导致转化率较低。
为了解决TAL催化活性低的问题,本研究利用大肠杆菌作为底盘细胞,通过构建组成型表达粘红酵母tal基因随机突变体文库,结合高通量筛选技术获得TAL酶活性显著提高的突变体,并对突变体进行突变验证,最终TAL高活性突变体提高了大肠杆菌合成对香豆酸的产量。
1 材料与方法 1.1 菌株与质粒菌株Escherichia coli BL21(DE3)、CP032和质粒pET-30a均为作者所在实验室保藏。
1.2 培养基LB培养基:10 g/L胰蛋白胨,5 g/L酵母提取物,10 g/L NaCl,固体培养基添加15 g/L琼脂粉。突变体筛选培养基[18]:2 g/L酪蛋氨基酸,6.8 g/L Na2HPO4,3 g/L KH2PO4,1 g/L NH4Cl,0.5 g/L NaCl,20 g/L无水葡萄糖,1 mmol/L MgSO4,0.5 μg/mL硫胺素,0.5 g/L L-酪氨酸。摇瓶发酵所用NBS培养基[19]:3.5g/L KH2PO4,6.5 g/L K2HPO4·3H2O,3.5 g/L (NH4)2HPO4,0.25 g/L MgSO4·3H2O,15 mg/L CaCl2·2H2O,0.5 mg/L硫胺素,1 mL微量元素,20 g/L无水葡萄糖。用0.1 mol/L HCl配置微量元素(1 000×液):1.6 g/L FeCl3,0.2 g/L CoCl2·6H2O,0.1 g/L CuCl2,0.2 g/L ZnCl2·4H2O,0.2 g/L Na2MoO4·2H2O,0.05 g/L H3BO3。
1.3 易错PCR流程根据已报道粘红酵母tal基因序列(GenBank登录号:KF765779.1)[16],通过全基因合成的方式获得tal模板序列。然后利用带有BamHⅠ和Hind Ⅲ酶切位点的引物TAL-F/TAL-R (5′-GGAT CCATGGCGCCGCGCCCG/AAGCTTTTATGCCAGCATCTTCA-3′),通过易错PCR扩增tal随机突变序列。反应体系为:10×Taq PCR缓冲液5 μL,dNTPs (2.5 mmol/L each) 4 μL,上下游引物各1 μL,模板50 ng,10 mmol/L MnCl2 0.5 μL,25 mmol/L MgCl2 5 μL,rTaq DNA polymerase (5 U/μL) 0.5 μL,补充超纯水至总体积50 μL。PCR反应条件:94 ℃ 5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 2 min,30个循环;72 ℃ 5 min。PCR结束后,利用DNA胶回收试剂盒回收tal片段,以回收的DNA产物为模板,再进行一轮易错PCR扩增,扩增条件同上,再次胶回收即获得tal随机突变文库。
1.4 TAL突变体文库的构建将大肠杆菌M1-93组成型强启动子序列[20]首先连接到pET-30a载体上的XbaⅠ和BamHⅠ之间,形成中间载体pET30a-M1-93。然后将易错PCR扩增得到的tal随机突变文库序列插入到pET30a-M1-93载体上的BamHⅠ和Hind Ⅲ酶切位点中间,转化E. coli BL21(DE3)菌株,涂布于含有50 μg/mL卡那霉素(Kan)的LB平板上,37 ℃过夜培养至形成单菌落,获得含有pET30a-tal质粒的大肠杆菌突变体文库。
1.5 对香豆酸标准曲线的测定利用对香豆酸在310 nm处有特异性吸收峰的性质,绘制对香豆酸标准曲线。对香豆酸标准品浓度依次为12.5 mg/L、25 mg/L、50 mg/L、100 mg/L、200 mg/L、400 mg/L。以对香豆酸浓度为横坐标,310 nm吸光度值为纵坐标,绘制标准曲线。标准曲线线性方程为y=0.007 3x+0.001 4,R2=0.999 5。
1.6 TAL突变体的筛选TAL突变体文库高通量初步筛选:从大肠杆菌TAL突变体菌落文库中随机挑取一定数量的单菌落于每孔装有200 μL LB+50 μg/mL Kan培养基的96孔深孔细胞培养板中,以含有空载体pET30a-M1-93的BL21(DE3)菌株作为空白对照,在37℃、800 r/min摇床中培养16 h作为种子液。然后每孔吸取20 μL种子液,转移至新的每孔装有500 μL筛选培养基+50 μg/mL Kan培养基的96孔深孔细胞培养板中,28 ℃、800 r/min继续培养6 h。将深孔板4 000 r/min离心5 min,取20 μL上清液于180 μL去离子水中,用多功能酶标仪检测310 nm处的吸光度。根据对香豆酸标准曲线计算对香豆酸产量。
TAL文库突变体复筛和氨基酸点突变菌株的筛选:挑取单菌落到含有3 mL LB+50 μg/mL Kan培养基的试管中,37 ℃、250 r/min培养12 h作为种子液。将种子液按4%接种量接种到含有5 mL筛选培养基+50 μg/mL Kan培养基的大试管中(25 mm×200 mm),28 ℃、250 r/min培养6 h。取500 μL发酵液,10 000 r/min离心2 min,取20 μL上清液于180 μL去离子水中,用多功能酶标仪检测并计算对香豆酸产量。
1.7 氨基酸单点饱和突变体和双位点组合突变体的构建以pET30a-tal质粒为模板,利用在突变点处设计的一对反向互补的引物,扩增质粒全长序列,然后通过平末端连接的方式构建相应氨基酸的点突变体。在一种单点突变的基础上,继续突变另外一种氨基酸位点即可形成双位点组合突变体。最后转入E. coli BL21(DE3)菌株进行对香豆酸产量检测。
1.8 摇瓶发酵评价对香豆酸的产量将突变体质粒和原始TAL质粒分别转入实验室前期构建的酪氨酸高产菌株CP032中,挑取单菌落到含有3 mL LB+50 μg/mL Kan培养基的试管中,37 ℃、250 r/min培养12 h作为种子液。将种子液按1%接种量接种到含有10 mL NBS+ 50 μg/mL Kan培养基的100 mL三角瓶中,37 ℃、250 r/min培养48 h,每隔12 h取适量发酵液进行对香豆酸产量检测。
2 结果与分析 2.1 随机突变体文库的构建易错PCR技术是酶定向进化研究中广泛使用的一种技术手段,成功应用于多种酶的催化活性改造研究[21-23]。为了提高TAL的催化活性,利用易错PCR技术对tal基因进行随机突变,构建随机突变体文库,通过组成型强启动子M1-93[19]在pET30a载体上组成型表达tal基因,筛选TAL催化活性提高的突变体。
首先,对易错PCR体系中Mn2+和Mg2+浓度进行优化,既能保证突变率,又能获得足够数目的转化子。通过实验比较分析,最终确定Mg2+浓度为2.5 mmol/L,Mn2+浓度为0.1 mmol/L。每条tal序列上有5–7个碱基突变,且编码氨基酸序列发生突变的序列占总数的90%以上。因此,所得文库可用于突变体筛选。
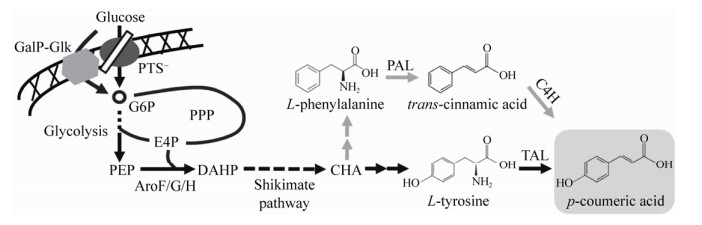
2.2 高通量筛选体系的优化HPLC检测对香豆酸产量的方法难以进行大规模的文库筛选。为了解决筛选通量的问题,本研究建立了高通量筛选方法,即利用96孔深孔细胞板培养菌体,通过酶标仪检测对香豆酸含量。由图 2A可以看出,当培养基中不含有对香豆酸时,310 nm处吸光度较低;当加入对香豆酸后,310 nm的吸光度明显升高(图 2B)。因此,通过检测310 nm测吸光度来表征培养基上清液中对香豆酸含量的方法是可行的。含有空载质粒pET30a-M1-93的大肠杆菌不能合成对香豆酸,其扫描图谱和培养基的扫描图谱一致。另外,为了验证HPLC与OD310检测结果的一致性,对不同浓度对香豆酸的HPLC与OD310检测值进行了拟合,由拟合曲线(图 2C)看出HPLC与OD310的检测结果拟合度较好,简便快速的OD310检测应该可以替代复杂费时的HPLC检测。
|
| 图 2 NBS培养基不同波长吸收值及对香豆酸OD310与HPLC检测拟合曲线(A:不含对香豆酸的培养基不同波长吸收值;B:含75 mg/L对香豆酸的培养基不同波长吸收值;C:OD310与HPLC检测拟合曲线) Fig. 2 Absorbance of NBS media under different wavelength and fit curve of p-coumaric acid via HPLC and OD310 detection. (A) Absorbance of NBS media without p-coumaric acid. (B) Absorbance of NBS media with 75 mg/L p-coumaric acid. (C) The fit curve of p-coumaric acid via HPLC and OD310 detection at 0, 1.56, 3.13, 6.25, 12.5, 15.6, 31.25, 50, 62.5, 100, 125, 200, 250 and 400 mg/L, respectively. |
| |
为了确保文库筛选的稳定性和可靠性,对高通量筛选方法进行优化。首先,优化种子液的培养时间。当种子液培养16 h时,种子液的OD600值达到最大值;继续培养,OD600值开始降低,可能会导致种子液细胞活性降低(图 3A)。因此,种子液培养时间选为16 h。其次,优化发酵培养时间。当发酵培养6 h时即可达到较高的香豆酸产量,菌株之间差异性较小;继续培养,产量提升不明显,且结果偏差逐渐增大(图 3B)。因此,发酵培养时间设定为6 h。

|
| 图 3 高通量筛选体系培养时间的优化(A:种子液培养时间的优化;B:发酵培养时间的优化) Fig. 3 Optimization of cultivation time for high-throughput screening system. (A) Optimization of seed cultivation time. (B) Optimization of fermentation time.All data and standard errors were derived from three independent biological replicates. |
| |
利用上述高通量筛选体系,对构建的TAL随机突变体文库进行筛选,获得正向突变的高催化活性TAL突变体。第一轮共筛选了10 000个突变体,初步筛选到9个对香豆酸产量提高大约1倍的菌株(MT-71E1、MT-71H1、MT73E1、MT73F1、MT73G1、MT73H1、MT74F1、MT74H1、MT75H1),并且这些突变体的OD600值与WT-TAL基本一致(图 4A)。

|
| 图 4 TAL突变体菌株的对香豆酸产量和OD600 (A:TAL突变体菌株对香豆酸产量和OD600的初筛检测;B:TAL突变体菌株对香豆酸产量和OD600的复筛检测) Fig. 4 p-coumaric acid titers and OD600 of TAL random mutants. (A) p-coumaric acid titers and OD600 of TAL random mutants in first round of screening. (B) p-coumaric acid titers and OD600 of TAL random mutants in second round of screening. All data and standard errors in (B) were derived from three independent biological replicates. |
| |
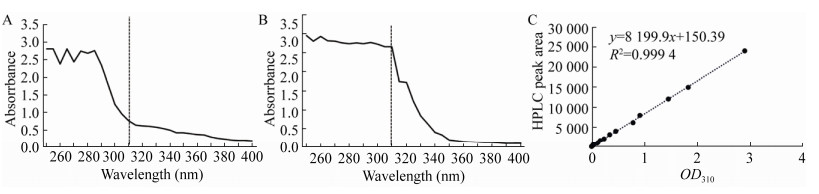
由于大规模筛选经常存在假阳性结果。为了进一步确认上述9个突变体的催化活性,对这些突变株进行复筛检测。通过检测,其中4个菌株(MT73E1、MT73F1、MT73G1、MT73H1)的对香豆酸产量仍然是对照菌株WT-TAL的2倍以上,分别为99.5 mg/L、103.8 mg/L、96.9 mg/L、108.2 mg/L;并且所有菌株的OD600值无明显差异(图 4B)。该结果进一步证明这4个TAL突变体的催化活性与WT-TAL相比得到显著提高。
对上述4个正向突变体进行测序和序列比对分析,发现这4个突变体的蛋白序列完全一致,包含3个氨基酸突变位点,分别为S9Y、A11N、E518A。分析原因可能是:(1)挑取单克隆的时候存在交叉污染,因为这4个突变体菌株在深孔板上的位置相邻;(2)在细胞转化时,复苏培养时间过长,细胞分裂次数过多,导致该突变体出现多个克隆;(3)在易错PCR过程中,可能是该突变发生后,又作为模板扩增了若干循环,导致该突变体数目较多,但是这种假设存在可能性较低。以上结果表明,这3个氨基酸位点突变可能是导致TAL催化活力提高的直接原因,但无法确定是哪个位点氨基酸突变导致酶活力发生变化。
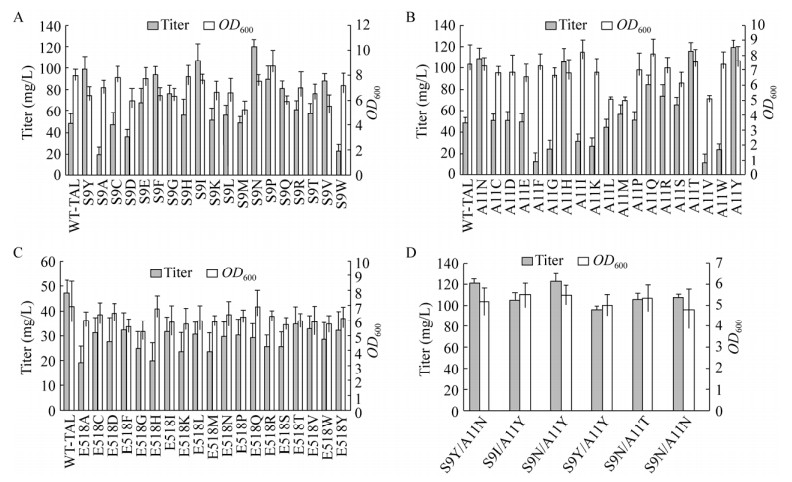
2.4 突变位点氨基酸的单点饱和突变和双位点组合突变对TAL催化活性的影响为了进一步验证TAL催化活力的提高是哪个位点氨基酸突变导致的,以及突变为其他氨基酸对酶催化活力的影响,对上述3个突变位点分别进行单点饱和突变和组合突变验证。通过单点饱和突变验证,当S9和A11位点发生突变时,对香豆酸产量发生显著升高或降低,表明这2个位点氨基酸突变可以显著影响TAL催化活力;当E518位点发生突变时,所有氨基酸类型突变体的对香豆酸产量均显著降低,表明该位点氨基酸突变会显著降低TAL催化活力,并且该位点原始氨基酸即为最佳氨基酸。其中,当S9位点突变为Y、I、N时,对香豆酸产量最高,分别为98.6 mg/L、107.1 mg/L、120.6 mg/L;而A11位点突变为N、T、Y时,对香豆酸产量最高,分别为109.1 mg/L、116.04 mg/L、119.3 mg/L;这2个位点均包含突变体文库筛选所获得的突变类型9Y和11N (图 5 A–C)。此外,这3个位点氨基酸突变导致TAL催化活力发生变化,最终会影响细胞的OD600值,并且对香豆酸产量与OD600值呈正相关关系(图 5 A–C)。
|
| 图 5 氨基酸点突变体和组合突变体的对香豆酸产量和OD600 (A:S9位点饱和突变体库的检测;B:A11位点饱和突变体库的检测;C:E518位点饱和突变体库的检测;D:S9和A11位点组合突变体的检测) Fig. 5 p-coumaric acid titers and OD600 of single and combinatorial TAL mutants. (A) Detection of S9 single saturated mutants. (B) Detection of A11 single saturated mutants. (C) Detection of E518 single saturated mutants. (D) Detection of S9 and A11 combinatorial mutants. All data and standard errors in (B) were derived from three independent biological replicates. |
| |
为了验证S9和A11位3种类型的组合突变对TAL催化活力的影响,分别构建这些位点的组合突变体,共获得6种突变体,分别为S9Y/A11N、S9I/A11Y、S9N/A11Y、S9Y/A11Y、S9N/A11T、S9N/A11N。通过组合突变验证,S9Y/A11N(同突变体文库筛选时得到的组合)和S9N/A11Y突变体的对香豆酸产量显著高于其他类型突变体,产量分别达121.2 mg/L和122.3 mg/L,并且该产量略高于单点突变体的产量(图 5D)。该结果表明S9Y/A11N和S9N/A11Y这2种组合突变确实可以提高TAL催化活力。
2.5 利用S9N/A11Y突变体提高对香豆酸发酵产量为了验证筛选的最佳TAL突变体S9N/A11Y是否能够提高对香豆酸从头合成的产量,将S9N/A11Y突变体质粒转入实验室前期构建的酪氨酸高产菌株CP0032中,以原始WT-TAL质粒作为对照。通过摇瓶发酵评价HPLC检测,S9N/A11Y突变体菌株在48 h时的对香豆酸产量达到394.2 mg/L,是对照WT-TAL菌株产量(180.5 mg/L)的2.2倍;这两个菌株的OD600无明显差别(图 6)。该结果进一步证明利用筛选的高催化活性TAL突变体可以进一步提高大肠杆菌从头合成对香豆酸的产量。

|
| 图 6 大肠杆菌CP032 S9N/A11Y和CP032 WT-TAL菌株的摇瓶发酵评价 Fig. 6 Shake flask fermentation of E. coli CP032 S9N/A11Y and CP032 WT-TAL strains.Filled circle with solid line: p-coumaric acid of S9N/A11Y; filled square with solid line: p-coumaric acid of WT-TAL; empty circle with dashed line: OD600 of S9N/A11Y; empty square with dashed line: OD600 of WT-TAL. All data and standard errors were derived from three independent biological replicates. |
| |
对香豆酸是一种具有多种药理活性和应用价值的天然酚类化合物。通过微生物合成生产对香豆酸相对于传统的化学合成和植物提取工艺来说具有许多优点。目前,已报道对香豆酸的微生物合成主要通过TAL催化酪氨酸脱氨基转化而来[16-17]。但是,这些研究报道的对香豆酸产量较低,可能是因为TAL酶催化活性较低。
为了解决TAL酶活性低的问题,本研究通过易错PCR技术对粘红酵母tal基因进行了随机突变,利用建立的高通量筛选方法从随机突变体文库中筛选了大约10 000个突变体。通过初筛和复筛验证,获得了4株TAL催化活性显著提高的突变体。但是通过测序和序列比对分析,发现这4个突变体的蛋白序列完全一致,包含3个氨基酸突变位点,分别为S9Y、A11N、E518A。通过分析,原因可能是挑取单克隆的时候存在交叉污染,或细胞转化时复苏培养时间过长,或易错PCR扩增的原因。不过可以肯定的是,这些位点突变确实提高了TAL的催化活性,导致对香豆酸产量提高。
为了进一步验证TAL催化活力的提高是哪个位点氨基酸突变导致的,对这3个突变位点氨基酸分别进行饱和突变验证。研究发现,当S9位点突变为Y、I、N和A11位点突变为N、T、Y时,TAL的催化活性显著提高,而当E518位点发生突变时,所有氨基酸类型突变体的催化活性均显著降低。进一步对这2个位点3种类型突变进行组合突变验证,发现S9Y/A11N和S9N/A11Y两种组合突变的TAL催化活力显著高于其他组合,并且高于单点突变。
通过TAL突变体筛选及突变验证发现,突变体的生物量与对香豆酸产量呈正相关趋势。当对香豆酸产量较高时,菌体生物量均正常;当生物量较低时,对香豆酸产量一定较低。这一结果暗示着生物量可以影响对香豆酸产量。若想获得较高的对香豆酸产量,需要获得足够多的生物量,而生物量直接影响TAL蛋白的表达水平。因此,通过实验室适应性进化手段,筛选对香豆酸耐受性较高的菌株作为宿主细胞,或许可以进一步提高对香豆酸的产量[24]。
本研究通过TAL突变体文库筛选以及单点和组合突变验证,获得TAL催化活力显著提高的突变体,该突变体可以显著提高大肠杆菌从头合成对香豆酸的产量。这一研究为微生物异源合成生产对香豆酸提供了重要参考。
| [1] |
Abdel-Wahab MH, El-Mahdy MA, Abd-Ellah MF, et al. Influence of p-coumaric acid on doxorubicin-induced oxidative stress in rat's heart. Pharmacol Res, 2003, 48(5): 461-465. DOI:10.1016/S1043-6618(03)00214-7 |
| [2] |
Ferguson LR, Zhu ST, Harriss PJ. Antioxidant and antigenotoxic effects of plant cell wall hydroxycinnamic acids in cultured HT-29 cells. Mol Nutr Food Res, 2005, 49(6): 585-593. DOI:10.1002/mnfr.200500014 |
| [3] |
Kim SI, Jeong YI, Jung ID, et al. p-coumaric acid inhibits indoleamine 2, 3-dioxygenase expression in murine dendritic cells. Int Immunopharmacol, 2007, 7(6): 805-815. DOI:10.1016/j.intimp.2007.01.020 |
| [4] |
Cheng JC, Dai F, Zhou B, et al. Antioxidant activity of hydroxycinnamic acid derivatives in human low density lipoprotein: mechanism and structure-activity relationship. Food Chem, 2007, 104(1): 132-139. DOI:10.1016/j.foodchem.2006.11.012 |
| [5] |
Taofiq O, González-Paramás AM, Barreiro MF, et al. Hydroxycinnamic acids and their derivatives: cosmeceutical significance, challenges and future perspectives, a review. Molecules, 2017, 22(2): 281. DOI:10.3390/molecules22020281 |
| [6] |
Pei K, Ou JY, Huang JQ, et al. p-coumaric acid and its conjugates: dietary sources, pharmacokinetic properties and biological activities. J Sci Food Agric, 2016, 96(9): 2952-2962. DOI:10.1002/jsfa.7578 |
| [7] |
Vannelli T, Xue ZX, Breinig S, et al. Functional expression in Escherichia coli of the tyrosine-inducible tyrosine ammonia-lyase enzyme from yeast Trichosporon cutaneum for production of p-hydroxycinnamic acid. Enzyme Microb Technol, 2007, 41(4): 413-422. DOI:10.1016/j.enzmictec.2007.03.013 |
| [8] |
Mavel S, Dikic B, Palakas S, et al. Synthesis and biological evaluation of a series of flavone derivatives as potential radioligands for imaging the multidrug resistance-associated protein 1 (ABCC1/MRP1). Bioorg Med Chem, 2006, 14(5): 1599-1607. DOI:10.1016/j.bmc.2005.10.009 |
| [9] |
Lin YH, Sun XX, Yuan QP, et al. Combinatorial biosynthesis of plant-specific coumarins in bacteria. Metab Eng, 2013, 18: 69-77. DOI:10.1016/j.ymben.2013.04.004 |
| [10] |
Santos CNS, Koffas M, Stephanopoulos G. Optimization of a heterologous pathway for the production of flavonoids from glucose. Metab Eng, 2011, 13(4): 392-400. DOI:10.1016/j.ymben.2011.02.002 |
| [11] |
Trantas E, Panopoulos N, Ververidis F. Metabolic engineering of the complete pathway leading to heterologous biosynthesis of various flavonoids and stilbenoids in Saccharomyces cerevisiae. Metab Eng, 2009, 11(6): 355-366. DOI:10.1016/j.ymben.2009.07.004 |
| [12] |
Yan YJ, Kohli A, Koffas MAG. Biosynthesis of natural flavanones in Saccharomyces cerevisiae. Appl Environ Microbiol, 2005, 71(9): 5610-5613. DOI:10.1128/AEM.71.9.5610-5613.2005 |
| [13] |
Cui PW, Zhong WH, Qin Y, et al. Characterization of two new aromatic amino acid lyases from actinomycetes for highly efficient production of p-coumaric acid. Bioprocess Biosyst Eng, 2020, 43(7): 1287-1298. DOI:10.1007/s00449-020-02325-5 |
| [14] |
Liang JL, Guo LQ, Lin JF, et al. Cloning, expression and enzymatic properties of tyrosine ammonia-lyase from Rhodotorula glutinis. J Chin Inst Food Sci Technol, 2017, 17(9): 213-219 (in Chinese). 梁景龙, 郭丽琼, 林俊芳, 等. 粘红酵母酪氨酸解氨酶基因的克隆表达与酶学性质. 中国食品学报, 2017, 17(9): 213-219. |
| [15] |
Watts KT, Mijts BN, Lee PC, et al. Discovery of a substrate selectivity switch in tyrosine ammonia-lyase, a member of the aromatic amino acid lyase family. Chem Biol, 2006, 13(12): 1317-1326. DOI:10.1016/j.chembiol.2006.10.008 |
| [16] |
Vannelli T, Wei QW, Sweigard J, et al. Production of p-hydroxycinnamic acid from glucose in Saccharomyces cerevisiae and Escherichia coli by expression of heterologous genes from plants and fungi. Metab Eng, 2007, 9(2): 142-151. |
| [17] |
Vannelli T, Xue ZX, Breinig S, et al. Functional expression in Escherichia coli of the tyrosine-inducible tyrosine ammonia-lyase enzyme from yeast Trichosporon cutaneum for production of p-hydroxycinnamic acid. Enzyme Microb Technol, 2007, 41(4): 413-422. DOI:10.1016/j.enzmictec.2007.03.013 |
| [18] |
Zhou SH, Liu PR, Chen J, et al. Characterization of mutants of a tyrosine ammonia-lyase from Rhodotorula glutinis. Appl Microbiol Biotechnol, 2016, 100(24): 10443-10452. DOI:10.1007/s00253-016-7672-8 |
| [19] |
Causey TB, Zhou S, Shanmugam KT, et al. Engineering the metabolism of Escherichia coli W3110 for the conversion of sugar to redox-neutral and oxidized products: homoacetate production. Proc Natl Acad Sci USA, 2003, 100(3): 825-832. DOI:10.1073/pnas.0337684100 |
| [20] |
Lu J, Tang JL, Liu Y, et al. Combinatorial modulation of galP and glk gene expression for improved alternative glucose utilization. Appl Microbiol Biotechnol, 2012, 93(6): 2455-2462. DOI:10.1007/s00253-011-3752-y |
| [21] |
Stephens DE, Singh S, Permaul K. Error-prone PCR of a fungal xylanase for improvement of its alkaline and thermal stability. FEMS Microbiol Lett, 2009, 293(1): 42-47. DOI:10.1111/j.1574-6968.2009.01519.x |
| [22] |
Cadwell RC, Joyce GF. Mutagenic PCR. PCR Methods Appl, 1994, 3(6): S136-S140. DOI:10.1101/gr.3.6.S136 |
| [23] |
Ling LL, Keohavong P, Dias C, et al. Optimization of the polymerase chain reaction with regard to fidelity: modified T7, Taq, and Vent DNA polymerases. PCR Methods Appl, 1991, 1(1): 63-69. |
| [24] |
Reyes LH, Gomez JM, Kao KC. Improving carotenoids production in yeast via adaptive laboratory evolution. Metab Eng, 2014, 21: 26-33. DOI:10.1016/j.ymben.2013.11.002 |
 2020, Vol. 36
2020, Vol. 36




