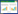中国科学院微生物研究所、中国微生物学会主办
文章信息
- 侯建屾, 高聪, 陈修来, 刘立明
- Hou Jianshen, Gao Cong, Chen Xiulai, Liu Liming
- 使用动态分子开关调控大肠杆菌生产莽草酸
- Using dynamic molecular switches for shikimic acid production in Escherichia coli
- 生物工程学报, 2020, 36(10): 2104-2112
- Chinese Journal of Biotechnology, 2020, 36(10): 2104-2112
- 10.13345/j.cjb.200078
-
文章历史
- Received: February 22, 2020
- Accepted: April 8, 2020
2. 江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122
2. Key Laboratory of Industrial Biotechnology, Ministry of Education, Jiangnan University, Wuxi 214122, Jiangsu, China
莽草酸(Shikimic Acid,SA)是植物和细菌中芳香族氨基酸生物合成途径的中间代谢物,也是抗流感药物磷酸奥司他韦(达菲)合成的中间体[1-2]。目前在大肠杆菌中合成莽草酸的策略包括中断莽草酸途径以减少消耗、增加前体物质浓度、强化生产路径等手段。例如刘向磊等通过中断莽草酸途径(敲除aroK和aroL基因)、强化生产路径(过表达基因aroB、aroE和aroG)、增加前体物质(过表达tktA和ppsA基因)等手段积累27.41 g/L莽草酸[3]。Rodriguez等通过中断莽草酸途径(敲除aroK和aroL基因),增加前体物质(过表达tktA和敲除pykF基因)同时过表达路径酶基因,生产43 g/L莽草酸[4]。以上策略面临的问题是生产莽草酸必须添加丰富的有机氮源,因为阻断莽草酸路径后,在基本培养基中细胞生长受到阻碍。而动态调控手段可以自主实现细胞生长与产物合成的分离,为莽草酸的生物合成提供了新的策略。
为了实现代谢流的动态调控,目前的转录水平的分子开关根据响应信号的不同分为3类[5],即环境回路[6] (温度,光照和pH)、细胞外回路[7] (群体感应)和细胞内回路[8-10] (感知生理状态和胞内代谢产物)。这些策略可以改变代谢状态[6],减少对细胞有害中间体积累[10]。例如,利用动态开关控制丙二酰辅酶A的供应使脂肪酸产量提高了2.1倍[8]。由于缺乏翻译后水平的调控,代谢流被定向到新的路径时,原路径的酶仍然存在于细胞中[11-13],导致转录水平调节的响应时间很长,代谢流的调节被延迟。
因此,可以将转录水平和翻译后水平的调控策略结合组建具备低能耗和响应快速的开关。在本文中,使用生长期的启动子和蛋白质降解标签[14-17]构建调控开关,并将调控开关应用到莽草酸合成中,菌株进入稳定期后动态降解莽草酸激酶并开始积累莽草酸,实现了细胞生长与产物合成之间的代谢流平衡分配。
1 材料与方法 1.1 菌种与质粒本研究使用的大肠杆菌Escherichia coli JM109和E. coli MG1655为实验室保藏,启动子Promoter J21339 (P9)的序列为TTGACAGCTAGC TCAGTCCTAGGTATAAT。本文所用质粒列于表 1。
| Strain | E. coli genotype/plasmid | Source |
| JM109 | E. coli, recA1 mcrB+ hsdR17 | Lab storage |
| rpsL-LAA | JM109, Ampr, pPMB1-PrpsL-egfp-LAA-PLac UV5-mKate2 | This study |
| rpsL-DAS | JM109, Ampr, pPMB1-PrpsL-egfp-DAS-PLac UV5-mKate2 | This study |
| rpsL-DAS+4 | JM109, Ampr, pPMB1-PrpsL-egfp-DAS+4-PLac UV5-mKate2 | This study |
| rpsL-DAS+8 | JM109, Ampr, pPMB1-PrpsL-egfp-DAS+8-PLac UV5-mKate2 | This study |
| rpsL-GSD | JM109, Ampr, pPMB1-PrpsL-egfp-GSD-PLac UV5-mKate2 | This study |
| rpsL | JM109, Ampr, pPMB1-PrpsL-egfp-PLac UV5-mKate2 | This study |
| rpsA-LAA | JM109, Ampr, pPMB1-PrpsA P1-egfp-LAA-PLac UV5-mKate2 | This study |
| rpsA-DAS | JM109, Ampr, pPMB1-PrpsA P1-egfp-DAS-PLac UV5-mKate2 | This study |
| rpsA-DAS+4 | JM109, Ampr, pPMB1-PrpsA P1-egfp-DAS+4-PLac UV5-mKate2 | This study |
| rpsA-DAS+8 | JM109, Ampr, pPMB1-PrpsA P1-egfp-DAS+8-PLac UV5-mKate2 | This study |
| rpsA-GSD | JM109, Ampr, pPMB1-PrpsA P1-egfp-GSD-PLac UV5-mKate2 | This study |
| rpsA | JM109, Ampr, pPMB1-PrpsA P1-egfp-PLac UV5-mKate2 | This study |
| rrnA-LAA | JM109, Ampr, pPMB1-PrrnA P1-egfp-LAA-PLac UV5-mKate2 | This study |
| rrnA-DAS | JM109, Ampr, pPMB1-PrrnA P1-egfp-DAS-PLac UV5-mKate2 | This study |
| rrnA-DAS+4 | JM109, Ampr, pPMB1-PrrnA P1-egfp-DAS+4-PLac UV5-mKate2 | This study |
| rrnA-DAS+8 | JM109, Ampr, pPMB1-PrrnA P1-egfp-DAS+8-PLac UV5-mKate2 | This study |
| rrnA-GSD | JM109, Ampr, pPMB1-PrrnA P1-egfp-GSD-PLac UV5-mKate2 | This study |
| rrnA | JM109, Ampr, pPMB1-PrrnA P1-egfp-PLac UV5-mKate2 | This study |
| rrnC-LAA | JM109, Ampr, pPMB1-PrrnC P1-egfp-LAA-PLac UV5-mKate2 | This study |
| rrnC-DAS | JM109, Ampr, pPMB1-PrrnC P1-egfp-DAS-PLac UV5-mKate2 | This study |
| rrnC-DAS+4 | JM109, Ampr, pPMB1-PrrnC P1-egfp-DAS+4-PLac UV5-mKate2 | This study |
| rrnC-DAS+8 | JM109, Ampr, pPMB1-PrrnC P1-egfp-DAS+8-PLac UV5-mKate2 | This study |
| rrnC-GSD | JM109, Ampr, pPMB1-PrrnC P1-egfp-GSD-PLac UV5-mKate2 | This study |
| rrnC | JM109, Ampr, pPMB1-PrrnC P1-egfp-PLac UV5-mKate2 | This study |
| rpsT-LAA | JM109, Ampr, pPMB1-PrpsT P1-egfp-LAA-PLac UV5-mKate2 | This study |
| rpsT-DAS | JM109, Ampr, pPMB1-PrpsT P1-egfp-DAS-PLac UV5-mKate2 | This study |
| rpsT-DAS+4 | JM109, Ampr, pPMB1-PrpsT P1-egfp-DAS+4-PLac UV5-mKate2 | This study |
| rpsT-DAS+8 | JM109, Ampr, pPMB1-PrpsT P1-egfp-DAS+8-PLac UV5-mKate2 | This study |
| rpsT-GSD | JM109, Ampr, pPMB1-PrpsT P1-egfp-GSD-PLac UV5-mKate2 | This study |
| rpsT | JM109, Ampr, pPMB1-PrpsT P1-egfp-PLac UV5-mKate2 | This study |
| SA1 | E. coli MG1655, Cmr, ΔptsHIcrr::Zmglf, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE | Lab storage |
| SA2 | E. coli MG1655, Cmr, ΔptsHIcrr::Zmglf, ΔaroL::tktA, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE | This study |
| SA3 | E. coli MG1655, Cmr, ΔptsHIcrr::Zmglf, ΔaroL::tktA, ΔaroK, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE | This study |
| SA31 | SA3, Ampr, Cmr, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE, pPMB1-PrpsA P1-aroK-LAA | This study |
| SA32 | SA3, Ampr, Cmr, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE, pPMB1-PrpsA P1-aroK-DAS+4 | This study |
| SA33 | SA3, Ampr, Cmr, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE, pPMB1-PrrnA P1-aroK-DAS+8 | This study |
| SA34 | SA3, Ampr, Cmr, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE, pPMB1-PrrnA P1-aroK-GSD | This study |
| SA35 | SA3, Ampr, Cmr, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE, pPMB1-PrrnC P1-aroK-DAS | This study |
| SA32S | SA3, Ampr, Cmr, p15A-PP9-aroGfbr-PP9-tktA-PP9-aroBopt-aroE, pPMB1-PrpsA P1-aroK | This study |
大肠杆菌的常规培养在LB培养基中进行,莽草酸发酵在无机盐培养基中进行。
LB培养基:5 g/L酵母粉,10 g/L蛋白胨,10 g/L NaCl,pH 7.0。LB固体培养基额外添加20 g/L的琼脂。
M9培养基:葡萄糖40 g/L、Na2HPO4·12H2O 15.11 g/L、KH2PO4 3 g/L、NH4Cl 1 g/L、NaCl 0.5 g/L、微量元素1 mL/L、MgSO4·7H2O 0.25 g/L。
NBS培养基:葡萄糖40 g/L、KH2PO4 3.5 g/L、K2HPO4 5.0 g/L、(NH4)2HPO4 3.5 g/L、CaCl2·2H2O 15 mg/L、微量元素1 mL/L、MgSO4·7H2O 0.25 g/L、维生素B1 0.5 mg/L、盐酸甜菜碱1 mmol/L。
AM1培养基:葡萄糖40 g/L、(NH4)2HPO4 2.63 g/L、NH4H2PO4 0.87 g/L、KCl 0.15 g/L、微量元素液1 mL/L、MgSO4·7H2O 0.37 g/L。
MM培养基:葡萄糖40 g/L、NH4Cl 2.0 g/L、(NH4)2SO4 5.0 g/L、KH2PO4 6.0 g/L、MOPS 8.4 g/L、NaCl 0.5 g/L、MgSO4 0.24 g/L、微量元素1 mL/L、维生素B1 1.01 g/L。
微量元素:FeCl3·6H2O 2.4 g/L、CoCl2·6H2O 0.3 g/L、CuCl2 0.15 g/L、ZnCl2·4H2O 0.3 g/L、NaMnO4 0.3 g/L、H3BO3 0.075 g/L、MnCl2·4H2O 0.5 g/L,溶于0.1 mol/L HCl。
1.3 动态分子开关的构建及表征 1.3.1 动态分子开关构建使用全质粒PCR等分子生物学方法[18]在GFP的C末端与5种degron融合组建GFP-LAA、GFP-DAS、GFP-DAS+4、GFP-DAS+8、GFP-GSD 5种质粒。使用SalⅠ和Hind Ⅲ限制性内切酶将融合有降解标签的GFP片段插入到含有生长偶联型启动子表达框的质粒中,构建25种动态分子开关。
1.3.2 荧光强度检测将相应的质粒转化到大肠杆菌JM109菌株中,挑取单菌落接种到含有100 mg/L氨苄青霉素的LB培养基中过夜培养。将过夜培养后的种子液,以1︰100 (V/V)的比例接种于含有50 mL LB的250 mL摇瓶中,在37 ℃、200 r/min下培养,将此时设为0 h,每隔2 h取200 μL培养液作为样品采用SpectraMax M3酶标仪检测样品的荧光强度。检测绿色荧光蛋白GFP时,激发波长为(488±10) nm,发射波长为(511±10) nm。检测红色荧光蛋白mKate2的激发波长为(588±10) nm,发射波长为(640±10) nm。
1.3.3 菌落水平检测为了在菌落水平上进行延时荧光成像,将构建的质粒转化至大肠杆菌JM109菌株中,将其接种到带有抗生素的LB培养基中过夜活化。然后取过夜活化后的菌液5 μL接种到含有琼脂的LB平板上,置于37 ℃培养,时间设为0 h。使用超灵敏荧光成像系统iBright FL1000 (Invitrogen,Thermo Fisher Scientific)在GFP (488/510 nm)和Texas Red (595/615 nm)下进行荧光成像。GFP和mKate2的曝光时间为50 ms,每隔2 h拍照一次。
1.4 莽草酸发酵条件 1.4.1 摇瓶发酵挑取单菌落到含有25 mL LB培养基的摇瓶中,37 ℃、200 r/min培养12 h,然后取200 μL种子液接种到含有氨苄青霉素100 mg/L和氯霉素30 mg/L的50 mL NBS培养基中,于37 ℃、200 r/min的条件下培养72 h,每隔12 h取样检测莽草酸。
1.4.2 5 L发酵罐补料发酵挑取单菌落到含有25 mL LB中,30 ℃、200 r/min培养12 h,再将培养液接种于50 mL种子培养基中,37 ℃、200 r/min培养12 h。然后将200 mL种子液接种到装有1.7 L发酵培养基、氨苄青霉素终浓度100 mg/L,氯霉素终浓度30 mg/L的5 L发酵罐中,在35 ℃、pH 7.0、溶氧30%的条件下发酵。以氨水调控pH。每隔4 h取样检测发酵液中的葡萄糖和莽草酸的含量。
1.5 分析方法 1.5.1 莽草酸激酶活性检测莽草酸激酶活性测定,将菌株在NBS培养基中培养,每12小时收集一次,12 000×g离心10 min,然后重悬于0.05 mol/L巴比妥缓冲液(pH 7.0)中。菌液通过超声处理裂解,12 000×g离心10 min。以牛血清蛋白为标准品,通过Bradford法测定蛋白质浓度。在含有4 μmol/L ATP、1 μmol/L SA、10 μmol/L NaF、5 μmol/L MgCl2、25 μmol/L巴比妥缓冲液(pH 9.0)的1 mL混合液中测量莽草酸激酶活性。1 U的酶活性定义为在上述条件下每分钟消耗1 μmol/L SA所需的量。
1.5.2 莽草酸HPLC检测方法采用高效液相色谱法(HPLC)分析莽草酸和葡萄糖的浓度,采用安捷伦1260高效液相系统(Agilent Technologies,USA),配备Aminex HPX-87H分析柱(300 mm×7.8 mm;9 μm;美国Bio-Rad)。样品在12 000×g离心10 min,然后使用0.22 μm过滤膜对收集的上清液进行过滤。流动相为5 mmol/L H2SO4,流速0.5 mL/min。用210 nm紫外检测器检测莽草酸,用示差折光检测器检测葡萄糖。
2 结果与分析 2.1 动态分子开关的组建和表征动态分子开关由生长偶联型启动子(GPP)、靶蛋白和降解标签(ssrA)组成(图 1A)。生长偶联型启动子在生长期开启靶蛋白转录,进入稳定期后停止转录。降解标签可以控制靶蛋白被大肠杆菌持续降解。在生长期启动子和降解标签的双重作用下,分子开关可以实现靶蛋白在生长期转录表达,进入稳定期后被逐渐降解。
|
| 图 1 动态分子开关的组建和表征 Fig. 1 Dynamic molecular switch design and characterization. (A) The dynamic turn-off switch design. (B) The integration of GFP expression level of 25 dynamic turn-off switches in the X-axis direction. (C) The fluorescence abundance curve of 5 selected representative combinations. (D) The time-lapse fluorescence imaging at the colony level. GFP is a fluorescence image in GFP channel, mKate2 is a fluorescence image in mKate2 channel. GFP+mKate2 is an image in two kinds of fluorescence channels. |
| |
在本研究中,通过将5种生长偶联型启动子(rpsL、rpsT P1、rpsA P1、rrnA P1和rrnC P1)[14-15]和5个强度的降解决定子(LAA、DAS+4、DAS+8、GSD和DAS)[16-17, 19]装配至GFP表达框中,获得25种动态分子开关。对25种分子开关的GFP荧光强度检测后,选择荧光曲线与X轴的积分作为评价指标,用于表征不同分子开关的强度,25种分子开关按照积分的强度一共可以分成5个梯度(图 1B)。在每种强度梯度中,选择代表性分子开关用于后续实验,即rrnC-DAS (积分:60 000)、rrnA-GSD (积分:40 000)、rrnA-DAS+8 (积分:30 000)、rpsA-DAS+4 (积分:10 000)、rpsA-LAA (积分:5 000) 5种分子开关(图 1C)。对选定的5种分子开关进行可视化的荧光成像表征,取过夜培养的菌液5 μL,点种到平板中,每隔2 h分别对GFP和mKate2进行荧光拍照,不携带降解标签的菌株(rrnA)作为对照菌株,由组成型启动子控制的红色荧光蛋白mKate2作为每种分子开关的对照蛋白。实验结果如图 1D所示,对照蛋白mKate2在培养阶段的荧光强度逐渐增加;随着5种分子开关的强度逐渐降低,可以观察到每种分子开关控制的GFP荧光强度逐渐减弱:rrnC-DAS和rrnA-GSD在培养8 h观察到GFP明显的降解现象,而强度更低的rrnA-DAS+8在培养6 h后可以观察到降解现象,最弱的rpsA-LAA在6 h后无法观察到绿色荧光。由此表明,可视化荧光表征的结果与分子开关的积分强度一致。
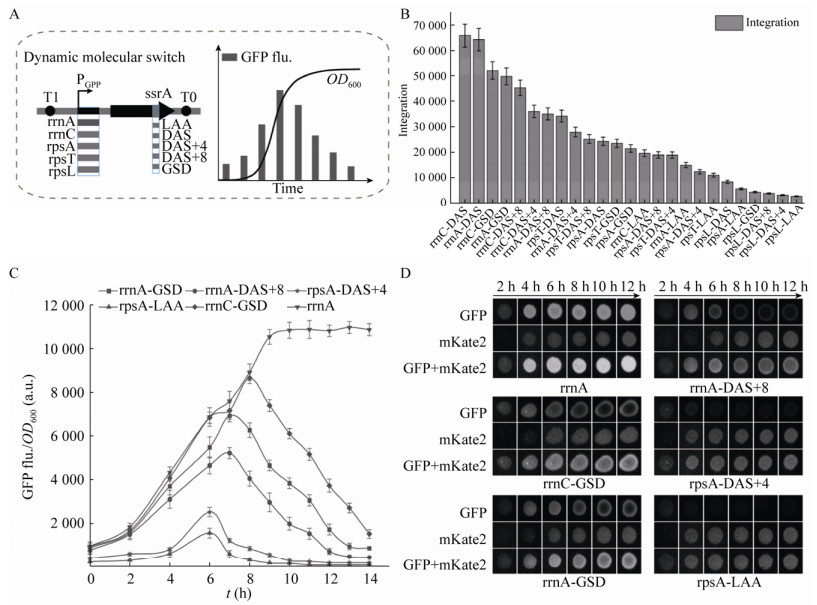
2.2 莽草酸生产菌株的构建大肠杆菌的莽草酸合成途径如图 2A所示,磷酸烯醇式丙酮酸(PEP)和赤藓糖-4-磷酸(E4P)经过DAHP合酶(aroG编码)催化为DAHP (3-脱氧-阿拉伯庚酮糖酸-7-磷酸),再经过3-脱氢奎尼酸合成酶(aroB编码)和莽草酸脱氢酶(aroE编码)等酶的催化最终合成莽草酸。莽草酸会被莽草酸激酶(aroL和aroK编码的同工酶)催化为莽草酸-3-磷酸,进而被合成为3种必需的芳香族氨基酸。为了获得莽草酸生产菌株,利用代谢工程手段对大肠杆菌MG1655进行改造:(1)使用来源于Z. mobilis的glf [20]在基因组上替换ptsHIcrr,构建菌株SA1,ptsHIcrr基因编码大肠杆菌的PTS系统(消耗PEP将葡萄糖转运至胞内),PTS系统被glf替换可以增加前体物质的浓度,同时回补由于PTS系统破坏导致的葡萄糖吸收速率下降;(2)以SA1为基础在基因组上敲除aroL,构建菌株SA2;(3)在SA2基础上在进一步敲除aroK构建菌株SA3。构建质粒BEGA,强化表达路径酶基因aroB、aroG、tktA和aroE。在质粒BEGA中aroB的第3个密码子经过优化(AGG突变为CGT),以增加表达量;aroG编码的第146位氨基酸Asp突变为Asn,消除反馈抑制。
|
| 图 2 构建莽草酸生产途径 Fig. 2 Engineering shikimic acid biosynthetic pathway. (A) The synthesis pathway of shikimic acid. (B) Shikimic acid production of engineered strain. PTS: phosphotransferase system; Zmglf: glucose facilitator; tktA: transketolase A; aroGfbr: DAHP synthase; aroBopt: DHQ synthase; aroD: DHQ dehydratase; aroE: shikimic acid dehydrogenase; aroK/aroL: shikimic acid kinase Ⅰ/Ⅱ; PYR: pyruvate; G6P: glucose 6-phosphate; PEP: phosphoenolpyruvate; E4P: erythrose-4-phosphate; DAHP: 3-deoxy-D-arabino-heptulosonate-7-phosphate; Ru5P: Ribulose-5-phosphate; DHQ: 3-dehydroquinic acid; DHS: 3-dehydroshikimate; S3P: shikimate-3-phosphate; CHA: chorismic acid. |
| |
将质粒BEGA分别转化至SA1、SA2、SA3三种菌株内并对其发酵性能进行评价,在补充有机氮源的M9培养基(额外添加酵母提取物5 g/L)中发酵72 h。实验结果如图 2B所示,SA1的产量为0.24 g/L,SA2的产量是0.31 g/L,SA3的产量为1.41 g/L。只敲除aroL的菌株SA2产量是SA1的1.29倍,仅为SA3的0.22倍,结果表明菌株仅敲除aroL无法彻底阻断莽草酸的下游代谢路径。而在其基础上改造的SA3通过彻底阻断莽草酸的代谢,使产量得到极大提升。
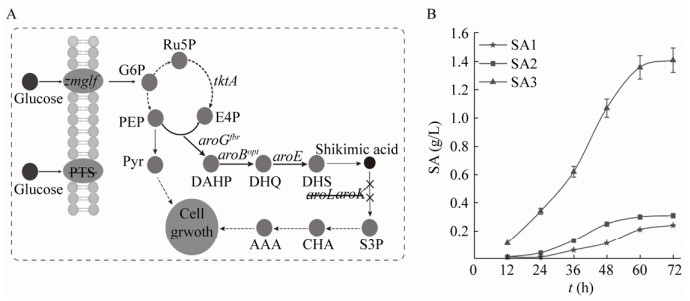
2.3 使用动态分子开关生产莽草酸将编码的莽草酸激酶的aroK作为靶基因,安装到先前构建的5种动态分子开关中(图 3A),再将5种分子开关和质粒BEGA转化至SA3菌株内,构建莽草酸生产菌株SA31、SA32、SA33、SA34和SA35。以SA3为对照组,经过72 h发酵后,结果如图 3B所示:(1)对照组SA3因为不能表达aroK导致不能在无机盐培养基中生长,OD600仅为0.04,几乎无法检测莽草酸浓度;(2)菌株SA31-SA35的OD600分别达到2.39、3.26、4.04、5.02和6.48,莽草酸产量分别为0.48 g/L、1.56 g/L、0.87 g/L、0.49 g/L和0.43 g/L;(3)菌株SA31-SA35所携带的分子开关强度逐渐增强,莽草酸激酶的表达量增加,导致最终OD600变高,而莽草酸产量呈现先增加再下降的趋势。由于SA32菌株实现了细胞生长和莽草酸合成的平衡,将用于后续的研究中。
|
| 图 3 使用动态分子开关生产莽草酸 Fig. 3 Production of shikimic acid using molecular switches. (A) Using molecular switches expressing aroK. (B) Effect of different combinations of the molecular switches on titer and cell growth in NBS medium. (C) and (D) The OD600 and shikimic acid titer in medium optimization test. |
| |
对最优菌株SA32在M9、NBS、AM1和MM等无机盐培养基中的生产性能进行评价,结果如图 3C和3D所示:经过24 h摇瓶发酵,M9、NBS、AM1和MM培养基中的莽草酸产量均在0.35 g/L左右;经过48 h的发酵,4种培养基中莽草酸的产量分别为1.21 g/L、1.12 g/L、0.82 g/L和0.54 g/L,M9培养基产量最高;经过60 h的发酵,4种培养基的莽草酸产量为别为1.39 g/L、1.48 g/L、1.01 g/L和0.73 g/L,NBS培养基的产量迅速增加,超过M9培养基;经过72 h的发酵,4种培养基的最终OD600分别为3.09、3.54、4.29和5.01,莽草酸的最终产量分别为1.51 g/L、1.64 g/L、1.24 g/L和1.01 g/L。上述结果表明NBS培养基是最适合莽草酸发酵的无机盐培养基。
2.4 分子开关的效率和适用性检测为了评价莽草酸生产过程中分子开关的效率,对发酵过程中莽草酸激酶的活性进行检测,实验在500 mL摇瓶中进行,因为对照组SA3菌株不含有动态分子开关,导致其无法在NBS培养基中正常生长,而对照组SA32S表达aroK时不携带降解标签,导致在整个培养阶段的莽草酸激酶活性都处于较高的状态(图 4A、图 4B)。对于最优菌株SA32,在动态分子开关的作用下,生长期(0–48 h)莽草酸激酶的活性从5.73 U/mg增加至25.06 U/mg,稳定期(48–72 h)后活性降至2.55 U/mg。由于莽草酸激酶失活,菌株无法在基本培养基中继续生长,导致生长停滞而莽草酸不断积累,这一结果证明动态分子开关可实现细胞生长与莽草酸的生产相互分离。

|
| 图 4 分子开关的效率和适用性 Fig. 4 Efficiency and applicability of molecular switches. (A, B) The OD600 and SA kinase activity in efficiency test. (C) Shikimic acid production of engineered strain SA32 in bioreactor. |
| |
为了验证分子开关在不同规模反应器中的适用性,最优菌株SA32在5 L发酵罐中进行发酵测试。结果如图 4C所示,菌株在发酵罐中经过24 h生长到达稳定期,同时莽草酸在发酵24 h后迅速积累,经过72 h发酵,产量升高到14.33 g/L (得率为0.22 g/g葡萄糖),比500 mL摇瓶发酵的产量提升8.73倍。结果表明,本研究中将细胞生长和化学品合成解偶联的动态分子开关适用于较大规模发酵。
3 讨论莽草酸的生产过程中面临着细胞生长受限制的问题,本文选择平衡细胞生长和产物合成的代谢流为解决策略。我们利用生长期启动子和内源蛋白降解系统构建了模块化、可调的动态分子开关。将GFP整合到动态分子开关内,可以实现GFP在生长期积累,进入稳定期后逐渐被降解。然后将该开关应用到大肠杆菌的体内代谢途径中,在5 L发酵罐上经过72 h发酵最终生产14.33 g/L莽草酸。
在调节水平方面,其他类型的调控如生物传感器[21]仅能在转录水平进行调节;而本文使用的动态开关可以在转录水平和翻译后水平同时调节蛋白丰度,具有高效和低能耗的优点。在组成元件方面,其他类型的调控回路如群体感应回路[7, 22]需要引入外源蛋白;而本文的开关根据细胞的生理状态和营养条件对代谢流进行调控[15, 23],组成开关的元件均为内源性转录因子和内源蛋白水解酶[14, 19],极大地减轻了宿主的代谢负担。在今后的研究中,可以利用合成生物学手段,组建启动子突变文库,进一步优化动态分子开关的动态范围和强度,为精确调控代谢流提供解决方案。
| [1] |
Farina V, Brown JD. Tamiflu: the supply problem. Angew Chem Int Ed, 2006, 45(44): 7330-7334. |
| [2] |
Kim HK, Park KJJ. A new efficient synthesis of oseltamivir phosphate (Tamiflu) from (-)-shikimic acid. Tetrahedron Lett, 2012, 53(13): 1561-1563. |
| [3] |
Liu XL. Production of shikimic acid and resveratrol in Escherichia coli based on synthetic biology[D]. Shanghai: China State Institute of Pharmaceutical Industry, 2016 (in Chinese). 刘向磊.合成生物学技术改造大肠杆菌生产莽草酸及白藜芦醇[D].上海: 中国医药工业研究总院, 2016. |
| [4] |
Rodriguez A, Martínez JA, Báez-Viveros JL, et al. Constitutive expression of selected genes from the pentose phosphate and aromatic pathways increases the shikimic acid yield in high-glucose batch cultures of an Escherichia coli strain lacking PTS and pykF. Microb Cell Fact, 2013, 12: 86. DOI:10.1186/1475-2859-12-86 |
| [5] |
Zhang FZ, Keasling J. Biosensors and their applications in microbial metabolic engineering. Trends Microbiol, 2011, 19(7): 323-329. DOI:10.1016/j.tim.2011.05.003 |
| [6] |
Zhou L, Niu DD, Tian KM, et al. Genetically switched D-lactate production in Escherichia coli. Metab Eng, 2012, 14(5): 560-568. DOI:10.1016/j.ymben.2012.05.004 |
| [7] |
Kim EM, Woo HM, Tian T, et al. Autonomous control of metabolic state by a quorum sensing (QS)-mediated regulator for bisabolene production in engineered E. coli. Metab Eng, 2017, 44: 325-336. DOI:10.1016/j.ymben.2017.11.004 |
| [8] |
Xu P, Li LY, Zhang FM, et al. Improving fatty acids production by engineering dynamic pathway regulation and metabolic control. Proc Natl Acad Sci USA, 2014, 111(31): 11299-11304. DOI:10.1073/pnas.1406401111 |
| [9] |
Chen XL, Liu LM. Gene circuits for dynamically regulating metabolism. Trends Biotechnol, 2018, 36(8): 751-754. DOI:10.1016/j.tibtech.2017.12.007 |
| [10] |
Dahl RH, Zhang FZ, Alonso-Gutierrez J, et al. Engineering dynamic pathway regulation using stress-response promoters. Nat Biotechnol, 2013, 31(11): 1039-1046. |
| [11] |
Larrabee KL, Phillips JO, Williams GJ, et al. The relative rates of protein synthesis and degradation in a growing culture of Escherichia coli. J Biol Chem, 1980, 255(9): 4125-4130. |
| [12] |
Belle A, Tanay A, Bitincka L, et al. Quantification of protein half-lives in the budding yeast proteome. Proc Natl Acad Sci USA, 2006, 103(35): 13004-13009. |
| [13] |
Greenbaum D, Colangelo C, Williams K, et al. Comparing protein abundance and mRNA expression levels on a genomic scale. Genome Biol, 2003, 4(9): 117. DOI:10.1186/gb-2003-4-9-117 |
| [14] |
Hirvonen CA, Ross W, Wozniak CE, et al. Contributions of UP elements and the transcription factor FIS to expression from the seven rrn P1 promoters in Escherichia coli. J Bacteriol, 2001, 183(21): 6305-6314. DOI:10.1128/JB.183.21.6305-6314.2001 |
| [15] |
Lemke JJ, Sanchez-Vazquez P, Burgos HL, et al. Direct regulation of Escherichia coli ribosomal protein promoters by the transcription factors ppGpp and DksA. Proc Natl Acad Sci USA, 2011, 108(14): 5712-5717. |
| [16] |
McGinness KE, Baker TA, Sauer RT. Engineering controllable protein degradation. Mol Cell, 2006, 22(5): 701-707. |
| [17] |
Shin Y, Davis JH, Brau RR, et al. Single-molecule denaturation and degradation of proteins by the AAA+ ClpXP protease. Proc Natl Acad Sci USA, 2009, 106(46): 19340-19345. DOI:10.1073/pnas.0910484106 |
| [18] |
Casini A, Storch M, Baldwin GS, et al. Bricks and blueprints: methods and standards for DNA assembly. Nat Rev Mol Cell Biol, 2015, 16(9): 568-576. DOI:10.1038/nrm4014 |
| [19] |
Hersch GL, Baker TA, Sauer RT. SspB delivery of substrates for ClpXP proteolysis probed by the design of improved degradation tags. Proc Natl Acad Sci USA, 2004, 101(33): 12136-12141. DOI:10.1073/pnas.0404733101 |
| [20] |
Tang JL, Zhu XN, Lu J, et al. Recruiting alternative glucose utilization pathways for improving succinate production. Appl Microbiol Biotechnol, 2013, 97(6): 2513-2520. DOI:10.1007/s00253-012-4344-1 |
| [21] |
Yang YP, Lin YH, Wang J, et al. Sensor-regulator and RNAi based bifunctional dynamic control network for engineered microbial synthesis. Nat Commun, 2018, 9: 3043. |
| [22] |
Soma Y, Hanai T. Self-induced metabolic state switching by a tunable cell density sensor for microbial isopropanol production. Metab Eng, 2015, 30: 7-15. DOI:10.1016/j.ymben.2015.04.005 |
| [23] |
Mallik P, Paul BJ, Rutherford ST, et al. DksA is required for growth phase-dependent regulation, growth rate-dependent control, and stringent control of fis expression in Escherichia coli. J Bacteriol, 2006, 188(16): 5775-5782. DOI:10.1128/JB.00276-06 |
 2020, Vol. 36
2020, Vol. 36