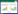中国科学院微生物研究所、中国微生物学会主办
文章信息
- 宋绍征, 张旻, 李丹, 陈朝军, 于康英, 张婷, 周鸣鸣, 成勇
- Song Shaozheng, Zhang Min, Li Dan, Chen Chaojun, Yu Kangying, Zhang Ting, Zhou Mingming, Cheng Yong
- 人乳铁蛋白(hLF)转基因山羊的遗传背景分析
- background of human lactoferrin transgenic goats
- 生物工程学报, 2020, 36(1): 67-76
- Chinese Journal of Biotechnology, 2020, 36(1): 67-76
- 10.13345/j.cjb.190173
-
文章历史
- Received: May 6, 2019
- Accepted: June 19, 2019
- Published: July 16, 2019
2. 扬州大学 兽医学院 江苏省转基因动物制药工程研究中心,江苏 扬州 225009
2. Jiangsu Provincial Research Center for Animal Transgenesis and Biopharming, College of Veterinary Medicine, Yangzhou University, Yangzhou 225009, Jiangsu, China
通过转基因技术生产的后代外源基因并不一定都能高效表达及稳定遗传,有可能产生基因沉默或外源基因拷贝数丢失,这些都是育种过程中无法回避的问题[1]。另外,相关文献报道,转基因技术可能会引起宿主染色体数量和结构的改变,易造成重构胚的损伤和重编程异常,从而造成异常的核型[2-4],直接导致胚胎在发育过程中死亡、流产或幼崽畸形[5]。Pursel等[6]和Reik等[7]相关研究也证实了当外源基因插入到宿主染色体上重要的功能基因中,就会导致宿主的畸形发育甚至死亡。因此,在转基因新品种培育的过程中,从分子遗传到染色体核型以及动物外在表型,都需要进行不断的遴选。
相关研究表明,外源基因的拷贝数在传递中会出现差异,有可能会影响外源基因的表达和遗传稳定,而且外源基因的拷贝在世代传递过程中还可能发生丢失[8-10]。Migliaccio等[11]通过体外筛选K256细胞发现,外源基因虽然能在许多位点整合和表达,但是只有在特定的位点才能稳定遗传给下一代,而其他位点处的外源基因就会发生丢失。此外,关于外源基因的拷贝数与表达水平之间的关系也说法不一。蒋冬花等[12]研究发现,转抗虫基因的烟草中,抗病效果与整合的拷贝数呈负相关性,单拷贝的植株比2–3个拷贝的植株抗病能力更强。Mansur等[13]构建了携带不同拷贝数(1、5、11)的胰岛素前体(MIP)基因的毕赤酵母载体,发现表达水平与拷贝数呈正相关性。
但是,目前关于转基因动物的遗传背景分析较少报道,尤其是人乳铁蛋白(hLF)转基因山羊的遗传背景分析尚未见报道,如刘长国等对陕西省境内5个山羊品种遗传背景的随机扩增多态性DNA (Radom amplified polymorphic DNA,RAPD)分析[14],毛凤显等对贵州地方山羊品种遗传背景的微卫星分析[15],这些均是针对非转基因普通山羊的遗传背景分析。
因此,本研究为了分析转基因山羊的遗传背景,选取扬州大学兽医学院江苏省转基因制药工程研究中心通过核移植制备的hLF转基因山羊作为研究对象,采用荧光原位杂交(Fluorescence in situ hybridization,FISH)、实时荧光定量PCR (qPCR)、酶联免疫吸附试验(Enzyme-linked immunosorbent assay,ELISA)和Western blotting作为研究手段,对转基因山羊的核型和外源基因拷贝数进行分析,旨在阐明转基因山羊整合的外源基因是否会对本身造成影响、是否能够稳定遗传给后代、拷贝数与外源基因表达水平的关系等一系列遗传背景问题,为今后转基因山羊及其他动物新品种培育提供良好的指导作用。
1 材料与方法 1.1 材料 1.1.1 主要试剂GoTaq qPCR Master Mix (Promega),GoTaq Green Master Mix (Promega),Wizard Genomic DNA Purification Kit (Promega),pGEM-T Easy Vector (Promega),SunShineclean质粒小量抽提试剂盒(SunShineBio),凝胶回收试剂盒(QIAGEN),GeneRuLer™ Low Range DNA ladders (Fermentas),鼠抗hLF单克隆抗体(Santa Cruz,sc-53498)、羊抗鼠单克隆抗体IgG-HRP (Santa Cruz,sc-2005),其余未说明试剂均为国产分析纯,分别购自生工生物工程(上海)股份有限公司和国药集团化学试剂有限公司等。
1.1.2 引物根据外源基因和管家基因(山羊β-actin,基因组拷贝数为2)序列用Primer Premier 5.0软件设计引物,由生工生物工程(上海)股份有限公司合成(表 1)。
| Primer name | Primer sequence (5'–3') |
| hLF-F | GTGGTGTCTCGGATGGATAAG |
| hLF-R | TTAGTAATGCCTGCGACATACTG |
| actin F | CTTCCTTCCTGGGTGAGTGAGA |
| actin R | ACAGCACCGTGTTGGCGTAAA |
BLC14载体构建并保存于本实验室,含有山羊β-乳球蛋白调控元件和hLF功能基因(图 2A)。
|
| 图 2 BLC14载体结构示意图与质粒酶切图谱 Fig. 2 The structure diagram of BLC14 vector (A) and enzyme digestion analysis of BLC14 (B). |
| |
F0 (标记L1–L6,BLC14载体转染山羊胎儿成纤维细胞作为供核细胞,通过体细胞核移植制备的转基因克隆山羊,整合hLF外源基因)及F1代(标记L1-1、L2-1、L3-1、L4-1、L5-1、L6-1,F0代与普通山羊杂交后代)的hLF转基因山羊和普通非转基因正常山羊(标记N0、N0-1)来自扬州大学实验农牧场,常规饲养。
1.2 方法 1.2.1 山羊血细胞染色体片的制备及核型分析选取6只原代hLF转基因山羊(F0,标记L1–L6)和2只正常山羊(标记N0、N0-1),1%肝素钠作抗凝剂,颈静脉无菌采血2 mL。37 ℃恒温箱中培养72 h,添加终浓度0.5 mol/mL的
秋水仙素处理后,继续培养,使细胞分裂停止在分裂中期。收集对数生长期细胞,使用终浓度0.05 μg/mL的秋水仙素处理6 h,然后依次5 mL低渗液(0.075 mol/L KCl) 37 ℃处理30 min、1 mL固定液(甲醇/冰醋酸3:1) 4 ℃放置20 min,重复固定2次后,−20 ℃过夜。次日,调整细胞浓度为1×105个/mL,滴片(20 cm高度滴到预冷冻的载玻片上),在空气中放置干燥。最后,添加1.5 g/L Giemsa工作液染色20 min,自来水冲洗载玻片,自然干燥。挑选良好的分裂相在显微镜下进行放大、摄影。利用Adobe Photoshop图像处理软件进行染色体的计数、剪切、排队,进行同源染色体的配对、测量。
1.2.2 荧光原位杂交(FISH)检测BLC14质粒经NotⅠ/SalⅠ酶切回收15 936 bp的片段作为BLC14探针。Digoxigenin-11-dUTP切口平移法标记探针,并进行探针免疫学检测:1 μL探针点尼龙膜;2 000 W,5 min,紫外交膜;马莱酸缓冲液,室温摇动2 min;2 mL封闭液(5% BSA),30 min;添加2 mL荧光抗体Anti-Dig-fluorescine,使其终浓度为200 μg/mL,孵育30 min;洗涤2次(洗涤液为0.1 mol/L马来酸+0.3% Tween20),15 min/次;添加10 mL检测缓冲液(0.1 mol/L Tris-HCl缓冲液,pH 9.5),平衡5 min;显色、拍照并保存结果。标记成功的探针,按照常规方法[16]进行探针杂交:玻片置于85 ℃变性液(70%甲酰胺/2×SSC)中3 min,立刻转移至70%冰乙醇,脱水,晾干;探针杂交液(2 μL 20×SSC,10 μL去离子甲酰胺,4 μL 50% DS,2 μL探针,2 μL超纯水)沸水浴5 min,转移至冰浴5 min;滴加变性后的杂交缓冲液20 μL于玻片上,封口,37 ℃湿盒杂交过夜;PBS洗净后,滴加100 μL抗体(anti-dig-antibody),37 ℃孵育2 h;PBS洗净、晾干后,滴加20 μL DAPI复染;使用荧光倒置显微镜(IX-71,Olympus,日本)观察,并拍照。
1.2.3 实时荧光定量PCR检测拷贝数选取6只原代hLF转基因山羊(F0,标记L1–L6)及其对应的后代(F1,标记L1-1、L2-1、L3-1、L4-1、L5-1、L6-1)和2只正常山羊(标记N0、N0-1),颈静脉无菌采血0.5 mL (加15% EDTA抗凝),提取基因组。根据外源基因和管家基因(β-actin,内参基因)序列,应用Primer Premier 5.0软件设计引物,PCR扩增、回收,与pGEM-T Easy Vector以3:1的比例进行连接,4 ℃反应8 h,转化感受态细胞,挑取单克隆菌落扩增,提取质粒,酶切和测序鉴定,正确结果分别命名actin-T和hLF-T。按照qPCR的操作说明进行转基因山羊和普通山羊基因组扩增,反应体系(20 μL):2×RT mix,10 μL;模板,1 μL;引物F和R (2 μmol/L),2 μL;无核酸酶水,7 μL。反应程序:95 ℃预变性2 min;95 ℃变性5 s,60 ℃退火及延伸30 s,40个循环。熔解曲线:温度为60–95 ℃,间隔为0.5 ℃,时间为5秒/个。设定好如上程序,Ct值、阈值等实验数据由软件自动收集生成。
1.2.4 ELISA检测选取F1代hLF转基因山羊(标记L1-1、L2-1、L3-1、L4-1、L5-1、L6-1)和正常山羊(标记N0-1)的乳清作为检测样品,每天用雌二醇(2 mg/只)、黄体酮(10 mg/只)进行人工诱导泌乳,连续催乳14 d后注射一支利血平(0.5 mg/只),2 d后收集乳汁。乳汁离心(10 000 r/min,30 min)去除上层脂肪及下层浑浊,吸取乳清,−20 ℃保存备用。以转基因山羊乳清作为样本、普通山羊乳清作为阴性对照、PBS作为空白对照、人乳清作为阳性对照,使用鼠抗hLF单克隆抗体作为一抗(sc-53498,Santa Cruz)、羊抗鼠单克隆抗体IgG-HRP (sc-2005,Santa Cruz)作为二抗进行ELISA检测,显色后酶标仪测定OD450值,并绘制标准曲线、计算hLF表达量。
1.2.5 Western blotting检测乳清样品以样本稀释液(125 mmol/L NaCl,25 mmol/L Tris,5 mmol/L KCl, 2 mmol/L苯甲基磺酰氟,pH 7.4)进行100倍稀释,按照1:1体积与电泳上样缓冲液(100 mmol/L Tris-HCl,pH 6.8,200 mmol/L 1, 4-二硫苏糖醇,4% SDS,0.2%溴酚蓝,20%甘油)混匀,100 ℃加热5 min,变性,进行12% SDS聚丙烯酰胺凝胶电泳(SDS-PAGE)[17]。使用转移缓冲液(1.93 g/L Tris,9 g/L glycine)将聚丙烯酰胺凝胶转移至PVDF膜,250 mA,转印3 h。超纯水冲洗后,37 ℃封闭(20 mmol/L Tris,137 mmol/L NaCl,0.1% Tween-20,10%胎牛血清,pH 7.6),2h。加入一抗稀释液(1:1 000稀释,鼠抗hLF单克隆抗体,sc-53498,Santa Cruz),37 ℃孵育2 h。TTBS (20 mmol/L Tris,137 mmol/L NaCl,1% Tween-20,pH 7.6)洗涤3次后,加入二抗-HRP稀释液(1:2 000稀释,羊抗鼠单克隆抗体IgG-HRP,sc-2005,Santa Cruz)中,37 ℃孵育2 h。取出PVDF膜,PBS洗净后,添加显色液(DAB 50 mg,0.05 mol/L TB 100 mL, 30 μL 30% H2O2,pH 7.6),室温15 min,晾干后拍照、记录并保存。
2 结果与分析 2.1 转基因山羊的染色体图谱分析普通健康山羊的染色体数目是60条,有29对是同源染色体的常染色体,最后一对是性染色体。在hLF转基因山羊染色体片中挑选分散良好、形态清晰、收缩适中的中期分裂相,通过与普通非转基因正常山羊染色体进行比较,结果显示:选取的6只F0代转基因山羊(L1、L2、L3、L4、L5、L6)的染色体和正常山羊的染色体十分相似,没有发现转基因山羊的染色体数目和形态结构异常变化。从图 1中可以看出染色体数目均为60条,形状均为马蹄形;而性染色体X也为端着丝粒染色体,相对长度较大;Y染色体形态较小,可以很明显地辨认。

|
| 图 1 山羊染色体核型分析图 Fig. 1 The karyotypes analysis map of goats. (A) Normal ram chromosome. (B) Normal ewe chromosome. (C) L1 chromosome. (D) L2 chromosome. (E) L3 chromosome. (F) L4 chromosome. (G) L5 chromosome. (H) L6 chromosome. |
| |
BLC14质粒(图 2A)酶切回收15 936 bp大小的片段,与预期的结果一致,如图 2B所示。按照切口平移法用Digoxigenin-11-dUTP标记BLC14探针的标记效率如图 3A所示。本实验共标记了0.5 ng/μL和1 ng/μL两种浓度的探针,均能够达到实验的预期目标,且标记效率均能达到要求。BLC14探针与转基因山羊杂交结果显示,染色体上有明显的绿色信号(图 3B1,L3转基因山羊,箭头所示),说明在该处染色体位点整合了外源目的基因(11号染色体);而正常非转基因普通山羊中未见到杂交绿色荧光信号(图 3B2),说明正常普通山羊中不含有该外源基因。

|
| 图 3 山羊染色体杂交检测结果 Fig. 3 The hybridization result of goat chromosomes. (A) Determination of the probe labeling efficiency (1–5: standard concentration (1 ng/μL, 0.5 ng/μL, 250 pg/μL, 100 pg/μL, 0 pg/μL); a: 0.5 ng/μL concentration labeled probe; b: 1 ng/μL concentration labeled probe). (B) Hybridization results of BLC14 probe in goat Chromosomes (B1: Chromosomes of hLF transgenic goats L3; B2: Chromosomes of normal goats). |
| |
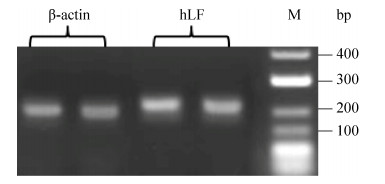
本研究检测的拷贝数是相对于内参基因的相对拷贝数,故内参基因的选择至关重要,我们选择山羊β-肌动蛋白(β-actin)作为内参基因,内参基因β-actin和外源目的基因hLF的PCR扩增产物进行1%凝胶电泳,分别得到193 bp和210 bp的目标片段,如图 4所示。根据qPCR的Conc (β-actin)= 10(–0.286*Ct+12.272)和Conc (hLF)=10(–3.05*Ct+13.070)公式,分别计算出内参基因和外源基因的平均拷贝数[18-19]。由于该内参基因在山羊体内是纯合二倍体[20],而转基因山羊的外源基因是纯合体的几率极小,因此,通过公式“外源基因拷贝数=(待测试样转基因拷贝数×2)/待测试样内标准基因拷贝数”计算出外源基因hLF的相对拷贝数。如表 2和表 3所示,L1–L6号转基因山羊的hLF及其相应的后代相对拷贝数分别是2、16、8、2、12、5,而正常普通山羊(标记N0、N0-1)的相对拷贝数是0,这说明了转基因山羊的外源基因能够稳定地遗传给后代,拷贝数不发生变化。
|
| 图 4 内参基因β-actin和外源基因hLF的PCR扩增电泳图 Fig. 4 The PCR amplification of reference gene β-actin and exogenous gene hLF. |
| |
| Gene sample | Average number of copies | 2×hLF/actin | Relative number of copies for hLF | |
| hLF | β-actin | |||
| L1 | 72.2 | 89.1 | 1.6 | 2 |
| L2 | 279.0 | 34.7 | 16.1 | 16 |
| L3 | 7.6 | 2.0 | 7.7 | 8 |
| L4 | 84.4 | 81.3 | 2.1 | 2 |
| L5 | 199.0 | 32.1 | 12.4 | 12 |
| L6 | 42.5 | 16.2 | 5.2 | 5 |
| N0 | 2.2 | 174.0 | 0.0 | 0 |
| Gene sample | Average number of copies | 2×hLF/actin | Relative number of copies for hLF | hLF expression level (g/L) | |
| hLF | β-actin | ||||
| L1-1 | 81.6 | 95.4 | 1.7 | 2 | 0 |
| L2-1 | 283.0 | 36.1 | 15.7 | 16 | 0.85 |
| L3-1 | 26.5 | 6.8 | 7.8 | 8 | 1.12 |
| L4-1 | 454.0 | 463.0 | 2.0 | 2 | 0 |
| L5-1 | 383.0 | 64.2 | 11.9 | 12 | 0.13 |
| L6-1 | 31.9 | 12.0 | 5.3 | 5 | 0.19 |
| N0-1 | 3.1 | 153.2 | 0.0 | 0 | 0 |
以人乳清标准品浓度(g/L)作为横坐标、OD450值作为纵坐标,制作标准曲线(如图 5A所示),6只F1代转基因山羊乳腺表达hLF含量检测结果见表 3。根据曲线计算,其中L1-1和L4-1转基因山羊乳清未检测出hLF,其余4只转基因山羊乳清检测表达hLF的浓度在0.13–1.12 g/L之间(L2-1:0.85 g/L,L3-1:1.12 g/L,L5-1:0.13 g/L,L6-1:0.19 g/L),最高表达水平达到1.12 g/L (L3-1)。从表 3的转基因山羊表达水平和相对拷贝数对照结果中,可以看出hLF相对拷贝数过低(2)的转基因山羊未表达hLF,而其他表达hLF的转基因山羊拷贝数与表达水平高低无直接明显的相关性。

|
| 图 5 转基因山羊乳腺表达hLF的检测结果 Fig. 5 Detection of hLF expression in mammary glands of transgenic goats. (A) Standard curve of the concentration of hLF in whey samples. The standard concentrations of lactoferrin in human whey were 0, 0.031 25, 0.062 5, 0.125, 0.25, 0.5, 1.0 and 2.0 g/L, respectively. L1-1–L6-1 were collected from F1 transgenic goats after lactagogue. (B) Western blotting analysis of the milk of transgenic goats (All samples were diluted 100 times). 1: positive control (human whey); 2: negative control (normal sheep whey); 3: L1-1 sheep whey; 4: L2-1 sheep whey; 5: L3-1 sheep whey; 6: L4-1 sheep whey; 7: L5-1 sheep whey; 8: L6-1 sheep whey; M: protein marker. |
| |
乳清Western blotting结果(图 5B)表明,L2-1、L3-1、L5-1、L6-1号转基因山羊的乳汁中均有人乳铁蛋白(分子量80 kDa)的表达,出现大小约80 kDa的条带,与预期目标一致;而L1-1、L4-1和普通山羊乳汁中并没有检测出人乳铁蛋白条带,这与ELSIA检测结果相一致。
3 讨论自转基因技术诞生以来,在生物医药、农业生产等领域产生了巨大的效益[1],但外源基因在转基因动物培育繁殖过程中无法稳定高效地遗传和表达是目前转基因新品种培育面临的主要困难之一,因此只有弄清外源基因的遗传背景才能更好地建立转基因新品系[21-22]。刘长国等分析了陕西省境内5个山羊品种(陕南白山羊、莎能奶山羊、关中奶山羊、安哥拉山羊、波尔山羊)的遗传变异及遗传关系[14],毛凤显等研究了4个贵州省地方山羊品种(贵州白山羊、贵州黑山羊、黔北麻羊、榕江小香羊)的遗传背景[15],均是针对普通山羊的遗传背景分析。关于转基因动物的遗传背景分析的研究较少,尤其是hLF转基因山羊的遗传背景分析少之又少。本文主要是从染色体的形态结构和数量、外源基因在染色体上的拷贝数和遗传性、拷贝数与表达水平间的关系等方面来研究hLF转基因山羊的遗传背景。
通过核移植和显微注射等方法制备的转基因动物很容易造成染色体数目和结构异常,致使胚胎在发育过程中自然流产或产生畸形个体[23]。而从我们获得的结果中发现,转基因山羊的染色体核型、数量与普通山羊没有明显的差异,染色体数目均是60条,其中29对为同源染色体的常染色体,最后一对为性染色体,形态结构呈现为马蹄形,性染色体X相对长度较大,Y染色体形态较小。转基因动物中的外源基因通常以头尾相连串联体的形式整合到宿主染色体基因组上,分为单拷贝单一位点的整合、多拷贝单一位点的整合及多拷贝多位点的整合[24]。本实验结果显示6只F0代hLF转基因山羊与其相应的F1后代外源基因的相对拷贝数相同,但整合拷贝数高低不同(2–16),这表明外源基因整合在宿主染色体拷贝数目不定,但是能够在转基因动物中稳定遗传,且拷贝数不变。同时,L3转基因山羊的FISH结果显示只有单一的荧光信号,说明该外源基因hLF是以多拷贝单一位点的整合方式进入山羊染色体中。此外,由于外源基因的绝对拷贝数较难测量,本文提出了“相对拷贝数”这一概念,主要是参照山羊β-actin基因,以此作为内参进行拷贝数计算[18-19]。
据有关文献报道,拷贝数与外源基因表达可以呈现正相关,也可以是负相关,或者拷贝数多少对表达无显著的影响[8, 25]。例如,Hobbs等[26]通过对转基因烟草的GUS基因表达研究发现表达量的高低取决于外源基因的插入位点而不是拷贝数。Meyer等[27]的研究发现,多拷贝时,90%的转基因玉米A1外源基因失活,在该基因启动子附近还出现甲基化现象,这一现象表明多拷贝可能会导致外源基因表达抑制或失活。Kohli等[28]的研究发现,外源基因多拷贝并未导致其表达量下降或转基因沉默,外源基因4–5个与1–2个拷贝的表达水平相差无几。本研究中发现,hLF转基因山羊的相对拷贝数过低时(2)不表达,但是表达水平的高低又和拷贝数无直接的相关性,最高表达水平(1.12 g/L)的转基因山羊拷贝数(8)并非最高。产生这一现象的原因可能是外源基因的表达受拷贝数在内的多种机制的调节,不能单独考虑某一个特定的因素。
总之,本实验主要通过对hLF转基因山羊的遗传背景分析研究发现:转基因山羊的染色体核型、数量与普通山羊没有明显的差异,这表明外源基因的插入未导致染色体出现断裂、缺失等现象;整合外源基因hLF的拷贝数高低不同(2–16),但F0和F1代拷贝数相一致,这表明转基因山羊外源基因能够稳定遗传给下一代;6只F1代hLF转基因山羊中,2只山羊的乳汁未检测到表达hLF (L1-1和L4-1,拷贝数均为2),其余4只表达hLF量在0.13–1.12 g/L,最高表达水平达到1.12 g/L (L3-1,拷贝数为8),这表明外源基因拷贝数多少和表达水平无直接明显的相关性,过低或过高在一定程度上均可能影响外源基因的表达水平。因此,本研究首次比较全面地分析了hLF转基因山羊的遗传背景,且该转基因山羊能够健康存活、正常繁殖和表达外源目的蛋白,为进一步转基因山羊遗传育种和转基因动物新品种培育奠定了基础。
| [1] |
Chansel-Debordeaux L, Bezard E. Local transgene expression and whole-body transgenesis to model brain diseases in nonhuman primate. Anim Model Exp Med, 2019, 2(1): 9-17. DOI:10.1002/ame2.12055 |
| [2] |
Chen JQ, Juan C, Xu XJ, et al. Effect of cytoplast on the development of inter-subspecies nuclear transfer reconstructed goat embryo. Mol Reprod Dev, 2007, 74(5): 568-573. DOI:10.1002/mrd.20647 |
| [3] |
Fischer K, Kind A, Schnieke A. Assembling multiple xenoprotective transgenes in pigs. Xenotransplantation, 2018, 25(6): e12431. DOI:10.1111/xen.12431 |
| [4] |
Li GL, Zhong CL, Mo JX, et al. Advances in site-specific integration of transgene in animal genome. Hereditas, 2017, 39(2): 98-109 (in Chinese). 李国玲, 钟翠丽, 莫健新, 等. 动物基因组定点整合转基因技术研究进展. 遗传, 2017, 39(2): 98-109. DOI:10.3760/cma.j.issn.1673-4386.2017.02.007 |
| [5] |
Di Meco A, Joshi YB, Lauretti E, et al. Maternal dexamethasone exposure ameliorates cognition and tau pathology in the offspring of triple transgenic AD mice. Mol Psychiatry, 2016, 21(3): 403-410. DOI:10.1038/mp.2015.78 |
| [6] |
Pursel VG, Pinkert CA, Miller KF. Genetic engineering of livestock. Science, 1989, 244(4910): 1281-1288. DOI:10.1126/science.2499927 |
| [7] |
Reik W, Römer I, Barton SC, et al. Adult phenotype in the mouse can be affected by epigenetic events in the early embryo. Development, 1993, 119(3): 933-942. |
| [8] |
Bervoets I, Charlier D. A novel and versatile dual fluorescent reporter tool for the study of gene expression and regulation in multi- and single copy number. Gene, 2018, 642: 474-482. DOI:10.1016/j.gene.2017.11.061 |
| [9] |
Bellec L, Katz LA. Analyses of chromosome copy number and expression level of four genes in the ciliate Chilodonella uncinata reveal a complex pattern that suggests epigenetic regulation. Gene, 2012, 504(2): 303-308. DOI:10.1016/j.gene.2012.04.067 |
| [10] |
Lucotte EA, Skov L, Jensen JM, et al. Dynamic copy number evolution of X- and Y-linked ampliconic genes in human populations. Genetics, 2018, 209(3): 907-920. |
| [11] |
Migliaccio AR, Bengra C, Ling JH, et al. Stable and unstable transgene integration sites in the human genome: extinction of the Green Fluorescent Protein transgene in K562 cells. Gene, 2000, 256(1/2): 197-214. |
| [12] |
Jiang DH, Guo ZJ. Disease resistance and inheritance analysis of Cryptogein transgenic tobacco plants. Acta Phytopathol Sin, 2003, 33(3): 237-242 (in Chinese). 蒋冬花, 郭泽建. 转隐地蛋白基因烟草的抗病性及遗传分析. 植物病理学报, 2003, 33(3): 237-242. DOI:10.3321/j.issn:0412-0914.2003.03.010 |
| [13] |
Mansur M, Cabello C, Hernández L, et al. Multiple gene copy number enhances insulin precursor secretion in the yeast Pichia pastoris. Biotechnol Lett, 2005, 27(5): 339-345. DOI:10.1007/s10529-005-1007-7 |
| [14] |
Liu CG, Luo J, Yang GS, et al. Study on the genetic background of five goat breeds in Shaanxi province using RAPD. J Northwest Sci-Tech Univ Agric For: Nat Sci Ed, 2003, 31(3): 19-24 (in Chinese). 刘长国, 罗军, 杨公社, 等. 陕西省境内5个山羊品种遗传背景的RAPD分析. 西北农林科技大学学报:自然科学版, 2003, 31(3): 19-24. |
| [15] |
Mao FX, Huangpu JY, Zhao YZ. Analysis of microsatellite loci on genetic background of the local goats in Guizhou province. Anim Husband Vet Med, 2006, 38(2): 13-15 (in Chinese). 毛凤显, 皇甫江云, 赵有璋. 贵州地方山羊品种遗传背景的微卫星分析. 畜牧与兽医, 2006, 38(2): 13-15. DOI:10.3969/j.issn.0529-5130.2006.02.005 |
| [16] |
An LY, Yuan YG, Yu BL, et al. Generation of human lactoferrin transgenic cloned goats using donor cells with dual markers and a modified selection procedure. Theriogenology, 2012, 78(6): 1303-1311. DOI:10.1016/j.theriogenology.2012.05.027 |
| [17] |
Song SZ, Ge X, Cheng YB, et al. High-level expression of a novel recombinant human plasminogen activator (rhPA) in the milk of transgenic rabbits and its thrombolytic bioactivity in vitro. Mol Biol Rep, 2016, 43(8): 775-783. DOI:10.1007/s11033-016-4020-0 |
| [18] |
Li K, Gao HL, Gao L, et al. Development of a real-time PCR for determination of foreign gene copy number in genome of Pichia pastoris. Acta Vet Zootech Sin, 2011, 42(5): 742-746 (in Chinese). 李凯, 高宏雷, 高立, 等. TaqMan实时荧光定量PCR检测毕赤酵母基因组中外源基因拷贝数. 畜牧兽医学报, 2011, 42(5): 742-746. |
| [19] |
Yu J, Zou J, Fu Q, et al. Detecting copy number of exogenous genes in transgenic tobacco by multiplex RT-PCR. Acta Tabacaria Sin, 2017, 23(4): 92-97 (in Chinese). 余婧, 邹颉, 付强, 等. 多重实时荧光定量PCR分析转基因烟草外源基因拷贝数. 中国烟草学报, 2017, 23(4): 92-97. |
| [20] |
Das A, Deng MY, Babiuk S, et al. Modification of two capripoxvirus quantitative real-time PCR assays to improve diagnostic sensitivity and include beta-actin as an internal positive control. J Vet Diagn Invest, 2017, 29(3): 351-356. DOI:10.1177/1040638717695609 |
| [21] |
Lavania D, Dhingra A, Siddiqui MH, et al. Current status of the production of high temperature tolerant transgenic crops for cultivation in warmer climates. Plant Physiol Biochem, 2015, 86: 100-108. DOI:10.1016/j.plaphy.2014.11.019 |
| [22] |
Wang JK, Cui K, Yang Z, et al. Transcriptome analysis of improved wool production in skin-specific transgenic sheep overexpressing ovine β-catenin. Int J Mol Sci, 2019, 20(3): 620. DOI:10.3390/ijms20030620 |
| [23] |
Scott GJ, Gruzdev A. Genome editing in mouse embryos with CRISPR/Cas9//Allen IC. Mouse Models of Innate Immunity. New York, NY: Humana Press, 2019: 23-40.
|
| [24] |
Saito H, Kurome M, Tomii R, et al. Foreign gene integration patterns in transgenic porcine fetuses produced by ICSI-mediated gene transfer. Reprod Fert Dev, 2007, 19(1): 319-320. |
| [25] |
Rukh G, Ericson U, Andersson-Assarsson J, et al. Dietary starch intake modifies the relation between copy number variation in the salivary amylase gene and BMI. Am J Clin Nutr, 2017, 106(1): 256-262. DOI:10.3945/ajcn.116.149831 |
| [26] |
Hobbs SLA, Kpodar P, DeLong CMO. The effect of T-DNA copy number, position and methylation on reporter gene expression in tobacco transformants. Plant Mol Biol, 1990, 15(6): 851-864. DOI:10.1007/BF00039425 |
| [27] |
Meyer P, Heidmann I. Epigenetic variants of a transgenic petunia line show hypermethylation in transgene DNA: an indication for specific recognition of foreign DNA in transgenic plants. Mol Gen Genet, 1994, 243(4): 390-399. DOI:10.1007/BF00280469 |
| [28] |
Kohli A, Griffiths S, Palacios N, et al. Molecular characterization of transforming plasmid rearrangements in transgenic rice reveals a recombination hotspot in the CaMV 35S promoter and confirms the predominance of microhomology mediated recombination. Plant J, 1999, 17(6): 591-601. DOI:10.1046/j.1365-313X.1999.00399.x |
 2020, Vol. 36
2020, Vol. 36