中国科学院微生物研究所、中国微生物学会主办
文章信息
- 潘梦妍, 徐显皓, 刘延峰, 李江华, 吕雪芹, 堵国成, 刘龙
- Pan Mengyan, Xu Xianhao, Liu Yanfeng, Li Jianghua, Lü Xueqin, Du Guocheng, Liu Long
- 甲壳素酶Chisb的定向进化及生物转化合成几丁寡糖
- Directed evolution of chitinase Chisb and biosynthesis of chitooligosaccharides
- 生物工程学报, 2019, 35(9): 1787-1796
- Chinese Journal of Biotechnology, 2019, 35(9): 1787-1796
- 10.13345/j.cjb.190069
-
文章历史
- Received: February 20, 2019
- Accepted: April 2, 2019
- Published: April 25, 2019
2. 江南大学 生物工程学院,江苏 无锡 214122
2. 2 College of Bioengineering, Jiangnan University, Wuxi 214122, Jiangsu, China
甲壳素酶(EC 3.2.1.14)来源广泛,存在于各种细菌、真菌和一些高等植物的组织中[1-2]。目前,甲壳素酶多应用于生物防治植物病虫害和降解甲壳素产几丁寡糖等方面。近年来,越来越多的国内外研究学者致力于研究甲壳素酶,但是野生型的菌株甲壳素酶产量低、活性也低,从而限制了甲壳素酶的实际应用[3]。因此,运用基因工程或酶工程等手段将甲壳素酶进行克隆表达、分子改造以提高甲壳素酶的催化活性,已经成为研究热点。
Huang等[4]成功在大肠杆菌中高效表达了来源于蜡样芽孢杆菌的甲壳素酶基因ChiCH和ChiCW,重组的甲壳素酶ChiCH和ChiCW通过GST融合标签进行纯化,纯化后的蛋白比酶活分别为7.48 U/mg和3.69 U/mg。随后又对纯化后的蛋白进行了生物化学特性的鉴定,结果发现ChiCH为外切甲壳素酶,ChiCW为内切甲壳素酶。2017年,Guo等[5]从来源于类芽孢杆菌属Paenibacillus pasadenensis CS0611的菌株中分离纯化了一种碱性甲壳素酶,纯化后比酶活达到10.28 U/mg,经过HPLC检测其水解产物发现,该酶可以将胶体甲壳素水解为甲壳二糖。2018年,Zhang等[6]从梅园几丁质分解细菌Chitinolyticbacter >meiyuanensis SYBC-H1中表达了一种新型甲壳素酶CmChi1,经鉴定属于糖苷水解酶18家族,通过胶体甲壳素亲和层析纯化后比酶活达到15.3 U/mg。CmChi1能水解胶体甲壳素且最终产物为GlcNAc,得率接近100%,产物分离后纯度达到98%。
此外,还有学者通过分子对接、定点突变等技术手段对甲壳素酶的结构进行了研究。Madhuprakash等[7]对来源于变形斑沙雷氏菌Serratia proteamaculans的甲壳素酶D的结构进行研究,结果表明,该酶同时具有水解活性和转糖基活性,且该酶具有糖苷水解酶18家族的特点,在其催化结构域中存在保守区域DXDXE结构,这是首次发现单个结构域的甲壳素酶同时具有两种活性。Fan等[8]通过对来源于舞毒蛾Lymantria dispar的甲壳素酶进行定点突变,并对其活性残基进行了功能分析。结果发现,野生型的酶比突变酶具有更高的催化效率,虽然突变位点都为负突变,但是证明了D143、D145和W146为该酶的关键活性位点。
我们在前期研究中,通过信号肽筛选、核糖体结合位点(RBS)优化和定点突变等手段,获得了一个具有较高催化能力的甲壳素酶Chisb,其比酶活达到249.62 U/mg。研究其酶学性质发现,该酶的最适pH为5.0,最适温度为60 ℃,Km和Kcat/Km分别为2.24 mmol/L和3.9 mmol/(L·s)。通过离子色谱检测其水解产物发现,以胶体甲壳素为底物,该酶最终可水解得到4种不同聚合度的几丁寡糖[9]。
如前所述,研究者已经运用多种手段对甲壳素酶进行了研究。但运用易错PCR[10]或DNA改组[11-12]的方式对甲壳素酶分子进行定向改造的研究较少。易错PCR是利用Taq DNA多聚酶不具有3′→5′校对功能的特性,在一定条件下(如不同的dNTPs浓度、Mg2+浓度和MnCl2存在)能够按较高的机率引入随机突变的一种技术。本研究旨在前期研究基础上,采用易错PCR技术将随机突变引入来源于芽孢杆菌属Bacillus sp. DAU101的甲壳素酶Chisb,经过96孔板初筛和摇瓶复筛,获得催化效率进一步提高的突变体,为实现其在高效生物转化合成几丁寡糖中的应用奠定基础。
1 材料与方法 1.1 菌株与质粒大肠杆菌Escherichia coli JM109、枯草芽孢杆菌Bacillus subtilis WB600和重组菌株R13NprB-C-SP-H[9]均为本实验室保藏;质粒pP43NMK_R13NprB-chisb-sp-his[9]为本实验室构建;质粒pP43NMK_MR13NprB-chisb-sp-his为本研究中构建。
1.2 试剂与培养基试剂:可调式易错PCR试剂盒(CAT#: 160903-100,北京天恩泽基因科技有限公司);几丁寡糖标准品(聚合度1–5)购自上海甄准生物科技有限公司;甲壳素粉末(C7170,SIGMA-ALDRICH);改良型Bradford法蛋白浓度测定试剂盒(生工生物工程(上海)股份有限公司);NuPAGE® Novex® Bis-Tris预制凝胶及标准蛋白Marker (Thermo Fisher scientific);氨苄青霉素(CAS 69-52-3)和卡那霉素(CAS 25389-94-0)购自生工生物工程(上海)股份有限公司;其他常用试剂均为分析纯。
培养基:种子培养基(Luria-Bertani,LB):酵母粉5 g/L,蛋白胨10 g/L,NaCl 10 g/L。固体培养基添加2%的琼脂粉。
发酵培养基(Terrific-Broth,TB):酵母粉24 g/L,蛋白胨12 g/L,甘油4 mL/L,K2HPO4 72 mmol/L,KH2PO4 17 mmol/L。
1.3 易错PCR扩增chisb基因片段以质粒pP43NMK_R13NprB-chisb-sp-his为模板,扩增chisb片段,所用引物如表 1所示。易错PCR反应体系(30 μL):Taq DNA聚合酶(5 U/mL) 1 μL,10×易错PCR Mix 3 μL,10×dNTPs 3 μL,MnCl2 4 μL,dGTP 4 μL,易错PCR引物各1 μL,DNA模板1 μL,超纯水12 μL。用1%的琼脂糖凝胶电泳鉴定PCR产物,并使用胶回收试剂盒回收PCR产物。反应条件:94 ℃ 3min;94 ℃ 1 min,45 ℃ 1 min,72 ℃ 1 min,重复30个循环;72 ℃ 5 min。
| Primer name | Primer sequence (5′–3′) |
| chisb1-F1 | GGTTATTACCCTTCTTGGGGTGCCTACGGACGGGATTTTCAGGTGTGGG |
| chisb1-R1 | CGCCAGCTTCCTTGTAGCGCTTCACCGTGCTCTCAATATTAAACGTTTCTGCG |
| chisb1-F2 | GGTTATTACCCTTCTTGGGGTGCCTACGGACGGG |
| chisb1-R2 | CGCCAGCTTCCTTGTAGCGCTTCACCGT |
| chisb1-F3 | GGTTATTACCCTTCTTGGGGTGCC |
| chisb1-R3 | GCTTCACCGTGCTCTCAATATTAAACG |
| chisb2-F1 | GTTCTATGACCCGAAGGCAAAAGAGGCAGGGGTGCCGAACGC |
| chisb2-R1 | CGCCAAGCTTTCATCATTACTTGCAGTCGCCTATTAACTTCCAAACGCCTGACTC |
| chisb2-F2 | GTTCTATGACCCGAAGGCAAAAGAGGCAGGGGTG |
| chisb2-R2 | CGCCAAGCTTTCATCATTACTTGCAGTCGCC |
| chisb2-F3 | GTTCTATGACCCGAAGGCAAAAGAGG |
| chisb2-R3 | CGCCAAGCTTTCATCATTACTTGCA |
| P43-F | GTTTGGAAGTTAATAGGCGACTGCAAGTAATGATGAAAGCTTGGCGTAATCATGG |
| P43-R | CACCTGAAAATCCCGT CCG TAGGCACCCCAAGAAGGGTAATAACC |
使用表 1中P43-F和P43-R引物进行线性化扩增质粒pP43NMK_R13NprB-chisb-sp-his。用1%的琼脂糖凝胶电泳鉴定PCR产物,并使用胶回收试剂盒回收PCR产物。回收的片段chisb1、chisb2和载体pP43NMK采用一步克隆试剂盒(ClonExpress® Ⅱ,Vazyme Biotech Co.,Ltd)连接,构建突变重组质粒pP43NMK_MR13NprB-chisb- sp-his。
1.5 重组质粒转化与平板菌落PCR鉴定将突变的重组质粒转化至E. coli JM109中,涂布氨苄青霉素抗性平板,于37 ℃培养8–12 h。待平板长出菌落后,随机挑选单菌落进行平板菌落PCR,验证转化子的阳性克隆概率。
1.6 96孔板初筛与摇瓶复筛将平板上的菌落接种到LB液体培养基中培养一段时间,提取质粒,转化至B. subtilis WB600中,涂布卡那霉素抗性平板,37 ℃培养12 h。待平板长出菌落后,首先将单菌落接种至96孔板中,用LB培养基进行种子培养,8 h后转接至TB培养基发酵培养12 h后测定酶活大小。
将初筛得到的正向突变体进行摇瓶复筛,培养方法与初筛一致。将复筛得到的正向突变体送至苏州金唯智有限公司进行测序,确定突变位点。
1.7 突变体活性与蛋白浓度的测定以胶体甲壳素作为底物测定酶活大小。500 μL反应体系包含66 μL 10%的胶体甲壳素、334 μL磷酸氢二钠-柠檬酸盐缓冲液(pH 5.0)和纯化的突变体的反应混合物在60 ℃振荡15 min后,100 ℃加热5 min终止反应,14 000 r/min离心5 min[9]。使用改进的3, 5-二硝基水杨酸(DNS)法在540 nm处测定上清液中还原糖的量[13]。一个单位的甲壳素酶酶活定义为在60 ℃、pH 5.0的条件下,每小时释放1 μmol还原糖所需的酶量。用GlcNAc测定标准曲线,加热灭活的酶作为对照。使用Bradford蛋白含量测定试剂盒(Sangon Biotech Co.,Ltd)[14],以牛血清白蛋白为标准对蛋白质含量进行测定。
1.8 突变体纯化与酶学性质研究 1.8.1 突变体纯化将发酵液于4 ℃、12 000 r/min离心30 min,收集上清液。上清液通过0.45 μm滤膜除去杂质,以0.1 mL/min的流速直接上样至His GraviTrap柱(GE Healthcare,Tokyo)。上样后,用缓冲液A (40 mmol/L Tris-HCl,150 mmol/L NaCl,pH 7.5)平衡柱子,流速为0.2 mL/min。然后用缓冲液B (40 mmol/L Tris-HCl,150 mmol/L NaCl,500 mmol/L咪唑,pH 7.5)从5%–100%进行梯度洗脱,流速为0.2 mL/min,最后使用HiTrapTM Desalting脱盐柱进行脱盐,纯化后的酶液保存在4 ℃以备使用。纯化的蛋白用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDS-PAGE)[15]进行检测。
1.8.2 突变体酶学性质研究为测定温度对突变体酶活性的影响,根据1.7中突变体酶活的测定方法,测定25 ℃–70 ℃等温度下,突变体的酶活,确定其最适反应温度。
为测定pH对突变体酶活性的影响,根据1.7中突变体酶活的测定方法,选择pH 3.0–12.0的各种缓冲液测定突变体的最适反应pH。使用的缓冲液如下:磷酸氢二钠-柠檬酸盐缓冲液(pH 3.0–6.0),磷酸氢二钾-磷酸二氢钾缓冲液(pH 6.0–9.0),甘氨酸-氢氧化钠缓冲液(pH 9.0–12.0)。使用的所有缓冲液浓度均为20 mmol/L。
为测定不同金属离子对突变体酶活性的影响,分别用1、5和10 mmol/L的CoCl2、MnCl2、MgCl2、FeCl3、CuCl2、ZnCl2、CaCl2、KCl、AlCl3、BaCl2、NiSO4、C2H3O2Li等金属离子的盐溶液在标准酶活测定条件下反应,以不加金属离子的反应为对照,研究金属离子对突变体酶活的影响。
以不同浓度的胶体甲壳素为底物,测定突变体的动力学参数。使用Matlab软件拟合曲线,计算米氏常数(Km)和最大反应速率(Vmax),进而推导出特征常数Kcat和催化效率比Kcat/Km。
以上所有实验均进行3次,并取平均值和方差进行计算。
1.9 高效液相色谱(HPLC)分析水解产物收集12 h的对照菌株与突变体的发酵上清液,在3 g/L的底物终浓度下分别于55 ℃反应2、4、6、8、10、12、20、24 h。14 000 r/min离心10 min水解后的反应液,上清液用于HPLC分析(Agilent 1260 system,USA)。色谱柱:Shodex Asahipak NH2P-50 4E柱(4.6 mm×250 mm),流动相:乙腈:水=70:30;流速是0.6 mL/min,检测器用紫外检测器(VWD),波长210 nm,进样量10 μL,柱温30 ℃。
2 结果与分析 2.1 易错PCR扩增结果使用表 1的引物扩增片段chisb1和chisb2,chisb基因全长1 692 bp,分为两段进行扩增。chisb1片段长767 bp,chisb2片段长925 bp,扩增结果如图 1所示。扩增chisb1片段的3对引物中,只有chisb1-F1/R1能扩增出目的条带,其余2对引物扩增都没有明显条带;而用引物chisb2- F2/R2扩增chisb2片段的效果没有chisb2-F1/R1和chisb2-F3/R3引物的扩增效果好。所以选用引物chisb1-F1/R1扩增chisb1片段,引物chisb2- F1/R1扩增chisb2片段,引入随机突变位点。
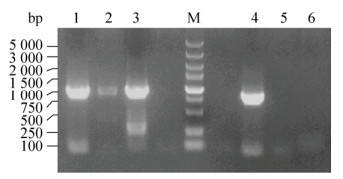
|
| 图 1 chisb1和chisb2的凝胶电泳图 Fig. 1 The gel electrophoresis of chisb1 and chisb2. M: marker; 1: chisb2-F1/R1; 2: chisb2-F2/R2; 3: chisb2-F3/R3; 4: chisb1-F1/R1; 5: chisb1-F2/R2; 6: chisb1-F3/R3. |
| |
引物P43-F和P43-R扩增得到的线性化质粒与回收的chisb1和chisb2片段采用一步克隆试剂盒进行连接,转化E. coli JM109,可在氨苄平板上得到约1 000个单菌落,随机挑选100个单菌落进行PCR验证,用1%的琼脂糖凝胶电泳检测条带大小,结果显示全部为阳性克隆。随机挑选8个单菌落的验证条带如图 2所示。可以看出,chisb片段已成功转入E. coli JM109中。将平板上的单菌落接种于LB液体培养基进行培养,然后提取重组质粒,转化至B. subtilis WB600,在卡那霉素平板上长出约700个阳性克隆。

|
| 图 2 基因chisb的凝胶电泳图 Fig. 2 The gel electrophoresis of chisb. M: marker; 1–8: chisb. |
| |
将卡那霉素平板上长出的约700个阳性克隆接种于含LB液体培养基的96孔板中,培养8 h后,再转接至TB发酵培养基中培养12 h,12 000 r/min离心10 min后取上清液进行酶活测定。结果显示,其中24株突变体的酶活高于对照。将这24株突变体进行摇瓶复筛鉴定,采用同样的方法进行培养和酶活测定,其中2株突变体的活力显著高于其他突变体。经过序列比对发现,分别是第43位半胱氨酸突变为天冬氨酸(C43D)和第336位谷氨酸突变为精氨酸(E336R)。
2.4 突变体的纯化为了进一步研究突变体的酶学性质,将摇瓶复筛得到的2个突变体进行了镍柱纯化。结果如图 3和表 2所示。由图 3可以看出,突变体C43D和E336R经过镍柱纯化后完全被10%的B液洗脱,在62 kDa处得到一条单一的条带。由表 2可见,经过镍柱纯化后的突变体酶与粗酶相比,比酶活都得到了提高,突变体C43D和E336R的纯化倍数分别是之前的3.05倍和3.69倍,得率分别为80.77%和85.45%。
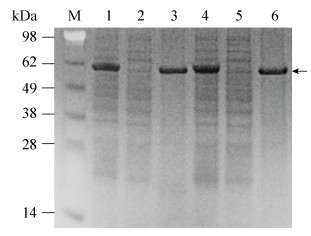
|
| 图 3 突变体的SDS-PAGE分析 Fig. 3 SDS-PAGE analysis of the mutants. M: marker; 1: the fermentation supernatant of C43D; 2: flow-through of C43D; 3: effluent of 10% buffer B of C43D; 4: the fermentation supernatant of E336R; 5: flow-through of E336R; 6: effluent of 10% buffer B of E336R. |
| |
| Mutants | Purification step | Enzyme activity (U/mL) | Protein concentration (mg/mL) | Specific activity(U/mg) | Purified fold | Recovery rate (%) |
| C43D | Crude enzyme | 58.97 | 0.68 | 86.72 | 1.00 | 100.00 |
| Ni-chelating affinity | 47.63 | 0.18 | 264.61 | 3.05 | 80.77 | |
| chromatography | ||||||
| E336R | Crude enzyme | 62.33 | 0.82 | 76.01 | 1.00 | 100.00 |
| Ni-chelating affinity | 53.26 | 0.19 | 280.32 | 3.69 | 85.45 | |
| chromatography |
由图 4A可以看出,突变体C43D和E336R的最适温度都为55 ℃,当温度高于55 ℃,突变体活性迅速降低。图 4B显示了突变体C43D的最适pH为5.0,pH大于5.0,突变体活性缓慢下降;而E336R的最适pH为9.0,当pH在3.0–6.0之间时,E336R的活性呈上升趋势,pH高于6.0活性下降,但在9.0时活性显著上升,pH高于9.0又迅速下降。
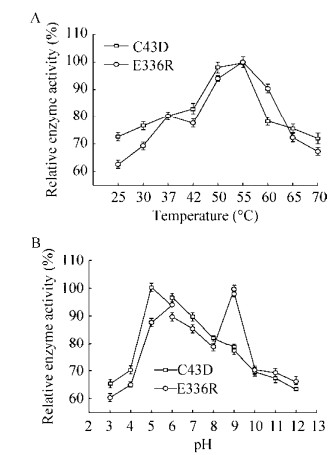
|
| 图 4 温度(A)和pH (B)对突变体活性的影响 Fig. 4 Effects of temperature (A) and pH (B) on mutants activity. |
| |
通过测定金属离子对突变体酶活的影响(如图 5所示),发现15 mmol/L的Mn2+对两株突变体的活性具有显著的促进作用,高浓度的Ba2+对突变体的活性也有促进作用。而Ni+、Mg2+、Ca2+和K+对突变体C43D的活性影响不大,但无论是高浓度还是低浓度的Fe3+、Cu2+和Al3+都对C43D突变体活性具有抑制作用;与C43D突变株一样,Fe3+、Cu2+和Al3+也会抑制E336R的活性。除此之外,高浓度的Zn2+也会抑制其活性。
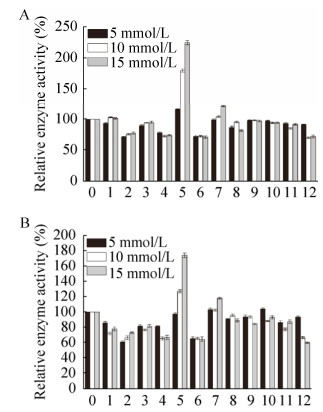
|
| 图 5 金属离子对突变体酶活的影响 Fig. 5 Effects of metal ions on mutants activity. (A) The mutant of C43D. (B) The mutant of E336R. 0–12: no metal ions, Ni+, Fe3+, Co2+, Cu2+, Mn2+, Al3+, Ba2+, Li+, Mg2+, Ca2+, K+, Zn2+. |
| |
以胶体甲壳素作为底物,对两个突变体的动力学参数也进行了测定。软件拟合曲线如图 6所示,计算结果如表 3所示。实验发现,与对照相比,突变体C43D和E336R的Km值降低,说明底物亲和力增加;催化常数Kcat升高,催化效率Kcat/Km相比对照分别提高了1.35倍和1.57倍。

|
| 图 6 突变体C43D (A)和E336R (B)的动力学拟合曲线 Fig. 6 Kinetic fitting curves of mutant C43D (A) and E336R (B). |
| |
结果如图 7所示。由图 7A可以看出,对照(原始酶)水解胶体甲壳素生成的几丁寡糖总量为0.89 g/L,底物转化率为29.7%,水解产物以(GlcNAc)2为主。而突变体E336R水解胶体甲壳素产生的几丁寡糖总量始终高于C43D突变体,其催化24 h后几丁寡糖总量达到最大,分别为2.53 g/L和2.06 g/L,转化率分别为84.3%和68.7% (图 7B,7C)。进一步分析发现,突变体的催化水解产物都为GlcNAc、(GlcNAc)2、(GlcNAc)3和(GlcNAc)5,但是反应8 h之后,检测不到(GlcNAc)3的生成。其中(GlcNAc)2含量最高,反应至24 h时,C43D和E336R突变体生成的(GlcNAc)2分别占总糖含量的71.8%和57.7%。

|
| 图 7 突变体的水解产物分析 Fig. 7 Analysis of the hydrolysates of the mutants. (A) The hydrolysates of original enzyme. (B) The hydrolysates of E336R. (C) The hydrolysates of C43D. |
| |
甲壳素酶在农业、医药和生物防治方面具有广泛的应用。但是野生菌产的甲壳素酶活性较低,对于工业化生产甲壳素酶有一定差距[16]。随着对甲壳素酶研究的深入,研究者多采用在大肠杆菌和毕赤酵母中进行异源表达的方式生产甲壳素酶[17-19]。除此之外,还对甲壳素酶的特性和结构进行了细致的研究。
目前,运用易错PCR和定点突变技术是对甲壳素酶进行改造的有效方式[20-21]。易错PCR是一种使复制的DNA序列出现错误的一种PCR技术,它通过保真度低的Taq DNA聚合酶,使DNA复制的保真度降低,从而在合成的新链中增加错配的碱基,达到产物出现多个点突变的效果[22]。而运用易错PCR对甲壳素酶进行定向进化,可能成为一种提高甲壳素酶催化活性的有效方式。本研究中,以前期获得的产甲壳素酶的重组菌R13NprB-C-SP-H为出发菌株,利用随机突变和高通量筛选的方法获得了底物亲和力、催化效率和比活性都明显提高的甲壳素酶突变体C43D和E336R。
通过对两种甲壳素酶突变体的酶学性质进行测定发现,突变体的最适温度(55 ℃)相比未突变的酶(60 ℃)要低,突变体在60 ℃时,活性显著降低,这可能是位于甲壳素酶催化结构域附近的天冬氨酸,其羧基在高温下不稳定而半胱氨酸的巯基更加稳定导致的。比较最适pH发现,C43D突变体没有发生变化,E336R突变体的最适pH为9.0,造成这一现象的原因其一可能是:由于336位的谷氨酸为酸性氨基酸,带负电荷,当突变为精氨酸后,精氨酸为碱性氨基酸,突变后原本结构中的酸性基团变为碱性基团,在pH为9.0的缓冲溶液中,蛋白带正电荷,在碱性环境下更稳定;其二是由于突变为Arg之后,Arg与周围相邻的其他氨基酸形成了新的氢键网络和盐桥结构,导致突变体的pH发生改变[23-24]。而C43D突变体中的天冬氨酸为酸性氨基酸,带负电荷,它会影响断裂位点周围电离基团的pKa值,在原本酸性的环境下,使得突变体C43D的最适pH保持不变[25]。除此之外,天冬氨酸在甲壳素酶的催化反应中还参与配体的催化和结合。2012年,Suginta等[26]报道了来源于哈氏弧菌的甲壳素酶A,其催化结构域附近的Asp313的羧基可以与N-乙酰基团中的-C=O (羰基)相互作用,从而使得生成的产物构像发生改变。本研究中43位的半胱氨酸位于甲壳素酶Chisb的催化结构域附近,当突变为天冬氨酸,其羧基可以与底物乙酰基团中的羰基发生相互作用,而半胱氨酸的巯基与羰基的相互作用较弱,因此突变后底物亲和力增加,说明催化结构域附近的天冬氨酸在甲壳素酶的底物结合中具有重要作用[27]。
当以胶体甲壳素作为底物对突变体的动力学参数进行测定,我们发现,相比未突变酶的米氏常数(Km)为2.24 mmol/L,突变体C43D和E336R的Km值降低,说明突变体对底物的亲和力增加;特别是突变体E336R,由于精氨酸结构中的侧链比谷氨酸的侧链长,会导致底物的结合口袋变大,增加了酶与底物的结合能力,更多的底物会进入到底物结合口袋,因此底物亲和力显著增加;另外,精氨酸结构中所含有的阳离子不会阻碍底物结合,底物可以自由进入到底物结合口袋,使底物亲和力增加。同时,突变体的催化效率(Kcat/Km)相比未突变的酶的催化效率(3.9 L/(s·mmol),也分别提高了1.35倍和1.57倍。分析甲壳素酶Chisb的三维蛋白结构[9, 28],C43D突变体中的天冬氨酸,它可以和几丁寡糖分子中(-1)位的非还原性糖基结合形成氢键,稳定甲壳素酶的催化活性;而E336R突变体中的精氨酸与甲壳素酶Chisb周围相邻的氨基酸形成了新的盐桥,从而提高了催化效率[29-30]。
综上所述,我们的研究证明了随机突变结合高通量筛选技术是提高甲壳素酶催化效率及其底物亲和力的一种有效手段,这为利用甲壳素酶高效生物转化合成几丁寡糖奠定了一定基础。
| [1] |
Boller T, Gehri A, Mauch F, et al. Chitinase in bean leaves: induction by ethylene, purification, properties, and possible function. Planta, 1983, 157(1): 22-31. |
| [2] |
Schlumbaum A, Mauch F, Vögeli U, et al. Plant chitinases are potent inhibitors of fungal growth. Nature, 1986, 324(6095): 365-367. DOI:10.1038/324365a0 |
| [3] |
Jiang HB, Zhang Y, Jiang QL, et al. Advances in the research of chitinase. Shandong Sci, 2000, 13(4): 41-45 (in Chinese). 蒋红彬, 张瀛, 蒋千里, 等. 几丁质酶的研究概况. 山东科学, 2000, 13(4): 41-45. |
| [4] |
Huang CJ, Chen CY. High-level expression and characterization of two chitinases, ChiCH and ChiCW, of Bacillus cereus 28-9 in Escherichia coli. Biochem Biophys Res Commun, 2005, 327(1): 8-17. |
| [5] |
Guo XX, Xu P, Zong MH, et al. Purification and characterization of alkaline chitinase from Paenibacillus pasadenensis CS0611. Chin J Cataly, 2017, 38(4): 665-672. |
| [6] |
Zhang AL, He YM, Wei GG, et al. Molecular characterization of a novel chitinase CmChi1 from Chitinolyticbacter meiyuanensis SYBC-H1 and its use in N-acetyl-D-glucosamine production. Biotechnol Biofuels, 2018, 11: 179. DOI:10.1186/s13068-018-1169-x |
| [7] |
Madhuprakash J, Singh A, Kumar S, et al. Structure of chitinase D from Serratia proteamaculans reveals the structural basis of its dual action of hydrolysis and transglycosylation. Int J Biochem Mol Biol, 2013, 4(4): 166-178. |
| [8] |
Fan XJ, Yang C, Zhang C, et al. Cloning, site-directed mutagenesis, and functional analysis of active residues in Lymantria dispar chitinase. Appl Biochem Biotechnol, 2017, 184(1): 12-24. |
| [9] |
Pan MY, Li JH, Lü XQ, et al. Molecular engineering of chitinase from Bacillus sp. DAU101 for enzymatic production of chitooligosaccharides. Enzyme Microb Technol, 2019, 124: 54-62. DOI:10.1016/j.enzmictec.2019.01.012 |
| [10] |
Xu HF, Zhang XE, Zhang YM, et al. Recent advances in vitro molecular directed evolution. Prog Biochem Biophys, 2002, 29(4): 518-522 (in Chinese). 徐卉芳, 张先恩, 张用梅, 等. 体外分子定向进化研究进展. 生物化学与生物物理进展, 2002, 29(4): 518-522. DOI:10.3321/j.issn:1000-3282.2002.04.005 |
| [11] |
Stemmer WP. DNA shuffling by random fragmentation and reassembly: in vitro recombination for molecular evolution. Proc Natl Acad Sci USA, 1994, 91(22): 10747-10751. DOI:10.1073/pnas.91.22.10747 |
| [12] |
Moore JC, Jin HM, Kuchner O, et al. Strategies for the in vitro evolution of protein function: enzyme evolution by random recombination of improved sequences. J Mol Biol, 1997, 272(3): 336-347. |
| [13] |
Lee YS, Park IH, Yoo JS, et al. Cloning, purification, and characterization of chitinase from Bacillus sp. DAU101. Bioresour Technol, 2007, 98(14): 2734-2741. DOI:10.1016/j.biortech.2006.09.048 |
| [14] |
Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72(1/2): 248-254. |
| [15] |
Laemmli UK. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature, 1970, 227(5259): 680-685. DOI:10.1038/227680a0 |
| [16] |
Brzezinska MS, Jankiewicz U, Walczak M. Biodegradation of chitinous substances and chitinase production by the soil actinomycete Streptomyces rimosus. Int Biodeter Biodegr, 2013, 84: 104-110. DOI:10.1016/j.ibiod.2012.05.038 |
| [17] |
Gao C, Zhang AL, Chen KQ, et al. Characterization of extracellular chitinase from Chitinibacter sp. GC72 and its application in GlcNAc production from crayfish shell enzymatic degradation. Biochem Eng J, 2015, 97: 59-64. DOI:10.1016/j.bej.2015.02.010 |
| [18] |
Wang D, Li AJ, Han HY, et al. A potent chitinase from Bacillus subtilis for the efficient bioconversion of chitin-containing wastes. Int J Biol Macromol, 2018, 116: 863-868. DOI:10.1016/j.ijbiomac.2018.05.122 |
| [19] |
Nguyen HQ, Vu VH, Le PD, et al. High-level expression, purification and properties of an Endochitinase gene without signal peptide from Lecanicillium lecanii 43H in Pichia pastoris. Mol Biol Rep, 2018, 45(5): 1067-1075. DOI:10.1007/s11033-018-4256-y |
| [20] |
Wang AX, Li W, Chen XL, et al. Optimization of the mutation conditions of Trichoderma harzianum chitinase gene using Error-prone PCR. J Northeast Agric Univ, 2013, 44(10): 134-138 (in Chinese). 王傲雪, 李微, 陈秀玲, 等. Error-prone PCR致哈茨木霉几丁质酶突变的条件优化. 东北农业大学学报, 2013, 44(10): 134-138. DOI:10.3969/j.issn.1005-9369.2013.10.024 |
| [21] |
Yun CS, Matsuda H, Kawamukai M. Directed evolution to enhance secretion efficiency and thermostability of chitosanase from Mitsuaria chitosanitabida 3001. Biosci Biotechnol Biochem, 2006, 70(2): 559-563. DOI:10.1271/bbb.70.559 |
| [22] |
Cadwell RC, Joyce GF. Randomization of genes by PCR mutagenesis. PCR Methods Appl, 1992, 2(1): 28-33. DOI:10.1101/gr.2.1.28 |
| [23] |
Liu YH, Lu FP, Yu L, et al. Acid stabilization of Bacillus licheniformis alpha amylase through introduction of mutations. Appl Microbiol Biotechnol, 2008, 80(5): 795-803. DOI:10.1007/s00253-008-1580-5 |
| [24] |
Binter A, Staunig N, Jelesarov I, et al. A single intersubunit salt bridge affects oligomerization and catalytic activity in a bacterial quinone reductase. FEBS J, 2009, 276(18): 5263-5274. DOI:10.1111/j.1742-4658.2009.07222.x |
| [25] |
Kolstad G, Synstad B, Eijsink VGH, et al. Structure of the D140N mutant of chitinase B from Serratia marcescens at 1.45 Å resolution. Acta Crystallogr D Biol Crystallogr, 2002, 58: 377-379. DOI:10.1107/S0907444901018972 |
| [26] |
Suginta W, Sritho N. Multiple roles of Asp313 in the refined catalytic cycle of chitin degradation by Vibrio harveyi chitinase A. Biosci Biotechnol Biochem, 2012, 76(12): 2275-2281. DOI:10.1271/bbb.120559 |
| [27] |
Watanabe T, Uchida M, Kobori K, et al. Site-directed mutagenesis of the Asp-197 and Asp-202 residues in chitinase A1 of Bacillus circulans WL-12. Biosci Biotechnol Biochem, 1994, 58(12): 2283-2285. DOI:10.1271/bbb.58.2283 |
| [28] |
Matsumoto T, Nonaka T, Hashimoto M, et al. Three-dimensional structure of the catalytic domain of chitinase A1 from Bacillus circulans WL-12 at a very high resolution. Proc Jpn Acad Ser B, 1999, 75(9): 269-274. DOI:10.2183/pjab.75.269 |
| [29] |
Yang HQ, Liu L, Shin HD, et al. Structure-guided systems-level engineering of oxidation-prone methionine residues in catalytic domain of an alkaline α-amylase from Alkalimonas amylolytica for significant improvement of both oxidative stability and catalytic efficiency. PLoS ONE, 2013, 8(3): e57403. DOI:10.1371/journal.pone.0057403 |
| [30] |
Gumpena R, Kishor C, Ganji RJ, et al. Glu121-Lys319 salt bridge between catalytic and N-terminal domains is pivotal for the activity and stability of Escherichia coli aminopeptidase N. Protein Sci, 2012, 21(5): 727-736. DOI:10.1002/pro.2060 |
 2019, Vol. 35
2019, Vol. 35




