中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘洋, 冯晋文, 刘万霖, 秦钧, 丁琛, 贺福初
- Liu Yang, Feng Jinwen, Liu Wanlin, Qin Jun, Ding Chen, He Fuchu
- 小鼠肝脏蛋白质组数据门户
- Mouse liver proteome database
- 生物工程学报, 2019, 35(9): 1715-1722
- Chinese Journal of Biotechnology, 2019, 35(9): 1715-1722
- 10.13345/j.cjb.190166
-
文章历史
- Received: April 28, 2019
- Accepted: June 21, 2019
2. 复旦大学 生命科学学院 上海 200438;
3. 国家蛋白质科学中心·北京,北京 102206
2. School of Life Sciences, Fudan University, Shanghai 200438, China;
3. National Center for Protein Sciences·Beijing, Beijing 102206, China
肝脏是脊椎动物体内最大的器官,它在维持代谢稳态、合成生物体必需的物质以及对外源物的解毒等方面起着非常关键的作用[1]。除了其生物学功能外,肝脏的生理学特征也很独特,例如肝脏的再生功能[2]、节律特征[3]等。研究肝脏不同功能的分子机制对认识和治疗肝脏疾病起着非常重要的作用。肝脏蛋白质组计划(The Human Liver Proteome Project)[4]作为第一个在器官层面的蛋白质组工程在过去的十几年中取得了很多的成果,越来越多的研究利用基于质谱的蛋白质组学技术[5-7]来描绘揭示肝脏在不同生理和病理条件下的蛋白质分子特征。
应用液相串联质谱技术,刘明伟等在亚细胞蛋白质组学层面揭示了脂滴在肝脏脂肪代谢平衡中的作用[8];Azimifar等在细胞分辨率水平揭示了肝脏中不同细胞类型所承担的功能[9];丁琛等进一步地揭示了它们通过信号传递来协作行使肝脏的各种生物学功能[10];王云之等和Wang等在昼夜节律中通过肝脏蛋白质组的动态变化揭示节律调控网络和机制[11-12];刘晓伟等研究了在脂多糖(LPS)刺激下肝损伤的形成机理[13];Hsieh等研究阐释了在部分肝切除后组织再生的分子机理[14-15]。目前,针对小鼠肝脏的功能性研究已经积累了数千组高质量的蛋白质组数据。上述研究均是通过比较不同条件下小鼠肝脏蛋白质表达谱的变化,从而获得和变化条件相关的蛋白质,然后去探究它们在不同条件下承担的功能。但是这些蛋白质表达谱中仍然有很多知识和新发现等待进一步挖掘,然而目前还没有可用的数据库系统性地整理、分析和展示小鼠肝脏的蛋白质组数据。
因此,我们建立了首个小鼠肝脏蛋白质组学门户,来呈现小鼠肝脏中的蛋白质在不同生理和病理状态下的表达情况,分析出不同条件下表达有差异的蛋白质以及它们参与的生物学功能和信号通路,为研究者提供和不同条件存在潜在关联的蛋白以供参考和验证,并为实验提供数据支持。
1 数据收集和处理 1.1 元数据和蛋白质组数据的收集门户网站共包含834组关于小鼠肝脏的蛋白质组实验数据,其中60组为2010–2017年间已发表的小鼠肝脏的蛋白质组数据集,从文献中筛选实验数据的标准是:研究对象是小鼠肝脏的蛋白质组;蛋白质组分析技术是基于质谱仪器;定量方法采用非标定量[16]技术。774组实验数据来自笔者的科研团队。这些实验包含464组蛋白全谱[17] (Profiling)和310组转录因子DNA结合活性谱[18] (catTFRE),catTFRE技术是利用转录因子可与序列特异性DNA元件结合的特点,合成了100种转录因子结合序列的串联多拷贝双链DNA结合元件,将其用生物素标记以包装成DNA诱饵富集细胞中内源性转录因子,将被DNA诱饵捕获的内源性转录因子利用串联质谱技术进行定性和定量。定量方法同样是非标定量。实验的基本信息按照表 1中提供的字段进行整理。
| Field | Example 1 | Example 2 |
| Strain | C57 | C57 |
| Sex | Male | Female |
| Age (d) | 70 | 70 |
| Tissue | Liver | Liver |
| Genotype | Wide type | Knock out Lep |
| Treatment | HighFatDiet | PHx |
| Duration (h) | 840 | 72 |
| Dosage | 50 mg/(kg·d) | |
| Circadian | ||
| Celltype | Whole tissue extract | Hepatocytes |
| Organelle | LD | Whole cell extract |
| Strategy | Profiling | catTFRE |
对本实验室产生的蛋白质组数据运算进行了严格的质量控制,利用Mascot[19]软件对质谱产生的谱图作鉴定,控制肽段和谱图匹配的错误发现率(FDR)小于1%,对匹配得到的肽段采用以基因为中心的蛋白定性和定量算法[20],用iBAQ[21]值作为基因在蛋白质层面的表达量。在这834组实验中一共鉴定到11 471个基因产物。再根据以下两个条件的:1)基因表达产物至少被鉴定到2个唯一性肽段;2)至少在5次实验中被鉴定到;筛选得到10 595个高可信度的小鼠肝脏表达的蛋白质。
从文献中收集得到的数据则根据它提供的质谱数据处理流程和最终的数据表格进行统一的处理,对利用MaxQuant[22]软件进行蛋白的定性和定量的实验数据,利用蛋白表达量列表里的峰度值计算iBAQ,用唯一的肽段数筛选高可信度的蛋白,对同一个基因表达的多个蛋白计算总和作为基因的表达量。
1.3 数据预处理由于这些数据来自的样本不同、处理方式不同、检测仪器不同,需要对所有实验的蛋白质表达量进行标准化,834组实验根据实验策略和实验材料分开进行数据的标准化。采用的标准化方法是分位数标准法[23],这一方法的假设前提是每组实验蛋白质表达量的分布一致,所以对实验策略一致并且实验材料为同一细胞类型或者同一细胞系的所有实验整合在一起对表达量iBAQ进行标准化,计算方法为:对整合在一起的实验计算每组实验的五分位数qk=(qk1, ···, qkn)
根据所有实验的每一个分位数值计算平均值:

|
将每次实验的5个五分位数值调整为所有实验对应的五分位数的平均值,再根据每次实验调整后的分位数值和原始值的倍数缩放每次实验所有蛋白质的表达量。
最后根据国家生物技术信息中心(NCBI)的基因信息数据库Entrez Gene[24],将数据集中采用不同数据库(Ensemble、Uniprot)的基因名都转换成Entrez Gene数据库中的基因名,并把Entrez GeneID作为基因的唯一标识符。
1.4 基因集功能分析方法基因集的功能分析方法是利用Gene Ontology[25] (GO)的生物学过程条目做富集分析[26]。该方法的输入数据是需要功能分析的基因名集合,计算过程首先是计算该基因集与在GO层次关系中处于最底层的GO条目之间的富集程度,用Fisher精确检验的P值来表征该富集程度,在计算上一层的GO条目时移除在子条目中出现的基因,然后再计算富集程度。最后挑选出富集程度较高的GO条目,作为基因集的功能。采用的程序来自R程序包topGO。
2 结果与分析 2.1 小鼠肝脏蛋白质组数据门户概览小鼠肝脏蛋白质组数据门户从质谱数据中获得了10 595个高可信度的小鼠肝脏蛋白质在不同生理和病理条件下的表达数据,在这些蛋白质中包含了660个转录因子。门户网站也包含了细胞核、线粒体和脂滴这3种细胞系中的蛋白质组图谱(图 1A)。根据GO的注释将门户网站中的小鼠肝脏蛋白质组作功能分析[27],发现这些蛋白质的功能主要集中在:生命体所需物质的代谢,如氨基酸和脂质的代谢、蛋白的成熟和降解、膜转运和能量代谢;维持生命体正常运转的功能,如细胞周期和凋亡;以及在免疫系统中起到一定的作用(图 1B)。

|
| 图 1 门户概览(A:小鼠肝脏中蛋白质鉴定情况;B:肝脏蛋白功能,每一块多边形代表一类蛋白的功能,多边形的不同颜色代表不同的GO功能类别,多边形大小代表蛋白承担功能的比重) Fig. 1 Overview of Mouse Liver Portal. (A) Identified genes products in mouse liver. (B) Mouse liver proteome function. Every tile represents one type of proteins. Tiles are arranged and colored according to the hierarchical GO terms. Tile sizes represent the mass fractions of proteins. |
| |
小鼠肝脏蛋白质组数据门户提供了操作方便的比较功能。如图 2所示,用户可以选择两种不同的条件,比较在这两种不同条件下蛋白表达的变化情况。通过点击基因搜索框右侧的加号按钮得到筛选不同条件的字段:Strain、Gender、Genotype、Gene、Cell Type、Organelle、Treatment、Time,通过选择不同的条件信息生成两种类型,然后点击搜索便能得到在两种条件下蛋白的表达情况。

|
| 图 2 小鼠肝脏不同生理和病理状态条件的选择页面 Fig. 2 Page of choosing different physiological and pathological conditions. |
| |
比较结果页面中会展示蛋白表达情况和功能分析结果,图 3展示了肝实质细胞和Kupffer细胞中蛋白质表达的比较情况,通过箱形图形象地展示了同一蛋白质在两种条件下表达的高低情况(表达量是标准化iBAQ值的log转换),也能看到蛋白质表达量的平均值,可以用来比较不同蛋白质表达的高低情况。比较功能的结果页还展示了在两种不同条件下蛋白质的表达量在统计学上是否有显著的差异,方法是对每个蛋白质表达量的两组数据进行t检验,对计算的P作做多重假设检验的矫正,将FDR值小于0.05的作为有显著差异的蛋白。
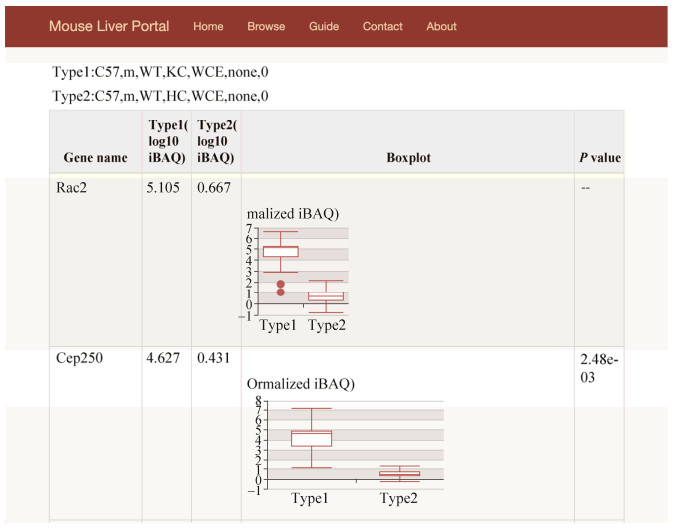
|
| 图 3 雄性C57小鼠肝脏的肝实质细胞和Kupffer细胞中蛋白质的表达情况和差异比较 Fig. 3 Proteins' expression and difference in liver parenchyma cells and Kupffer cells of C57 male mice. |
| |
比较结果页面还会提供表达量差异倍数在5倍以上并且该差异在统计上显著的蛋白质功能,采用的是topGO基因集功能分析方法,图 4展示了Kupffer细胞中相较于肝实质细胞特异性高表达蛋白质的功能,这些蛋白质会参与免疫应答、呈递抗原等功能。比较结果分析页面还会展示潜在的转录因子和靶基因的调控作用网络,如果用户选择的条件有采用catTFRE实验策略产生的数据,门户网站会筛选出catTFRE实验中有差异的转录因子和profiling中有差异表达的蛋白,根据CellNet[28]提供的转录因子和靶基因的调控关系,展示这些变化的蛋白之间存在的调控网络。
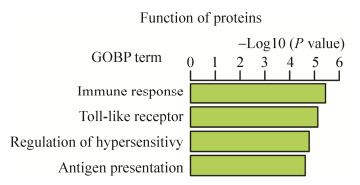
|
| 图 4 Kupffer细胞中特异性高表达蛋白在GO生物学过程条目中富集得到的功能 Fig. 4 The functions of specific expressed proteins in Kupffer cell which enriched with GO biological process terms. |
| |
在网站的首页用户可以输入感兴趣的蛋白质,在结果展示页面(图 5)会显示该蛋白质的基本信息,这些信息来自UniProt数据库,页面下方会展示该蛋白质在不同小鼠品系、不同性别、不同年龄阶段、不同细胞类型、不同细胞系中的表达水平,以及它在高脂饮食、节律和部分肝切除实验中不同时间点的表达水平。除了蛋白质在每个属性不同条件下的表达高低情况,搜索结果页面还提供了该蛋白表达量在每种属性下不同条件的两两之间是否存在显著差异,根据计算得到的P值大小显示不同深浅的红色。从图 5中可以看到Hnf4a在细胞核提取物中的浓度要显著高于它在全细胞中的浓度。

|
| 图 5 Hnf4a在不同条件下(两种品系的小鼠、雌雄小鼠、多种细胞类型、多种细胞器和肝切除后多个时间点)的表达情况和差异显著性 Fig. 5 Hnf4a's expression and significance of difference under different condition (two strains of mice, male and female mice, multiple cell types, multiple organelles, and multiple time points after hepatectomy). |
| |
用户查询的基因如果是转录因子并且该基因在我们推测的调控网络中,那么除了基因在不同实验条件下的表达情况,用户还可以得到与该基因存在潜在调控关系的靶基因,靶基因的推测方法是Context likelihood of relatedness (CLR)算法[29],该算法是基于相关性网络,首先计算基因两两之间在所有实验中表达量的相关性系数,将每个基因当作网络中的节点,基因之间的相关性作为基因之间连接权重,删除连接权重低于某一阈值的连接,从而生成基因之间的连接网络。CLR在此基础上利用基因之间的相关性计算了统计似然性作为背景分布,根据背景分布挑选相关性显著高于其他基因之间的连接对,与转录因子存在高连接度的基因就是该转录因子的潜在靶基因,再结合ENCODE数据库中转录因子和基因的结合信息作进一步筛选。采用CLR算法我们得到了肝脏中蛋白质之间潜在的调控关系,帮助用户进一步认识小鼠肝脏中的转录调控网络。
2.2.3 数据上传和下载功能门户网站可以支持用户将自己产生的关于小鼠肝脏蛋白质组数据上传到数据库中,现阶段支持上传已经完成数据库搜索的质谱数据,用户需要根据表 1的字段填写实验的基本情况并且写明数据库搜索条件和质控情况以及对应产生的数据表,然后将实验信息和数据表格打包成压缩文件进行上传,我们获得数据后会根据质控流程将数据存入到数据库中,这样可以不断提高数据库的全面性。门户网站同样也支持用户下载不同条件下的蛋白质表达数据以便用户进行后续的处理和分析。
3 总结小鼠肝脏蛋白质组数据门户为用户提供了当前最全面的小鼠肝脏蛋白质组数据库。该数据库包含小鼠肝脏基因的蛋白产物的表达量和实验条件的基本信息,并且支持用户进行查看比较不同实验条件下蛋白表达谱的变化情况和查询自己感兴趣的蛋白在不同实验条件下的表达量以及和实验条件的相关性。门户网站还提供了差异蛋白的功能分析以及潜在的转录因子调控的作用网络,为用户提供可能的研究方向。例如在小鼠肝脏部分切除的实验中,网站提供了在处理前后的不同时间点发生显著变化的转录因子以及其下游发生显著变化的靶基因和它们之间存在的相互作用以及它们富集出的生物学功能。
基于质谱的蛋白质组学已经越来越成熟,未来会有更多的研究产生大量关于小鼠肝脏的蛋白质组数据,该门户网站会不断地将发表的数据进行处理和质控后加入到数据库中,同时用户也可以将自己实验室产生的数据提交给门户网站。随着数据库中数据量的不断增长,门户网站可以提供更多的分析角度和更加可靠的分析结果。
| [1] |
Falcón-Pérez JM, Lu SC, Mato JM. Sub-proteome approach to the knowledge of liver. Proteomics Clin Appl, 2010, 4(4): 407-415. DOI:10.1002/prca.200900123 |
| [2] |
Fausto N. Liver regeneration. J Hepatol, 2000, 32(S1): 19-31. |
| [3] |
Stokkan KA, Yamazaki S, Tei H, et al. Entrainment of the circadian clock in the liver by feeding. Science, 2001, 291(5503): 490-493. DOI:10.1126/science.291.5503.490 |
| [4] |
He FC. Human liver proteome project: plan, progress, and perspectives. Mol Cell Proteomics, 2005, 4(12): 1841-1848. DOI:10.1074/mcp.R500013-MCP200 |
| [5] |
Gillet LC, Leitner A, Aebersold R. Mass spectrometry applied to bottom-up proteomics: entering the high-throughput Era for hypothesis testing. Ann Rev Anal Chem, 2016, 9: 449-472. DOI:10.1146/annurev-anchem-071015-041535 |
| [6] |
Aebersold R, Mann M. Mass-spectrometric exploration of proteome structure and function. Nature, 2016, 537(7620): 347-355. DOI:10.1038/nature19949 |
| [7] |
Sinitcyn P, Rudolph JD, Cox J. Computational methods for understanding mass spectrometry-based shotgun proteomics data. Ann Rev Biomed Data Sci, 2018, 1: 207-234. DOI:10.1146/annurev-biodatasci-080917-013516 |
| [8] |
Liu MW, Ge R, Liu WL, et al. Differential proteomics profiling identifies LDPs and biological functions in high-fat diet-induced fatty livers. J Lipid Res, 2017, 58(4): 681-694. DOI:10.1194/jlr.M071407 |
| [9] |
Azimifar SB, Nagaraj N, Cox J, et al. Cell-type-resolved quantitative proteomics of murine liver. Cell Metab, 2014, 20(6): 1076-1087. DOI:10.1016/j.cmet.2014.11.002 |
| [10] |
Ding C, Li YY, Guo FF, et al. A cell-type-resolved liver proteome. Mol Cell Proteomics, 2016, 15(10): 3190-3202. DOI:10.1074/mcp.M116.060145 |
| [11] |
Wang JK, Mauvoisin D, Martin E, et al. Nuclear proteomics uncovers diurnal regulatory landscapes in mouse liver. Cell Metab, 2017, 25(1): 102-117. DOI:10.1016/j.cmet.2016.10.003 |
| [12] |
Wang YZ, Song L, Liu MW, et al. A proteomics landscape of circadian clock in mouse liver. Nat Commun, 2018, 9(1): 1553. |
| [13] |
Liu XW, Lu FG, Zhang GS, et al. Proteomics to display tissue repair opposing injury response to LPS-induced liver injury. World J Gastroenterol, 2004, 10(18): 2701-2705. DOI:10.3748/wjg.v10.i18.2701 |
| [14] |
Hsieh HC, Chen YT, Li JM, et al. Protein profilings in mouse liver regeneration after partial hepatectomy using iTRAQ technology. J Proteome Res, 2009, 8(2): 1004-1013. DOI:10.1021/pr800696m |
| [15] |
Sun YW, Deng XY, Li WR, et al. Liver proteome analysis of adaptive response in rat immediately after partial hepatectomy. Proteomics, 2007, 7(23): 4398-4407. DOI:10.1002/pmic.200600913 |
| [16] |
Cox J, Hein MY, Luber CA, et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Mol Cell Proteomics, 2014, 13(9): 2513-2526. DOI:10.1074/mcp.M113.031591 |
| [17] |
Ding C, Jiang J, Wei JY, et al. A fast workflow for identification and quantification of proteomes. Mol Cell Proteomics, 2013, 12(8): 2370-2380. DOI:10.1074/mcp.O112.025023 |
| [18] |
Ding C, Chan DW, Liu WL, et al. Proteome-wide profiling of activated transcription factors with a concatenated tandem array of transcription factor response elements. Proc Natl Acad Sci USA, 2013, 110(17): 6771-6776. DOI:10.1073/pnas.1217657110 |
| [19] |
Perkins DN, Pappin DJC, Creasy DM, et al. Probability-based protein identification by searching sequence databases using mass spectrometry data. Electrophoresis, 1999, 20(18): 3551-3567. DOI:10.1002/(SICI)1522-2683(19991201)20:18<3551::AID-ELPS3551>3.0.CO;2-2 |
| [20] |
Saltzman AB, Leng M, Bhatt B, et al. gpGrouper: a peptide grouping algorithm for gene-centric inference and quantitation of bottom-up proteomics data. Mol Cell Proteomics, 2018, 17(11): 2270-2283. DOI:10.1074/mcp.TIR118.000850 |
| [21] |
Schwanh usser B, Busse D, Li N, et al. Global quantification of mammalian gene expression control. Nature, 2011, 473(7347): 337-342. DOI:10.1038/nature10098 |
| [22] |
Cox J, Mann M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat Biotechnol, 2008, 26(12): 1367-1372. DOI:10.1038/nbt.1511 |
| [23] |
Bolstad BM, Irizarry RA, Åstrand M, et al. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics, 2003, 19(2): 185-193. DOI:10.1093/bioinformatics/19.2.185 |
| [24] |
Maglott D, Ostell J, Pruitt KD, et al. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Res, 2007, 35: D26-D31. DOI:10.1093/nar/gkl993 |
| [25] |
Harris MA, Clark J, Ireland A, et al. The gene ontology (GO) database and informatics resource. Nucleic Acids Res, 2004, 32: D258-D261. DOI:10.1093/nar/gkh036 |
| [26] |
Alexa A, Rahnenfuhrer J, Lengauer T. Improved scoring of functional groups from gene expression data by decorrelating GO graph structure. Bioinformatics, 2006, 22(13): 1600-1607. DOI:10.1093/bioinformatics/btl140 |
| [27] |
Liebermeister W, Noor E, Flamholz A, et al. Visual account of protein investment in cellular functions. Proc Natl Acad Sci USA, 2014, 111(23): 8488-8493. DOI:10.1073/pnas.1314810111 |
| [28] |
Cahan P, Li H, Morris SA, et al. CellNet: network biology applied to stem cell engineering. Cell, 2014, 158(4): 903-915. DOI:10.1016/j.cell.2014.07.020 |
| [29] |
Faith JJ, Hayete B, Thaden JT, et al. Large-scale mapping and validation of Escherichia coli transcriptional regulation from a compendium of expression profiles. PLoS Biol, 2007, 5(1): e8. DOI:10.1371/journal.pbio.0050008 |
 2019, Vol. 35
2019, Vol. 35




