中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王远山, 郝文辉, 吴哲明, 牛坤, 龚美华
- Wang Yuanshan, Hao Wenhui, Wu Zheming, Niu Kun, Gong Meihua
- 原位显微镜在细胞生物量在线监测中的发展与应用
- Development and application of in-situ microscopy in on-line monitoring of cell biomass
- 生物工程学报, 2019, 35(9): 1607-1618
- Chinese Journal of Biotechnology, 2019, 35(9): 1607-1618
- 10.13345/j.cjb.190056
-
文章历史
- Received: January 31, 2019
- Accepted: April 25, 2019
随着近几十年生物技术的迅猛发展,生物发酵已经广泛应用于食品、制药、环境保护、能源等行业中[1],酸奶、啤酒、生物乙醇、抗生素、氨基酸、单克隆抗体等发酵产品层出不穷,生物发酵已与人们的生活息息相关。发酵过程的高效调控是确保生物发酵产品产量与质量的关键。截至目前,多种传感器已被开发用于监测发酵过程中的一些重要参数,比如pH、溶氧、温度、底物浓度、产物浓度、呼吸商、生物量等[2],为发酵过程的实时调控提供切实可靠的数据,保障产品的最终产量与质量。
在发酵过程中,通过活体细胞代谢将底物转化为产物是生物反应过程中的关键,因此对生物量的监测,尤其是对活细胞生物量的监测就显得尤为重要[3-5]。生物量是指单位体积培养基中所含细胞的质量或细胞的数目[6]。通过对生物量的监测,可以对发酵过程中细胞的生长状态进行评估,从而采取合适的调控策略,以保证发酵过程的正常运行。目前关于生物量监测的主流方法多是采用离线的方式来进行,如干重法、光密度法(OD600)、计数法、离心体积法等,虽然这些方法操作简单、成本低廉,但是过程繁琐、耗时费力,对培养液中的活细胞、死细胞以及不溶性物料无法区分,更重要的是不能实时反映生物量,使得对发酵过程的调控具有严重的滞后性[7-10],并且频繁的取样过程会使得发酵罐中的培养体系发生改变,增加染菌的风险[11]。除了生物量的监测之外,对细胞的活力鉴定也极为重要。通常,根据原理,细胞活力鉴定可以分为如下几类[12-19]:第1类是基于细胞在固体平板上生长的能力;第2类是基于细胞膜的完整性;第3类是基于细胞膜的功能性;第4类是基于细胞中的某些酶的活性;第5类是基于流式细胞仪的筛选鉴定。这些方法对发酵过程的调控同样具有滞后性。为了克服检测滞后性,学者们做了大量工作,开发了荧光法、电容法和原位显微镜、近红外光谱、激光反射光谱、拉曼光谱等多种在线监测技术[1, 20-27]。
荧光法是通过荧光传感器对细胞中自发荧光基团,如NADPH,或者与荧光试剂相络合的基团进行监测,收集荧光信号,构建多元线性回归模型和偏最小二乘(Partial least squares, PLS)回归模型,对发酵过程中的生物量浓度、底物浓度等发酵参数进行在线监测[28]。荧光传感器种类繁多,有二维荧光传感器、多维荧光传感器等。Haack等[28]利用多维荧光传感器对酿酒酵母细胞生物量浓度进行了在线监测,Hantelmann等[24]将二维荧光传感器应用于酿酒酵母的分批发酵。然而荧光法是基于细胞中自发荧光基团或者需要向培养基中添加荧光试剂,并且在监测过程中存在荧光信号重叠问题,使得荧光法在活细胞生物量在线监测过程中难以实施[24]。
电容法是基于在交变电场中具有完整生物膜的细胞会发生极化,通过测定发酵液在一定频率范围内的电容和电导率来衡量发酵液中的生物量浓度[7]。生物反应器中的发酵液可以视为由3个独立的部分组成,分别为细胞质、外部质膜、悬浮介质。细胞质是由内部细胞器和蛋白质、核酸以及小分子物质组成的高度复杂的混合物;质膜是细胞导电的核心,其本身不能导电;悬浮介质由水和带电离子基团组成。因此,可以将发酵液视为包含众多球形电容器的悬浮液[29]。向发酵液施加一个交变电场,使细胞内部和外部带正电荷的离子和带负电荷的离子向相反的方向移动,直至遇到质膜,带正电荷的离子与带负电荷的离子分别聚集在细胞膜的两侧,导致细胞发生极化。这种由电场所引起的细胞极化的量级可以通过电容来进行测量,进而对发酵液中生物量浓度进行监测。Lan等[30]将电容法应用于真氧产碱杆菌Ralstonia eutropha发酵聚羟基链烷酸酯过程中的活细胞量检测;Xiong等[31]在酿酒酵母高密度分批补料发酵谷胱甘肽中利用电容法测定发酵液中的活细胞生物量浓度。尽管存在一些不足,如价格昂贵、细胞质中的膜结构对整个细胞的介电性质产生影响,同时电极的极化使得整体的电容测量偏大等[21],电容法仍然是应用最为广泛的活细胞生物量在线监测方法。华东理工大学生物反应器国家重点实验室等研究机构在这方面做了大量工作,在发酵过程控制与优化方面也取得了良好的效果[27, 30-31]。
原位显微镜技术是基于在线采集生物反应器中发酵液的显微图片,通过对显微图片的处理分析,在线获取发酵液中的生物量浓度,并对细胞形态进行分析[32],目前已成为一种具有较好应用前景的在线生物量分析技术。
笔者所在团队长期从事工业生物技术领域的研究,其中涉及到大量的微生物发酵和细胞培养过程,对过程控制与优化有着较好的积累,深知细胞生物量和形态在线检测的重要性。同时,笔者及所在团队也承担了生物工程专业主干课程生物工程设备(国家精品资源共享课程http://www.icourses.cn/sCourse/course_2616.html)的教学工作,生物培养过程的检测与控制是教学重要内容,因而对生物量在线检测技术也有较好的了解。鉴于目前在国内还没有系统介绍原位显微镜发展及其应用的文献,本文结合笔者科研和教学工作的积累就此进行综述。
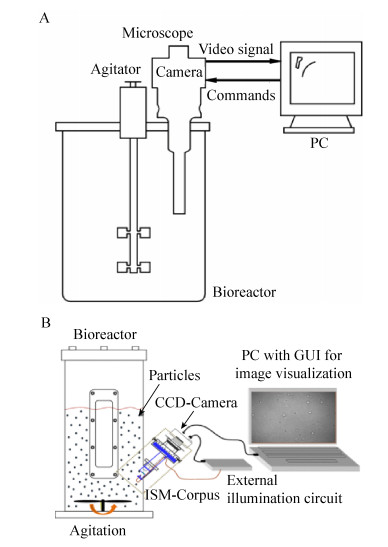
1 原位显微镜的结构1990年,Suhr等首次描述了原位显微镜的概念。它由显微成像系统、控制系统、图像处理系统3部分组成[33],其结构示意图如图 1所示[43],具体描述将在下文展开。根据原位显微镜在发酵过程中对样本量的定义方式,可以将其划分为两类:一类是光学采样原位显微镜,通过估计显微照片的景深来定义虚拟的样本量。第二类是机械采样原位显微镜,通过机械装置重复封装体积相等的样品来定义样本量[6]。对于光学采样原位显微镜因其需要从显微照片中计算景深,所以其算法结构相对于机械采样原位显微镜要更复杂。而对于机械采样原位显微镜,其机械装置和命令系统更复杂。
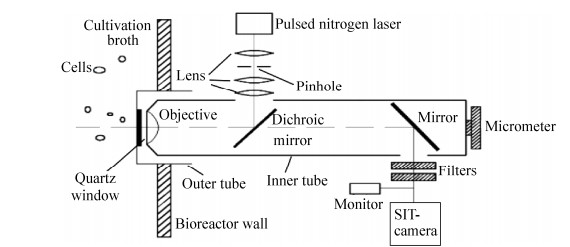
1995年,Suhr等第一次详细描述了光学采样原位显微镜,其结构简图如图 2所示[6]。一个直径为25 mm的不锈钢外管安装在生物反应器上,其末端是一个密封的透明石英玻璃窗,作为隔绝发酵罐内部环境与外部环境的屏障,从而保障发酵罐内部的无菌环境。一个直射光显微镜集成在不锈钢内管中,在发酵时将内管插入到外管中,与SIT照相机一起组成显微成像系统。发酵结束后可以将内管从外管中取出单独保存。
显微镜由物镜(Olympus D APO l00×UV,油浸,NA=0.95)、二向色镜以及外耦合镜组成,并且可以通过调节千分尺螺旋来实现显微镜的正确聚焦。氮激光光源发射波长为337 nm的光,经过二向色镜反射后,照亮细胞。随后被照射的细胞激发出450 nm波长的荧光,透过二向色镜,经过外耦合镜,将细胞的图像呈递给照相机记录。之后显微照片传输至搭载了图像分析算法的电脑中被储存和分析。在发酵开始的阶段,图像获取的频率为每2秒一张图像,到了后期频率变为每20秒一张图像。图像的处理速度为每分钟一张。显然图像的分析速度慢,无法为发酵过程调控提供大量实时的具有统计学意义的数据。
Camisard等[34]、Wiedemann等[35]对Suhr等开发的光学采样原位显微镜进行了改进。他们以发光二极管(LED)作为光源,通过光纤传导,照亮物镜前的区域。在纤维末端和石英玻璃窗之间,存在0.7 mm的间隙,允许发酵液自由流动。此外,图像获取频率和图像处理的速度也得到了极大的发展。在Camisard等开发的算法中,图像处理速度达到了0.6 s/张[34]。目前市场上光学采样显微镜结构简图和实物图如图 3所示[36, 41]。
1998年,Bittner等[33]基于光学采样原位显微镜开发了机械采样原位显微镜,其结构简图如图 4所示[38, 45]。安装在发酵罐的前端具有透明石英窗的不锈钢外管,可以自由移动带有直射光显微镜的内管,一个CCD照相机组成显微成像系统。在不锈钢外管的前端有可以自由滑动的密封的照明系统,由一个滑块、聚光器、发光二极管组成。采样时,电机驱动照明系统向外管的前端滑动,从而与外管共同组成体积固定的样品小室。在这个封闭的小室中完成样品的显微成像后,照明系统回到原位,样品小室重新打开,封装的样品被释放,发酵液可以自由地通过采样区。
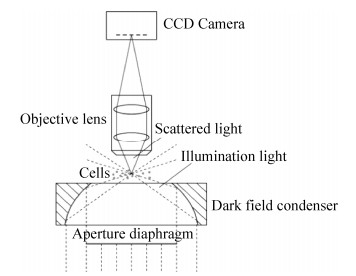
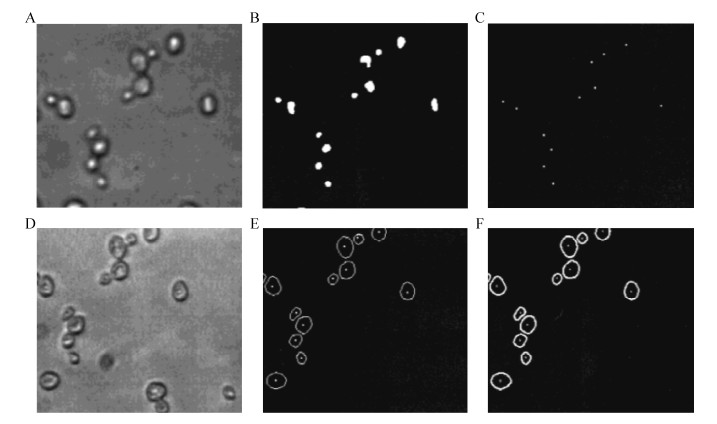
2007年,Wei等[38]在机械采样原位显微镜的基础上对照明系统进行改进,开发出了暗场原位显微镜。在暗场显微镜中,正常的聚光器被暗场聚光器所取代,这样可以使显微照片的背景变得更暗,目标细胞变得更亮,从而增加照片的清晰度,有利于后续的细胞识别,其照明原理如图 5所示[39]。准直透镜将LED光源发射的光整合成平行光,位于准直透镜与石英玻璃窗中间的暗场聚光器对中间部分的平行光进行限制,同时改变边缘部分平行光的路径,使其投射于采样区的细胞。从而光源发射出的光无法进入物镜,而被照亮的细胞发出的散射光被物镜收集,从而将细胞的图像呈递给照相机记录。同时,在物镜与相机之间添加一个中继透镜,通过中继透镜的二次成像,可以使照相机记录的显微照片更清晰,有中继透镜和无中继透镜拍摄的图像如图 6所示[39]。
对图像的处理和分析是原位显微镜技术应用的关键环节。近几十年来,随着人工智能的发展,应用于图像处理和分析的软件及算法越来越完善。尽管针对不同的发酵条件,其图像处理的过程不尽相同,但是可以将其划分为3个部分[33]。首先是图像预处理,其目的是消除图像捕获期间的所有背景噪声并增加细胞和背景之间的对比度。其次是细胞识别,这也是图像处理过程中最核心的步骤,它的目的是将细胞像素从背景中的非细胞像素中识别和分离出来,因此如何正确识别细胞像素对于算法是至关重要的,在下文将详细介绍。最后是细胞表征,通过从显微照片中分离出的细胞像素来估计细胞的直径、形态和大小,从而对细胞的活力进行表征。
对于一个算法的优良性,可以用灵敏度(Sensitivity, SE)和正向预测值(Positive predictive value, PPV)来进行评估[38, 40],其中灵敏度(SE)表示算法能够正确识别细胞的比例;正向预测值(PPV)表示算法检测到的细胞是真实细胞的比例。高的灵敏度和正向预测值可以保证通过算法得到的数据的真实性和可靠性。用于细胞生物量和细胞活力表征的图像处理技术将在后文中分别进行叙述。此外,图像的处理速度也是至关重要的,因为在监测过程中会产生大量的显微照片,处理速度慢就无法提供具有统计学意义的数据来实现在线监测。在初期,Suhr等[6]使用的图像处理器的处理速度为每分钟一张图像。2017年,Belini等[41]使用WiT和MATLAB对显微照片进行处理,其速度为每秒6张图像,提高了360倍。
2.1 细胞生物量检测的图像分析1995年,在Scholz等[42]开发的算法中,包含有5个步骤,分别为:1)背景消除;2)平滑;3)生成模糊敏感特征向量;4)确定景深;5)计算浓度。虽然它可以对发酵液中的细胞生物量进行检测和计算,但是不能提供关于细胞的特定信息,而且图像处理速度很慢。随后,1998年Bittner等[33]基于机械采样原位显微镜开发出比较复杂的算法,同时利用酿酒酵母的光学特性即酵母细胞的细胞质具有比发酵液更高的折射率来提高算法的准确性。作者使用两台CCD相机分别记录聚焦细胞和离焦细胞,其中离焦细胞用于细胞定位,而聚焦细胞用于细胞像素与背景中的非细胞像素分离。细胞识别后,使用中值滤波对图像进行进一步降噪处理从而表征细胞形态,并且构建三维模型用于计算细胞体积。其图像分析过程如图 7所示[33]。
2016年,Dias等[43]利用Matlab8.1运行图像处理算法对废水中的丝状细菌进行在线监测,图像经过预处理、二值化、菌丝鉴定后实现菌丝长度的量化,其中菌丝鉴定的过程如图 8[43]所示。同年,为了实现对毕赤酵母高浓度发酵过程的在线监测,Marquard等[44]采用两种算法对图像进行分析。在发酵过程的初始阶段,发酵液中的细胞浓度低,作者使用毕赤酵母计数算法(Pichia counter algorithm, PCA)对发酵液中的细胞进行监测。之后经过指数期,细胞生长进入到稳定期,发酵液当中的细胞浓度很高并且聚集形成块状,此时通过PCA无法获得发酵液中的细胞浓度,而聚类识别算法(Cluster recognition algorithm, CRA)可以解决这个问题,即通过计算块状的体积来建立与细胞浓度之间的关系。2017年,Belini等[41]利用WiT和MATLAB分析酵母细胞的原位显微图像,利用圆形霍夫转化(Circular hough transform, CHT)对近似圆形的酵母细胞进行在线监测。此外还报道,利用原位显微镜与光纤pO2传感器、中红外光谱以及三维全息技术协同工作,监测乙醇发酵过程中细胞密度、葡萄糖、乙醇等的变化[45-47]。
2007年,在支持向量机(Support vector machine, SVM)的帮助下,Wei等[38]在没有使用任何染料的条件下实现了细胞的生物量检测和细胞的活力鉴定。用两个不同的实验组对支持向量机(SVM)进行训练,第一个实验组的细胞处于对数期,第二个实验组为死细胞。遗憾的是,细胞活力分类是在离线装置中进行的,无法在发酵过程中进行实时监控。2010年,Burgemeister等[40]采用相同的方法,在腺病毒EI基因改造的人神经细胞系AGEI.HN培养过程中,利用CellViCAM算法对细胞的生物量和细胞的活力进行鉴定。该算法分两步进行:首先,在图像经过预处理后,利用CHT对图像中的细胞像素进行定位;其次,利用已经建立好的4套训练集,分别为活细胞训练集、坏死细胞训练集、早期凋亡细胞训练集、晚期凋亡细胞训练集对SVM分类器进行训练。同时,应用3级树状分类架构,用于区分显微照片中相应的4个细胞状态。然而,使用细胞匹配和圆形相似性对细胞活力进行的鉴定仅限于具有规则细胞形态的细胞。
除了机器学习方法之外,还可以基于不同的细胞形态对细胞活力进行鉴定,在细胞生长最后阶段,细胞形态的异质性就会表现出来。图 9展示了处于生长期和死亡期的哺乳动物细胞形态[35]。通过对细胞形态的异质性进行表征,实现细胞的活力鉴定。2011年,Wiedemann等[35]基于显微图像细胞像素灰度值的信息熵方差和对比度差异,对杂交瘤细胞的活力进行鉴定。旨在获得单细胞的熵信息并计算低熵细胞的百分比,其中低熵意味着均匀的细胞形态,高熵意味着不均匀的细胞形态。2018年,Marbà-Ardébol等[48]基于非芽殖和芽殖细胞的不同形态,对酵母细胞活力进行鉴定,图 10展示了酵母细胞在芽殖过程中的形态异质性变化[48]。利用人工神经网络(Artificial neural networks, ANN)将显微图像中的细胞像素分为出芽细胞和非出芽细胞,同时采用训练的贝叶斯分类器对结果进行验证。最后,根据鉴定结果计算出芽指数,从而判定酵母细胞的活力。
原位显微镜多应用于酵母细胞以及动物细胞的生化反应过程中,仅有少量文献报道其在丝状细菌、大肠杆菌以及藻类中的应用。
3.1 在酵母菌培养过程中的应用1995年Suhr等[6]应用光学采样原位显微镜对酿酒酵母的发酵过程进行监控。1998年Bittner等[33]同样将机械采样原位显微镜应用于酿酒酵母的发酵过程,此后Wei等[38]开发出暗场显微镜并且尝试对酿酒酵母的活力进行鉴定;Marquard等[44]应用不同的算法尝试将原位显微镜应用于巴氏毕赤酵母的高密度发酵过程;Belini等[41]将原位显微镜应用于生物乙醇的工业生产中;Marbà-Ardébol等[48]利用原位显微镜来计算酿酒酵母细胞的出芽指数从而判定细胞的活力。表 1汇总了原位显微镜技术在酵母菌培养过程中的应用情况。由表 1可知,原位显微镜在酵母细胞的生物发酵过程中应用前景广泛。无论是高密度发酵还是工业生产,都实现了原位显微镜的在线监测,并且在线监测数据稳定,与离线方法的相关系数达到0.9以上。
| Sampling technique |
Test strain |
Cell concentration |
Correlation coefficient with off-line data |
Cell viability classification |
Image processing speed |
Reference |
| Optical | S. cerevisiae | 5×108 | Agree well | No | 1 min/image | [6] |
| Mechanical | S. cerevisiae | 106–109 cells/mL | 0.99 | Yes | 15 s/image | [33] |
| Mechanical | S. cerevisiae | / | 0.98 | Yes | / | [38] |
| Mechanical | P. pastoris | 74 g/L(CRA); Below 35 g/L(PCA) | All above 0.96 | No | 2 s/image | [44] |
| Optical | S. cerevisiae | 2×108 cells/mL | / | No | / | [41] |
| Mechanical | S. cerevisiae | / | / | Yes | / | [48] |
原位显微镜在动物细胞中的应用相对较晚,直到2002年,Joeris等[49]利用机械采样原位显微镜对CHO细胞进行在线监测,2004年,Guez等[50]利用原位显微镜对杂交瘤细胞的高密度发酵进行在线监控。此后,Rudolph等[37]首次将原位显微镜应用于贴壁生长的小鼠成纤维细胞中,首先基于显微图像开发出一种算法,分割微载体与细胞;其次基于神经网络算法估测微载体的分段图像区域的电镀效率和细胞定植水平;2010年,Burgemeister等[40]基于CellViCAM算法以及半自动支持向量机(SVM)训练,利用暗场显微镜对腺病毒EI基因改造的人神经细胞系AGEI.HN进行在线监测,其算法识别细胞的准确率达到86%,对细胞活力鉴定的准确性达到了87%。因支持向量机不是完全自动,2011年Wiedemann等[35]通过计算显微照片中高熵细胞的比例判定细胞的活力。此后Lüder等[46]将原位显微镜与中红外光谱连用,监测CHO细胞系的发酵过程。表 2汇总了原位显微镜技术在动物细胞培养过程中的应用情况。由表 2可知,在动物细胞的发酵过程中原位显微镜是一种有力的在线监测工具,其准确性高,并且与离线方法能够良好拟合。
| Sampling technique |
Test strain | Cell concentration |
Correlation coefficient with off-line data |
Cell viability classification |
Image processing speed |
Reference |
| Mechanical | CHO | 9.5×106 cells/mL | Agree well | No | / | [49] |
| Optical | Hybridoma cells | 4.5×106 cells/mL | 0.99 | No | / | [50] |
| Optical | Hybridoma cell | 106 cells/mL | Good correlations | No | 10 frames/s | [37] |
| Optical | Human cell line | / | Accuracy 87% | Yes | 30–60 s/image | [40] |
| Optical | Mammalian cells | / | / | Yes | 5 s/image | [35] |
| Mechanical | CHO-K1 | 1.4×107 cells/mL | R2=0.98 | Yes | / | [46] |
截至目前,原位显微镜多用于近圆形细胞的发酵过程中,在其他形态细胞中仅有少量文献报道。2016年Dias等[43]利用原位显微镜技术量化丝状细菌的菌丝长度;2017年Marquard等[51]利用原位显微镜对大肠杆菌Escherichia coli的发酵过程进行监控;同年Marbà-Ardébol等[47]利用原位显微镜与三维全息图像技术对寇氏隐甲藻Crypthecodinium cohnii细胞中的脂质积累进行监测。表 3汇总了原位显微镜技术在其他细胞培养过程中的应用情况。
| Sampling technique |
Test strain | Cell concentration | Correlation coefficient with off-line data |
Cell viability classification |
Image processing speed |
Reference |
| Optical | filamentous bacteria |
Total extended filament length detection |
/ | No | 0.7 s/image | [43] |
| Mechanical | E. coli | 10 g/L to 70 g/L dcw | All above 0.99 | No | / | [51] |
| Optical | C. cohnii | / | / | / | / | [47] |
综上所述,原位显微镜是生物量浓度和细胞活力鉴定的有效工具,其非侵入性和非破坏性操作对于现代生物发酵至关重要。原位显微镜的研发主要集中于机械设备研制和算法优化及软件的研发。鉴于原位显微镜主要用于动物细胞和酵母等近圆形细胞的培养过程,为拓展其应用范围,可以从以下几个方面着手:1)对原位显微镜的硬件设施进行改进,如添加透镜、改进照明方式等,以获得更加清晰的显微照片。2)对原位显微镜的软件进行研发,开发新的图像处理技术和算法,提高识别效果和图像处理速度,使得原位显微镜能够应用于更多发酵过程和更加复杂的发酵环境。3)增加原位显微镜的放大倍数,以便更多应用于细菌发酵过程。随着这些新技术的开发,可以有效促进原位显微镜技术的应用,为生物量和细胞形态等的在线监测提供有效的工具,对发酵过程的高效调控具有一定意义。
| [1] |
Assawajaruwan S, Reinalter J, Hitzmann B. Comparison of methods for wavelength combination selection from multi-wavelength fluorescence spectra for on-line monitoring of yeast cultivations. Anal Bioanal Chem, 2017, 409(3): 707-717. |
| [2] |
Vann L, Sheppard J. Use of near-infrared spectroscopy (NIRs) in the biopharmaceutical industry for real-time determination of critical process parameters and integration of advanced feedback control strategies using MIDUS control. J Ind Microbiol Biotechnol, 2017, 44(12): 1589-1603. DOI:10.1007/s10295-017-1984-2 |
| [3] |
Petiot E, El-Wajgali A, Esteban G, et al. Real-time monitoring of adherent Vero cell density and apoptosis in bioreactor processes. Cytotechnology, 2012, 64(4): 429-441. DOI:10.1007/s10616-011-9421-2 |
| [4] |
Olsson L, Nielsen J. On-line and in situ monitoring of biomass in submerged cultivations. Trends Biotechnol, 1997, 15(12): 517-522. DOI:10.1016/S0167-7799(97)01136-0 |
| [5] |
Workman J, Koch M, Veltkamp DJ. Process analytical chemistry. Anal Chem, 2003, 75(12): 2859-2876. DOI:10.1021/ac0301393 |
| [6] |
Suhr H, Wehnert G, Schneider K, et al. In situ microscopy for on-line characterization of cell-populations in bioreactors, including cell-concentration measurements by depth from focus. Biotechnol Bioeng, 1995, 47(1): 106-116. |
| [7] |
Fernandez RE, Lebiga E, Koklu A, et al. Flexible bioimpedance sensor for label-free detection of cell viability and biomass. IEEE Trans Nanobiosci, 2015, 14(7): 700-706. DOI:10.1109/TNB.2015.2451594 |
| [8] |
Holland T, Blessing D, Hellwig S, et al. The in-line measurement of plant cell biomass using radio frequency impedance spectroscopy as a component of process analytical technology. Biotechnol J, 2013, 8(10): 1231-1240. |
| [9] |
Belini V L, Wiedemann P, Suhr H. In situ microscopy: a perspective for industrial bioethanol production monitoring. J Microbiol Methods, 2013, 93(3): 224-232. DOI:10.1016/j.mimet.2013.03.009 |
| [10] |
Thomson K, Bhat A, Carvell J. Comparison of a new digital imaging technique for yeast cell counting and viability assessments with traditional methods. J Inst Brewing, 2015, 121(2): 231-237. DOI:10.1002/jib.v121.2 |
| [11] |
Rathore AS, Bhambure R, Ghare V. Process analytical technology (PAT) for biopharmaceutical products. Anal Bioanal Chem, 2010, 398(1): 137-154. |
| [12] |
Bouchez JC, Cornu M, Danzart M, et al. Physiological significance of the cytometric distribution of fluorescent yeasts after viability staining. Biotechnol Bioeng, 2004, 86(5): 520-530. DOI:10.1002/(ISSN)1097-0290 |
| [13] |
Attfield PV, Kletsas S, Veal DA, et al. Use of flow cytometry to monitor cell damage and predict fermentation activity of dried yeasts. J Appl Microbiol, 2000, 89(2): 207-214. |
| [14] |
Beck AE, Hunt KA, Carlson RP. Measuring cellular biomass composition for computational biology applications. Processes, 2018, 6(5): 38. DOI:10.3390/pr6050038 |
| [15] |
Hernlem B, Hua SS. Dual fluorochrome flow cytometric assessment of yeast viability. Curr Microbiol, 2010, 61(1): 57-63. |
| [16] |
Chandler WL, Yeung W, Tait JF. A new microparticle size calibration standard for use in measuring smaller microparticles using a new flow cytometer. J Thromb Haemost, 2011, 9(6): 1216-1224. DOI:10.1111/j.1538-7836.2011.04283.x |
| [17] |
Mirek MK, Zadrag-Tecza R. Comparison of methods used for assessing the viability and vitality of yeast cells. FEMS Yeast Res, 2014, 14(7): 1068-1079. |
| [18] |
Longo VD, Shadel GS, Kaeberlein M, et al. Replicative and chronological aging in Saccharomyces cerevisiae. Cell Metab, 2012, 16(1): 18-31. DOI:10.1016/j.cmet.2012.06.002 |
| [19] |
Essary BD, Marshall PA. Assessment of FUN-1 vital dye staining: yeast with a block in the vacuolar sorting pathway have impaired ability to form CIVS when stained with FUN-1 fluorescent dye. J Microbiol Methods, 2009, 78(2): 208-212. DOI:10.1016/j.mimet.2009.05.018 |
| [20] |
Bluma A, Höpfner T, Lindner P, et al. In-situ imaging sensors for bioprocess monitoring: state of the art. Anal Bioanal Chem, 2010, 398(6): 2429-2438. DOI:10.1007/s00216-010-4181-y |
| [21] |
Justice C, Brix A, Freimark D, et al. Process control in cell culture technology using dielectric spectroscopy. Biotechnol Adv, 2011, 29(4): 391-401. DOI:10.1016/j.biotechadv.2011.03.002 |
| [22] |
Hall JW, McNeil B, Rollins MJ, et al. Near-infrared spectroscopic determination of acetate, ammonium, biomass, and glycerol in an industrial Escherichia coli fermentation. Appl Spectrosc, 1996, 50(1): 102-108. |
| [23] |
Expósito PL, Suárez AB, lvarez CN. Laser reflectance measurement for the online monitoring of Chlorella sorokiniana biomass concentration. J Biotechnol, 2017, 243: 10-15. DOI:10.1016/j.jbiotec.2016.12.020 |
| [24] |
Hantelmann K, Kollecker M, Hüll D, et al. Two-dimensional fluorescence spectroscopy: a novel approach for controlling fed-batch cultivations. J Biotechnol, 2006, 121(3): 410-417. |
| [25] |
Agarwal UP. 1064 nm FT-Raman spectroscopy for investigations of plant cell walls and other biomass materials. Front Plant Sci, 2014, 5: 490. |
| [26] |
Kiviharju K, Salonen K, Moilanen U, et al. Biomass measurement online: the performance of in situ measurements and software sensors. J Ind Microbiol Biotechnol, 2008, 35(7): 657-665. DOI:10.1007/s10295-008-0346-5 |
| [27] |
Wang ZJ, Wang P, Zhang Q, et al. Principle and application of physiological parameters detection biosensor technology in microbial fermentation process optimization. Biotechnol Busin, 2018, 63(1): 19-32 (in Chinese). 王泽建, 王萍, 张琴, 等. 微生物发酵过程生理参数检测传感器技术与过程优化. 生物产业技术, 2018, 63(1): 19-32. DOI:10.3969/j.issn.1674-0319.2018.01.004 |
| [28] |
Haack MB, Eliasson A, Olsson L. On-line cell mass monitoring of Saccharomyces cerevisiae cultivations by multi-wavelength fluorescence. J Biotechnol, 2004, 114(1/2): 199-208. |
| [29] |
Carvell JP, Dowd JE. On-line measurements and control of viable cell density in cell culture manufacturing processes using radio-frequency impedance. Cytotechnology, 2006, 50: 35-48. DOI:10.1007/s10616-005-3974-x |
| [30] |
Lan L, Wang ZJ, Chen XJ, et al. Optimization of polyhydroxyalkanoates fermentations with on-line capacitance measurement. Bioresourc Technol, 2014, 156: 216-221. DOI:10.1016/j.biortech.2014.01.042 |
| [31] |
Xiong ZQ, Guo MJ, Guo YX, et al. Real-time viable-cell mass monitoring in high-cell-density fed-batch glutathione fermentation by Saccharomyces cerevisiae T65 in industrial complex medium. J Biosci Bioeng, 2008, 105(4): 409-413. DOI:10.1263/jbb.105.409 |
| [32] |
Suhr H, Speil P, Wehnert G, et al. In situ Mikroskopsonde und Meßverfahren, DE 4032002A1, 1991.
|
| [33] |
Bittner C, Wehnert G, Scheper T. In situ microscopy for on-line determination of biomass. Biotechnol Bioeng, 1998, 60(1): 24-35. |
| [34] |
Camisard V, Brienne JP, Baussart H, et al. Inline characterization of cell concentration and cell volume in agitated bioreactors using in situ microscopy: application to volume variation induced by osmotic stress. Biotechnol Bioeng, 2002, 78(3): 353. |
| [35] |
Wiedemann P, Guez JS, Wiegemann HB, et al. In situ microscopic cytometry enables noninvasive viability assessment of animal cells by measuring entropy states. Biotechnol Bioeng, 2011, 108(12): 2884-2893. DOI:10.1002/bit.v108.12 |
| [36] |
Wiedemann P, Guez JS, Cassar JP, et al. Optical sampling in-situ microscopy for on-line monitoring of animal cell cultures[C]. 11th International Symposium on Computer Applications in Biotechnology. Leuven, Belgium, July 7-9, 2010
|
| [37] |
Rudolph G, Lindner P, Gierse A, et al. Online monitoring of microcarrier based fibroblast cultivations with in situ microscopy. Biotechnol Bioeng, 2008, 99(1): 136-145. |
| [38] |
Wei N, You J, Friehs K, et al. An in situ probe for on-line monitoring of cell density and viability on the basis of dark field microscopy in conjunction with image processing and supervised machine learning. Biotechnol Bioeng, 2007, 97(6): 1489-1500. DOI:10.1002/(ISSN)1097-0290 |
| [39] |
Wei N, You J, Friehs K, et al. In situ dark field microscopy for on-line monitoring of yeast cultures. Biotechnol Lett, 2007, 29(3): 373-378. |
| [40] |
Burgemeister S, Nattkemper TW, Noll T, et al. CellViCAM-Cell viability classification for animal cell cultures using dark field micrographs. J Biotechnol, 2010, 149(4): 310-316. |
| [41] |
Belini VL, Caurin GAP, Wiedemann P, et al. Yeast fermentation of sugarcane for ethanol production: can it be monitored by using in situ microscopy?. Braz J Chem Eng, 2017, 34(4): 949-959. |
| [42] |
Scholz T. Ein depth from focus-verfahren zur online-bestimmung der zellkonzentration bei fermentationsprozessen[D]. Heidelberg: Universität Heidelberg, 1995.
|
| [43] |
Dias PA, Dunkel T, Fajado DAS, et al. Image processing for identification and quantification of filamentous bacteria in in situ acquired images. BioMed Eng Online, 2016, 15: 64. DOI:10.1186/s12938-016-0197-7 |
| [44] |
Marquard D, Enders A, Roth G, et al. In situ microscopy for online monitoring of cell concentration in Pichia pastoris cultivations. J Biotechnol, 2016, 234: 90-98. DOI:10.1016/j.jbiotec.2016.07.024 |
| [45] |
Akin M, Prediger A, Yuksel M, et al. A new set up for multi-analyte sensing: At-line bio-process monitoring. Biosens Bioelectron, 2011, 26(11): 4532-4537. DOI:10.1016/j.bios.2011.05.018 |
| [46] |
Lüder C, Lindner P, Bulnes-Abundis D, et al. In situ microscopy and MIR-spectroscopy as non-invasive optical sensors for cell cultivation process monitoring. Pharm Bioprocess, 2014, 2(2): 157-166. DOI:10.4155/pbp.14.13 |
| [47] |
Marbà-Ardébol AM, Emmerich J, Neubauer P, et al. Single-cell-based monitoring of fatty acid accumulation in Crypthecodinium cohnii with three-dimensional holographic and in situ microscopy. Process Biochem, 2017, 52: 223-232. DOI:10.1016/j.procbio.2016.11.003 |
| [48] |
Marbà-Ardébol AM, Emmerich J, Muthig M, et al. Real-time monitoring of the budding index in Saccharomyces cerevisiae batch cultivations with in situ microscopy. Microb Cell Fact, 2018, 17: 73. DOI:10.1186/s12934-018-0922-y |
| [49] |
Joeris K, Frerichs JG, Konstantinov K, et al. In-situ microscopy: online process monitoring of mammalian cell cultures. Cytotechnology, 2002, 38(1/3): 129-134. DOI:10.1023/A:1021170502775 |
| [50] |
Guez JS, Cassar JP, Wartelle F, et al. Real time in situ microscopy for animal cell-concentration monitoring during high density culture in bioreactor. J Biotechnol, 2004, 111(3): 335-343. |
| [51] |
Marquard D, Schneider-Barthold C, Düsterloh S., et al. Online monitoring of cell concentration in high cell density Escherichia coli cultivations using in situ microscopy. J Biotechnol, 2017, 259: 83-85. DOI:10.1016/j.jbiotec.2017.08.003 |
 2019, Vol. 35
2019, Vol. 35