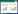中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李雅君, 李爱宁, 孟繁凡, 张宏宇, 钱韦, 贺伟, 邓超颖
- Li Yajun, Li Aining, Meng Fanfan, Zhang Hongyu, Wei Qian, He Wei, Deng Chaoying
- MarR家族转录因子HpaR和XC0449协同调控野油菜黄单胞菌致病性
- MarR family transcription regulator HpaR and XC0449 coordinately regulate the virulence of Xanthomonas campestris pv. campestris
- 生物工程学报, 2019, 35(8): 1500-1510
- Chinese Journal of Biotechnology, 2019, 35(8): 1500-1510
- 10.13345/j.cjb.190057
-
文章历史
- Received: January 31, 2019
- Accepted: February 28, 2019
2. 中国科学院微生物研究所 植物基因组学国家重点实验室,北京 100101
2. State Key Laboratory of Plant Genomics, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
MarR (Multiple antibiotic resistance regulator)家族转录因子作为控制多重抗生素抗性的一种转录抑制子,于1983年首次在大肠杆菌中被鉴定[1-2],这些抗生素包括四环素、氯霉素、嘌呤霉素、萘啶酸、青霉素、氟喹诺酮等[3-4]。MarR家族转录因子广泛存在于细菌及古生菌中[5]。根据Pfam数据库的统计,结合已有的遗传及生化证据,目前在44 000种细菌和古生菌中分布着至少335 000个MarR家族转录因子[6-7]。结构上,MarR家族转录因子是保守的有翼的螺旋-转角-螺旋(wHTH)结合蛋白,以二聚体的形式结合在启动子内的回文序列上[8]。功能上,它们作为感应环境变化的传感器,能够结合小分子化合物并快速启动转录水平的适应性调节[9],包括控制毒力因子产生、对抗生素和氧化应激的反应以及环境芳香族化合物的分解代谢等。MarR作为转录调控因子,既可以抑制也可以激活下游基因的表达。在特定的信号或者配体分子存在的情况下,MarR蛋白与DNA的结合被改变,从而控制不同基因的表达[10]。
野油菜黄单胞菌(Xanthomonas campestris pv. campestris,Xcc)是一种杆状、极生单鞭毛、营化能异养型革兰氏阴性细菌,是十字花科植物的主要病原菌,也是植物病理学研究领域的主要模式生物。它能够侵染模式植物拟南芥以及包括甘蓝、白菜、花椰菜、芥菜、萝卜、油菜等多种重要十字花科作物并引起黑腐病,每年造成巨大的经济损失[11-12]。目前,已有3个Xcc菌株完成了全基因组序列测定,它们的全基因组大小约5.1 Mb,编码4 000余个基因[13-14]。功能基因组学分析结合遗传学证据发现,Xcc中至少有100个基因参与了细菌的致病过程,涉及胞外多糖合成、胞外酶合成、各类分泌系统相关蛋白、铁锌离子转运、细胞解毒途径、纤毛和菌毛组装以及芳香族氨基酸等大分子合成等多种生理生化途径,并受到精细、灵活及严格的转录水平的调控[15-16]。
已有研究表明,MarR家族转录因子HpaR (XC2827)的失活会显著降低细菌对于寄主甘蓝的致病力和非寄主辣椒的超敏反应,同时会导致胞外蛋白酶的过量表达[17]。此外,HpaR能作为转录激活子或抑制子直接调控约448个下游基因的表达,涉及细菌毒力及相关的多种生理生化过程[18]。本研究进一步发现,Xcc 8004基因组共编码9个MarR家族转录因子,其中XC0449基因编码的转录因子能与HpaR相互作用,并协同调控下游致病相关基因XC0705的表达,最终调控细菌毒力及胞外蛋白酶合成。
1 材料与方法 1.1 材料 1.1.1 实验菌株大肠杆菌Escherichia coli DH5α、BL21 (DE3)购自Invitrogen公司,TB1购自BioLabs公司。野油菜黄单胞菌(Xanthomonas campestris pv. campestris 8004,Xcc)为中国科学院微生物研究所植物基因组学国家重点实验室保存。
1.1.2 质粒载体克隆载体pGEM-T Easy购自Promega公司。敲除载体pK18mob和pK18mobSacB,广寄主互补载体pHMI、pHMII均为中国科学院微生物研究所植物基因组学国家重点实验室保存。表达载体pET30a购自Novagen公司。pMAL-p2X购自BioLabs公司。实验中所用引物见表 1。
| Primer name | Sequence (forward/reverse, 5′–3′) |
| △XC0449-A | TCTAGACATCTTGCGGAAGTAGTCGTG |
| △XC0449-B | CATATGCACGCTGAGGCGATAGGG |
| △XC0449-C | CATATGAGCGCCGAAGAACAGCAGAC |
| △XC0449-D | AAGCTTCGGGAAGCGACGCAGTG |
| XC0449-F | CATATGATGAGCGATCTCGACACCCC |
| XC0449-R | AAGCTTGTCCTTGCTGGCCATCCG |
| tmRNA-F | CCAACGACGACAACTACGGT |
| tmRNA-R | ACGAGCACGGGCACAAG |
| qRT-0705-F | TCAATGCGGCGTACTTCC |
| qRT-0705-R | TCGCCGCCCATCACAT |
| P0705-F | GCCAAGTAACGCCACGCAC |
| P0705-R | GACGTAGTCACCCGAAGAAGAAGA |
| GUS-0705-F | GAATTCTCGTTTCCTTGCCAAGTAACG |
| GUS-0705-R | GGATCCCCCGAAGAAGAAGAGACGCG |
各种DNA限制性内切酶、T4 DNA连接酶购自TaKaRa和Promega公司。各种Taq酶购自Qiagen公司。其他常用分子生物学及生化试剂均为国产或进口分析纯。
1.2 方法 1.2.1 分子生物学基本操作PCR扩增、酶切、连接、转化大肠杆菌感受态细胞等常规分子生物学基本操作均按Molecular Cloning[19]进行。
1.2.2 细菌的培养条件LB培养基(蛋白胨10 g/L,酵母提取物5 g/L,NaCl 10 g/L)用于培养大肠杆菌,培养温度为37 ℃;NYG培养基(蛋白胨5 g/L,酵母提取物3 g/L,甘油20 g/L)用于培养野油菜黄单胞菌(X. campestris pv. campestris 8004,Xcc),培养温度为28 ℃;在特定的诱导条件下需要用到MMX培养基(葡萄糖5 g/L,(NH4)2SO4 2 g/L,柠檬酸三钠1 g/L, K2HPO4 4 g/L,KH2PO4 5 g/L,MgSO4 0.2 g/L)以及XVM2培养基(NaCl 1.2 g/L,(NH4)2SO4 1.32 g/L,MgSO4 1.23 g/L,CaCl2 0.11 g/L,K2HPO4 0.022 g/L, KH2PO4 0.073 g/L,FeSO4 0.003 g/L,果糖1.8 g/L,蔗糖3.43 g/L)。抗生素的配制浓度为利福平(Rifampicin,25 µg/mL),卡那霉素(Kanamycin,50 µg/mL)和壮观霉素(Spectinomycin,150 µg/mL)。
1.2.3 读框内删除突变体以及双突菌株的构建按照内删除引物设计原则设计引物,具体引物设计见表 1。分别扩增两条片段,酶切连接到pK18mobSacB质粒上,测序酶切正确后,电击到野油菜黄单胞菌野生型感受态中,进行二次交换后,通过扩增片段大小判断菌株是否正确。
双突菌株的构建即用成功构建的单突变体菌株制备感受态,将pK18mobSacB上构建的另一基因重组载体转化到单突变体感受态中,进行二次交换,并筛选出正确的双突变体菌株。
1.2.4 植物致病力、胞外酶表型分析野油菜黄单胞菌Xcc 8004对于寄主(Brassica oleracea cv. Jingfeng 1)的致病力通过剪叶法来进行观测[20]。
胞外酶检测:将菌液摇至OD600=0.4,取1.5 µL于含有0.1%可溶性淀粉的NYG平板上,置于28 ℃。培养48 h后用1︰100 I2/KI (0.08 mol/L I2,3.2 mol/L KI)溶液染色15 min。用清水将染色液洗净后观察淀粉酶产生的透明圈即可,每个实验3次重复。
1.2.5 总RNA制备、RT-PCR以及qRT-PCR总RNA的制备:将菌液培养过夜至OD600 = 0.4后,用XVM2培养基洗菌2次,用XVM2培养基诱导10 min,之后采用Trizol方法提取RNA。
RT-PCR:用反转录试剂盒(Promega, catalog # A3500)反转录RNA为单链cDNA。
qRT-PCR:反转后的cDNA作为模板,参考上海翊圣生物科技有限公司HieffTM qPCR SYBR® Green Master Mix说明书配制反应体系。
1.2.6 GUS转录融合构建及酶活性测定实验构建GUS转录融合突变体:以Xcc 8004的DNA作为模板,扩增XC0705基因起始密码子ATG上游除去SD (Shine-Dalgarno)序列的200 bp基因启动子,然后通过酶切,将其与gus基因、pHMⅡ载体连接,得到的重组质粒电击转化至Xcc野生型或突变体菌株感受态细胞中,提质粒,酶切验证正确即为正确菌株。
GUS酶活测定:过夜培养细菌将其摇到OD600为0.4,用XVM2培养基洗菌2次后,在XVM2环境中诱导10 min,取2 mL菌液于1.5 mL进口EP管中,离心收菌放入液氮中,保存于–80 ℃。在每管中加入300 µL PBS,重悬菌体后加入终浓度为1 mmol/L的苯甲基磺酰氟(PMSF),进行非接触式超声,开关各30 s,共超声15个循环,高速离心取上清为蛋白样品。用Coomassie Brilliant Blue G-250 Protein Assay (Bio-Rad)试剂进行蛋白定量,根据实际情况稀释蛋白样品。取稀释好的蛋白10 µL,加入到96孔板中,迅速加入反应液(1 mmol/L 4-甲基伞形酮- β-D-葡萄糖苷与PBS的混合液),加入时即开始计时,37 ℃培养5 min、10 min、15 min,时间到时立即加入200 μL的0.4 mol/L Na2CO3至60 μL的反应液中终止反应。用酶标仪(激发波长为360 nm,吸收波长为460 nm)测定荧光值,用不同浓度的4-MUG的反应产物4-甲基伞形酮(4-MU)作为标准品制作标准曲线,计算样品中GUS的酶活性[21]。
1.2.7 蛋白的原核表达及纯化扩增目的片段、连接转化到pGEM T-easy载体,测序正确后,酶切连接pET30a载体,挑取正确的质粒电击到BL21感受态细胞中,提质粒进行酶切验证,正确的菌株即为构建好的带有His6标签的融合蛋白表达菌株。
蛋白的表达:构建好的菌株摇培到OD600为0.6左右加入终浓度0.3 mmol/L的异丙基硫代半乳糖苷(IPTG),16 ℃过夜诱导,收集的菌体按3 mL/g的比例加入含20 mmol/L咪唑的1×Ni-NTA结合缓冲液中重悬。冰上超声处理至菌体透光后,4 ℃收集上清,即可溶性蛋白提取物。按5−10 mg/mL融合蛋白Ni-NTA His·Bind (Novagen)树脂的量加入树脂悬液至手动装好的层析柱中,将离心后的上清加入到平衡好的树脂中,4 ℃结合2−3 h。加入含45 mmol/L咪唑的4 mL 1×Ni-NTA漂洗缓冲液洗涤树脂4次以洗去杂蛋白,最后用含250 mmol/L咪唑的1×Ni-NTA洗脱缓冲液洗脱特异性吸附于树脂上的带有His6标签的融合蛋白,收集此样品,进行SDS-PAGE,考马斯亮蓝染色验证。
1.2.8 Western blotting半干转膜:SDS-PAGE中分离完毕后,在半干转膜仪(Trans-Blot® SD Semi-Dry Electrophoretic Transfer Cell)进行半干转膜,小心取出膜用于Western blotting。
Western blotting:用TBST (10 mmol/L Tris-HCl,pH 7.5,150 mmol/L NaCl,1 mmol/L EDTA,pH 8.0,0.1% Tween)加5%脱脂奶粉作为封闭液,将转膜后的PVDF放入其中常温封闭1 h,或4 ℃封闭过夜。后进行一抗孵育1 h,用TBST洗膜3次,每次10 min,之后进行二抗孵育1 h,再用TBST洗3次,每次10 min,根据二抗标记物HRP,选择用ECL底物发光压X片显影。
1.2.9 体外Pull-down实验分别纯化蛋白带有His6标签的HpaR和带有MBP标签的XC0449蛋白,分别将两个蛋白和混合蛋白加入到Co-IP缓冲液中(50 mmol/L Tris- HCl,pH 7.5,50 mmol/L NaCl,0.2% Triton X-100,1 mmol/L EDTA,pH 8,10% glycerol)和50 µL能特异性吸附MBP标签的磁珠中,混合为终浓度为10 mmol/L的1 mL混合物,4 ℃过夜结合。15 min后离心取上清,将上清和过夜结合的磁珠以及MBP抗体4 ℃结合1 h以上。弃上清,用PBS洗脱2次,再用含1% Triton和1 mmol/L EDTA的PBS洗2次后,加入1×SDS上样缓冲液煮8 min后,取上清为Elute的样品。将每一步洗脱的上清加入1×SDS上样缓冲液后煮,和Elute一起点样进行Western blotting分析。
1.2.10 凝胶迁移阻滞试验(EMSA)以基因启动子区为靶标设计引物,以野油菜黄单胞菌Xcc 8004基因组DNA为模板进行PCR扩增,回收得到目的片段。在40 µL体系中,加入1 pmol的探针,4 µL T4多聚核苷酸激酶(PNK)缓冲液以及1 µL PNK酶,其余部分用水补齐后,37 ℃标记1 h。将纯化蛋白、10×EMSA结合缓冲液(100 mmol/L Tris-HCl,pH 7.5, 500 mmol/L KCl,10 mmol/L DTT)以及适量的非竞争DNA探针混合在一起,混匀后室温反应30 min。加入300 fmol标记好的探针至20 µL的反应体系中,室温反应30 min。上样于5%的EMSA非变形PAGE胶,电泳缓冲液为0.5×TBE,100 V预电泳30 min后上样,120 V电泳1 h左右。将胶块置于滤纸上,放入到透明袋中,置于磷屏上,压3 h左右后扫描检测。
1.2.11 微量热涌动实验(MST)按照Monolith NTTM Protein Labeling Kit RED-NHS (Cat Nr: L001)试剂盒的方法标记纯化的蛋白。用MST缓冲液(50 mmol/L Tris-HCl,pH 7.8,150 mmol/L NaCl,10 mmol/L MgCl2,0.05% Tween-20)成倍稀释标记好的蛋白,使荧光值在800–1 200之间。准备16个离心管,第2−16管每个管子中加入10 µL MST缓冲液,每次需要换枪头取小分子或者是受体蛋白等配体10 µL加入到第一个管子中,再取10 µL到第2个管子中,轻轻吸打混匀后梯度稀释,直到最后一个管子中吸打混匀后,吸出10 µL弃之。在每个管子中加入10 µL稀释好的标记蛋白,每次需要更换枪头。将每个管子中的溶液混匀,避免产生气泡后,用毛细管吸取液体,上机测量Monolith NT. Label Free (Nano Temper Technologies GMBH, Germany),LED power根据蛋白的荧光值调整,MST power为80%。扫描结束后,用Nano Temper Analysis Software对实验结果进行分析,导出数据并作图。
2 结果与分析 2.1 野油菜黄单胞菌Xcc 8004编码9个MarR家族转录因子为确定野油菜黄单胞菌Xcc 8004基因组中含MarR家族蛋白的数目,利用HMMER2算法[6]和Xcc 8004蛋白组序列信息搜索Pfam数据库(E-value≤1.0)。在结果中共搜索得到9个编码MarR家族的蛋白(XC0273、XC0363、XC0449、XC0601、XC1457、XC2827 (HpaR)、XC2840、XC3419和XC4254),这些蛋白都包含典型的MarR家族的DNA结合结构域,其E值在9.10E-06至8E-20之间,推测均能发挥转录因子功能。使用ClustalX软件对这9个MarR家族转录因子进行聚类分析,根据MEGA7.0中的最大似然法构建系统发育树,结果如图 1所示。系统发育树分析结果表明,XC2827 (HpaR)与XC2840聚在一个分支上,而XC0449与XC0601聚在一个分支上。

|
| 图 1 野油菜黄单胞菌中MarR家族转录因子系统发育树分析 Fig. 1 The phylogenetic tree of MarR family in X. campestris pv. campestris 8004 was inferred using the Maximum Likelihood method. The analysis involved 9 amino acid sequences. All positions containing gaps and missing data were eliminated. There were a total of 135 positions in the final dataset. Evolutionary analyses were conducted in MEGA 5 |
| |
利用大肠杆菌BL21 (DE3)菌株和pET30a载体系统表达并纯化了MarR家族转录因子。重组蛋白在开放读码框(Open-reading-rame,ORF) C端带上His6标签,16 ℃、1 mmol/L IPTG诱导表达,Western blotting检测其表达结果。用Ni-NTA His-Bind树脂亲和层析,从表达菌株的粗提液中纯化上述重组蛋白,用含40 mmol/L咪唑的磷酸缓冲液冲洗杂蛋白,含250 mmol/L咪唑的磷酸缓冲液收集洗脱液,10 kDa浓缩柱浓缩并置换至蛋白保存液中。如图 2所示,除HpaR蛋白[18]以外,同时还纯化得到纯度在95%以上的MarR家族转录因子XC0449,其理论大小分别是19.7 kDa (XC0449-His6)和20.0 kDa (HpaR-His6)。
|
| 图 2 MarR家族转录因子的纯化蛋白XC0449和HpaR Fig. 2 The purified MarR family transcription factors XC0449 and HpaR. The final amount of protein is 10 µg, and the gel was stained with Coomassie Blue |
| |
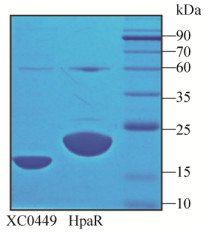
实验室前期研究结果表明,系统敲除MarR家族转录因子后,除HpaR[18]以外,XC0449突变体同样导致细菌致病力显著下降。既然HpaR和XC0449都参与到细菌致病途径中,那么作为转录因子,它们可能通过相互作用调控同一个致病相关基因,并在转录水平发生协同或拮抗效应。鉴于以上推测,我们首先设计了检测HpaR和XC0449互作的体外实验。如图 3A所示,微量热泳动(Microscale thermophoresis,MST)实验表明,体外XC0449蛋白与HpaR能发生中等强度的特异性结合,其解离常数(Kd)为(1.03±0.3) μmol/L,而与其他的MarR家族转录因子在体外并不结合(图 3B)。推测XC0449蛋白可能在体内对HpaR的转录活性有特异性的影响。进一步表达并纯化N端带有MBP标签的0449融合蛋白,进行体外验证蛋白-蛋白互作的Pull-down实验。分别将纯化的HpaR-his6蛋白、MBP-XC0449蛋白以及能特异性吸附MBP标签的磁珠三者混合并在低温下孵育过夜,通过蛋白-蛋白相互作用获得结合在磁珠上的蛋白复合体,中等离子强度的缓冲液清洗后,SDS上样缓冲液裂解蛋白复合体,用鼠源His6单克隆抗体,Western blotting检测复合体中HpaR-his6蛋白的存在。实验结果同样表明,在体外XC0449能与HpaR相互作用(图 3C)。
|
| 图 3 微量热泳动实验 (MST) 和体外Pull-down实验证明XC0449和HpaR之间存在相互作用 Fig. 3 Identification of interaction between HpaR and XC0449 by Microscale thermophoresis (MST) and Pull-down assays. (A) Microscale thermophoresis (MST) analysis revealed the XC0449 binds HpaR. (B) MST analysis revealed the XC0449 does not bind XC0273, which is another MarR family protein and acts as the negative control. Labeled XC0449 protein and diluted HpaR or XC0273 proteins as ligand. The solid curves fit the data points to the standard KD-Fit function. Black bars represented standard deviations. Kd=dissociation constant. The experiments were repeated three times. (C) Pull-down experiment revealed the interaction between HpaR-His6 and MBP-XC0449. Mix protein represent the fusion protein of HpaR and XC0449. The gel was transferred to PVDF membrane and incubated with different antibodies |
| |
为进一步探究XC0449基因功能以及与hpaR可能存在的遗传上下位关系,构建了XC0449基因读码框删除突变体以及XC0449与hpaR基因双突变体并进行致病力等表型分析。结果如图 4A所示,XC0449单突变体能显著降低Xcc对寄主甘蓝的致病性,其致病水平下降至野生型的50%左右,而XC0449与hpaR双突变体致病表型则与hpaR单突变体相似,几乎完全丧失。这一结果提示在遗传上,XC0449位于hpaR基因的上游或处于另外一条致病力调控的途径上。胞外蛋白酶活性检测发现,XC0449与hpaR双突变体表型与hpaR单突变体相似,与野生型相比,胞外蛋白酶活性均显著上升,而XC0449单突变体则无明显变化,表明XC0449不参与调控蛋白酶(图 4B)。

|
| 图 4 hpaR与XC0449表型分析 Fig. 4 The phenotypic characterization of hpaR and XC0449. (A) Bacterial virulence against host plant B. oleracea cv. Jingfeng No.1. Virulence scores were estimated 10 days after inoculation. Sterile MgCl2 (10 mmol/L) was inoculated as a negative control. Virulence scores of bacterial strains represent the virulence levels, and were estimated by a semi-quantitative standard. A sterisk indicates significant difference compared with the WT strain (P < 0.05, n=12). (B) Assay of extracellular protease production. Error bars were calculated by Student's t-test and represented standard deviations (n=6) |
| |
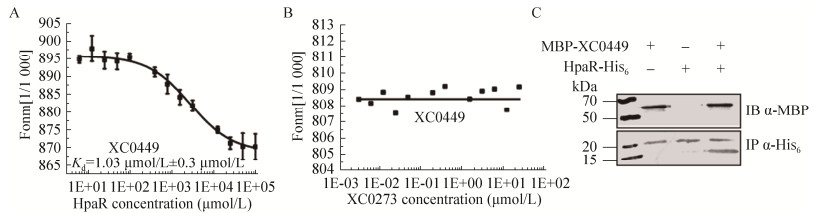
既然HpaR和XC0449在体外能相互作用,并都参与到细菌致病过程中,那它们是否可能共同调控同一个致病相关基因?结合XC0449与hpaR单突和双突的表型结果以及实验室已发表的hpaR调控元的ChIP-seq组学数据,初步确定编码多聚半乳糖醛酸内切酶的XC0705作为下游靶基因做后续验证。如图 5A所示,凝胶迁移实验(Electrophoretic mobility shift assay,EMSA)表明,体外XC0449和HpaR蛋白均能直接结合基因XC0705的启动子区。进一步分析XC0449和HpaR与启动子结合后的竞争条带发现,在加入100×未标记的PXC0705探针后,与HpaR结合的标记探针条带强度明显减弱,而与XC0449结合的标记探针条带则没发生明显变化,说明HpaR竞争XC0705启动子的能力要强于XC0449。在加入1 000×未标记的PXC0705探针后,与XC0449结合的标记条带才发生强度明显减弱且位置下移,也说明XC0449与PXC0705的结合能力更强。
|
| 图 5 EMSA、qPCR及GUS检测HpaR和XC0449对于XC0705的直接调控 Fig. 5 Identification that HpaR and XC0449 bind to and positively regulate XC0705 in vitro and in vivo. (A) Electrophoretic motility shift assay (EMSA) revealed that HpaR and XC0449 protein both can bind the promoter of XC0705. The PCR products of the promoter regions of XC0705 operons was labeled with [γ-32P] ATP and used as DNA probes in the EMSA. Increasing amounts of unlabeled DNA probes were used as competitors (from 100× to 1 000×). Each experiment was repeated three times. (B) qRT-PCR was used to quantify the mRNA amounts of these genes in different bacterial strains. The amplification of the cDNA of tmRNA was used as an internal control. (C) and (D) GUS activity assay. Detect the GUS activity after inducing in MMX medium or XVM2 medium for 10 min |
| |
结合遗传上位分析的结果,推测体内在转录因子通过相互作用调控同一下游基因的过程中,HpaR起着主要作用,而XC0449则协同调控HpaR发挥功能。如图 5B所示的qPCR结果表明,在三型分泌系统诱导培养基XVM2中诱导10 min后,和野生型相比,在XC0449和hpaR单突变体中,XC0705的表达量均显著下降,分别降至野生型中XC0705表达量的78%和58%,且双突变体中XC0705表达量下降最为严重,降至野生型的37%,说明XC0449和HpaR均能正调控XC0705的表达,且两者能协同作用使得对下游基因的调控具有叠加效应。我们进一步构建了XC0705的GUS转录融合突变体,在XVM2培养基或MMX基本培养基中诱导生长10 min后,hpaR单突变体中的GUS酶活均显著降低,XC0449单突变体在XVM2中有显著降低,在MMX中无显著变化,但XC0449和hpaR双突变体表现则与qPCR类似,即协同调控使得下游基因的表达出现了叠加效应。
3 讨论MarR家族转录因子早在古生菌和细菌分化之前就已出现,并且在这些物种中普遍存在。在Ensembl (http://asia.ensembl.org/index.html)中MarR家族转录因子已在超过44 000种细菌和古生菌中收录了超过335 000条注释,平均每个基因组大约有7个MarR家族转录因子。且物种的基因组越大,生活方式越复杂,包含的MarR家族转录因子一般就越多[5]。这说明MarR家族转录因子在微生物进化过程中,在响应环境变化并维持自身生存方面发挥着重要作用。本研究生物信息学分析发现,Xcc 8004基因组一共编码9个MarR家族转录因子,提示在植物病原细菌致病过程中面临的复杂环境及其可能的依赖于MarR家族转录因子的应对策略。MarR家族转录因子作为感应各种环境变化的传感器,可以调控多种细胞过程,包括毒力、抗生素、抗胁迫反应、活性氧反应以及有害化合物的降解等等[22-28]。如弗氏志贺菌Shigella flexneri中MarR家族转录因子SlyA在酸胁迫环境下通过激活下游谷氨酸脱酸系统相关基因的表达,最终维持细菌的生存[29]。当前,MarR家族转录因子相关研究的热点在于MarR蛋白与环境刺激相关的小分子配体的结合影响其与DNA双链的结合并改变其结构及活性,从而导致下游基因的表达量的适应性变化[30]。
除了小分子,MarR家族转录因子还有可能与哪些生物大分子结合并影响其生化活性?已有研究表明,MarR家族转录因子HpaR (XC2827)在植物病原细菌Xcc 8004侵染寄主甘蓝并致病的过程中发挥关键作用[17]。本研究系统性分析了Xcc 8004基因组中编码的9个MarR家族转录因子,并利用亲和层析纯化了其中的XC0449和HpaR (XC2827)[18]。体外MST和Pull-down实验均表明,XC0449基因编码的转录因子能与HpaR相互作用,并协同调控下游致病相关基因XC0705的表达。
在体外验证XC0449蛋白能结合XC0705启动子区的EMSA实验中,在加入1 000×未标记的PXC0705探针后与XC0449结合的标记条带发生位置下移,而非探针位移条带的曝光量减小或游离的标记探针条带。查阅文献发现,EMSA胶中出现多个条带或者条带位置的变化,是由于蛋白与探针结合过程中,往往会形成单聚体、二聚体甚至多聚体的形式,因而能在非变形胶上处于不同位置[33]。对此,推测加入1 000×竞争性探针后蛋白构象发生变化,由高聚体解聚成低聚体,因而在变性胶中电泳时泳动速度更快,显示为条带位置的下降。
XC0705编码多聚半乳糖醛酸内切酶(Endopolygalacturonase),在植物病原真菌及细菌中的主要功能是在病原菌侵染寄主过程中通过分泌到胞外、降解植物细胞壁从而保证细菌的成功入侵,因而是细菌重要的毒力因子[31-32]。本实验室的系统研究发现了直接调控XC0705表达的两个MarR家族转录因子,在细菌致病过程中能协同激活XC0705的表达,最终调控细菌毒力及胞外蛋白酶合成,从而形成完整的调控通路。
本研究的结果为今后研究和了解MarR家族转录因子打下了基础,同时也为研究其他重要调控基因的相互作用提供了方法和思路。
| [1] |
George AM, Levy SB. Gene in the major cotransduction gap of the Escherichia coli K-12 linkage map required for the expression of chromosomal resistance to tetracycline and other antibiotics. J Bacteriol, 1983, 155(2): 541-548. |
| [2] |
Seoane AS, Levy SB. Characterization of MarR, the repressor of the multiple antibiotic resistance (mar) operon in Escherichia coli. J Bacteriol, 1995, 177(12): 3414-3419. DOI:10.1128/jb.177.12.3414-3419.1995 |
| [3] |
George AM, Levy SB. Amplifiable resistance to tetracycline, chloramphenicol, and other antibiotics in Escherichia coli: involvement of a non-plasmid-determined efflux of tetracycline. J Bacteriol, 1983, 155(2): 531-540. |
| [4] |
Cohen SP, McMurry LM, Hooper DC, et al. Cross-resistance to fluoroquinolones in multiple-antibiotic-resistant (Mar) Escherichia coli selected by tetracycline or chloramphenicol: decreased drug accumulation associated with membrane changes in addition to OmpF reduction. Antimicrob Agents Chemother, 1989, 33(8): 1318-1325. DOI:10.1128/AAC.33.8.1318 |
| [5] |
Deochand DK, Grove A. MarR family transcription factors: dynamic variations on a common scaffold. Crit Rev Biochem Mol Biol, 2017, 52(6): 595-613. DOI:10.1080/10409238.2017.1344612 |
| [6] |
Finn RD, Coggill P, Eberhardt RY, et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Res, 2016, 44(D1): 279-285. DOI:10.1093/nar/gkv1344 |
| [7] |
Wang D, Guo CJ, Gu LJ, et al. Comparative study of the marR genes within the family Enterobacteriaceae. J Microbiol, 2014, 52(6): 452-459. DOI:10.1007/s12275-014-3586-2 |
| [8] |
Alekshun MN, Levy SB, Mealy TR, et al. The crystal structure of MarR, a regulator of multiple antibiotic resistance, at 2.3 resolution. Nat Struct Biol, 2001, 8(8): 710-714. DOI:10.1038/90429 |
| [9] |
Wilkinson SP, Grove A. Ligand-responsive transcriptional regulation by members of the MarR family of winged helix proteins. Curr Issues Mol Biol, 2006, 8(1): 51-62. |
| [10] |
Perera IC, Grove A. Molecular mechanisms of ligand-mediated attenuation of DNA binding by MarR family transcriptional regulators. J Mol Cell Biol, 2010, 2(5): 243-254. DOI:10.1093/jmcb/mjq021 |
| [11] |
Vicente JG, Holub EB. Xanthomonas campestris pv. campestris (cause of black rot of crucifers) in the genomic era is still a worldwide threat to brassica crops. Mol Plant Pathol, 2013, 14(1): 2-18. |
| [12] |
Typas A, Becker G, Hengge R. The molecular basis of selective promoter activation by the σS subunit of RNA polymerase. Mol Microbiol, 2007, 63(5): 1296-1306. DOI:10.1111/j.1365-2958.2007.05601.x |
| [13] |
Qian W, Jia Y, Ren SX, et al. Comparative and functional genomic analyses of the pathogenicity of phytopathogen Xanthomonas campestris pv. campestris. Genome Res, 2005, 15(6): 757-767. DOI:10.1101/gr.3378705 |
| [14] |
Da Silva AC, Ferro JA, Reinach FC, et al. Comparison of the genomes of two Xanthomonas pathogens with differing host specificities. Nature, 2002, 417(6887): 459-463. DOI:10.1038/417459a |
| [15] |
Rott P, Fleites L, Marlow G, et al. Identification of new candidate pathogenicity factors in the xylem-invading pathogen Xanthomonas albilineans by transposon mutagenesis. Mol Plant Microbe Interact, 2011, 24(5): 594-605. DOI:10.1094/MPMI-07-10-0156 |
| [16] |
Ryan RP, Vorh lter FJ, Potnis N, et al. Pathogenomics of Xanthomonas: understanding bacterium-plant interactions. Nat Rev Microbiol, 2011, 9(5): 344-355. DOI:10.1038/nrmicro2558 |
| [17] |
Wei K, Tang DJ, He YQ, et al. hpaR, a putative marR family transcriptional regulator, is positively controlled by HrpG and HrpX and involved in the pathogenesis, hypersensitive response, and extracellular protease production of Xanthomonas campestris pathovar campestris. J Bacteriol, 2007, 189(5): 2055-2062. DOI:10.1128/JB.01331-06 |
| [18] |
Pan Y, Liang F, Li RJ, et al. MarR-family transcription factor HpaR controls expression of the vgrR-vgrS operon of Xanthomonas campestris pv. campestris. Mol Plant Microbe Interact, 2018, 31(3): 299-310. DOI:10.1094/MPMI-07-17-0187-R |
| [19] |
Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. New York: Cold Spring Harbor Laboratory Press, 1989.
|
| [20] |
Dow JM, Clarke BR, Milligan DE, et al. Extracellular proteases from Xanthomonas campestris pv. campestris, the black rot pathogen. Appl Environ Microbiol, 1990, 56(10): 2994-2998. |
| [21] |
Wang L, Pan Y, Yuan ZH, et al. Two-component signaling system VgrRS directly senses extracytoplasmic and intracellular iron to control bacterial adaptation under iron depleted stress. PLoS Pathog, 2016, 12(12): e1006133. DOI:10.1371/journal.ppat.1006133 |
| [22] |
Davis JR, Brown BL, Page R, et al. Study of PcaV from Streptomyces coelicolor yields new insights into ligand-responsive MarR family transcription factors. Nucleic Acids Res, 2013, 41(6): 3888-3900. DOI:10.1093/nar/gkt009 |
| [23] |
Kallscheuer N, Vogt M, Kappelmann J, et al. Identification of the phd gene cluster responsible for phenylpropanoid utilization in Corynebacterium glutamicum. Appl Microbiol Biotechnol, 2016, 100(4): 1871-1881. |
| [24] |
Kim Y, Joachimiak G, Bigelow L, et al. How aromatic compounds block DNA binding of HcaR catabolite regulator. J Biol Chem, 2016, 291(25): 13243-13256. DOI:10.1074/jbc.M115.712067 |
| [25] |
Weatherspoon-Griffin N, Wing HJ. Characterization of SlyA in Shigella flexneri identifies a novel role in virulence. Infec Immun, 2016, 84(4): 1073-1082. DOI:10.1128/IAI.00806-15 |
| [26] |
Ellison DW, Miller VL. Regulation of virulence by members of the MarR/SlyA family. Curr Opin Microbiol, 2006, 9(2): 153-159. DOI:10.1016/j.mib.2006.02.003 |
| [27] |
Liu GJ, Liu X, Xu HJ, et al. Structural insights into the redox-sensing mechanism of MarR-type regulator AbfR. J Am Chem Soc, 2017, 139(4): 1598-1608. DOI:10.1021/jacs.6b11438 |
| [28] |
Palm GJ, Chi BK, Waack P, et al. Structural insights into the redox-switch mechanism of the MarR/DUF24-type regulator HypR. Nucleic Acids Res, 2012, 40(9): 4178-4192. DOI:10.1093/nar/gkr1316 |
| [29] |
Zhang BY, Ran LH, Wu M, et al. Shigella flexneri regulator SlyA controls bacterial acid resistance by directly activating the glutamate decarboxylation system. Front Microbiol, 2018, 9: 2071. DOI:10.3389/fmicb.2018.02071 |
| [30] |
Grove A. MarR family transcription factors. Curr Biol, 2013, 23(4): R142-R143. DOI:10.1016/j.cub.2013.01.013 |
| [31] |
Flego D, Marits R, Eriksson ARB, et al. A two-component regulatory system, pehR-pehS, controls endopolygalacturonase production and virulence in the plant pathogen Erwinia carotovora subsp. carotovora. Mol Plant Microbe Interact, 2000, 13(4): 447-455. DOI:10.1094/MPMI.2000.13.4.447 |
| [32] |
Ten Have A, Mulder W, Visser J, et al. The endopolygalacturonase gene Bcpg1 is required for full virulence of Botrytis cinerea. Mol Plant Microbe Interact, 1998, 11(10): 1009-1016. DOI:10.1094/MPMI.1998.11.10.1009 |
| [33] |
Hellman LM, Fried MG. Electrophoretic mobility shift assay (EMSA) for detecting protein-nucleic acid interactions. Nat Protoc, 2007, 2(8): 1849-1861. DOI:10.1038/nprot.2007.249 |
 2019, Vol. 35
2019, Vol. 35