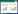中国科学院微生物研究所、中国微生物学会主办
文章信息
- 郭敏, 赵子雅, 王瑞宁, 郑晓宁, 彭永东, 刘铮铸, 李祥龙, 巩元芳
- Guo Min, Zhao Ziya, Wang Ruining, Zheng Xiaoning, Peng Yongdong, Liu Zhengzhu, Li Xianglong, Gong Yuanfang
- 北极狐β-防御素103基因(CBD103)启动子活性及转录调控元件分析
- Activity and transcriptional regulatory elements of the promoter in Arctic fox (Vulpes lagopus) β-defensin103 gene
- 生物工程学报, 2019, 35(8): 1469-1477
- Chinese Journal of Biotechnology, 2019, 35(8): 1469-1477
- 10.13345/j.cjb.190063
-
文章历史
- Received: February 13, 2019
- Accepted: June 19, 2019
2. 河北农业大学 动物科技学院,河北 保定 071000
2. College of Animal Science and Technology, Agricultural University of Hebei Province, Baoding 071000, Hebei, China
多彩的毛皮不仅是动物在自然选择下所遗留的成果,更为动物界增添了靓丽。毛色作为一种重要的表型遗传标记,在确定品种纯度、亲缘关系等方面发挥着至关重要的作用,同时对毛皮品质也有着重要的影响[1-2]。黑色素是位于动物表层结构中最重要的色素[3-4]。防御素(Defensins)是一种阳离子抗菌肽,根据其空间结构的不同可分为α、β和θ防御素[5];其中β-防御素103基因主要在哺乳动物上皮组织中表达,其编码的防御素是富含精氨酸残基的分泌型多肽。哺乳动物CBD103基因通常含有1个内含子和2个外显子,第一个外显子编码5′端非编码序列、信号序列和部分前导序列,第二个外显子编码剩余前导序列、3′端非编码序列以及成熟肽[6]。CBD103基因在一些哺乳动物毛色类型转换的相关研究中表现出重要作用,该基因编码蛋白结构与Agouti基因编码蛋白结构相似,其蛋白产物竞争性结合MC1R且亲和力极高,可改变黑色素合成通路,最终改变动物的毛色[7]。这一结果拓展了对β-防御素作用的认识,其不仅对哺乳动物具有免疫作用[8],还可通过黑皮质素受体信号最终改变动物毛色,成为毛色调控的候选基因。在基因表达调控过程中起“总指挥”作用的启动子具有决定转录起始和调控基因表达的功能[9]。目前尚未见北极狐CBD103基因调控毛色的报道。本研究利用先进的分子生物学技术结合生物信息学分析[10-11]等方法对CBD103基因启动子区及调控元件进行预测及分析,探明该基因的核心启动子区域及重要的调控元件,为深入探究该基因对北极狐毛色形成的调控作用提供了理论支持。
1 材料与方法 1.1 材料与试剂北极狐皮肤组织样品取自河北省秦皇岛市昌黎县金岛育种场。LA Taq DNA Polymerase、pMD19-T Vector、大肠杆菌Escherichia coli DH5α感受态细胞、反转录试剂盒和T4 DNA连接酶购自大连TaKaRa公司;2×TransStart FastPfu PCR SuperMix和TransStart Taq DNA Polymerase购自北京全式金生物技术有限公司;TRIzol试剂盒、胰蛋白酶、限制性内切酶、血清、Lipofectamine 2000转染试剂及Opti-MEM培养基均购自Thermo Fisher Scientific公司(美国);质粒DNA小量提取抽提试剂盒、无内毒素质粒DNA大量提取试剂盒和DNA胶回收试剂盒购自天根生化科技(北京)有限公司;D-PBS和DMEM培养基(高糖型)购自Hyclone公司(美国)[12]。本实验室保藏有人恶性黑色素瘤细胞系(A375)、人肾上皮细胞系(293T)、质粒载体pRL-TK和pGL3-Basic。
1.2 北极狐CBD103基因启动子的克隆扩增课题组前期扩增获得北极狐CBD103基因序列2 327 bp (包括5′侧翼区2 071 bp、CDS区204 bp、3′侧翼区52 bp),并利用生物信息学的方法对启动子区及调控元件进行了预测[13]。根据预测结果,选取起始密码子上游2 000 bp左右的序列作为候选启动子区并设计引物(表 1),PCR扩增4个缺失片段(图 1)。。使用Primer Premier 5.0软件分析限制性内切酶位点。PCR扩增体系(50 μL):其中基因组DNA 1 μL,10×PCR缓冲液(Mg2+ Plus) 5 μL,dNTP Mixture (2.5 mmol/L) 8 μL,上游引物(20 μmol/L) 1 μL,下游引物(20 μmol/L) 1 μL,LA Taq DNA聚合酶(5 U/μL) 0.5 μL,灭菌ddH2O 33.5 μL。使用JASPAR、AliBaba 2.1、Nsite、Cluster和Signal SCAN在线网站预测启动子区的转录因子结合位点(表 2)。
| Primer name | Primer sequence (5′–3′) | Annealing temperature (℃) | Extension time (min) |
| 2123F | GGACGCGTCCCCAGTAGAGGAAAAGTG | 57 | 2.0 |
| 1656F | GGACGCGTACTCGCTTCCTCCCTCATTT | 57 | 2.0 |
| 1178F | GGACGCGTCACGGCACTTGTCTATCTGT | 56 | 2.0 |
| 559F | GGACGCGTATTTCCATTTCAGGCACACC | 58 | 2.0 |
| R | CCAAGCTTCAGGCATCAAGAACAGCAAG | ||
| Mut-Sp1-1-F | GGCACCTGAGTTGCTCATTAG | 58 | 0.5 |
| Mut-Sp1-1-R | CTAATGAGCAACTCAGGTGCC | ||
| Mut-Sp1-2-F | TCTGTGCTTACTCACGCATCT | 57 | 0.5 |
| Mut-Sp1-2-R | AGATGCGTGAGTAAGCACAGA | ||
| Mut-Sp1-3-F | CCCAAGAAGTAGGATGTTTTC | 57 | 0.5 |
| Mut-Sp1-3-R | GAAAACATCCTACTTCTTGGG | ||
| Note: the underlined and thickened bases represent restriction sites and mutant bases, respectively. | |||
|
| 图 1 北极狐CBD103基因预测启动子片段 Fig. 1 The predicted promoter fragment of Arctic fox CBD103 gene. |
| |
| Software | Website |
| JASPAR | http://jaspar.binf.ku.dk/ |
| AliBaba 2.1 | http://gene-regulation.com/pub/programs/alibaba2/index.html |
| Nsite | http://www.softberry.com/berry.phtml?topic=nsite&group=programs&subgroup=promoter |
| Cluster | http://zlab.bu.edu/cluster-buster/cbust.html |
| Signal SCAN | http://www-bimas.cit.nih.gov/molbio/signal/ |
将不同缺失片段的PCR扩增产物连接到pMD19-T载体中构建重组克隆载体pMD19-X。经过菌液PCR鉴定为阳性的样品送至生工生物工程(上海)股份有限公司测序。重组克隆载体pMD19-X及pGL3-Baisc载体同时进行双酶切,将回收产物连接至pGL3-Baisc中构建pGL3-Baisc-X重组报告载体。
1.4 构建及鉴定突变载体分别以1656F和Mut-Sp1-1-R、Mut-Sp1-1-F和R、1656F和Mut-Sp1-2-R、Mut-Sp1-2-F和R、1656F和Mut-Sp1-3-R、Mut-Sp1-3-F和R为引物(表 1),以pGL3-Baisc-1656质粒为模板,通过重叠延伸PCR技术扩增上下游产物。重叠延伸PCR扩增体系(40 μL):野生型质粒模板2 μL,2×TransStart FastPfu Super Mix 20 μL,上下游引物(20 μmol/L)各1 μL,灭菌ddH2O 16 μL;将上述扩增的产物进行回收并进行等比例混合,用此混合物为模板,以引物1656F和R扩增突变载体全长片段,PCR扩增体系(50 μL):其中混合物模板2.5 μL,10×TransStart Taq缓冲液5 μL,TransStart Taq DNA聚合酶0.5 μL,dNTP Mixture (10 mmol/L) 1 μL,上下游引物(20 μmol/L)各1 μL,灭菌ddH2O 39 μL。
1.5 双荧光素酶活性鉴定利用Lipofectamine 2000脂质体分别将荧光素酶报告基因质粒pGL3-Baisc-X和内参质粒pRL-TK一起转染至293T和A375细胞中,转染细胞48 h后收集细胞,按照双荧光素酶检测试剂盒说明书对基因启动子活性进行检测。使用发光仪检测pGL3质粒中萤火虫荧光素酶活性,读出数值为F,检测pRL-TK质粒中海肾荧光素酶活性,读出数值为R。用F/R的值代表基因启动子相对活性。
1.6 统计分析利用SPSS 24.0软件中的单因素方差分析对不同检测片段的启动子活性数据进行统计学分析,P < 0.05表示差异显著性,P < 0.01表示差异极显著性。
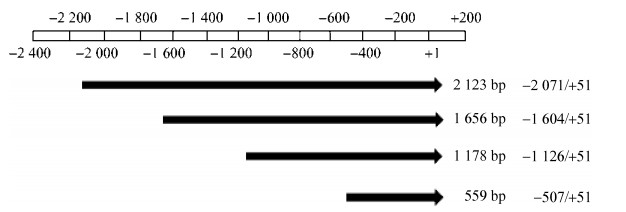
2 结果与分析 2.1 北极狐CBD103基因启动子缺失载体的扩增以基因组DNA为模板,对北极狐CBD103基因候选启动子区域进行不同缺失片段的扩增,分别扩增出片段1-4:2 123 bp (-2 017/+51)、1 656 bp (-1 604/+51)、1 178 bp (-1 126/+51)和559 bp (-507/+51),1.2%琼脂糖凝胶电泳检测结果见图 2,扩增出的条带大小与预期相一致。

|
| 图 2 北极狐CBD103基因启动子缺失片段PCR产物凝胶电泳 Fig. 2 Agarose gel electropherogram of PCR products of deletion fragments in Arctic fox CBD103 gene promoter. M1: DL5000 marker; 1: 2 123 bp; 2: 1 656 bp; 3: 1 178 bp; 4: 559 bp; M2: DL2000 marker. |
| |
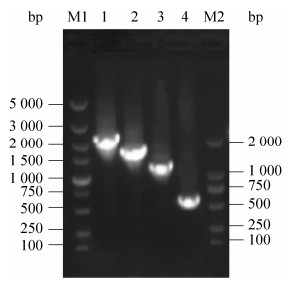
使用T4 DNA连接酶将双酶切后的片段连接到pGL3-Basic质粒上,将构建完成的荧光素酶表达载体进行鉴定(图 3和图 4)。
|
| 图 3 北极狐CBD103基因荧光素酶表达载体PCR产物凝胶电泳 Fig. 3 Agarose gel electropherogram of PCR products of luciferase expression vector in Arctic fox CBD103 gene. M1: DL5000 marker; 1: 2 123 bp; 2: 1 656 bp; 3: 1 178 bp; 4: 559 bp; M2: DL2000 marker. |
| |

|
| 图 4 北极狐CBD103基因荧光素酶表达载体双酶切产物凝胶电泳 Fig. 4 Agarose gel electropherogram of double-enzyme digestion of luciferase expression vector in Arctic fox CBD103 gene. M: DL5000 marker; 1: 2 123 bp; 2: 1 656 bp; 3: 1 178 bp; 4: 559 bp. |
| |
将启动子荧光素酶表达载体质粒与pRL-TK质粒共同转染至A375和293T细胞中,检测双荧光素酶活性(图 5),结果显示4个启动子缺失片段荧光素酶表达载体质粒转染两种细胞的活性值高低趋势一致;除报告基因2 123 (–2 071/+51)与pGL3-basic对照组相比差异显著(P < 0.05)外,其他3个启动子片段的活性与对照组比较均差异极显著(P < 0.01);报告基因1 656 (-1 604/+51)在两种细胞间的活性均最高,且与其他3个片段活性相比,均差异极显著(P < 0.01),表明该区域为该基因的核心启动子区域。

|
| 图 5 北极狐CBD103基因启动子表达载体相对荧光素酶活性值 Fig. 5 Relative luciferase activity of Arctic fox CBD103 gene promoter expression vector. *: significant difference compared with pGL3-basic control group (P < 0.05). **: the difference was extremely significant compared with the pGL3-basic control group (P < 0.01). |
| |
利用多种转录因子在线分析网站对核心启动子区(-1 604/+51)的转录因子结合位点进行预测,为了提高预测的准确性,综合考虑至少2个预测网站的结果(表 3),分析结果表明在-1 552/-1 564、-1 439/-1 454和-329/-339位置存在Sp1结合位点。
| Transcription factor binding sites | Software and database | |||
| Cluster | Nsite | AliBaba2.1 | Signal scan | |
| -1 586/-1 575 | Mef-2 | |||
| -1 552/-1 564 | Sp1 | Sp1 | ||
| -1 439/-1 454 | Sp1 | Sp1 | Sp1 | Sp1 |
| -1 169/-1 160 | OCT-1 | |||
| -681/-672 | USF | |||
| -564/-555 | NF-1 | NF-1 | ||
| -329/-339 | Sp1 | Sp1 | ||
| -146/-137 | CREB | CREB | ||
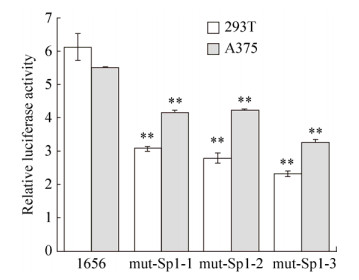
以pGL3-Baisc-1656为模板对预测的3个Sp1结合位点分别进行点突变(图 6),通过重叠延伸PCR技术扩增上下游引物并构建突变载体(图 7),突变前后序列对比见表 4,分别将pGL3-Baisc-1656载体和突变载体转染至A375和293T细胞中,并对启动子活性进行检测,结果表明,相比于野生型载体1656 (-1 604/+51),3个Sp1结合位点突变后的载体启动子活性均极显著下降(P < 0.01) (图 8)。

|
图 6 北极狐CBD103基因转录因子结合位点突变
Fig. 6 llustration of the mutation of transcription factor binding sites of Arctic fox CBD103 gene.   |
| |

|
| 图 7 北极狐CBD103基因启动子突变体的扩增 Fig. 7 Amplification of CBD103 gene promoter mutant in Arctic fox. M: DL-2000 marker; 1: Mut-Sp1-1 upstream fragment; 2: Mut-Sp1-1 downstream fragment; 3: Mut-Sp1-2 upstream fragment; 4: Mut-Sp1-2 downstream fragment; 5: Mut-Sp1-3 upstream fragment; 6: Mut-Sp1-3 downstream fragment; 7: Mut-Sp1-1; 8: Mut-Sp1-2; 9: Mut-Sp1-3. |
| |
| Gene | Transcription factor binding site | Pre mutation sequence | Mutation sequence |
| CBD103 | Sp1 (-1 552/-1 564) | GGCACCTGGGCGGCTCATTAG | GGCACCTGAGTTGCTCATTAG |
| Sp1 (-1 439/-1 454) | TCTGCCCTTCCTCCCGCCTCT | TCTGTGCTTACTCACGCATCT | |
| Sp1 (-329/-339) | CCCAAGAGGAAGGGGGTTTTC | CCCAAGAAGTAGGATGTTTTC | |
| Note: the underlined and thickened bases represent mutant bases. | |||
|
| 图 8 北极狐CBD103基因突变启动子表达载体相对荧光素酶活性值 Fig. 8 Relative luciferase activity of expression vector carrying mutant promoter in Arctic fox CBD103 gene. **: the promoter activity of wild-type 1 656(-1 604/+51) was significantly different from that of the luciferase reporting system (P < 0.01). |
| |
狐皮被称为世界毛皮产业三大台柱之一,其毛色性状[14]将直接影响皮张品质和其经济价值。动物毛色的形成主要受遗传因素控制,基因调控动物毛色形成的机理十分复杂[15]。启动子是RNA聚合酶识别、结合和开始转录的一段DNA序列,就像“开关”决定基因的活动,其克隆方式也越来越多,大致分为基因组文库筛选法、利用启动子探针载体筛选启动子法和基于PCR技术克隆启动子法等,这些技术在生物实验中得到了不同程度的应用[16]。研究表明克隆片段若仅含有转录起始位点上游区域易出现假阳性结果,可选择包含TSS、ATG及部分CDS区来提高阳性率[17]。本研究克隆获得了北极狐CBD103基因启动子序列,构建了4个不同长度的启动子缺失片段表达载体,通过双荧光素酶系统检测启动子活性,以确定启动子的功能序列位置。经检测确定-1 604/+51区域为北极狐CBD103基因的核心启动子区,同时对此区域的调控元件进行预测,结果显示该区域存在3个Sp1 (-329/-339;-1 439/-1 454;-1 552/-1 564)转录因子结合位点。现已在坝上长尾鸡TYR基因[18],北极狐、水貂TYRP1基因[19-20],坝上长尾鸡、北极狐、山羊、水貂PMEL基因[21-24]中的启动子区域发现存在Sp1这一转录因子。Sp1已被证实是一种能影响转录起始的反式激活子[25]。启动子中存在多种顺式作用元件参与基因的转录,如CAAT框、TATA框和GC框等。CAAT框是真核生物基因中普遍存在的调控区,控制着转录起始的频率[26];TATA决定基因转录起始的选择,并能影响转录的速率;GC框主要分布在转录起始位点前-128/-23 bp区域内,一般位于CAAT框的两侧和TATA框上游,能够结合调节蛋白Sp1[27]。有学者指出,某基因中CAAT框若存在于TATA框上游,则该基因的表达量将出现大幅提升[28]。对北极狐CBD103基因启动子区调控元件进行预测发现,该基因在同一区域存在GC框(-1 560)和Sp1 (-1 552/-1 564)转录因子结合位点,推测GC框可能与转录因子Sp1结合而发挥转录激活作用,为北极狐CBD103基因核心启动子区的确定提供参考。研究发现Sp1的抑制表达能够显著降低猪成肌细胞中SKIP启动子活性,Sp1通过GC框对猪成肌细胞分化过程中SKIP基因的转录激活起正调控作用[29]。田宏攀[30]在乳腺癌的研究中发现,Sp1对FOXF2基因启动子转录激活的控制受到DNA甲基化的影响,Sp1可以增强无甲基化和部分甲基化的FOXF2基因启动子转录活性,对完全甲基化的启动子转录活性无影响。3个Sp1转录因子结合位点的突变(-1 552/-1 564、-1 439/-1 454和-329/-339)都能够使得北极狐CBD103基因启动子的活性显著下降,推断以上3个Sp1转录因子结合位点为北极狐CBD103基因的正调控区域。为了进一步探究转录因子Sp1与北极狐CBD103基因之间的转录调控关系,接下来可利用染色质免疫共沉淀(ChIP)和凝胶迁移实验(EMSA)进行验证,并与其他毛色调控基因启动子序列中的转录因子Sp1结合位点在基因转录调控中的具体作用做对比以发现这其间的共性,为进一步探究动物毛色的调控机制做铺垫。
4 结论本研究构建了4个包含北极狐CBD103基因5′端不同长度缺失启动子片段的荧光素酶报告基因表达载体,确定了-1 604/+51区域为北极狐CBD103基因的启动子区;通过生物信息学方法预测出-1 552/-1 564、-1 439/-1 454和-329/-339处存在Sp1转录因子结合位点,并对以上3个结合位点进行点突变和活性检测,结果显示以上3个转录因子Sp1的结合位点为北极狐CBD103基因的正调控区域。
| [1] |
Zhang JZ, Dong CS, Fan RW, et al. Advance in pigment melanin of mammalian. Progr Vet Med, 2006, 27(S1): 65-68 (in Chinese). 张俊珍, 董常生, 范瑞文, 等. 哺乳动物毛色形成研究进展. 动物医学进展, 2006, 27(S1): 65-68. |
| [2] |
Xu GL, Liu ZZ, Gong YF, et al. Research progress on the controlling genes for the coat colors of fur animals. Heilongjiang Anim Sci Vet Med, 2015(13): 52-54 (in Chinese). 徐桂利, 刘铮铸, 巩元芳, 等. 毛皮动物毛色调控基因的研究进展. 黑龙江畜牧兽医, 2015(13): 52-54. |
| [3] |
Krauss J, Geiger-Rudolph S, Koch I, et al. A dominant mutation in TYRP1A leads to melanophore death in zebrafish. Pigment Cell Melanoma Res, 2014, 27(5): 827-830. DOI:10.1111/pcmr.12272 |
| [4] |
Ollmann MM, Lamoreux ML, Wilson BD, et al. Interaction of Agouti protein with the melanocortin 1 receptor in vitro and in vivo. Genes Dev, 1998, 12(3): 316-330. DOI:10.1101/gad.12.3.316 |
| [5] |
Wang W, Cole AM, Hong T, et al. Retrocyclin, an antiretroviral the θ-defensin, is a lectin. J Immunol, 2003, 170(9): 4708-4716. DOI:10.4049/jimmunol.170.9.4708 |
| [6] |
Lehrer RI, Ganz T. Antimicrobial peptides in mammalian and insect host defence. Curr Opin Immunol, 1999, 11(1): 23-27. |
| [7] |
Candille SI, Kaelin CB, Cattanach BM, et al. A β-defensin mutation causes black coat color in domestic dogs. Science, 2007, 318(5855): 1418-1423. DOI:10.1126/science.1147880 |
| [8] |
Jin X, Zhang M, Fan YR, et al. The role of β- defensin in the process of infection and inflammation. Heilongjiang Anim Sci Vet Med, 2015(13): 55-57 (in Chinese). 金鑫, 张曼, 范燕茹, 等. β-防御素在感染和炎症过程中的作用. 黑龙江畜牧兽医, 2015(13): 55-57. |
| [9] |
Li SY, Lang ZH, Huang DF. Research progress on eukaryotic promoter. Curr Biotechnol, 2014, 4(3): 158-164 (in Chinese). 李圣彦, 郎志宏, 黄大昉. 真核生物启动子研究概述. 生物技术进展, 2014, 4(3): 158-164. DOI:10.3969/j.issn.2095-2341.2014.03.02 |
| [10] |
Long X, Chai J, Zhao JG, et al. Cloning, transcriptional activity and regulatory elements analysis of porcine EIF2S3 gene promoter. Acta Agric Bor-Sin, 2018, 33(S1): 38-44 (in Chinese). 龙熙, 柴捷, 赵久刚, 等. 猪EIF2S3基因启动子的克隆、活性及转录调控元件分析. 华北农学报, 2018, 33(S1): 38-44. DOI:10.7668/hbnxb.2018.S1.007 |
| [11] |
Ma TF, Wang JY, Hou JC, et al. Regulating role of hsa-miR-4443 in expression of TIMP2 detected by dual-luciferase reporter system. Acta Med Univ Sci Technol Huazhong, 2018, 47(3): 269-273 (in Chinese). 马腾飞, 王金焱, 侯俊宸, 等. 双荧光素酶报告系统鉴定hsa-miR-4443对TIMP2基因的调控作用. 华中科技大学学报:医学版, 2018, 47(3): 269-273. |
| [12] |
Guo M. Study on promoter activity of fox fur gene TYR、CBD103 and MITF [D]. Qinhuangdao: Hebei Normal University of Science Technology, 2018. (in Chinese) 郭敏.狐狸毛色基因TYR、CBD103、MITF启动子活性探究[D].秦皇岛: 河北科技师范学院, 2018. |
| [13] |
Guo M, Niu D, Liu YJ, et al. Bioinformatics analysis and promoter prediction on CBD103 gene in Arctic fox. Heilongjiang Anim Sci Vet Med, 2018(1): 52-56 (in Chinese). 郭敏, 牛迪, 刘艳军, 等. 北极狐CBD103基因序列生物信息学分析及启动子预测. 黑龙江畜牧兽医, 2018(1): 52-56. |
| [14] |
Ghosh TC, Gupta SK, Majumdar S. Studies on codon usage in Entamoeba histolytica. Int J Parasitol, 2000, 30(6): 715-722. DOI:10.1016/S0020-7519(00)00042-4 |
| [15] |
Suzuki H. Evolutionary and phylogeographic views on MC1r and ASIP variation in mammals. Genes Genet Syst, 2013, 88(3): 155-164. DOI:10.1266/ggs.88.155 |
| [16] |
Tang F, Tu HZ. Advances in eukaryotic promoter. China Forest Sci Technol, 2015, 29(2): 7-12 (in Chinese). 汤方, 涂慧珍. 真核启动子研究进展. 林业科技开发, 2015, 29(2): 7-12. |
| [17] |
Hou DF, Guan YJ, Guan R, et al. Cloning of human NPCEDRG core promoter and preliminary identification of its CCAAT/NFY binding site. Progr Biochem Biophys, 2011, 38(8): 713-723 (in Chinese). 侯德富, 关勇军, 关瑞, 等. 人NPCEDRG基因启动子的克隆及CCAAT/NFY结合位点初步分析. 生物化学与生物物理进展, 2011, 38(3): 713-723. |
| [18] |
Liu XH, Zhou RY, Zhang CS, et al. Identification of the core promoter and SNPs analysis of TYR gene in Bashang long-tail chicken (Gallus gallus). J Agricul Biotechnol, 2018, 26(6): 959-969 (in Chinese). 刘小辉, 周荣艳, 张传生, 等. 坝上长尾鸡TYR基因核心启动子鉴定与单核苷酸多态性分析. 农业生物技术学报, 2018, 26(6): 959-969. |
| [19] |
Zheng XN, Wang RN, Wang YQ, et al. Promoter and transcription regulatory region in fox TYRP1 gene. Chin J Vet Sci, 2018, 38(12): 2366-2373 (in Chinese). 郑晓宁, 王瑞宁, 王亚琪, 等. 北极狐TYRP1基因启动子活性及转录调控区域分析. 中国兽医学报, 2018, 38(12): 2366-2373. |
| [20] |
Li LS, Li LH, Li XL, et al. Promoter activity and Sp1 binding sites of mink TYRP1 gene. Chin J Vet Sci, 2018, 38(5): 991-997 (in Chinese). 李丽莎, 李兰会, 李祥龙, 等. 水貂TYRP1基因启动子活性和Sp1结合位点. 中国兽医学报, 2018, 38(5): 991-997. |
| [21] |
Liu XH, Zhou RY, Peng YD, et al. Identification of the core promoter of the pmel gene of Bashang long-tail chickens. Chin J Biotech, 2018, 34(11): 1750-1759 (in Chinese). 刘小辉, 周荣艳, 彭永东, 等. 坝上长尾鸡pmel基因核心启动子的鉴定. 生物工程学报, 2018, 34(11): 1750-1759. |
| [22] |
Zheng XN, Wang YQ, Wang RN, et al. Activity and regulatory region analysis of PMEL gene promoter in Vulpes lagopus. J Agric Biotechnol, 2018, 26(8): 1351-1360 (in Chinese). 郑晓宁, 王亚琪, 王瑞宁, 等. 北极狐PMEL基因启动子活性及转录调控区域研究. 农业生物技术学报, 2018, 26(8): 1351-1360. |
| [23] |
Li LS, Peng YD, Zheng XN, et al. Analysis of the promoter activity and transcriptional regulatory elements of goat PMEL gene. Chin J Anim Vet Sci, 2017, 48(5): 826-835 (in Chinese). 李丽莎, 彭永东, 郑晓宁, 等. 山羊PMEL基因启动子活性及转录调控元件分析. 畜牧兽医学报, 2017, 48(5): 826-835. |
| [24] |
Li LS, Li LH, Li XL, et al. Analysis of the promoter activity and transcriptional regulatory elements of PMEL gene in mink. J Agricul Biotechnol, 2017, 25(6): 911-920 (in Chinese). 李丽莎, 李兰会, 李祥龙, 等. 水貂PMEL基因启动子活性及转录调控元件分析. 农业生物技术学报, 2017, 25(6): 911-920. |
| [25] |
Zhang X, Luo J, Li JH, et al. Cloning and activity determination of fatty acid synthase (FAS) gene promoter of Xinong Saanen Dairy Goat. Sci Agric Sin, 2010, 43(3): 640-647 (in Chinese). 张晓, 罗军, 李建华, 等. 西农萨能奶山羊脂肪酸合酶基因启动子的克隆及活性测定. 中国农业科学, 2010, 43(3): 640-647. DOI:10.3864/j.issn.0578-1752.2010.03.027 |
| [26] |
Wang Y, Mai WJ, Liang CY, et al. Advances on studies of plant promoters. Acta Bot Bor-Occid Sin, 2003, 23(11): 2040-2048 (in Chinese). 王颖, 麦维军, 梁承邺, 等. 高等植物启动子的研究进展. 西北植物学报, 2003, 23(11): 2040-2048. DOI:10.3321/j.issn:1000-4025.2003.11.037 |
| [27] |
Du XX, Gao XG, Chen PH, et al. Research on gene promoter of fish. Biotechnol Bull, 2013(8): 12-16 (in Chinese). 杜小溪, 高祥刚, 陈潘海, 等. 鱼类基因启动子的研究进展. 生物技术通报, 2013(8): 12-16. |
| [28] |
Sreenivasulu G, Senthilkumaran B, Sudhakumari CC, et al. 20 β-hydroxysteroid dehydrogenase gene promoter: potential role for cyclic AMP and xenobiotic responsive elements. Gene, 2012, 509(1): 68-76. |
| [29] |
Xiong Q, Chai J, Zhang N, et al. Role of transcription factors Sp1 in the expressional regulation of SKIP gene in porcine C2C12 myoblasts. Hubei Agric Sci, 2012, 51(8): 4147-4151 (in Chinese). 熊琪, 柴进, 张年, 等. 转录因子Sp1对成肌细胞中猪SKIP的表达调控作用. 湖北农业科学, 2012, 51(8): 4147-4151. |
| [30] |
Tian HP. DNA Methylation affects the SP1 transcription factor-regulated functions of FORKHEADFOX F2 in breast cancer cell[D]. Tianjin: Tianjin Medical University, 2015 (in Chinese). 田宏攀.乳腺癌中DNA甲基化对Sp1调控FOXF2转录及其功能的影响[D].天津: 天津医科大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10062-1016704099.htm |
 2019, Vol. 35
2019, Vol. 35