中国科学院微生物研究所、中国微生物学会主办
文章信息
- 柴昊男, 张会图, 袁飞燕, 刘焕, 路福平
- Chai Haonan, Zhang Huitu, Yuan Feiyan, Liu Huan, Lu Fuping
- 枯草芽孢杆菌中一种新型双向启动子的功能鉴定
- Characterization of a novel bidirectional promoter in Bacillus subtilis
- 生物工程学报, 2019, 35(7): 1326-1334
- Chinese Journal of Biotechnology, 2019, 35(7): 1326-1334
- 10.13345/j.cjb.190028
-
文章历史
- Received: January 15, 2019
- Accepted: April 2, 2019
枯草芽孢杆菌表达系统作为基因工程表达系统,因具有外源蛋白易分离纯化[1]、适应大规模流线发酵过程[2]、安全性(GRAS认证)[3]及无偏倚密码子的使用[4]等多种优势,使其在表达异种蛋白方面处于首选位置,因此枯草杆菌在酶工业应用中得到了广泛的应用[5]。
工业酶的全球市场估计为16亿美元,其中包括食品酶(29%)、饲料酶(15%)和通用技术酶(56%)[6]。目前市场上使用的大多数洗涤剂蛋白酶是芽孢杆菌丝氨酸蛋白酶[7]。据估计,芽孢杆菌产酶约占整个酶市场的50%[8]。外源蛋白在芽孢杆菌中的高效表达是实现其在工业应用上的重要途径,启动子是影响外源蛋白在芽孢杆菌中高效表达的关键因素之一[9]。
蛋白质的相互作用是实现功能的基础。对蛋白质的功能研究需要考虑蛋白质之间的相互作用,特别是对二聚体的研究,往往需要在同一载体中表达两种蛋白质[10]。构建过程通常将两个不同的启动子插入同一载体,或将两种蛋白质构建成融合蛋白,但都有可能增加转化的难度或影响外源基因的表达。利用双向启动子的启动功能,可以为蛋白质之间的相互作用的研究提供新思路[11]。
双向启动子是反向转录基因对的共享启动子序列,可以调控两个方向的基因的转录[12]。双向启动子在真核生物基因组中广泛分布,大多数的双向启动子具有较高的GC含量和丰富的CpG岛,而缺少TATA盒。双向启动子的双向转录机制一种解释是两个RNA聚合酶同时聚集在无核小体的复制起始区,然后在两个方向上起始转录[13]。尽管大部分双向启动子在两个方向上都有表达活性,但是有约1/10都只在一个方向上进行表达。大多数双向启动子调控双向基因对的共表达,少部分双向启动子一个方向促进转录,另一个方向抑制[14]。双向启动子在原核生物中少有报道,主要集中在大肠杆菌表达系统。常用的原核生物双向启动子是MaZel团队在霍乱弧菌中发现的Pc/Pint[15]。廖昱泓等发现真核生物酵母基因中的双向启动子即MAL1基因启动子区在原核生物大肠杆菌中仍具有双向启动的功能[11]。新的双向启动子的发现,实现了芽孢杆菌基因工程的“一箭双星”的功能,为原核生物双向启动子的研究补充了参考数据,对于加速芽孢杆菌基因工程的研究进展具有重要意义[16]。
目前,国内外由基因组中筛选获取新型启动子的方法主要包括运用启动子探针载体筛选[17]、常规PCR法[18]、序列特异性引物PCR法[19]和基因组文库筛选法[20]等。本研究从地衣芽孢杆菌中筛选获得一种新型的双向启动子,即在地衣芽孢杆菌转录组测序结果的基础上,运用生物信息学等分析获得的lanA1和lanA2基因(编码lichenicidin prepeptide)的启动子pLA/pLB,以强启动子pShuttle-09为对照,以克劳氏芽孢杆菌碱性蛋白酶基因为报告基因,鉴定新型启动子的启动强度。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒大肠杆菌Escherichia coli EC135来自南开大学,枯草芽孢杆菌B. subtilis WB600、地衣芽孢杆菌B. licheniformis 2709和大肠杆菌-枯草芽孢杆菌穿梭表达载体pWH1520均为本实验室保藏。
1.1.2 试剂和培养基DNA聚合酶、限制性内切酶、T4 DNA连接酶购自宝生物工程(大连)有限公司;GeneRuler 1 kb DNA Ladder购自赛默飞世尔科技公司;基因组提取试剂盒、质粒提取试剂盒、DNA切胶回收试剂盒购自Omega Biotek核酸纯化公司;福林酚、酪蛋白底物、三氯乙酸等购自生工生物工程(上海)股份有限公司。
牛奶平板培养基(100 mL):酵母粉0.5 g,蛋白胨1 g,氯化钠1 g,琼脂1.5 g,脱脂奶粉1 g;种子培养基100 mL:酵母粉0.5 g,蛋白胨1 g,氯化钠1 g。
发酵产酶培养基(100 mL):玉米粉6.4 g,豆饼粉4 g,磷酸氢二钠0.4 g,磷酸二氢钾0.03 g,高温淀粉酶0.07 g。
枯草芽孢杆菌感受态制备培养基:电转化感受态制备重悬液(溶液A):D-山梨醇0.5 mol/L,D-甘露醇0.5 mol/L,甘油10%,加去离子水定容,121 ℃灭菌20 min。电转化复苏液(溶液B):蛋白胨10 g/L,酵母粉5 g/L,氯化钠5 g/L,D-山梨醇0.5 mol/L,D-甘露醇0.38 mol/L,加去离子水定容,121 ℃灭菌20 min。
1.2 方法 1.2.1 地衣芽孢杆菌启动子筛选接种地衣芽孢杆菌2709于无菌LB平板上,37 ℃培养过夜,挑取单菌落于种子培养基中,220 r/min过夜培养,按2%接种量分别接种于100 mL发酵培养基,37 ℃、220 r/min振荡培养,每隔12 h进行取样,离心收集上清液,进行产酶活力测定,对蛋白酶表达量较高的样品进行转录组测序分析。在地衣芽孢杆菌的转录组测序结果的基础上,运用生物信息学结合BPROM软件预测基因的转录起始位点,并将转录起始位点上游500 bp的核苷酸进行分析,寻找与之匹配的σ因子和转录因子的识别位点,从而发现启动子候选基因。
1.2.2 地衣芽孢杆菌基因组提取地衣芽孢杆菌基因组DNA使用OMEGA公司Bacterial DNA Kit提取,步骤如下:
1) 收集过夜培养于LB培养基中的菌液,13 000 r/min离心3 min,弃上清,重悬菌体于200 μL无菌水中;2)加40 μL溶菌酶,37 ℃恒温水浴30 min,加100 μL BTL Buffer和30 μL蛋白酶K,轻轻颠倒混匀,55 ℃振荡水浴45 min;3)加5 μL RNase A,颠倒混匀,室温静置5 min,12 000 r/min离心2 min;4)取上清,加入220 μL BDL Buffer,颠倒混匀,65 ℃恒温水浴10 min;5)加入220 μL无水乙醇,最大速度振荡20 s,转移所有液体(包括沉淀)至吸附柱中,12 000 r/min离心1 min,弃废液;6)加入500 μL HBC Wash buffer,12 000 r/min离心1 min,弃废液;7)加入600 μL DNA Wash buffer,12 000 r/min离心1 min,弃底液,重复1次;8) 12 000 r/min空离2 min,将吸附柱取出,放入一个新的1.5 mL无菌EP管中,开盖晾干;9)加入40 μL预热的无菌双蒸水,静置2 min,12 000 r/min离心1 min,重复2次,基因组即被收集到EP管中。
1.2.3 启动子片段、蛋白酶基因的克隆由转录组分析结果可知,基因lanA1和lanA2之间有一段137 bp的序列,能够分别驱动这两段基因的表达(图 1)。通过在线分析软件BPROM预测这段启动子区域的-35区和-10区,发现正义链启动子pLA与反义链启动子pLB的-10区有重叠。
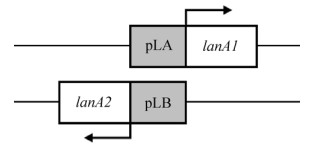
|
| 图 1 基因lanA1和lanA2以及双向启动子在基因组中的相对位置 Fig. 1 The map of lanA1, lanA2 and the bidirectional promoter pLA/pLB |
| |
分别通过引物pLA-F/pLA-Alk-R和pLB-F/ pLB-Alk-R (表 1),以该地衣芽孢杆菌基因组为模板,PCR获得大小为137 bp的pLA和pLB片段。PCR反应体系为:ddH2O 33.8 μL,基因组模板2 μL,上游引物和下游引物各2 μL,10×Pyrobest缓冲液5 μL,dNTP Mixture (2.5 mmol/L) 5 μL,Pyrobest DNA聚合酶0.2 μL。PCR反应条件为:95 ℃预变性5 min;95 ℃变性30 s,51 ℃退火30 s,72 ℃延伸8 s,共30个循环;72 ℃延伸10 min。0.8%琼脂糖凝胶电泳检测,胶回收分离纯化PCR产物。
| Primer name | Primer sequence (5′–3′) | Size (bp) |
| pLA-F | gACTAGTATATTCCACCTCCTCCTAG | 26 |
| pLA-Alk-R | GTTTCTTCATGATTCTCACCTCCTAGAACGG | 31 |
| pLB-F | gACTAGTGATTCTCACCTCCTAGAACGG | 28 |
| pLB-Alk-R | GAGAGAGGAAACCATATATTCCACCTCCTAG | 31 |
| pS-F | gACTAGTGTCACAATGCGCCATCAAAC | 27 |
| pS-Alk-R | GTTTCTTCATGTTTCCTCTCTCCCCTCTAATCG | 33 |
| Alk-pLA-F | GGTGAGAATCATGAAGAAACCGTTGGGG | 28 |
| Alk-pLB-F | GGTGGAATATATGAAGAAACCGTTGGGG | 28 |
| Alk-pS-F | GAGAGAGGAAACATGAAGAAACCGTTGGGG | 30 |
| alk-R | acatGCATGCGATTAGCGTGTTGCCGCT | 28 |
| The lowercase: protective base sequence. | ||
启动子pShuttle-09 (以下简写为pS)通过引物pS-F/pS-Alk-R (表 1)经PCR扩增获得,片段大小为215 bp。
报告基因为克劳氏芽孢杆菌碱性蛋白酶基因alk (GenBank Sequence ID:FJ940727.1)。根据编码基因序列,使用引物Alk-pLA-F、Alk-pLB-F、Alk-pS-F和alk-R通过PCR扩增获得大小为1 143 bp的基因片段。重叠PCR获得大小为1 280 bp、1 280 bp和1 358 bp的3个片段pLA-Alk、pLB-Alk和pS-Alk。
1.2.4 重组表达载体的构建PCR产物胶回收得到的启动子片段与报告基因片段使用SpeⅠ和SphⅠ酶切,与使用SpeⅠ和SphⅠ双酶切的pWH1520表达载体连接后转化大肠杆菌EC135,提取验证成功的重组载体再通过如下方法转入枯草芽孢杆菌WB600细胞中。
1) 挑取新活化的枯草芽孢杆菌单克隆菌株于20 mL LB液体培养基中,37 ℃、220 r/min摇床振荡培养12 h;2)取600 µL培养物分别接种至含有不同Gly浓度(0、0.5%、0.75%、1%、1.25%、1.5%、1.75%、2%)的30 mL LB液体培养基中,37 ℃、220 r/min培养16 h,测定OD600,计算不同浓度Gly对菌体生长的抑制率;3)取Gly抑制率在60%–75%的菌悬液2.0 mL转接至50 mL含相同浓度Gly的LB培养基,37 ℃、220 r/min继续培养至OD600为0.5–0.6;4)将上述培养好的菌液冰浴10 min,4 ℃、6 000 r/min离心10 min,弃上清,倒置,使培养液流尽;5)用预冷的溶液A重悬菌体,轻轻混匀,4 ℃、6 000 r/min离心10 min,重复3次;6)用预冷的溶液A重新悬浮细胞,无菌条件下分装于预冷的1.5 mL EP管中,每管80 μL,-80 ℃保存备用;7)取制备好的感受态细胞,无菌条件下加入重组质粒,轻轻吹吸混匀;8)吸取感受态细胞与DNA的混合物至2 mm预冷的电转杯中,冰浴3 min,电击(电压2 500 V),迅速加入1 mL溶液B,混匀,37 ℃、220 r/min复苏3 h;9) 4 000 r/min离心5 min,取适量涂布于20 μg/mL四环素抗性筛选平板,37 ℃倒置培养,12 h后挑取转化子单菌落于5 mL LB试管,培养后提取质粒DNA并验证。
重组蛋白酶表达载体分别命名为pS-alk- pWH1520、pLA-alk-pWH1520、pLB-alk-pWH1520,对应的重组基因工程菌分别为pS-alk-pWH1520- WB600、pLA-alk-pWH1520-WB600、pLB-alk- pWH1520-WB600。
1.2.5 重组蛋白酶基因工程菌的表达将新鲜平板上的3株重组基因工程菌的单菌落点接牛奶板培养,选取透明圈较大的转化子分别点接脱脂牛奶平板并接入50 mL四环素抗性种子培养基中,37 ℃、220 r/min振荡培养12 h,以相同的接种量转接于含有四环素的发酵培养基中,于37 ℃、220 r/min发酵培养,每隔12 h收集发酵液,4 ℃、12 000 r/min离心取上清。
碱性蛋白酶酶活测定参照GB/T 23527-2009附录B福林酚法进行,1个酶活力单位(U/mL)定义为1 mL酶液在40 ℃、pH为10.5条件下反应1 min水解酪蛋白产生1 μg酪氨酸所需要的酶量[21]。
2 结果与分析 2.1 地衣芽孢杆菌启动子筛选对地衣芽孢杆菌2709进行发酵培养,分别取12、48、60 h的发酵液,由苏州金唯智生物科技有限公司进行转录组测序。从测序结果中发现存在两段“头对头”[22]的基因(部分数据见表 2),且两段基因的表达强度较高。将两段基因序列在NCBI (http://www.ncbi.hlm.nih.gov/BLAST)中进行同源性比较,其编码基因为lanA1和lanA2基因。
| RPKM | lanA1 | lanA2 |
| 12h-1 | 34 665.46 | 9 727.69 |
| 12h-2 | 34 242.21 | 10 200.22 |
| 12h-3 | 26 796.22 | 3 962.31 |
| 48h-1 | 17 886.43 | 4 623.28 |
| 48h-2 | 51 120.94 | 9 242.01 |
| 48h-3 | 23 368.72 | 4 916.26 |
| 60h-1 | 71 214.72 | 15 014.39 |
| 60h-2 | 43 646.51 | 11 255.09 |
| 60h-3 | 36 419.9 | 7 749.53 |
| RPKM: reads per kilobase per million mapped reads. | ||
各个重组表达载体的构建过程如图 2所示。通过PCR扩增以及DNA切胶回收,获得大小为1 280 bp、1 280 bp和1 358 bp的3个片段pLA-Alk、pLB-Alk和pS-Alk,及通过SpeⅠ和SphⅠ双酶切并切胶回收获得的7 694 bp的pWH1520载体片段。重组载体连接产物分别转化大肠杆菌EC135,提取质粒验证正确后电转化WB600感受态。
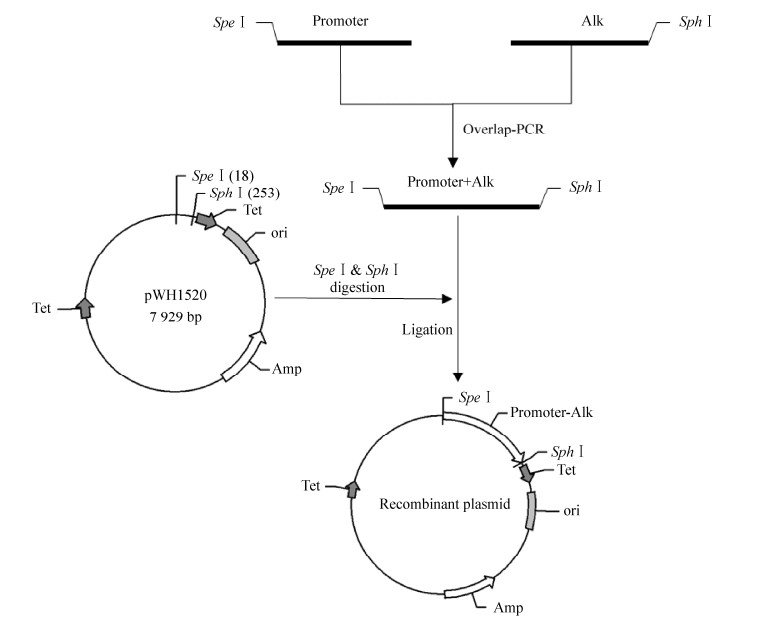
|
| 图 2 重组载体的构建过程 Fig. 2 The construction of recombinant plasmids |
| |
通过四环素抗性平板筛选转化子提取质粒DNA,再次进行验证并由苏州金唯智生物科技有限公司进行测序,测序分析与实验结果完全一致,证明报告基因和启动子片段成功克隆到载体上。重组蛋白酶表达载体pS-alk-pWH1520、pLA-alk-pWH1520、pLB-alk-pWH1520,以及所对应的工程菌pS-alk-pWH1520-WB600、pLA-alk- pWH1520-WB600、pLB-alk-pWH1520- WB600构建成功。
2.3 不同启动子对克劳氏碱性蛋白酶基因启动功能的分析将3种重组菌株在牛奶板上培养24 h,结果如图 3所示,pLA-alk-pWH1520-WB600的透明圈大小明显大于另外两者。

|
| 图 3 三种重组工程菌株对蛋白质的分解能力 Fig. 3 Decomposition of protein by three recombination strains |
| |
测定不同时间发酵上清液中碱性蛋白酶的酶活,每组实验作3个平行,结果取平均值(图 4)。发酵培养36 h后,蛋白酶产量大幅提升,60 h后,蛋白酶产量趋于稳定。发酵培养84 h,3种启动子pS、pLA、pLB表达克劳氏碱性蛋白酶的活性分别达到114、164、111 U/mL,说明pLA的启动强度明显高于pS和pLB,pLA启动子与pLB启动子均可表达碱性蛋白酶。

|
| 图 4 三种启动子对碱性蛋白酶基因的表达活性 Fig. 4 Effects of three promoters in alkaline protease expression |
| |
真核生物中大部分双向启动子缺少TATA盒,而均具有较高的GC含量并与CpG岛存在高度的相关性,小于1 kb的双向启动子的数量与基因组大小大致成负相关[13]。原核生物双向启动子样本较少,目前已知数据很难归类总结,有待进一步的研究发现。就启动子pLA/pLB个体而言,它是启动子序列反向互补的双向启动子,启动子序列重叠,双向转录基因对5′端不重叠[23] (见图 1)。序列相对于其他已知原核双向启动子较短,GC含量约为32.85%,不含有CpG岛。
通过在线分析软件BPROM对筛选所获得的启动子pLA与pLB进行结构预测和分析,结果如图 5所示。pLA与pLB均具有能被σA因子识别的保守序列-35区和-10区,且SD序列与起始密码子之间的距离均为8 bp。启动子pLA有4个转录调控因子结合位点:两个RNA聚合酶σ因子RpoD17 (结合位点为TTTATAAT和CAATTCTA)、精氨酸抑制因子ArgR (TTATAATT)和脱氧核糖核酸调节因子DeoR (AATTCTAA)的结合序列。启动子pLB有2个转录调控因子结合位点:RpoD17 (ATACTATA)和整合宿主因子IHF (Integration host factor,binding sites:ACAAAAAA)的结合序列。

|
| 图 5 双向启动子结构分析 Fig. 5 Structure analysis of the bidirectional promoter. (A) Sequence of promoter pLA. (B) Sequence of promoter pLB. The transcription start site (+1), the Shine-Dalgarno sequence, the -10 and -35 boxes are indicated with underline |
| |
启动子pShuttle-09来源于地衣芽孢杆菌,是一个杂合启动子,其表达强度是枯草芽孢杆菌强启动子P43的8倍[24]。含有两对典型的保守序列,均被σA因子识别。有4个转录调控因子结合位点:磷酸盐调节子转录调控蛋白PhoB (TCATAAAA)、ArgR (CATAAAAA)、嘌呤调节子阻遏蛋白PurR (ATAAAAAG)和RNA聚合酶σ因子RpoD16 (TGATATAA)的结合序列。
启动子的启动强度主要取决于启动子的结构,一般来说,两保守区序列-35区和-10区间隔为(17±1) bp时启动活性最强,而SD序列与起始密码子之间的距离也是决定启动子强度的关键因素,通常为10 bp[25]。pLA和pS两保守区之间间隔为17 bp和18 bp,pLB为14 bp,这可能是造成pLB的启动活性低于pLA的主要原因。而pLA、pLB中SD序列与起始密码子均相距8 bp,pS中二者相距4 bp,这可能是pLA表达活性高于pS的原因之一。
构建的工程菌株碱性蛋白酶产量仍然较低,构建用于工业生产的菌株时可选择分泌强度更高的信号肽及拷贝数更高的表达载体。
4 结论本研究将启动子pLA/pLB以两种不同的方向克隆进同一种质粒中,以克劳氏碱性蛋白酶基因作为报告基因并转化枯草芽孢杆菌细胞,成功构建了3种重组表达载体pS-alk-pWH1520、pLA-alk-pWH1520、pLB-alk-pWH1520及对应的3种工程菌株。通过检测碱性蛋白酶的酶活力,证实了该启动子在原核生物枯草芽孢杆菌中有双向启动能力,即lanA基因启动子区可进行两个方向的转录,且5′–3′方向的启动子启动强度明显大于3′–5′方向的启动子。从而为枯草芽孢杆菌表达系统中异源基因的表达提供一个新的方向,也为原核生物中共同表达两种基因提供新的思路。
| [1] |
Miao M, Huang KL, Liang ZH. The characters and advances of Bacillus expression system.
Sci Technol Food Ind, 2017, 38(18): 312–316, 322.
(in Chinese). 苗苗, 黄昆仑, 梁志宏. 芽孢杆菌表达系统的特点及研究进展. 食品工业科技, 2017, 38(18): 312-316, 322. |
| [2] | Harwood CR, Cranenburgh R. Bacillus protein secretion: an unfolding story. Trends Microbiol, 2008, 16(2): 73–79. DOI: 10.1016/j.tim.2007.12.001 |
| [3] | Westers L, Westers H, Quax WJ. Bacillus subtilis as cell factory for pharmaceutical proteins: a biotechnological approach to optimize the host organism. Biochim Biophys Acta, 2004, 1694(1/3): 299–310. |
| [4] | Shields DC, Sharp PM. Synonymous codon usage in Bacillus subtilis reflects both translational selection and mutational biases. Nucl Acids Res, 1987, 15(19): 8023–8040. DOI: 10.1093/nar/15.19.8023 |
| [5] |
Yu XX, Tian J, Liu XQ, et al. Research progress of Bacillus subtilis expression system and its promoter regulatory elements.
Biotechnol Bull, 2015, 31(2): 35–44.
(in Chinese). 余小霞, 田健, 刘晓青, 等. 枯草芽孢杆菌表达系统及其启动子研究进展. 生物技术通报, 2015, 31(2): 35-44. |
| [6] | Outtrup H, Jorgensen ST. Chapter 14 The importance of Bacillus species in the production of industrial enzymes. In Applications and Systematic of Bacillus and Relatives. New Jersey: Wiley-Blackwell Press, 2002: 206-218. |
| [7] | Rao MB, Tanksale AM, Ghatge MS, et al. Molecular and biotechnological aspects of microbial proteases. Microbiol Mol Biol Rev, 1998, 62(3): 597–635. |
| [8] | Schallmey M, Singh A, Ward OP. Developments in the use of Bacillus species for industrial production. Canad Microbiol, 2004, 50(1): 1–17. DOI: 10.1139/w03-076 |
| [9] |
Xiong HT, Wei YT. Research progress of Bacillus subtilis expression system and its promoter.
Guangxi Sci, 2018, 25(3).
(in Chinese). 熊海涛, 韦宇拓. 枯草芽孢杆菌表达系统及其启动子的研究进展. 广西科学, 2018, 25(3). |
| [10] |
Ding JB, Cui ZZ, Sun SH, et al. A bi-directional promoter at the upstream of pp38 gene from Marek's Disease.
Acta Microbiol Sin, 2004, 44(2): 162–166.
(in Chinese). 丁家波, 崔治中, 孙淑红, 等. 马立克氏病病毒pp38基因上游的一个双向启动子研究. 微生物学报, 2004, 44(2): 162-166. DOI:10.3321/j.issn:0001-6209.2004.02.007 |
| [11] |
Liao YH, Tu GH, Zhao DG. Cloning of MAL1 promoter region and analysis of promoter function in E. coli.
Biotechnology, 2005, 15(6): 2–5.
(in Chinese). 廖昱泓, 涂桂洪, 赵德刚. MAL1基因启动子区的克隆及其在大肠杆菌中的启动功能分析. 生物技术, 2005, 15(6): 2-5. DOI:10.3969/j.issn.1004-311X.2005.06.002 |
| [12] |
Qi K. Analysis and functional study of transcription regulatory elements in bidirectional promoter[D]. Heilongjiang: Harbin Institute of Technology, 2014 (in Chinese). 奇克.双向启动子中转录调控元件的分析与功能研究[D].黑龙江: 哈尔滨工业大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10213-1014083844.htm |
| [13] |
Liu SJ, Zheng CC. Structure and functions of eukaryotic bidirectional promoters.
Chin J Biochem Mol Biol, 2011, 27(10): 894–900.
(in Chinese). 刘石娟, 郑成超. 真核生物双向启动子的结构与功能. 中国生物化学与分子生物学报, 2011, 27(10): 894-900. |
| [14] |
Yan XH. Research of identification of alternative promoters and bidirectional promoters based on new-generation sequencing technology[D]. Heilongjiang: Harbin Institute of Technology, 2014 (in Chinese). 闫晓惠.基于新一代测序技术的选择性启动子和双向启动子识别研究[D].黑龙江: 哈尔滨工业大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10213-1014081790.htm |
| [15] | Baharoglu Z, Krin E, Mazel D. Connecting environment and genome plasticity in the characterization of transformation-induced SOS regulation and carbon catabolite control of the Vibrio cholerae integron integrase. J Bacteriol, 2012, 194(7): 1659–1667. DOI: 10.1128/JB.05982-11 |
| [16] |
Zhang CX, Gai Y, Zhu YY, et al. Construction of bidirectional promoter and its transient expression in Populus tomentosa.
J Beijing Fores Univ, 2007, 29(1): 119–122.
(in Chinese). 张春晓, 盖颖, 朱艳燕, 等. 双向启动子构建及在毛白杨中瞬时表达研究. 北京林业大学学报, 2007, 29(1): 119-122. DOI:10.3321/j.issn:1000-1522.2007.01.021 |
| [17] | Phan TT, Nguyen HD, Schumann W. Establishment of a simple and rapid method to screen for strong promoters in Bacillus subtilis. Protein Expr Purif, 2010, 71(2): 174–178. DOI: 10.1016/j.pep.2009.11.010 |
| [18] |
Su N, Sun M, Li YN, et al. Isolation and modification of rice chloroplast 16S promoter, construction of expression vector and transformation.
Chin Bull Bot, 2003, 20(3): 295–301.
(in Chinese). 苏宁, 孙萌, 李轶女, 等. 水稻叶绿体16S启动子克隆改造、载体构建及转化研究. 植物学通报, 2003, 20(3): 295-301. DOI:10.3969/j.issn.1674-3466.2003.03.006 |
| [19] | Shyamala V, Ames GFL, Wu R. Genome walking by single specific primer-polymerase chain reaction. Gene, 1989, 84(1): 1–8. DOI: 10.1016/0378-1119(89)90132-7 |
| [20] |
Nie LN, Xia LQ, Xu ZS, et al. Progress on cloning and functional study of plant gene promoters.
J Plant Genet Res, 2008, 9(3): 385–391.
(in Chinese). 聂丽娜, 夏兰琴, 徐兆师, 等. 植物基因启动子的克隆及其功能研究进展. 植物遗传资源学报, 2008, 9(3): 385-391. |
| [21] |
General Administration of Quality Supervision, Inspection and Quarantine of China, Standardization Administration of China. GB/T 23527-2009 Proteinase Preparations. Beijing: China Standard Press, 2009 (in Chinese). 中华人民共和国国家质量监督检验检疫总局, 中国国家标准化管理委员会. GB/T 23527-2009蛋白酶制剂.北京: 中国标准出版社, 2009. |
| [22] | Thomas G, Meike H, Marc S, et al. Expression vector: US, 10196645B2. 2019-02-05. |
| [23] | Li YY, Yu H, Guo ZM, et al. Systematic analysis of head-to-head gene organization: evolutionary conservation and potential biological relevance. PLoS Comput Biol, 2006, 2(7): e74. DOI: 10.1371/journal.pcbi.0020074 |
| [24] | Zhang AL, Liu H, Yang MM, et al. Assay and characterization of a strong promoter element from B. subtilis. Biochem Biophys Res Commun, 2007, 354(1): 90–95. DOI: 10.1016/j.bbrc.2006.12.137 |
| [25] |
Chen K, Yuan FY, Chai HN, et al. Screening and study of a new promoter with effective expression of alkaline protease.
Biotechnol Bull, 2018, 34(1): 208–214.
(in Chinese). 陈坤, 袁飞燕, 柴昊男, 等. 一种高效表达碱性蛋白酶的新型启动子的筛选及研究. 生物技术通报, 2018, 34(1): 208-214. |
 2019, Vol. 35
2019, Vol. 35




