中国科学院微生物研究所、中国微生物学会主办
文章信息
- 黄传玺, 马洁, 吴琛, 朱云平
- Huang Chuanxi, Ma Jie, Wu Chen, Zhu Yunping
- 基于TCGA数据预测肿瘤代表性新抗原的一种生物信息学新方案
- A new bioinformatics approach for prediction of potential tumor neoantigens based on the cancer genome atlas dataset
- 生物工程学报, 2019, 35(7): 1295-1306
- Chinese Journal of Biotechnology, 2019, 35(7): 1295-1306
- 10.13345/j.cjb.190025
-
文章历史
- Received: January 12, 2019
- Accepted: March 25, 2019
- Published: April 2, 2019
2. 军事科学院军事医学研究院生命组学研究所,北京 102206;
3. 蛋白质组学国家重点实验室 国家蛋白质科学中心(北京) 北京蛋白质组研究中心 蛋白质药物国家工程研究中心,北京 102206
2. Beijing Institute of Life Omics, Beijing 102206, China;
3. State Key Laboratory of Proteomics, Beijing Proteome Research Center, National Center for Protein Sciences (Beijing), Beijing 102206, China
肿瘤的发生与发展伴随着基因突变的产生与选择[1-3]。肿瘤病变区域往往包含免疫细胞,这种免疫反应在一定程度上反映了机体免疫系统根除肿瘤的尝试,越来越多的证据也表明多种类型的肿瘤具有抗免疫反应[4]。在肿瘤的治疗过程中,现行的放疗和化疗方案不可避免地损伤正常人体细胞。对于部分靶向药物,由于肿瘤细胞的高度异质性及其基因组的不稳定性,肿瘤细胞可以减少对特定功能通路的依赖,通过改变自身的部分性状,从而产生获得性耐药[5]。免疫疗法期望对患者免疫系统进行重编程,提高自身免疫能力,从而发挥抗肿瘤作用,理论上有可能避免常规药物疗法带来的抗药性[6]。
肿瘤新抗原的寻找是免疫治疗推进过程中的重大挑战[7-8]。肿瘤细胞产生的基因突变在健康组织中缺乏表达,而且具有高度免疫原性,但从肿瘤细胞发生基因突变到可以被成熟T细胞谱系识别[9]需要经历复杂的生物学过程。即使突变基因产生了异常蛋白,但仅有部分水解后的肽段可以被呈递到细胞表面并被免疫细胞所识别。因此,寻找正确有效的肿瘤新抗原是亟待解决的问题。目前已经有大量的研究通过生物信息学方法,发展寻找肿瘤新抗原的新工具,并利用基因测序数据或质谱数据预测具有治疗效用的肿瘤新抗原,如Jurtz等发展的NetMHCpan工具[10]、Stranzl等发展的NetCTLpan工具[11]、Bais等发展的CloudNeo工具[12]等;Kreiter等利用NetMHCpan工具成功设计了特定肿瘤小鼠模型的免疫疫苗[7],Pritchard等利用肿瘤患者外周血细胞的基因突变数据筛选了个体化的免疫肽[13],这两项研究的成果均取得了良好的应用效果。
肿瘤患者的突变基因组高度特异[14],但每一种肿瘤都具有区别于其他肿瘤的代表性的基因突变特征,因此,有可能开发出针对部分人群适用的免疫治疗策略。基于这种想法,文中提出了一种预测肿瘤代表性新抗原的生物信息学新方案,兼顾抗原在胞内呈递和与胞外组织相容性复合物(Major histocompatibility complex,MHC)结合的能力,并利用肿瘤基因组图谱数据库(The Cancer Genome Atlas,TCGA,https://cancergenome.nih.gov/) 中的大规模肿瘤基因组测序数据[15],寻找肿瘤代表性新抗原,旨在为后续的研究提供可信的肿瘤新抗原参考列表。
1 材料与方法 1.1 构建肿瘤新抗原预测策略基于基因突变产生肿瘤新抗原的过程可以概括如下:肿瘤细胞发生基因突变,突变基因被转录并翻译出异常的蛋白质,异常蛋白质的部分肽段被细胞选择性呈递到细胞表面从而被免疫系统识别。综上,基于基因突变产生肿瘤新抗原呈递的过程主要取决于以下几个方面[16-19]:1)突变基因能否翻译成异常蛋白质;2)基因突变产生的异常蛋白质能否被蛋白酶体选择性酶解;3)酶解后的肽段能否被抗原加工相关转运体(Transporter associated with antigen processing,TAP)选择性转运;4)肽段与MHC分子结合并呈递,呈递后的抗原能否被T细胞所识别。基于上述过程,文中构建了兼顾抗原在胞内呈递和与胞外MHC分子结合的肿瘤新抗原的生物信息学预测流程,完整的工作流程如图 1所示。

|
| 图 1 肿瘤新抗原生物信息学预测的工作流程 Fig. 1 Workflow of bioinformatics prediction of new tumor antigens |
| |
在肿瘤新抗原预测流程中,通过联用NetCTLpan和NetMHCpan两个生物信息学软件针对突变氨基酸序列进行预测,从而兼顾MHCⅠ类抗原在胞内呈递和MHC与抗原胞外结合能力两个关键过程。两个软件均采用人工神经网络模型学习现有免疫抗原肽段序列特征,学习的数据集均来自高可信的免疫抗原数据库(IEDB, http://www.iedb.org/home_v3.php;SYFPEITHI,http://www.syfpeithi.de/)。NetCTLpan软件(https://swissmodel.expasy.org/)整合了人类MHCⅠ类分子与肽段结合能力、蛋白酶体C端选择性剪切以及TAP转运效率3个方面的信息,对8–11个氨基酸长度的肽段能否被呈递到MHC分子上进行预测。该软件考虑了异常蛋白质在胞内完整的呈递过程[11]。NetMHCpan软件(http://www.cbs.dtu.dk/services/NetMHCpan/)用于预测已知序列的肽段与MHC分子的结合能力,与此同时,该软件方法整合了天然洗脱配体数据,抗原与MHC分子结合亲和力数据的信息,可以预测给定肽段成为天然配体的可能性以及与T细胞的结合亲和能力[10]。
1.3 数据来源与数据处理根据2018年中国癌症中心发布的2014年中国不同地区恶性肿瘤发病和死亡分析报告[20],以及2018年美国癌症协会发布的2014年美国癌症发病率、死亡率和存活率的数据[21],选取以下7种发病率或复发率较高的癌症作为研究对象,包括膀胱癌、乳腺癌、结肠癌、肝癌、胃癌及肺癌,其中肺癌的数据进一步划分为肺鳞状细胞癌与肺腺癌两类。测序数据均来源于TCGA中基因组测序数据,突变基因对应的氨基酸序列来自SwissPort数据库(https://www.uniprot.org/, 序列下载日期为2018年11月)[22]。基因组参考序列为hg19,测序平台为IlluminaGA或IlluminaHiSeq。基因突变数据信息如表 1所示。更多信息见补充附件1 (可在网络版中下载)。
| Cancer type | Bladder cancer | Breast cancer | Colon cancer | Liver cancer | Stomach cancer | Lung adeno- carcinoma | Lung squamous cell carcinoma |
| Number of missense mutation | 82 606 | 55 063 | 63 519 | 32 554 | 81 771 | 144 453 | 42 890 |
| Categories of amino acid length | 4 | 4 | 4 | 4 | 4 | 4 | 4 |
| Number of allele | 20 | 20 | 20 | 20 | 20 | 20 | 20 |
| Number of patients | 396 | 982 | 217 | 373 | 379 | 543 | 178 |
| Peptides involved in prediction | 62 780 560 | 41 847 880 | 48 274 440 | 24 741 040 | 62 145 960 | 109 784 280 | 32 596 400 |
| Candidate epitope | 698 | 1 021 | 1 314 | 245 | 436 | 6 460 | 704 |
| Candidate epitope involved genes | 92 | 121 | 208 | 25 | 59 | 640 | 65 |
基于构建的肿瘤新抗原生物信息学预测流程,对7种肿瘤基因突变数据进行统一处理。以乳腺癌为例,首先利用TCGA中基因突变数据筛选出非同义单点突变,然后将基因突变信息对应到SwissProt数据库特定氨基酸序列上,选择HLA-A、HLA-B (Human leukocyte antigen,HLA)中翻译产物与肽段结合能力排名前20的代表基因作为预测使用的等位基因,肽段长度限定为8–11个氨基酸。NetCTLpan软件预测参数设置为:排序阈值≤1%、C端氨基酸残基类别的权重占比为0.225、TAP转运效率权重占比为0.025。NetMHCpan软件预测参数设置为:排序阈值≤2%。分别采用NetMHCpan与NetCTLpan对包含突变位点的肽段序列进行预测;然后将NetMHCpan与NetCTLpan的输出结果进行整合,选取突变位点与呈递序列在两个软件预测结果中重复出现的序列,此时得到的候选结果相当于同时进行了胞内呈递过程与胞外结合能力的预测。
统计各基因突变位点的突变频率,基因突变频率越低,提示该突变越可能是个体高度特异的突变,基因突变频数越高,对应的基因越可能与该肿瘤的发生与发展密切相关。因此,我们结合基因突变位点的突变频数对上述结果进行分析,过滤个体高度特异的突变序列(肺鳞状癌数据量较小,以≥2为筛选标准;肺腺癌数据量较大,以≥5为筛选标准;其余肿瘤数据均以≥3为筛选标准),进一步筛选得到的结果称为候选肿瘤新抗原。
1.4 生物信息学功能注释及蛋白质结构模型预测通过对预测获得的候选肿瘤新抗原对应的基因进行功能注释与通路分析,过滤非肿瘤因素引起的基因突变。候选肿瘤新抗原对应的基因通过DAVID (https://david.ncifcrf.gov/home.jsp)进行包括细胞成分、代谢过程和生物学通路等方面的功能注释[23]。最后,结合突变位点在肿瘤人群的覆盖度、MHC结合位点数目两方面的信息进一步筛选出肿瘤代表性免疫肽段列表。采用SWISS- MODEL (https://swissmodel.expasy.org/)进行蛋白质三维结构模拟,展示突变带来的构象变化[24],对结果进行进一步验证。
2 结果与分析 2.1 肿瘤新抗原的预测结果在肿瘤的发生与发展过程中,为获得生存优势,肿瘤细胞产生了部分候选优势突变[25],由于抗原呈递过程涉及复杂的生物学过程,基因突变引起的异常蛋白在抗原呈递过程中可能存在不同的结果,从而给肿瘤新抗原的实验筛选带来大量重复性工作以及高额的经济成本。文中构建了肿瘤新抗原新的预测策略,并利用TCGA中7种肿瘤突变数据进行肿瘤新抗原的预测,共计382 170 560条氨基酸序列参与了肿瘤新抗原的预测。基于两款软件的预测结果,选取基因突变位点相同、序列信息相同的肽段作为候选肿瘤新抗原列表,完整的候选肿瘤新抗原列表见补充附件2 (可在网络版中下载),详细结果统计信息如表 1和图 2所示。

|
| 图 2 TCGA数据库中7种肿瘤基因突变数据的生物信息学预测结果 Fig. 2 Bioinformatics prediction results of seven cancer genomics datasets in TCGA database. (A) Distribution of the proportion of candidate mutation loci in all mutation loci. (B) Distribution of candidate peptides in all peptides involved in prediction. (C) Distribution of the candidate peptides predicted by NetMHCpan and NetCTLpan. (D) Distribution of the population coverage of candidate peptides in cancer dataset. Lung_AD and Lung_SCC are the abbreviations of Lung Adenocarcinoma and Lung Squamous cell carcinoma respectively |
| |
基于兼顾胞内呈递和与胞外MHC分子结合的肿瘤新抗原生物信息学预测流程,膀胱癌突变数据通过NetMHCpan和NetCTLpan软件预测,分别获得1 924条和782条序列结果,取两者交集获得最终候选肿瘤新抗原列表,共698条,对应92个基因。肺腺癌、结肠癌、乳腺癌、肺鳞状细胞癌、胃癌、肝癌数据集分别获得6 460条、1 314条、1 021条、704条、436条和245条候选肿瘤新抗原,对应640个、208个、121个、65个、59个和25个基因。如图 2A和图 2B所示,通过预测流程,95.0%以上肿瘤患者高度特异性的突变位点及99.9%以上包含个体高度特异突变位点的氨基酸序列可以被排除,极大减少了实验验证的工作量与经济成本。从理论预测的结果来看,胞内免疫肽的多个呈递过程对最终的结果影响巨大(图 2C)。
目前,NetMHCpan是最常用的肿瘤新抗原预测工具,但NetMHCpan的预测原理仅关注胞外MHC分子与相关肽段的亲和能力,以及其形成的复合体与T细胞受体的结合能力,忽略了肽段在胞内的一系列处理过程,这也是两款软件预测结果重叠性不高的原因。如肝癌、结肠癌、肺腺癌数据集结果所示,通过文中提出的兼顾抗原胞内呈递和与胞外MHC分子结合信息的肿瘤新抗原筛选流程,有效整合两个软件的预测结果,可以得到更为准确的肿瘤新抗原候选结果验证区间。如图 2D所示,即使两个软件的整合结果排除了大部分NetMHCpan的预测结果,但得到的肿瘤新抗原列表仍然在肿瘤患者中占有相当的比率,进一步佐证了数据挖掘的可靠性。
2.2 基因功能富集分析基因功能的重要程度与其转录翻译的频率呈正相关趋势,那么基因的突变频率将与其功能的重要程度密切相关[26-27]。如表 2所示,以膀胱癌和结肠癌为例,展示了候选肿瘤新抗原列表中突变频率排名前5位的位点及其相关信息。可以看到这些突变位点对应的基因在前人的工作中已被证明与肿瘤的发生与发展密切相关[28-34]。
| Cancer type | Gene | Amino acid mutation site | Frequency | Cumulative sample number | Percentage (%) | Number of HLA alleles |
| Bladder cancer | FGFR3 | S249C | 30 | 29 | 7.32 | 3 |
| PIK3CA | E545K | 28 | 50 | 12.63 | 6 | |
| PIK3CA | E542K | 17 | 65 | 16.41 | 9 | |
| ZNF814 | D404E | 15 | 77 | 19.44 | 2 | |
| TP53 | E285K | 12 | 85 | 21.46 | 3 | |
| Colon cancer | BRAF | V600E | 25 | 25 | 11.57 | 3 |
| PIK3CA | E545K | 19 | 44 | 20.37 | 6 | |
| KRAS | G12V | 17 | 61 | 28.24 | 1 | |
| NEFH | E645K | 15 | 74 | 34.26 | 3 | |
| OPRD1 | C27F | 17 | 83 | 38.43 | 2 |
针对每一种癌症,人群中普遍存在的突变往往在维持肿瘤生存能力方面具有重大的影响[35]。基于候选肽段涉及的基因,对除肝癌(肝癌数据集得到的候选肽段仅对应25个基因,数量过少,未进行分析)以外的6种肿瘤关联的基因进行了功能富集分析,以膀胱癌基因突变数据为例,候选肿瘤新抗原肽段分布在92个基因上,这些基因功能富集结果如图 3所示,其他肿瘤数据功能富集信息见补充附件3 (可在网络版中下载)。通路富集结果与基因条目注释结果均显示这些基因所涉及的通路与生物学过程与癌症密切相关,并反映了人体生理学变化,如血小板激活、焦点粘连、癌症的核心碳代谢、血管内皮生长因子通路、癌症的蛋白聚糖等,进一步佐证了预测流程的可靠性。

|
| 图 3 膀胱癌数据集候选免疫肽段对应基因的功能富集分析 Fig. 3 Functional enrichment analysis of candidate neoantigen related genes in bladder cancer dataset. (A) Pathway enrichment analysis. (B) Go annotation analysis |
| |
同时,不同癌症数据集的预测结果中可以观察到与特定癌症相关的功能通路明显富集,显示出肿瘤类型的特异性,这可能为特定癌症的治疗提示新的方向。以膀胱癌数据集为例,其分析结果中血小板激活通路显著富集,原因可能有以下两个:1)血小板对肿瘤细胞的促进作用。有研究表示,血小板数量和活性的增加与肿瘤转移相关,血小板可以通过促进免疫逃避、血管生成来参与肿瘤转移过程[36]。体内外实验证明,肿瘤细胞能将突变的RNA转入血小板中。研究者从神经胶质瘤和前列腺癌患者的血小板中发现了癌症相关的RNA生物标记物,如egfrviii等[37]。血小板还能够捕获和激活tgf-β,从而辅助侵袭性肿瘤细胞抑制T细胞的生物学功能[38]。2)参与凝血过程。参与血液循环中的凝血过程是血小板重要的生理功能之一,而约80%-90%的膀胱癌患者伴有血尿的症状。血小板数量及活性的增加可能归因于血尿的出现,也有可能与术后复高发率[39]的现象有所关联。
此外,在乳腺癌数据结果中核心碳代谢通路、蛋白聚糖通路相对活跃;结肠癌数据结果中,血管生成信号通路、鞘脂信号通路相对活跃;胃癌数据结果中致病性大肠杆菌感染与细菌侵袭上皮细胞、蛋白聚糖出现基因富集现象;肺鳞状细胞癌数据结果中,rap1信号通路、jak-stat信号通路明显富集。有意思的是在两种肺癌数据中(肺腺癌、肺鳞状细胞癌),均发现大量嗅觉转导密切相关的基因发生突变,虽然这些基因突变频率不高,但总体数目较多,这提示这些基因在肺癌患者中可能介导着其他的生理作用。同时,癌症相关的蛋白聚糖、鞘脂信号等通路在多种肿瘤数据集的分析结果中反复出现,提示涉及这些通路的治疗方案有可能对多种癌症都具有治疗效果。
2.3 免疫肽的进一步过滤及结构信息的辅助确认两种软件理论预测的结果经过整合,候选肿瘤新抗原的数量与比例已经大幅减少,但其绝对数目仍然较大,进行候选新抗原实验验证依然存在挑战。因此,结合突变位点的频率、突变位点在人群中的占比以及HLA涉及的等位基因数目3个指标作为主要评价标准,进一步筛选出每种肿瘤预测结果中排名前20的候选肿瘤新抗原,这些候选肿瘤新抗原被认为是高可信的结果,详细信息参见补充附件4 (可在网络版中下载)。
基因突变可以通过对蛋白质三维结构产生影响进而调控相关的生物学功能,最终促进肿瘤的发生与发展。结构异常的蛋白质相对于胞内其他蛋白是新蛋白,具有相对高的免疫原性,异常蛋白被降解后形成的肽段被呈递在细胞表面的几率较高。采用SWISS-MODEL对预测结果进行三维蛋白质结构模拟,可以进一步确认预测结果是否与上述假设相符合。如图 4所示,TP53蛋白的第175个氨基酸位点和PIK3CA蛋白的第542、545个氨基酸位点由正常状态到发生突变后,其结构模拟示意图差别明显。TP53蛋白175氨基酸位点发生突变后,该区域由原来的开放式结构转变为了环形结构,这可能是肿瘤患者体内P53蛋白抑癌功能缺失的部分原因。PIK3CA蛋白的第542、545个氨基酸位点发生突变后,该位点由原来利于结合的构象转为不利于结合的构象,提示PIK3CA蛋白催化下游底物的效率可能减弱,肿瘤细胞信号调节受到影响,进而影响细胞生命活动。
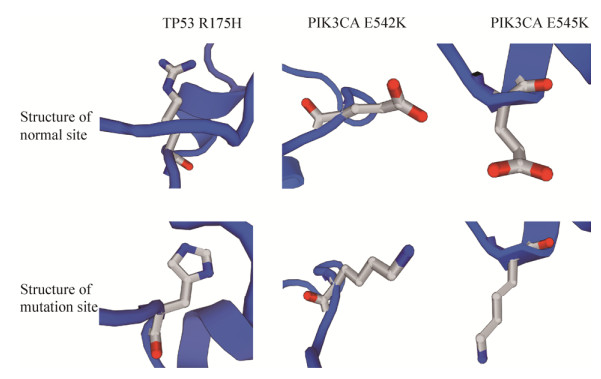
|
| 图 4 蛋白TP53和PIK3CA氨基酸位点突变引起的蛋白质三维结构变化的模拟示意图 Fig. 4 The simulation of changes in three-dimensional structure of protein TP53 and PIK3CA induced by mutations |
| |
基因突变为肿瘤细胞提供了进化的来源[3],某些突变基因型赋予细胞亚克隆选择性优势,使其在局部组织环境中生长并最终占优势[40]。基因突变给肿瘤细胞提供了生存的基础,也给肿瘤治疗带来了新的治疗靶点。近年来,肿瘤免疫治疗如火如荼地开展,但现阶段免疫治疗仅对20%–30%的患者有明显疗效。令人欣慰的是,这个领域还在以方兴未艾的态势发展[41-43]。2018年,来自美国MD安德森癌症研究中心的Allison和日本京都大学的Tasuku关于负性免疫调节治疗癌症的疗法荣获诺贝尔生理学奖,进一步推动免疫治疗的研究。虽然目前确认有效的肿瘤抗原位点有限[44-46],但肿瘤疫苗的研究仍不断取得新的进展。
Teku等于2018年使用NetMHCpan 4.0对30种癌症的蛋白质组数据进行了分析,其分析结果显示,单一的NetMHCpan预测并不合理[8]。肽段与HLA分子的紧密结合是引发免疫反应的必要不充分条件,实际生物学过程中,蛋白酶体和其他蛋白酶对抗原呈递细胞中前体蛋白的加工、TAP复合物将肽段从胞质转运至内质网的效率都将影响抗原呈递的结果。本文将NetCTLpan与NetMHCpan工具结合,构建了肿瘤新抗原预测的生物信息学策略,充分考虑了突变产生的新肽段在胞内的及胞外的呈递过程,数据分析流程的设计更为合理,预测的免疫肽组合可以覆盖相当比例的人群。如图 5所示,应用新的肿瘤新抗原生物信息学流程分析结肠癌、膀胱癌、肺鳞状细胞癌、乳腺癌、肝癌、胃癌和肺腺癌数据集,预测得到的候选肿瘤新抗原组合分别可以覆盖各个数据集71.76%、40.66%、33.52%、33.23%、29.22%、27.97%和22.28%的肿瘤患者。
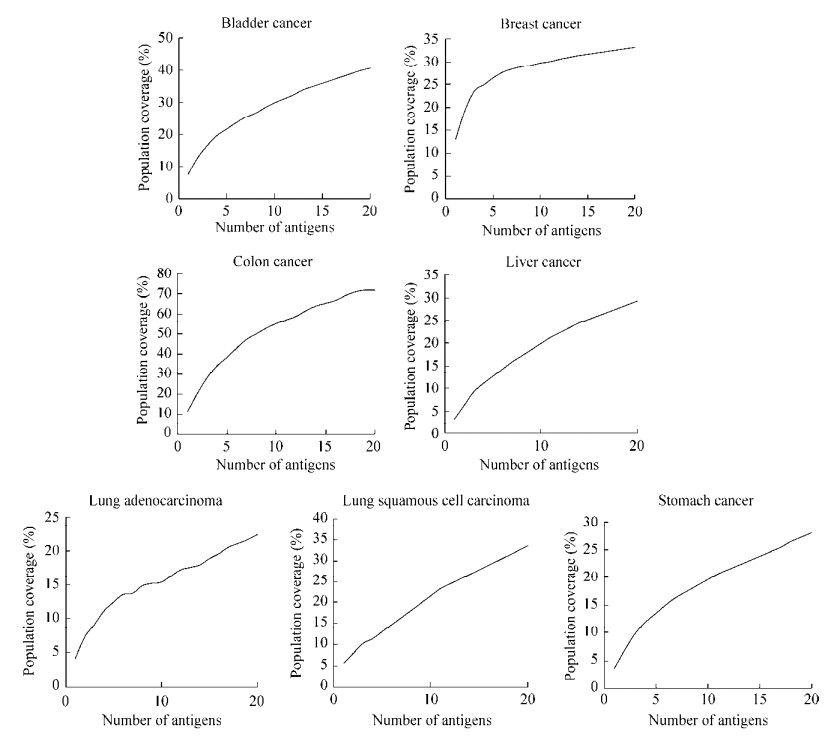
|
| 图 5 不同癌症数据集中排名前20的候选肿瘤新抗原肽段在患者样本中的累积覆盖度 Fig. 5 Cumulative population coverage of the top 20 candidate peptides in different cancer datasets |
| |
此外,在7种癌症数据集最终预测结果中,我们还发现了部分基因在肿瘤中普遍发生基因突变,这些基因有可能在多种癌症的免疫治疗中产生效果,如tp53 (7/7)与pik3ca (5/7)以及部分锌指蛋白基因等。其中TP53蛋白的第175个氨基酸位点突变形式在4种癌症中都出现,该突变位点在人群中的占比分别为6.91% (结肠癌)、2.90% (胃癌)、2.14% (乳腺癌)和1.68%(肺鳞状细胞癌)。在175位点附近,176/163/157等位点的突变在人群中也占有一定频率。PIK3CA蛋白的第545 (7/7)、542 (5/7)氨基酸位点在7种癌症中也频繁出现,两个位点共占比例为11.41% (乳腺癌)、11.36% (膀胱癌)、8.76% (结肠癌)、7.30%(肺鳞状细胞癌)、2.90% (胃癌)和2.39% (肺腺癌)。除了上述基因之外,我们还发现了一批仅在特定肿瘤中常见的突变。如仅在结肠癌与肺腺癌中突变频率较高的kras基因突变、膀胱癌中突变频率最高的fgfr3基因突变、结肠癌中突变频率较高的opprd1、nefh基因突变等等在其他几种癌症中几乎未发现。基因突变的选择性提示我们由于机体器官组成的规律不同,不同器官形成肿瘤后,肿瘤细胞的功能需求可能不同。
文中构建的肿瘤新抗原新的预测策略虽然考虑了更多的生物学过程,但每个预测过程相对独立,仍存在较大的改进空间。此外,预测策略分析TCGA数据获得了部分肿瘤新抗原,但不是所有MHC呈递的抗原都能引起免疫反应,肿瘤新抗原具有高免疫原性,但免疫耐受的现象仍可能存在,文中提到的不同肿瘤新抗原组合有可能改善这一情况。同时,由于预测工具本身可能存在一定程度的假阳性和假阴性结果,后续分子实验验证、动物模型验证、临床实验仍是必不可少的部分。因此,从肿瘤新抗原的预测到实际的临床应用仍然道阻且长,免疫治疗的进展需要多个领域的研究人员共同推进。
综上,文中构建了兼顾抗原在胞内呈递和与胞外组织相容性复合物结合能力的肿瘤新抗原新的预测策略,有望大幅减少实验工作量,同时也为后续研究者提供了一个有参考价值的数据分析流程。应用该策略系统分析了TCGA中多种肿瘤基因组测序数据,得到了一批可靠度较高的肿瘤新抗原;与此同时,最终预测的部分MHCⅠ类肿瘤新抗原组合在人群中占有一定的覆盖度,存在潜在的临床应用价值。
| [1] | Greenman C, Stephens P, Smith R, et al. Patterns of somatic mutation in human cancer genomes. Nature, 2007, 446(7132): 153–158. DOI: 10.1038/nature05610 |
| [2] | Salk JJ, Fox EJ, Loeb LA. Mutational heterogeneity in human cancers: origin and consequences. Annu Rev Pathol, 2010, 5: 51–75. DOI: 10.1146/annurev-pathol-121808-102113 |
| [3] | Stratton MR, Campbell PJ, Futreal PA. The cancer genome. Nature, 2009, 458(7239): 719–724. DOI: 10.1038/nature07943 |
| [4] | Pagès F, Galon J, Dieu-Nosjean MC, et al. Immune infiltration in human tumors: a prognostic factor that should not be ignored. Oncogene, 2010, 29(8): 1093–1102. DOI: 10.1038/onc.2009.416 |
| [5] | Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell, 2011, 144(5): 646–674. DOI: 10.1016/j.cell.2011.02.013 |
| [6] | 张毓. 开启肿瘤治疗的新时代——2018年诺贝尔生理学或医学奖评介. 中国生物化学与分子生物学报, 2018, 34(11): 1135-1137. |
| [7] | Kreiter S, Vormehr M, van de Roemer N, et al. Mutant MHC class Ⅱ epitopes drive therapeutic immune responses to cancer. Nature, 2015, 520(7549): 692–696. DOI: 10.1038/nature14426 |
| [8] | Teku GN, Vihinen M. Pan-cancer analysis of neoepitopes. Sci Rep, 2018, 8: 12735. DOI: 10.1038/s41598-018-30724-y |
| [9] | Yadav M, Jhunjhunwala S, Phung QT, et al. Predicting immunogenic tumour mutations by combining mass spectrometry and exome sequencing. Nature, 2014, 515(7528): 572–576. DOI: 10.1038/nature14001 |
| [10] | Jurtz V, Paul S, Andreatta M, et al. NetMHCpan-4.0: improved peptide-MHC class Ⅰ interaction predictions integrating eluted ligand and peptide binding affinity data. J Immunol, 2017, 199(9): 3360–3368. DOI: 10.4049/jimmunol.1700893 |
| [11] | Stranzl T, Larsen MV, Lundegaard C, et al. NetCTLpan: pan-specific MHC class Ⅰ pathway epitope predictions. Immunogenetics, 2010, 62(6): 357–368. DOI: 10.1007/s00251-010-0441-4 |
| [12] | Bais P, Namburi S, Gatti DM, et al. CloudNeo: a cloud pipeline for identifying patient-specific tumor neoantigens. Bioinformatics, 2017, 33(19): 3110–3112. DOI: 10.1093/bioinformatics/btx375 |
| [13] | Pritchard AL, Burel JG, Neller MA, et al. Exome sequencing to predict neoantigens in melanoma. Cancer Immunol Res, 2015, 3(9): 992–998. DOI: 10.1158/2326-6066.CIR-15-0088 |
| [14] | Negrini S, Gorgoulis VG, Halazonetis TD. Genomic instability--an evolving hallmark of cancer. Nat Rev Mol Cell Biol, 2010, 11(3): 220–228. DOI: 10.1038/nrm2858 |
| [15] | The Cancer Genome Atlas Research Network, Weinstein JN, Collisson EA, et al. The cancer genome atlas pan-cancer analysis project. Nat Genet, 2013, 45(10): 1113–1120. DOI: 10.1038/ng.2764 |
| [16] |
Gao H, Han Y, Zhai XX, et al. The research progress of antigen presentation by MHC molecules.
Chin Bull Life Sci, 2017, 29(5): 450–461.
(in Chinese). 高花, 韩勇, 翟晓鑫, 等. MHC分子抗原递呈机制的研究进展. 生命科学, 2017, 29(5): 450-461. |
| [17] | Tenzer S, Peters B, Bulik S, et al. Modeling the MHC class Ⅰ pathway by combining predictions of proteasomal cleavage, TAP transport and MHC class Ⅰ binding. Cell Mol Life Sci, 2005, 62(9): 1025–1037. DOI: 10.1007/s00018-005-4528-2 |
| [18] | Paul S, Weiskopf D, Angelo MA, et al. HLA class Ⅰ alleles are associated with peptide-binding repertoires of different size, affinity, and immunogenicity. J Immunol, 2013, 191(12): 5831–5839. DOI: 10.4049/jimmunol.1302101 |
| [19] | Larsen MV, Lundegaard C, Lamberth K, et al. Large-scale validation of methods for cytotoxic T-lymphocyte epitope prediction. BMC Bioinf, 2007, 8: 424. DOI: 10.1186/1471-2105-8-424 |
| [20] |
Chen WQ, Sun KX, Zheng RS, et al. Report of cancer incidence and mortality in different areas of China, 2014.
China Cancer, 2018, 27(1): 1–14.
(in Chinese). 陈万青, 孙可欣, 郑荣寿, 等. 2014年中国分地区恶性肿瘤发病和死亡分析. 中国肿瘤, 2018, 27(1): 1-14. |
| [21] | Siegel RL, Miller KD, Jemal A. Cancer statistics, 2018. Cancer J Clin, 2018, 68(1): 7–30. DOI: 10.3322/caac.21442 |
| [22] | The UniProt Consortium. UniProt: the universal protein knowledgebase. Nucleic Acids Res, 2018, 46(5): 2699. DOI: 10.1093/nar/gky092 |
| [23] | Jiao XL, Sherman BT, Huang DW, et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics, 2012, 28(13): 1805–1806. DOI: 10.1093/bioinformatics/bts251 |
| [24] | Waterhouse A, Bertoni M, Bienert S, et al. SWISS-MODEL: homology modelling of protein structures and complexes. Nucleic Acids Res, 2018, 46(W1): W296–W303. DOI: 10.1093/nar/gky427 |
| [25] |
Zheng X. Significance of mutanome in tumor immunotherapy.
Chin J Cancer Biother, 2015, 22(6): 794–798.
(in Chinese). 郑晓. 肿瘤基因突变组学在肿瘤免疫治疗中的意义. 中国肿瘤生物治疗杂志, 2015, 22(6): 794-798. |
| [26] | Martincorena I, Seshasayee ASN, Luscombe NM. Evidence of non-random mutation rates suggests an evolutionary risk management strategy. Nature, 2012, 485(7396): 95–98. DOI: 10.1038/nature10995 |
| [27] | Baugh EH, Ke H, Levine AJ, et al. Why are there hotspot mutations in the TP53 gene in human cancers?. Cell Death Differ, 2018, 25(1): 154–160. DOI: 10.1038/cdd.2017.180 |
| [28] | Cao W, Ma EG, Zhou L, et al. Exploring the FGFR3-related oncogenic mechanism in bladder cancer using bioinformatics strategy. World J Surg Oncol, 2017, 15: 66. DOI: 10.1186/s12957-017-1125-4 |
| [29] | Koundouros N, Poulogiannis G. Phosphoinositide 3-kinase/akt signaling and redox metabolism in cancer. Front Oncol, 2018, 8: 160. DOI: 10.3389/fonc.2018.00160 |
| [30] | Ma DX, Yang JY, Wang Y, et al. Whole exome sequencing identified genetic variations in Chinese hemangioblastoma patients. Am J Med Genet A, 2017, 173(10): 2605–2613. DOI: 10.1002/ajmg.a.v173.10 |
| [31] | Mishra A, Brat DJ, Verma M. P53 tumor suppression network in cancer epigenetics. Methods Mol Biol, 2015, 1238: 597–605. DOI: 10.1007/978-1-4939-1804-1 |
| [32] | Marranci A, Jiang Z, Vitiello M, et al. The landscape of BRAF transcript and protein variants in human cancer. Mol Cancer, 2017, 16: 85. DOI: 10.1186/s12943-017-0645-4 |
| [33] | Kawada K, Toda K, Sakai Y. Targeting metabolic reprogramming in KRAS-driven cancers. Int J Clin Oncol, 2017, 22(4): 651–659. DOI: 10.1007/s10147-017-1156-4 |
| [34] | van Vlodrop IJH, Joosten SC, de Meyer T, et al. A four-gene promoter methylation marker panel consisting of GREM1, NEURL, LAD1, and NEFH predicts survival of clear cell renal cell cancer patients. Clin Cancer Res, 2017, 23(8): 2006–2018. DOI: 10.1158/1078-0432.CCR-16-1236 |
| [35] | Castle JC, Kreiter S, Diekmann J, et al. Exploiting the mutanome for tumor vaccination. Cancer Res, 2012, 72(5): 1081–1091. DOI: 10.1158/0008-5472.CAN-11-3722 |
| [36] |
Jia J. Functions of platelets in tumor growth and metastasis.
J Int Oncol, 2013, 18(11): 1033–1036.
(in Chinese). 贾静. 血小板在肿瘤转移中的作用. 临床肿瘤学杂志, 2013, 18(11): 1033-1036. |
| [37] | Joosse SA, Pantel K. Tumor-educated platelets as liquid biopsy in cancer patients. Cancer Cell, 2015, 28(5): 552–554. DOI: 10.1016/j.ccell.2015.10.007 |
| [38] | Rachidi S, Metelli A, Riesenberg B, et al. Platelets subvert T cell immunity against cancer via GARP-TGFβ axis. Sci Immunol, 2017, 2(11): eaai7911. DOI: 10.1126/sciimmunol.aai7911 |
| [39] | Kamat AM, Hahn NM, Efstathiou JA, et al. Bladder cancer. Lancet, 2016, 388(10061): 2796–2810. DOI: 10.1016/S0140-6736(16)30512-8 |
| [40] | Cleary AS. Teamwork: The tumor cell edition. Science, 2015, 350(6265): 1174–1175. DOI: 10.1126/science.aad7103 |
| [41] | Le DT, Hubbard-Lucey VM, Morse MA, et al. A blueprint to advance colorectal cancer immunotherapies. Cancer Immunol Res, 2017, 5(11): 942–949. DOI: 10.1158/2326-6066.CIR-17-0375 |
| [42] | O'Donnell TJ, Rubinsteyn A, Bonsack M, et al. MHCflurry: open-source class Ⅰ MHC binding affinity prediction. Cell Syst, 2018, 7(1): 129–132. DOI: 10.1016/j.cels.2018.05.014 |
| [43] | Boegel S, Löwer M, Bukur T, et al. A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines. Oncoimmunology, 2014, 3(8): e954893. DOI: 10.4161/21624011.2014.954893 |
| [44] | Park TS, Rosenberg SA, Morgan RA. Treating cancer with genetically engineered T cells. Trends Biotechnol, 2011, 29(11): 550–557. DOI: 10.1016/j.tibtech.2011.04.009 |
| [45] | Porter DL, Levine BL, Kalos M, et al. Chimeric antigen receptor-modified T cells in chronic lymphoid leukemia. N Engl J Med, 2011, 365(8): 725–733. DOI: 10.1056/NEJMoa1103849 |
| [46] | Lee DW, Kochenderfer JN, Stetler-Stevenson M, et al. T cells expressing CD19 chimeric antigen receptors for acute lymphoblastic leukaemia in children and young adults: a phase 1 dose-escalation trial. Lancet, 2015, 385(9967): 517–528. DOI: 10.1016/S0140-6736(14)61403-3 |
 2019, Vol. 35
2019, Vol. 35




