中国科学院微生物研究所、中国微生物学会主办
文章信息
- 周正雄, 堵国成, 康振
- Zhou Zhengxiong, Du Guocheng, Kang Zhen
- 3‘-磷酸腺苷-5’-磷酸硫酸的高效合成及其应用
- Production and application of 3'-phosphoadenosine-5'-phosphosulfate
- 生物工程学报, 2019, 35(7): 1222-1233
- Chinese Journal of Biotechnology, 2019, 35(7): 1222-1233
- 10.13345/j.cjb.180527
-
文章历史
- Received: December 20, 2018
- Accepted: February 18, 2019
- Published: March 13, 2019
2. 江南大学 生物工程学院,江苏 无锡 214122
2. School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China
硫酸化反应广泛存在于生物体内源性物质代谢及外源性物质化学修饰过程中,对细胞发育、分化、免疫、解毒等生物学功能作用显著。在生物体中,大部分硫酸转移酶催化3'-磷酸腺苷-5'-磷酸硫酸(3'-phosphoadenosine-5'-phosphosulfate,PAPS)的硫酸基团转移到对应的底物中合成硫酸化的代谢产物。上述硫酸化反应机制也用于生物体或体外酶法制备肝素、硫酸软骨素、羟胺硝喹等化合物。但PAPS是一类高能磷酸化合物,在胞内不能大量积累,在体外不能稳定保存。因此,构建稳定、高效的PAPS合成体系对肝素、硫酸软骨素、羟胺硝喹等高附加值化合物的生成尤为重要。
在过去十年,研究者从PAPS的代谢途径、合成方式及其生物学应用对其进行探究。文中主要综述了全细胞合成PAPS、体外酶法制备PAPS及PAPS的生物学应用,为PAPS的高效制备及其应用提供潜在的方向。
1 PAPS的合成 1.1 利用全细胞合成PAPS从单细胞生物到哺乳动物细胞中都存在ATP到PAPS的合成途径。ATP经ATP硫酸化酶催化形成5'-磷酸腺苷硫酸(Adenosine-5'-phosphosulfate,APS),再经APS激酶催化形成PAPS (图 1),或由双功能酶PAPS合酶催化ATP合成PAPS。PAPS合成过程中存在3个关键因素:1)硫酸基团的转运和供给;2) ATP的合成速度;3) ATP合成PAPS的转化效率。
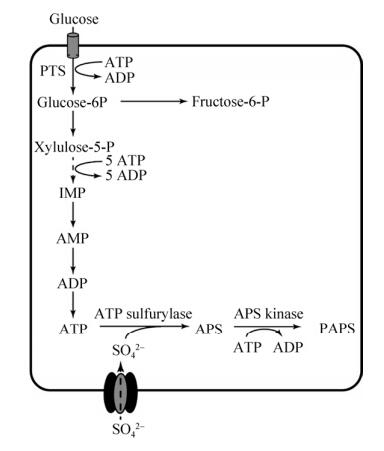
|
| 图 1 PAPS合成途径 Fig. 1 Biosynthetic pathway of PAPS. PTS: phosphoenolpyruvate-dependent glucose phosphotransferase system; IMP: inosine monophosphate; PAPS: 3'-phosphoadenosine-5'-phosphosulfate. |
| |
1) 硫酸基团的转运和供给。在生物体内硫酸基团的转运方式有两种:钠离子偶联型及钠离子非偶联型离子通道。所谓钠离子偶联型通道是通过钠离子偶联转运体转运钠离子的同时按照3︰1的比例转运硫酸基团等-2价阴离子,这种转运方式主要存在于肾脏细胞及肠道细胞中[1]。钠离子非偶联型离子通道又称硫酸根离子通道Ⅰ,主要用于非特异性的转运如草酰乙酸、琥珀酸等有机离子。其中钠离子偶联型离子通道对硫酸根的转运能力比钠离子非偶联型离子通道转运能力高50倍以上[2]。
2) ATP的合成速度。ATP是细胞内的能量物质,主要由氧化磷酸化和糖酵解途径产生。氧化磷酸化发生在线粒体中(真核生物)或细胞质中(原核生物),糖酵解途径在细胞质中进行。在有氧呼吸条件下,氧化磷酸化提供细胞生长代谢所需的绝大部分ATP。膜内外电位差及ΔpH驱动ADP和Pi合成ATP[3]。降低细胞有氧呼吸的程度即降低细胞中的溶解氧水平能显著降低ATP的合成速度[4]。而Ca2+能够促进线粒体的有氧呼吸代谢从而提高ATP的合成速度[5]。在正常条件下,细胞内的ATP浓度约为5 nmol/mg蛋白,ATP的合成速度约为400 nmol/(min·mg蛋白)[4]其中绝大部分的ATP用于细胞自身的能量代谢,ATP浓度过高或过低都会造成细胞的代谢紊乱[6]。
3) ATP合成PAPS的转化效率。目前,生物体内催化ATP合成PAPS的途径有两种。在植物和微生物细胞中ATP经ATP硫酸化酶及APS激酶共同催化形成PAPS。在哺乳动物细胞中存在一种双功能酶PAPS合酶能直接催化ATP合成PAPS。其N端序列表现为APS激酶活性,C端序列表现为ATP硫酸化酶活性。ATP分子结合ATP硫酸化酶后形成U型结构并与硫酸根结合催化形成APS[7]。但催化形成的APS占据ATP硫酸化酶的底物结合位点阻止ATP、硫酸基团与酶的结合从而抑制ATP硫酸化酶的活性。同时APS对APS激酶也产生抑制作用。APS激酶的催化机理也遵从如ATP硫酸化酶一样的乒乓机制,即APS激酶结合Mg2+、ATP后再与APS结合形成PAPS和ADP,然后释放出PAPS最后释放ADP还原成活性酶。但过量的APS能在ADP释放前与其结合形成APS激酶-APS-ADP的复合物阻止ADP的释放和Mg2+、ATP的结合从而抑制APS激酶活性[8]。对于双功能酶PAPS合成酶而言,其硫酸化活性远大于其激酶活性,这也就意味着APS很容易过量积累从而抑制PAPS合酶的活性。
综上所述,全细胞合成PAPS要解决的关键问题是:①增强硫酸基团转运和供给能力:通过异源表达钠离子依赖性离子通道增强硫酸基团转运能力。②增强ATP的合成速度,同时增加ATP流向PAPS的代谢通量维持机体正常的ATP供给水平。③解除APS的反馈抑制、底物抑制作用:提高APS激酶活性和对ADP的释放能力从而消除APS对APS激酶的抑制作用,同时提高ATP硫酸化酶对APS的释放能力达到提高ATP到PAPS的转化率。
1.2 体外酶法制备PAPS作为高能化合物PAPS在细胞内的积累水平相对较低,因此由细胞制备PAPS距离工业化应用还较远。研究人员一般采用体外酶法制备PAPS供给后续应用。体外酶法制备PAPS主要包括两种方法:ATP硫酸化酶和APS激酶催化ATP制备PAPS (图 2A)、酰基磺酸转移酶(Arylsulfotransferase Ⅳ,ASTIV)催化3', 5'-二磷酸腺苷(3′-phosphoadenosine- 5′-phosphate,PAP)合成PAPS (图 2B)。

|
| 图 2 PAPS再生系统 Fig. 2 Regeneration system of PAPS. (A) PAPS production based on ATP. (B) PAPS production based on PAP. PNPS: para-nitrophenylsulfate; PNP: pare-nitrophenol; PAP: 3′-phosphoadenosine-5′-phosphate. |
| |
1) 基于ATP硫酸化酶和APS激酶制备PAPS。不同来源的ATP硫酸化酶序列相似性较低,约为20%–30%,空间结构也复杂多样,包括单体、同源二聚体、同源四聚体、同源六聚体和异源多聚体[7, 9-16]。但其单体的空间结构却呈保守的不规则球形,其底物结合口袋由一条宽阔的通道与球体表面相通,Arg、His等碱性氨基酸与ATP的β、γ-磷酸基团相结合固定底物ATP和硫酸基团,HXXH残基直接决定其硫酸化活性[13, 17]。在体外酶催化反应过程中,ATP硫酸化酶也遵循乒乓机制,即ATP硫酸化酶先与Mg2+、ATP结合再与硫酸根结合后合成并释放出APS,最后释放出PPi。
从动力学的角度来说,ATP硫酸化酶反应释放出的副产物PPi能与大环荧光探针3-(9-蒽甲基)-3, 6, 9, 15-四氮杂双环十五-1, 11, 13-三烯和Zn2+复合物结合发生荧光淬灭,达到实时检测ATP硫酸化酶的活性的能力[18]。ATP硫酸化酶的催化速度可以达到0.1-0.4 μmol/(min·mg蛋白)[19]。但植物来源的ATP硫酸化酶催化活性相对较低,约为0.1-0.3 nmol/(min·mg蛋白)[20]。镁离子是ATP硫酸化酶反应的必需金属离子,而氯酸盐却能在一定程度上抑制ATP硫酸化酶活性[21]。这为随机突变ATP硫酸化酶提供良好的检测基础及实施的可能性。
不同来源的APS激酶在氨基酸序列和空间结构上也存在保守序列或保守空间。APS激酶在生物体内以二聚体的形式存在,其N端的Cys之间形成二硫键(蓝藻来源APS激酶除外)。其N端的二硫键改变APS激酶N端序列的空间结构,降低APS造成的底物抑制作用,且还原态的APS激酶活性高于氧化态的APS激酶活性[22-24]。蛋白质的二级结构分析表明,Arg93是底物识别的重要氨基酸,突变体R93A完全失去了底物抑制作用,同时也降低了ADP物质的亲和作用(降低217倍),达到提高APS激酶活性的目的[23]。
在理想条件下,APS激酶的催化速度可以达到10-900 nmol/(min·mg蛋白)[25]。ATP转化为PAPS的转化率为47%,与此同时生成等量的副产物ADP。An等[26]通过引入丙酮酸激酶催化ADP合成ATP进一步提高ATP合成PAPS的转化率至超过90%。
较高的转化率为PAPS的高效合成提供有力的保障。想要体外酶法高效制备PAPS还需要较高的酶量。目前报道的ATP硫酸化酶有30 000多种,APS激酶有3 200多种(www.ncbi.nlm.nih.gov),其中能在微生物中进行表达的屈指可数。大肠杆菌和酿酒酵母是ATP硫酸化酶或APS激酶表达的常用工业宿主。来自于拟南芥等的ATP硫酸化酶可在酿酒酵母中进行活性表达[27]。来自酿酒酵母[28]、洋葱[29]、山茶花[30]、产黄青霉[31]、乳酸克鲁维酵母[32]等的ATP硫酸化酶和APS激酶可在大肠杆菌中进行活性表达。但目前关于上述来源的ATP硫酸化酶和APS激酶没有产量的报道。从酶的空间结构来说,动物来源和植物来源的ATP硫酸化酶是同源二聚体,而微生物来源的ATP硫酸化酶是由基因cysD、cysN共同编码的异源二聚体。在大肠杆菌中共表达cysD、cysN时得到的ATP硫酸化酶活性是单独表达cysD的ATP硫酸化酶的活性的40倍[33]。APS激酶在N端形成分子内的二硫键使APS激酶处于氧化态[23],因此要选择合适的表达宿主如大肠杆菌Rosetta等[34]或在N端融合促进二硫键形成的短肽如TrxA[35],或共表达促进蛋白质折叠的伴侣分子DsbL等帮助蛋白质折叠[36-37],或者进行分泌表达,将蛋白分泌到周质空间中提供二硫键形成的环境[38]或者选用大肠杆菌以外的合适的表达宿主。
综上所述,以ATP为底物催化合成PAPS时,需要解决的问题包括:①提高ATP硫酸化酶释放产物APS的速度降低APS的产物抑制作用:通过改变底物结合口袋的氨基酸序列,在底物结合口袋中引入酸性或中性氨基酸减少酶与产物间的引力作用,从而加快APS的释放。②提高APS激酶释放产物PAPS和ADP的速度降低APS的底物抑制作用:通过在U型通道后面引入更多疏水性氨基酸扩大底物结合口袋U型通道,加快PAPS和ADP的释放速度,从而降低酶-ADP-APS复合物形成的可能性即降低APS的底物抑制作用,达到提高APS激酶活性的目的。③ATP硫酸化酶和APS激酶的表达:筛选合适的表达宿主及基因来源提高上述两个酶的表达量,以大肠杆菌为宿主进行表达上述基因时,除了常规的优化启动子、RBS、终止子、诱导剂浓度、诱导温度等手段提高表达量外,还要在N端或C端融合相应的蛋白提高二硫键的形成概率达到提高蛋白正确折叠的目的。
2) 基于ASTIV催化PAP合成PAPS。来自于哺乳动物和微生物的酰基磺酸转移酶能催化PAP合成PAPS。目前人来源ASTIV共有3种同工酶,分别是hSULT1A1、hSULT1A2、hSULT1A3,这3种同工酶的序列相似性在90%以上,且与大鼠来源的ASTIV序列相似性也达到约80%。其晶体呈粗糙球形,PAP的底物结合口袋在球体内部,通往底物结合口袋的通道入口有一段柔性区域像整个口袋的帽子(又称作门控序列,Gating loop),阻挡底物与酶的结合,从而降低PAPS的合成速率(图 3)。因此小分子底物或者偏中性底物相对容易进入底物结合口袋通道,而大分子的带负电荷的物质相对较难进入通道,也就意味着ASTIV对大分子PAP的亲和力相对较弱。但该门控序列所属类型(包括翼门型(Wing gate)、双开门型(Swinging door gate)、光圈孔型(Aperture gate)、吊门和双吊门型(Drawbridge and double drawbridge gates)、外壳门型(Shell gate))及作用方式目前研究还较少,因此目前对该段柔性Loop还没有较多理性的改造方式,后期研究过程中可以对其进行随机突变来提高PAPS的合成速度[39]。

|
| 图 3 人来源ASTIV (SULT1A1)晶体结构 Fig. 3 3D structure of human SULT1A1. (A) 3D structure of human SULT1A1. The gray loop was the gating loop, and the black ball was the substrate, PAP. (B) Amine acids in SULT1A1-PAP interactions on a 2D diagram. |
| |
此外,底物口袋表面多为亲水性、碱性氨基酸包括Lys、His、Arg等用于与PAP的磷酸基团结合固定底物,其催化氨基酸为His[40]。然而ASTIV的底物结合口袋具有很强的可塑性以适应不同的底物,因此其底物谱较广[41]。除了能催化PAP硫酸化为PAPS外,还能催化其他酚类、醇类、含氮类化合物硫酸化,且对小分子物质的亲和性相对较强但催化活性都较低,其中hSULT1A1主要催化小分子酚类物质,hSULT1A3主要催化胺类物质[42]。同时其底物结合口袋背面的氨基酸决定底物结合口袋的相对大小,从而调节催化活性及底物谱,如Y149F能提高hSULT1A2对对硝基苯酚的Km值近40倍[43],A146E能够改变hSULT1A1的底物谱类似hSULT1A3[44]。
与ATP硫酸化酶及APS激酶催化ATP合成PAPS一样,影响ASTIV催化形成PAPS应用于工业生产的因素除了其比酶活外还有得到ASTIV的成本,即ASTIV的表达水平。根据前期本课题组得到的数据表明,不同温度条件下ASTIV可溶表达的水平相差不大,但随着温度的升高,不可溶表达的ASTIV含量越高。换言之,影响ASTIV活性表达的因素不是蛋白的翻译水平而是蛋白的折叠水平,本课题组在ASTIV N端融合促融标签麦芽糖融合蛋白(Maltose binding protein,MBP),但效果不够显著[45]。分析其结构表明,ASTIV内部有一对二硫键,而在大肠杆菌胞内合成和折叠不利于二硫键的形成,从而导致可溶表达的水平较低[46]。因此,可以采取同APS激酶表达同样的策略提高ASTIV的可溶表达。
综上所述,以PAP为底物催化合成PAPS时,需要解决的问题包括:①减少底物结合口袋帽子结构的酸性氨基酸含量:通过点突变减少帽子结构中Glu的含量,从而减少帽子结构对于带负电荷的底物PAP的排斥作用,提高酶对PAP的亲和力。②理性设计突变底物结合口袋的氨基酸序列:通过增加碱性氨基酸的数量进一步提高底物结合口袋对于PAP的正负电荷引力作用,同时扩大底物结合口袋提高PAP进入底物结合口袋的速率从而达到提高PAPS合成速率的目的。③提高ASTIV二硫键形成的速率:通过融合蛋白或者共表达伴侣分子等策略提供二硫键形成的环境提高ASTIV二硫键形成的速率达到提高ASTIV可溶表达水平。
2 PAPS的生物学应用 2.1 芥子油苷的生物合成芥子油苷也称硫代葡萄糖苷或硫苷(Glucosinolate,GLS),是十字花科植物中的一类次级代谢物,主要存在于植物细胞的液泡中,当组织受到损伤时与黑芥子酶发生水解反应形成具有实际活性的物质异硫氰酸盐、硫氰酸盐和吲哚,从而达到保护植物的目的[47-48]。目前研究表明芥子油苷通过肠道内的微生物分解后形成异硫氰酸盐还具有抗癌的功效。研究其结构表明其含有1个R侧链和1个硫原子相连的D-砒喃葡萄糖。根据其R侧链差异分为脂肪族、芳香族和吲哚族芥子油苷。
在芥子油苷合成过程中,硫酸根经硫酸根转运通道(Sulfate transporter,SULTR)转运进细胞后经过质体膜跨膜蛋白转运进入质体。在细胞质中,硫酸根经ATP硫酸化酶(由cyt-ATPS2编码)及APS激酶(由APK3编码)共同催化形成PAPS;质体中ATP硫酸化酶由ATPS1、ATPS2、ATPS3、ATPS4编码,APS激酶由APK1、APK2、APK4编码。质体中合成的PAPS经跨膜通道进入细胞质中,催化脱硫酸芥子油苷(Desulfo-glucosinolate,Desulfo-GLS)合成芥子油苷(图 4)。因此,提高土壤中硫酸盐的供给或者细胞膜对硫酸根的转运能力能提高PAPS的合成能力从而提高芥子油苷的合成能力[49]。此外,Yatusevich等[50]研究表明提高转录因子R2R3-MYB的表达量能增加ATP硫酸化酶及APS激酶的表达量,加速PAPS的合成从而增加芥子油苷的合成量。

|
| 图 4 植物细胞中芥子油苷的合成机制 Fig. 4 Biosynthesis of glucosinolate in plant cell. SULTR: sulfate transporter; desulfo-GLS: desulfo- glucosinolate; cyt-ATPS2 is the ATPS2 in cytosol. |
| |
糖胺聚糖是一类广泛存在于细胞表面和胞外基质中的多糖类物质,主要包括肝素、硫酸乙酰肝素、硫酸软骨素、硫酸皮肤素、硫酸角质素、透明质酸等6类,除了透明质酸外其他5类都需要硫酸化修饰形成具有生物学活性的产物(图 5)。糖胺聚糖具有信号传递、免疫等生物学功能,在临床上被广泛用于术后抗凝血(肝素)、骨关节炎的治疗(硫酸软骨素)等[51]。但临床上使用的糖胺聚糖主要从动物组织中提取,含有较多的杂多糖,降低相应药物的疗效甚至危及生命[52]。酶法制备糖胺聚糖具有产物单一的优势,因此被认为是最有可能替代动物组织提取被应用于临床的糖胺聚糖生产方式[53]。
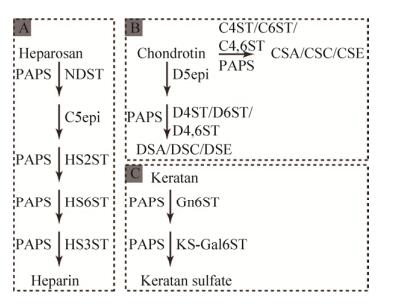
|
| 图 5 酶法合成糖胺聚糖 Fig. 5 Enzymatic synthesis of glycosaminoglycan. (A) Enzymatic synthesis of heparin base on heparosan. NDST, N-deacetylase/N-sulfotransferase; C5epi, glucuronyl C5 epimerase; HS2ST, heparan sulfate 2-O-sulfotransferase; HS6ST, heparan sulfate 6-O-sulfotransferase; HS3ST, heparan sulfate 3-O-sulfotransferase. (B) Enzymatic synthesis of chondroitin sulfate and dermatan sulfate. C4ST, chondroitin 4-O-sulfotransferase; C6ST, chondroitin 6-O-sulfotransferase; C4, 6ST, chondroitin 4, 6-O-sulfotransferase; D5epi, dermatan epimerase; D4ST, dermatan 4-O-sulfotransferase; D6ST, dermatan 6-O-sulfotransferase; D4, 6ST, dermatan 4, 6-O- sulfotransferase. (C) Enzymatic synthesis of keratan sulfate. Gn6ST, corneal N-acetylglucosaminyl-6- sulfotransferase; KS-Gal6ST, keratan sulfate galactosyl- 6-sulfotransferase. |
| |
在肝素和硫酸乙酰肝素合成过程中,肝素前体经N-脱乙酰/N-硫酸转移酶(N-deacetylase/ N-sulfotransferase,NDST)、葡萄糖醛酸变构酶(Glucuronyl C5 epimerase,C5epi)、肝素2-O-硫酸转移酶(Heparan sulfate 2-O-sulfotransferase,HS2ST)、肝素6-O-硫酸转移酶(Heparan sulfate 6-O-sulfotransferase,HS6ST)、肝素3-O-硫酸转移酶(Heparan sulfate 3-O-sulfotransferase,HS3ST)等催化合成具有抗凝血活性的肝素,在此过程中,PAPS是肝素前体硫酸化修饰的直接硫酸基供体(图 5A)[54-55]。在硫酸软骨素合成过程中,软骨素经软骨素4-O-硫酸转移酶(Chondroitin 4-O-sulfotransferase,C4ST)或软骨素6-O-硫酸转移酶(Chondroitin 6-O-sulfotransferase,C6ST)或软骨素4, 6-O-硫酸转移酶(Chondroitin 4, 6-O- sulfotransferase,C4,6ST)催化形成硫酸软骨素A (Chondroitin sulfate A,CSA)、硫酸软骨素C (Chondroitin sulfate C,CSC)或硫酸软骨素E (Chondroitin sulfate E,CSE),在此过程中所用的硫酸基供体还是PAPS (图 5B)[56]。在软骨素经软骨素葡萄糖醛酸变构酶(Dermatan epimerase,D5epi)、皮肤素4-O-硫酸转移酶(Dermatan 4-O-sulfotransferase,D4ST)、皮肤素6-O-硫酸转移酶(Dermatan 6-O-sulfotransferase,D6ST)、皮肤素4,6-O-硫酸转移酶(Dermatan 4, 6-O-sulfotransferase,D4,6ST)催化形成多种硫酸皮肤素(Dermatan sulfate,DS)及角质素经角膜N-乙酰氨基6-硫酸转移酶(Corneal N-acetylglucosaminyl-6-sulfotransferase,Gn6ST)、半乳糖苷-6-硫酸转移酶(Keratan sulfate galactosyl-6-sulfotransferase,KS-Gal6ST)催化形成硫酸角质素(Keratan sulfate,KS)过程中,所用的硫酸基供体全是PAPS (图 5C)[57-58]。
20年前人们研究上述糖胺聚糖酶法合成一般采用外源添加PAPS,造成合成糖胺聚糖的价格昂贵[59]。且在硫酸化反应过程中产生大量的副产物PAP,而PAP能够进一步与硫酸转移酶中的PAPS底物结合位点进行结合抑制硫酸转移酶的活性,降低相应产物的硫酸化程度。由ATP硫酸化酶和APS激酶催化ATP合成PAPS的再生系统引入糖胺聚糖酶法催化系统,在一定程度上降低了PAPS的合成成本实现化学酶法合成糖胺聚糖[32, 45],但没有解决副产物抑制的问题。由ASTIV催化PAP合成PAPS的再生循环系统降低了硫酸化修饰过程中副产物PAP的抑制作用。虽然上述两种PAPS再生的方式在一定程度上降低了酶法制备糖胺聚糖的成本,但酶法合成糖胺聚糖还是成本较高,PAPS合成成本依旧是制约化学酶法或酶法合成糖胺聚糖的关键因素,距离酶法合成糖胺聚糖的工业化应用还有一段距离。
2.3 羟胺硝喹疗效激活目前全世界有近3亿人正在饱受血吸虫病的折磨,大约有20万人因此而丧命[60]。同时血吸虫病能够诱导产生很多其他的并发症,如肝纤维化[61]、尿血症[60]等。20世纪80年代之前,治疗血吸虫病的主要药物为羟胺硝喹,但随着此药物的大量使用,血吸虫病的病原菌埃及血吸虫Schistosoma heamatobium、日本血吸虫Schistosoma japonicum和曼氏血吸虫Schistosoma mansoni对此产生了耐药性[62]。研究发现S. mansoni中存在一种硫酸转移酶能够催化羟胺硝喹的羟基硫酸化修饰为硫胺硝喹并抑制S. mansoni的生长,从而达到治疗S. heamatobium、S. mansoni引起的血吸虫病(由这两种病菌造成全世界99%的血吸虫病)[63-64],在此过程中使用的硫酸基供体也为PAPS。
此外,PAPS再生系统还应用于黄酮类[65]、胆碱类[66-67]、雌激素类[68-69]、固醇类[70-71]和倍酸类化合物[67]的硫酸化修饰及蛋白质酪氨酸硫酸化修饰等蛋白质翻译后修饰系统[72-73]等。
3 展望PAPS再生系统在整个化合物的硫酸化修饰过程中占据无比重要的位置,且对于工业化应用来说是成本控制的关键。目前不论从ATP合成PAPS还是从PAP合成PAPS的合成成本都离工业化还有一段距离。因此,未来可以从提高酶的活性表达和酶的转化效率上来进一步提高合成PAPS的速度达到降低生产成本的目的。提高酶的活性表达水平包括:1)优化酶的转录和翻译元件,如优化启动子、RBS、终止子序列等。2)提高二硫键形成速率帮助酶正确折叠,如共表达伴侣分子DsbL、在N端融合硫氧还蛋白、筛选合适的信号肽将酶分泌到周质空间等。3)优化诱导剂浓度、诱导温度等培养条件提高酶的表达。提高酶的转化效率是指提高比酶活,是根据ATP硫酸化酶、APS激酶和ASTIV的底物结合口袋和催化机制来理性设计调控底物结合口袋和通道序列,达到提高底物结合能力及产物释放速度。
| [1] | Markovich D, Murer H. The SLC13 gene family of sodium sulphate/carboxylate cotransporters. Pflügers Arch, 2004, 447(5): 594–602. DOI: 10.1007/s00424-003-1128-6 |
| [2] | Bissig M, Hagenbuch B, Stieger B, et al. Functional expression cloning of the canalicular sulfate transport system of rat hepatocytes. J Biol Chem, 1994, 269(4): 3017–3021. |
| [3] | Turina P, Petersen J, Gräber P. Thermodynamics of proton transport coupled ATP synthesis. Biochim Biophys Acta, 2016, 1857(6): 653–664. DOI: 10.1016/j.bbabio.2016.02.019 |
| [4] | Vanasco V, Magnani ND, Cimolai MC, et al. Endotoxemia impairs heart mitochondrial function by decreasing electron transfer, ATP synthesis and ATP content without affecting membrane potential. J Bioenerg Biomembr, 2012, 44(2): 243–252. DOI: 10.1007/s10863-012-9426-3 |
| [5] | Tarasov AI, Griffiths EJ, Rutter GA. Regulation of ATP production by mitochondrial Ca2+. Cell Calcium, 2012, 52(1): 28–35. DOI: 10.1016/j.ceca.2012.03.003 |
| [6] | Rangaraju V, Calloway N, Ryan TA. Activity-driven local ATP synthesis is required for synaptic function. Cell, 2014, 156(4): 825–835. DOI: 10.1016/j.cell.2013.12.042 |
| [7] | Ullrich TC, Blaesse M, Huber R. Crystal structure of ATP sulfurylase from Saccharomyces cerevisiae, a key enzyme in sulfate activation. EMBO J, 2001, 20(3): 316–329. DOI: 10.1093/emboj/20.3.316 |
| [8] | Mueller JW, Shafqat N. Adenosine-5'-phosphosulfate - a multifaceted modulator of bifunctional 3'-phospho-adenosine-5'-phosphosulfate synthases and related enzymes. FEBS J, 2013, 280(13): 3050–3057. DOI: 10.1111/febs.12252 |
| [9] | Mougous JD, Lee DH, Hubbard SC, et al. Molecular basis for G protein control of the prokaryotic ATP sulfurylase. Mol Cell, 2006, 21(1): 109–122. DOI: 10.1016/j.molcel.2005.10.034 |
| [10] | Taguchi Y, Sugishima M, Fukuyama K. Crystal structure of a novel zinc-binding ATP sulfurylase from Thermus thermophilus HB8. Biochemistry, 2004, 43(14): 4111–4118. DOI: 10.1021/bi036052t |
| [11] | MacRae IJ, Segel IH, Fisher AJ. Allosteric inhibition via R-state destabilization in ATP sulfurylase from Penicillium chrysogenum. Nat Struct Biol, 2002, 9(12): 945–949. DOI: 10.1038/nsb868 |
| [12] | Beynon JD, MacRae IJ, Huston SL, et al. Crystal structure of ATP sulfurylase from the bacterial symbiont of the hydrothermal vent tubeworm Riftia pachyptila. Biochemistry, 2001, 40(48): 14509–14517. DOI: 10.1021/bi015643l |
| [13] | Herrmann J, Ravilious GE, McKinney SE, et al. Structure and mechanism of soybean ATP sulfurylase and the committed step in plant sulfur assimilation. J Biol Chem, 2014, 289(15): 10919–10929. DOI: 10.1074/jbc.M113.540401 |
| [14] | Lalor DJ, Schnyder T, Saridakis V, et al. Structural and functional analysis of a truncated form of Saccharomyces cerevisiae ATP sulfurylase: C-terminal domain essential for oligomer formation but not for activity. Protein Eng, 2003, 16(12): 1071–1079. DOI: 10.1093/protein/gzg133 |
| [15] | MacRae IJ, Segel IH, Fisher AJ. Crystal structure of ATP sulfurylase from Penicillium chrysogenum: insights into the allosteric regulation of sulfate assimilation. Biochemistry, 2001, 40(23): 6795–6804. DOI: 10.1021/bi010367w |
| [16] | Ullrich TC, Huber R. The complex structures of ATP sulfurylase with thiosulfate, ADP and chlorate reveal new insights in inhibitory effects and the catalytic cycle. J Mol Biol, 2001, 313(5): 1117–1125. DOI: 10.1006/jmbi.2001.5098 |
| [17] | Jaramillo ML, Abanto M, Quispe RL, et al. Cloning, expression and bioinformatics analysis of ATP sulfurylase from Acidithiobacillus ferrooxidans ATCC 23270 in Escherichia coli. Bioinformation, 2012, 8(15): 695–704. DOI: 10.6026/bioinformation |
| [18] | Wang XB, Zhang ZY, Ma XY, et al. Real-time fluorescence assays of alkaline phosphatase and ATP sulfurylase activities based on a novel PPi fluorescent probe. Talanta, 2015, 137: 156–160. DOI: 10.1016/j.talanta.2015.01.028 |
| [19] | Mugford SG, Matthewman CA, Hill L, et al. Adenosine-5'-phosphosulfate kinase is essential for Arabidopsis viability. Febs Lett, 2010, 584(1): 119–123. DOI: 10.1016/j.febslet.2009.11.014 |
| [20] | Schiffmann S, Schwenn JD. APS-sulfotransferase activity is identical to higher plant APS-kinase (EC 2.7.1.25). Febs Lett, 1994, 355(3): 229–232. DOI: 10.1016/0014-5793(94)01193-1 |
| [21] | Ravilious GE, Herrmann J, Lee SG, et al. Kinetic mechanism of the dimeric ATP sulfurylase from plants. Biosci Rep, 2013, 33(4): e00053. |
| [22] | Herrmann J, Nathin D, Lee SG, et al. Recapitulating the structural evolution of redox regulation in adenosine 5ʹ-phosphosulfate kinase from cyanobacteria to plants. J Biol Chem, 2015, 290(41): 24705–24714. DOI: 10.1074/jbc.M115.679514 |
| [23] | Ravilious GE, Westfall CS, Jez JM. Redox-linked gating of nucleotide binding by the N-terminal domain of adenosine 5ʹ-phosphosulfate kinase. J Biol Chem, 2013, 288(9): 6107–6115. DOI: 10.1074/jbc.M112.439414 |
| [24] | Ravilious GE, Nguyen A, Francois JA, et al. Structural basis and evolution of redox regulation in plant adenosine-5'-phosphosulfate kinase. Proc Natl Acad Sci USA, 2012, 109(1): 309–314. DOI: 10.1073/pnas.1115772108 |
| [25] | Schriek U, Schwenn JD. Properties of the purified APS-kinase from Escherichia coli and Saccharomyces cerevisiae. Arch Microbiol, 1986, 145(1): 32–38. DOI: 10.1007/BF00413024 |
| [26] | An CY, Zhao L, Wei ZJ, et al. Chemoenzymatic synthesis of 3'-phosphoadenosine-5'-phosphosulfate coupling with an ATP regeneration system. Appl Microbiol Biotechnol, 2017, 101(20): 7535–7544. DOI: 10.1007/s00253-017-8511-2 |
| [27] | Logan HM, Cathala N, Grignon C, et al. Cloning of a cDNA encoded by a member of the Arabidopsis thaliana ATP sulfurylase multigene family: expression studies in yeast and in relation to plant sulfur nutrition. J Biol Chem, 1996, 271(21): 12227–12233. DOI: 10.1074/jbc.271.21.12227 |
| [28] | Luo J, Wu WJ, Zou BJ, et al. Expression and purification of ATP sulfurylase from Saccharomyces cerevisias in Escherichia coli and its application in pyrosequencing//Zhou GH, Song QX, Eds. Advances and Clinical Practice in Pyrosequencing. New York: Springer, 2016: 187-195. |
| [29] | Cumming M, Leung S, McCallum J, et al. Complex formation between recombinant ATP sulfurylase and APS reductase of Allium cepa (L.). Febs Lett, 2007, 581(22): 4139–4147. DOI: 10.1016/j.febslet.2007.07.062 |
| [30] | Zhu L, Deng WW, Ye AH, et al. Cloning of two cDNAs encoding a family of ATP sulfurylase from Camellia sinensis related to selenium or sulfur metabolism and functional expression in Escherichia coli. Plant Physiol Bioch, 2008, 46(8/9): 731–738. |
| [31] | Bao FF, Yan HH, Sun HJ, et al. Hydrolysis of by-product adenosine diphosphate from 3'-phosphoadenosine-5'-phosphosulfate preparation using Nudix hydrolase NudJ. Appl Microbiol Biotechnol, 2015, 99(24): 10771–10778. DOI: 10.1007/s00253-015-6911-8 |
| [32] | Zhou XX, Chandarajoti K, Pham TQ, et al. Expression of heparan sulfate sulfotransferases in Kluyveromyces lactis and preparation of 3'-phosphoadenosine-5'-phosphosulfate. Glycobiology, 2011, 21(6): 771–780. DOI: 10.1093/glycob/cwr001 |
| [33] | Leyh TS, Taylor JC, Markham GD. The sulfate activation locus of Escherichia coli K12: cloning, genetic, and enzymatic characterization. J Biol Chem, 1988, 263(5): 2409–2416. |
| [34] | Gopal GJ, Kumar A. Strategies for the production of recombinant protein in Escherichia coli. Protein J, 2013, 32(6): 419–425. DOI: 10.1007/s10930-013-9502-5 |
| [35] | Bessette PH, slund F, Beckwith J, et al. Efficient folding of proteins with multiple disulfide bonds in the Escherichia coli cytoplasm. Proc Natl Acad Sci USA, 1999, 96(24): 13703–13708. DOI: 10.1073/pnas.96.24.13703 |
| [36] | Inaba K. Disulfide bond formation system in Escherichia coli. J Biochem, 2009, 146(5): 591–597. DOI: 10.1093/jb/mvp102 |
| [37] | Ito K, Inaba K. The disulfide bond formation (Dsb) system. Curr Opin Struct Biol, 2008, 18(4): 450–458. DOI: 10.1016/j.sbi.2008.02.002 |
| [38] | Salinas G, Pellizza L, Margenat M, et al. Tuned Escherichia coli as a host for the expression of disulfide-rich proteins. Biotechnol J, 2011, 6(6): 686–699. DOI: 10.1002/biot.201000335 |
| [39] | Marques SM, Daniel L, Buryska T, et al. Enzyme tunnels and gates as relevant targets in drug design. Med Res Rev, 2017, 37(5): 1095–1139. DOI: 10.1002/med.2017.37.issue-5 |
| [40] | Malojčić G, Owen RL, Grimshaw JPA, et al. A structural and biochemical basis for PAPS-independent sulfuryl transfer by aryl sulfotransferase from uropathogenic Escherichia coli. Proc Natl Acad Sci USA, 2008, 105(49): 19217–19222. DOI: 10.1073/pnas.0806997105 |
| [41] | Berger I, Guttman C, Amar D, et al. The molecular basis for the broad substrate specificity of human sulfotransferase 1A1. PLoS ONE, 2011, 6(11): e26794. DOI: 10.1371/journal.pone.0026794 |
| [42] | Gamage NU, Tsvetanov S, Duggleby RG, et al. The structure of human SULT1A1 crystallized with estradiol. An insight into active site plasticity and substrate inhibition with multi-ring substrates. J Biol Chem, 2005, 280(50): 41482–41486. DOI: 10.1074/jbc.M508289200 |
| [43] | Lu JH, Li HT, Zhang JP, et al. Crystal structures of SULT1A2 and SULT1A1*3: Insights into the substrate inhibition and the role of Tyr149 in SULT1A2. Biochem Biophys Res Commun, 2010, 396(2): 429–434. DOI: 10.1016/j.bbrc.2010.04.109 |
| [44] | Brix LA, Barnett AC, Duggleby RG, et al. Analysis of the substrate specificity of human sulfotransferases SULT1A1 and SULT1A3: site-directed mutagenesis and kinetic studies. Biochemistry, 1999, 38(32): 10474–10479. DOI: 10.1021/bi990795q |
| [45] | Zhou ZX, Li Q, Huang H, et al. A microbial-enzymatic strategy for producing chondroitin sulfate glycosaminoglycans. Biotechnol Bioeng, 2018, 115(6): 1561–1570. DOI: 10.1002/bit.v115.6 |
| [46] | Grimshaw JPA, Stirnimann CU, Brozzo MS, et al. DsbL and DsbI form a specific dithiol oxidase system for periplasmic arylsulfate sulfotransferase in uropathogenic Escherichia coli. J Mol Biol, 2008, 380(4): 667–680. DOI: 10.1016/j.jmb.2008.05.031 |
| [47] | Grubb CD, Abel S. Glucosinolate metabolism and its control. Trends Plant Sci, 2006, 11(2): 89–100. DOI: 10.1016/j.tplants.2005.12.006 |
| [48] |
Wang JS, Gu HH, Yu HF, et al. Network of regulation and metabolism of indole glucosinolate in plants.
Acta Agric Zhejiangensis, 2012, 24(4): 739–747.
(in Chinese). 王建升, 顾宏辉, 虞慧芳, 等. 植物吲哚族芥子油苷的代谢调控网络. 浙江农业学报, 2012, 24(4): 739-747. DOI:10.3969/j.issn.1004-1524.2012.04.038 |
| [49] | Aziz M, Nadipalli RK, Xie XT, et al. Augmenting sulfur metabolism and herbivore defense in Arabidopsis by bacterial volatile signaling. Front Plant Sci, 2016, 7: 458. |
| [50] | Yatusevich R, Mugford SG, Matthewman C, et al. Genes of primary sulfate assimilation are part of the glucosinolate biosynthetic network in Arabidopsis thaliana. Plant J, 2010, 62(1): 1–11. DOI: 10.1111/j.1365-313X.2009.04118.x |
| [51] | Kang Z, Zhou ZX, Wang Y, et al. Bio-based strategies for producing glycosaminoglycans and their oligosaccharides. Trends Biotechnol, 2018, 36(8): 806–818. DOI: 10.1016/j.tibtech.2018.03.010 |
| [52] | Szajek AY, Chess E, Johansen K, et al. The US regulatory and pharmacopeia response to the global heparin contamination crisis. Nat Biotechnol, 2016, 34(6): 625–630. DOI: 10.1038/nbt.3606 |
| [53] | Zhang X, Pagadala V, Jester HM, et al. Chemoenzymatic synthesis of heparan sulfate and heparin oligosaccharides and NMR analysis: paving the way to a diverse library for glycobiologists. Chem Sci, 2017, 8(12): 7932–7940. DOI: 10.1039/C7SC03541A |
| [54] | Liu J, Linhardt RJ. Chemoenzymatic synthesis of heparan sulfate and heparin. Nat Prod Rep, 2014, 31(12): 1676–1685. DOI: 10.1039/C4NP00076E |
| [55] |
Li XY. Study on efficient preparation of several key enzymes and its mutienzymatic reaction engineering for heparin biosynthesis[D]. Zhejiang: Zhejiang University, 2016 (in Chinese). 李晓燕.生物酶法合成肝素的多种关键酶制备和酶反应工程研究[D].浙江: 浙江大学, 2016. |
| [56] | Mikami T, Kitagawa H. Biosynthesis and function of chondroitin sulfate. Biochim Biophys Acta, 2013, 1830(10): 4719–4733. DOI: 10.1016/j.bbagen.2013.06.006 |
| [57] | Habicher J, Haitina T, Eriksson I, et al. Chondroitin/ dermatan sulfate modification enzymes in zebrafish development. PLoS ONE, 2015, 10(3): e0121957. DOI: 10.1371/journal.pone.0121957 |
| [58] | Pomin VH. Keratan sulfate: an up-to-date review. Int J Biol Macromol, 2015, 72: 282–289. DOI: 10.1016/j.ijbiomac.2014.08.029 |
| [59] | Kuberan B, Lech MZ, Beeler DL, et al. Enzymatic synthesis of antithrombin Ⅲ-binding heparan sulfate pentasaccharide. Nat Biotechnol, 2003, 21(11): 1343–1346. DOI: 10.1038/nbt885 |
| [60] | Colley DG, Bustinduy AL, Secor WE, et al. Human schistosomiasis. Lancet, 2014, 383(9936): 2253–2264. DOI: 10.1016/S0140-6736(13)61949-2 |
| [61] | Carson JP, Ramm GA, Robinson MW, et al. Schistosome-induced fibrotic disease: the role of hepatic stellate cells. Trends Parasitol, 2018, 34(6): 524–540. DOI: 10.1016/j.pt.2018.02.005 |
| [62] | Anderson TJC, LoVerde PT, Le Clec'h W, et al. Genetic crosses and linkage mapping in schistosome parasites. Trends Parasitol, 2018, 34(11): 982–996. DOI: 10.1016/j.pt.2018.08.001 |
| [63] | Valentim CL, Cioli D, Chevalier FD, et al. Genetic and molecular basis of drug resistance and species-specific drug action in schistosome parasites. Science, 2013, 342(6164): 1385–1389. DOI: 10.1126/science.1243106 |
| [64] | Taylor AB, Roberts KM, Cao XH, et al. Structural and enzymatic insights into species-specific resistance to schistosome parasite drug therapy. J Biol Chem, 2017, 292(27): 11154–11164. DOI: 10.1074/jbc.M116.766527 |
| [65] | Chu LL, Dhakal D, Shin HJ, et al. Metabolic engineering of Escherichia coli for enhanced production of naringenin 7-sulfate and its biological activities. Front Microbiol, 2018, 9: 1671. DOI: 10.3389/fmicb.2018.01671 |
| [66] | Rivoal J, Hanson AD. Choline-O-sulfate biosynthesis in plants (identification and partial characterization of a salinity inducible choline sulfotransferase from species of limonium (plumbaginaceae). Plant Physiol, 1994, 106(3): 1187–1193. DOI: 10.1104/pp.106.3.1187 |
| [67] | Hirschmann F, Krause F, Papenbrock J. The multi-protein family of sulfotransferases in plants: composition, occurrence, substrate specificity, and functions. Front Plant Sci, 2014, 5: 556. |
| [68] | Guo Y, Hu BF, Huang H, et al. Estrogen sulfotransferase is an oxidative stress responsive gene that gender-specifically affects liver ischemia/ reperfusion injury. J Biol Chem, 2015, 290(23): 14754–14764. DOI: 10.1074/jbc.M115.642124 |
| [69] | Mungenast F, Aust S, Vergote I, et al. Clinical significance of the estrogen-modifying enzymes steroid sulfatase and estrogen sulfotransferase in epithelial ovarian cancer. Oncol Lett, 2017, 13(6): 4047–4054. DOI: 10.3892/ol.2017.5969 |
| [70] | Mueller JW, Idkowiak J, Gesteira TF, et al. Human DHEA sulfation requires direct interaction between PAPS synthase 2 and DHEA sulfotransferase SULT2A1. J Biol Chem, 2018, 293(25): 9724–9735. DOI: 10.1074/jbc.RA118.002248 |
| [71] | Foster PA, Mueller JW. Sulfation pathways: insights into steroid sulfation and desulfation pathways. J Mol Endocrinol, 2018, 61(2): T271–T283. DOI: 10.1530/JME-18-0086 |
| [72] | Teramoto T, Fujikawa Y, Kawaguchi Y, et al. Crystal structure of human tyrosylprotein sulfotransferase-2 reveals the mechanism of protein tyrosine sulfation reaction. Nat Commun, 2013, 4: 1572. DOI: 10.1038/ncomms2593 |
| [73] | Hartmann-Fatu C, Bayer P. Determinants of tyrosylprotein sulfation coding and substrate specificity of tyrosylprotein sulfotransferases in metazoans. Chem-biol Interact, 2016, 259: 17–22. DOI: 10.1016/j.cbi.2016.04.006 |
 2019, Vol. 35
2019, Vol. 35




