中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张雷, 雷林超, 张光亚, 李夏兰
- Zhang Lei, Lei Linchao, Zhang Guangya, Li Xialan
- 基于foldon介导的寡聚化以提高阿魏酸酯酶催化效率
- Oligomerization triggered by foldon to enhance the catalytic efficiency of feruloyl esterase
- 生物工程学报, 2019, 35(5): 816-826
- Chinese Journal of Biotechnology, 2019, 35(5): 816-826
- 10.13345/j.cjb.180458
-
文章历史
- Received: November 7, 2018
- Accepted: January 17, 2019
- Published: April 16, 2019
阿魏酸酯酶(Feruloyl esterase E.C 3.1.1.73,FAE)是羧酸酯酶的一个亚类,它能水解植物细胞壁中阿魏酸与多糖之间连接的酯键,释放阿魏酸[1-2]。FAE可以协同木质纤维降解酶,如木聚糖酶、纤维素酶及木质素酶,破坏木质纤维的致密网状结构,促进木质纤维降解[1-4]。但报道的FAE的催化性能有待提高,需对酶进行改造[5-7]。以定点突变和定向进化为代表的蛋白质工程技术已经成功地筛选了具有优化特性的酶,但这些方法在改造酶时仍存在困难,如突变体库的建立和大量筛选工作[8]。而通过固定化或化学修饰,有时也能提高酶的性能,但存在酶在固定化过程中会引起酶失活、首次固定化成本高、与大分子底物反应较困难等缺点[9]。
蛋白质的结构决定其功能,可通过蛋白质工程对蛋白质进行改造以获取蛋白质的新功能[10]。寡聚化是许多蛋白质自我联合成寡聚体以获得功能优势的一种常用方式[11]。寡聚化能够为目标酶提供多种功能优势,如提高热稳定性、pH耐受性、蛋白质分子结构稳定性及催化性能等[12-14]。Foldon是来源于T4噬菌体纤维蛋白C-末端,由27个氨基酸(GYIPEAPRDGQAYVRKDGEWVLLSTFL)组成,该结构域由3个相同亚基构成,每个亚基包含1个β-发夹结构[15]。通过基因融合,该结构域可与目标酶人工连接以改变其性质。几种工程蛋白的热力学稳定性,如短胶原纤维[16]、HIV1包膜糖蛋白[17],均已通过foldon结构域的附着而得到增强。由COMP和foldon诱导形成的寡聚体通常可导致热稳定性的提高[11, 18]。Can等[13]将foldon与抗冻蛋白进行连接后形成寡聚化同型抗冻蛋白且提高了冰晶表面结合的抗冻蛋白浓度,使其活力显著增加。本课题组在FAE的C-末端融合foldon,从而诱导酶自发形成三聚体,构成寡聚化阿魏酸酯酶,并对重组FAE的表达和其酶学性质进行研究。
1 材料与方法 1.1 主要材料 1.1.1 菌株与质粒大肠杆菌Escherichia coli DH5α购自生工生物工程(上海)股份有限公司;巴斯德毕赤酵母Pichia pastoris GS115、载体pPIC9K购自美国Invitrogen公司。
1.1.2 主要试剂和培养基限制性内切酶(SnaBⅠ、NotⅠ、SacⅠ)、T4 DNA连接酶购自美国Thermo Fisher公司;质粒抽提试剂盒、胶回收试剂盒购自生工生物工程(上海)股份有限公司;标准蛋白质Marker (10–170 kDa)、SDS-PAGE凝胶配制试剂盒、蛋白质快速银染试剂盒购自上海碧云天生物技术研究所;阿魏酸甲酯购自美国Sigma公司;其他试剂均为国产或进口分析纯产品。MD培养基、YPD种子培养基、BMGY种子培养基、BMMY诱导培养基按美国Invitrogen公司的毕赤酵母操作表达手册配制。
1.1.3 主要器材纳米激光粒度仪(英国Malvern仪器公司);200目铜网(中科镜仪有限公司);透射电子显微镜(日本电子株式社);超高效液相色谱仪(美国安捷伦科技有限公司)。
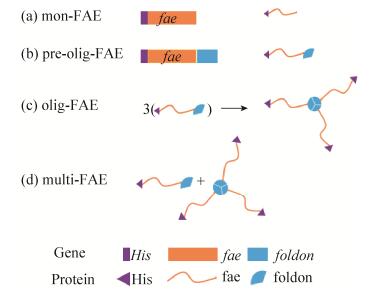
1.2 表达载体的构建在UniPort数据库中筛选酶活较高的FAEO42807,该FAE是从黑曲霉Aspergillus niger CBS120.49中分离得到的[1]。根据FAEO42807的氨基酸序列,以毕赤酵母标准密码子优化设计FAE的基因序列,并在基因序列N端设计His标签后,两端引入SnaBⅠ、NotⅠ酶切位点,化学合成基因序列His-fae。用SnaBⅠ、NotⅠ分别双酶切合成基因序列His-fae和质粒pPIC9K,胶回收双酶切片段后用T4 DNA连接酶连接并转化至E. coli DH5α感受态细胞。筛选出的阳性克隆子由Sangon测序,测序正确的重组质粒命名为pPIC9K/His-fae。另,将foldon基因与fae序列的C-末端直接融合,用相同方法构建重组质粒,命名为pPIC9K/His- fae-foldon。基因与蛋白质示意如图 1所示。
|
| 图 1 基因与蛋白质示意图 Fig. 1 A schematic of gene and corresponding protein structures. Olig-FAE is a trimer structure formed by the monomer pre-olig-FAE while protein is expressed; multi-FAE contains both mon-FAE and olig-FAE. |
| |
重组质粒用SacⅠ线性化,电转化至P. pastoris GS115感受态后,涂布于MD平板上筛选重组子,取MD平板上生长良好的菌落用牙签点种至YPD摇瓶中,过夜培养后提取其基因组DNA,利用通用引物5′AOX、3′AOX进行PCR鉴定。重组质粒的诱导表达参见毕赤酵母操作表达手册。4 ℃、10 000 r/min离心10 min,上清液即为粗酶液,粗酶液用镍层析柱进行纯化,纯化后用Bradford法测定蛋白质量浓度,SDS-PAGE分析纯化后的酶液。
1.4 重组FAE酶活力测定高效液相色谱法测定FAE酶活力[19]。空白对照为煮沸失活的酶液。酶活力单位为在25 ℃、pH 6.0的条件下1 min生成1 µmol阿魏酸所需的酶量为1个酶活力单位。
1.5 重组FAE酶学性质的研究以下实验所用的酶液,mon-FAE酶活力为(251±7.61) U/L,比活为(0.36±0.011) U/mg,multi-FAE酶活力(441±12.7) U/L,比活为(1.63±0.045) U/mg。
1.5.1 最适反应温度的测定取250 μL酶液保温5 min后,加入250 μL阿魏酸甲酯溶液(由pH值为6.0的Na2HPO4-柠檬酸缓冲溶液配制),在40–65 ℃中反应10 min,测定重组FAE的酶活力。以所测的最高酶活力为100%,计算相对酶活。
1.5.2 温度稳定性的测定将酶液置于30–50 ℃下保温12 h,每隔3 h取样测定残留酶活力,以保温0 h所测定的酶活力为100%,计算相对酶活。
1.5.3 最适反应pH值的测定取250 μL酶液于50 ℃保温5 min后,加入250 μL的阿魏酸甲酯溶液(0.2 mol/L的Na2HPO4-柠檬酸缓冲溶液调节pH值在3.0–8.0之间),50 ℃下反应10 min,测定重组FAE的酶活力。以所测的最高酶活力为100%,计算相对酶活。
1.5.4 pH值稳定性的测定将酶液置于0.2 mol/L、pH值为3.0–8.0的Na2HPO4-柠檬酸缓冲液中,50 ℃保温2 h,取250 μL保温酶液,向其加入250 μL阿魏酸甲酯溶液,50 ℃下反应10 min,测定重组FAE残留酶活力,以各pH值的0 h酶活力为100%,计算相对酶活。
1.5.5 金属离子对重组FAE酶活力的影响将酶液与含有10 mmol/L的各种金属离子(Na+,K+,Mn2+,Fe2+,Cu2+,Mg2+,Ca2+,Zn2+)的Na2HPO4-柠檬酸缓冲溶液(pH 6.0)混合,于50 ℃保温2 h,测定残留酶活,以不添加金属离子所测得的酶活力为100%,计算相对酶活。
1.5.6 动力学常数的测定米氏常数的测定:取8支试管,每管都加好200 g/L阿魏酸酯甲酯溶液和0.2 mol/L、pH 6.0的Na2HPO4-柠檬酸缓冲液,在25 ℃下预热3 min,用同一管酶液依次加样,依次在酶作用1–30 min时测定酶活力,然后算出酶活力与反应时间的比值,在一定时间内比值保持稳定,则在此时间内酶作用为一级反应,此时间即可确定为测Km值和Vmax的反应时间。用不同浓度的底物,在0.2 mol/L、pH 6.0的Na2HPO4-柠檬酸缓冲体系中,25 ℃下反应一定时间,测定酶活,计算相应的反应速度,利用米氏方程双倒数法求得Km值及Vmax。
1.6 重组FAE蛋白粒径的测定重组FAE的粒径大小由纳米激光粒度仪测定[20]。配置的参数:散射角173°、HeNe激光器633 nm、输出功率10 mW,使用内置温度将样品控制在特定温度(25–65 ℃),并在进行测定之前将样品在相应温度下稳定5 min;粒径大小为样品颗粒大小平均值。
1.7 重组FAE透射电子显微镜分析将5 µL适量浓度蛋白质样品滴加到碳支持膜涂覆的200目铜网格上并温育5 min,过量的蛋白质样品用滤纸进行印记干燥。网格用两个20 µL水洗涤并印记干燥,25 µL 2%乙酸铀酰滴30 s,印记干燥后使用透射电子显微镜在100 kV下观察样品,并使用CCD照相机记录电子显微照片。
2 结果与分析 2.1 重组FAE转化子的PCR鉴定与筛选分别以pPIC9K/His-fae和pPIC9K/His-fae- foldon的基因组为模板,用5′AOX1和3′AOX1引物进行PCR验证,PCR产物用1%的凝胶电泳检测,结果如图 2所示。以pPIC9K/His-fae转化子基因组为模板进行PCR,泳道1、2、4、6–8得到一条约800 bp的His-fae片段,显示目的基因His-fae已经成功整合到这些转化子基因组中,以pPIC9K/His-fae-foldon转化子基因组为模板进行PCR,泳道11、15–18得到一条约900 bp的His-fae-foldon片段,显示目的基因His-fae-foldon已经成功整合到这些转化子基因组中。

|
| 图 2 重组FAE转化子的PCR电泳图 Fig. 2 Electrophoresis map of the PCR analysis of recombinant FAE. Lane M: marker; lane 1–8: pPIC9K/His-fae transformants as template; lane 9: pPIC9K/His-fae plasmid as template; lane 10: sterile water as control; lane 11–18: pPIC9K/His-fae-foldon transformants as template; lane 19: pPIC9K/His-fae-foldon plasmid as template. |
| |
挑取上述阳性转化子,按照1.3进行培养和诱导表达,测定其酶活力,挑选实验中产酶活力最高的转化子。取其发酵上清液纯化后进行SDS-PAGE分析。对于pPCI9K/His-fae,如图 3A所示,pPCI9K/His-fae发酵上清液纯化后,40 kDa处得到一条清晰条带,而且几乎没有杂条带,初步说明mon-FAE得到成功表达。图 3A显示mon-FAE的表观相对分子质量明显大于其理论值(29.97 kDa),主要原因是FAE蛋白序列的N端有一个糖基化位点,而毕赤酵母表达外源蛋白时,会对其进行糖基化修饰,从而使相对分子质量增大[21]。对于pPCI9K/His-fae-foldon,如图 3B所示,pPCI9K/His-fae-foldon发酵上清液纯化后,SDS-PAGE结果显示的pre-olig-FAE表观相对分子质量明显大于其理论值(33.04 kDa);olig-FAE表观相对分子质量明显大于其理论值99.12 kDa,其原因与mon-FAE表观分子质量大于其理论值原因相同。且理论上,pPCI9K/His-fae-foldon上清发酵液应仅在三聚体位置即110 kDa处出现一条条带,而pPCI9K-/His-fae-foldon发酵上清液在单体(pre-olig-FAE) 45 kDa处和三聚体(olig-FAE) 110 kDa处均出现条带,原因可能是由于空间位阻效应,导致部分pre-olig-FAE未寡聚化,仍以pre-olig-FAE单体存在。

|
| 图 3 SDS-PAGE分析产物表达 Fig. 3 SDS-PAGE analysis of the recombinant FAE. (A) pPCI9K/His-fae. (B) pPCI9K/His-fae-foldon. |
| |
比较了mon-FAE和multi-FAE的酶活力及比活性(表 1),发现multi-FAE的酶活力和比活力相比mon-FAE有小幅度提高。这些结果表明foldon在FAE的C-末端融合有利于提高FAE的酶活力及比活力。
| Recombinant protein | Activity (U/L) | Total protein (mg/L) | Specific activity (U/mg) |
| Mon-FAE | 251±7.61 | 698±12.8 | 0.36±0.011 |
| Multi-FAE | 441±12.7 | 271±8.83 | 1.63±0.045 |
文献报道,foldon结构域与蛋白进行融合表达时,寡聚化的蛋白极易受到温度的影响,并在40 ℃开始发生少量解聚,当温度上升至60 ℃以上时,寡聚化结构蛋白完全解聚,当温度介于二者之间时,寡聚蛋白部分解聚,两种蛋白同时存在[22],本课题组也证明了此结果。在本实验过程中,为考察olig-FAE在不同温度条件下的解聚情况,分别将目标蛋白与蛋白上样缓冲液按5:1 (V/V)混合并在25 ℃、40 ℃、55 ℃、60 ℃、65 ℃、70 ℃、100 ℃条件下分别水浴5 min后进行SDS-PAGE分析,结果如图 4所示。结果表明环境温度为25 ℃时,45 kDa处的蛋白条带为由于空间位阻作用未形成寡聚化结构的pre-olig-FAE,且蛋白条带较细;110 kDa处为形成寡聚化结构的olig-FAE,该温度下,olig-FAE还未发生解聚。当温度为40 ℃时,45 kDa处的蛋白条带pre-olig-FAE相比于25 ℃时的蛋白条带较粗,说明olig-FAE开始发生部分解聚,且随着温度的升高,45 kDa处的蛋白条带pre-olig-FAE逐渐加粗,说明olig-FAE解聚量不断增加。在60 ℃及以上温度时110 kDa处的蛋白条带olig-FAE已完全消失,SDS-PAGE显示仅有一条45 kDa的条带,说明寡聚化酶完全解聚。但实验过程发现,解聚过程中在45 kDa单体位置有2条非常靠近的条带(图 4),因此,将经镍柱纯化后的浓度为1 mg/mL的酶液送样至上海中科新生命生物科技有限公司进行MOTIF/TOF质谱分析,结果显示仅在425 174.0出现一个峰,故判定图 4单体位置的两条非常靠近的条带为同一物质。

|
| 图 4 olig-FAE随温度变化聚集情况SDS-PAGE分析 Fig. 4 SDS-PAGE of olig-FAE aggregation as temperature change. M: protein molecular weight marker; lane 1: 25 ℃; lane 2: 40 ℃; lane 3: 55 ℃; lane 4: 60 ℃; lane 5: 65 ℃; lane 6: 70 ℃; lane 7: 100 ℃. |
| |
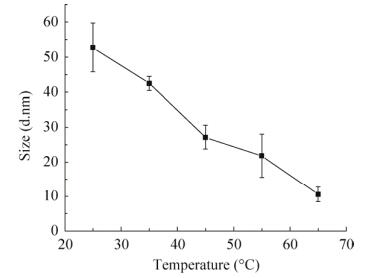
使用动态光散射(DLS)对不同温度时重组FAE的粒径进行了测定,结果显示在25 ℃时分别在6.57 d.nm处以及52.74 d.nm处有2个峰,且随温度的升高6.57 d.nm处的粒径出峰值变化不大,范围在6.5–8.5 d.nm之间,而52.74 d.nm处的粒径出峰值会随着温度的变化而逐渐变小,由于寡聚化结构foldon随温度的升高而逐渐解聚,平均粒径逐渐变小,所以判断该处为olig-FAE出峰处,olig-FAE随温度变化的粒径结果见图 5。从图中可以看出,随着温度升高,olig-FAE的平均粒径逐渐变小,且温度升高至65 ℃时,olig-FAE的平均粒径由25 ℃时的52.74 d.nm降低为10.62 d.nm,与mon-FAE所测得的平均粒径(9.54±2.12) d.nm相近,证明65 ℃时olig-FAE已全部解聚为单体结构。
|
| 图 5 温度对olig-FAE粒径的影响 Fig. 5 Effect of temperature on the particle size of olig-FAE. |
| |
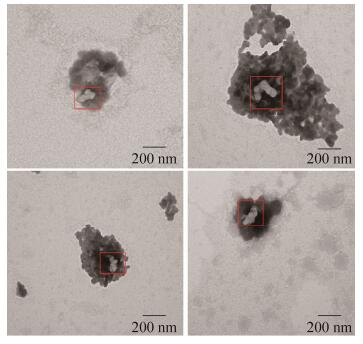
本实验还采用透射电子显微镜图像进一步探究在25 ℃时olig-FAE的蛋白结构性质,为了便于观察olig-FAE的蛋白结构,将样品稀释至0.15 mg/mL,电镜结果如图 6所示。图 6中显示在负染色液包裹范围中有接近球形形态的颗粒聚集在一起,形成寡聚化结构。
|
| 图 6 Olig-FAE的TEM图像 Fig. 6 TEM image of olig-FAE. |
| |
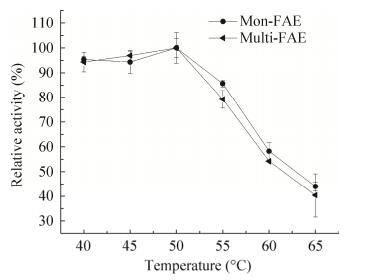
探究在40–65 ℃时酶反应的最适温度,结果如图 7所示。由图 7可知,mon-FAE与multi-FAE最适温度均为50 ℃,且50 ℃之前,两者的酶活均较高,相对酶活都在90%以上。超过50 ℃后,两者的酶活力下降均较快,可能是随着温度的升高,酶出现失活现象。
|
| 图 7 重组FAE的最适温度 Fig. 7 Effect of temperature on the activity of recombinant FAE. |
| |
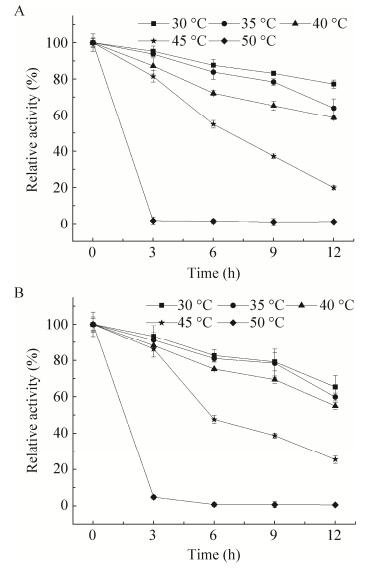
将重组FAE在30–55 ℃保温0、3、6、9、12 h后,测定其残余酶活,结果如图 8所示。mon-FAE及multi-FAE在30–55 ℃温度下,随着保温时间的延长,酶活力均逐渐下降。30–40 ℃保温12 h后,酶活力为初始酶的70%左右,且multi-FAE与mon-FAE酶活力相比较高一些,在40 ℃以后,multi-FAE及mon-FAE的酶活力均下降较快,且在50 ℃保温3 h后,基本丧失酶活。
|
| 图 8 重组FAE的温度稳定性 Fig. 8 Effect of temperature on the stability of recombinant FAE. (A) mon-FAE. (B) multi-FAE. |
| |
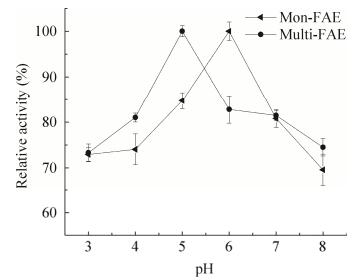
重组酶的最适pH结果如图 9所示。由图 9可知,mon-FAE的最适pH值为6.0;multi-FAE的最适pH值为5.0。重组酶的pH值稳定性结果如图 10所示。由图 10可知,pH值为3.0–6.0时,随着pH增大,mon-FAE的稳定性随之增大,pH值为6.0时,mon-FAE的pH稳定性最高,pH值大于6.0时,mon-FAE稳定性逐渐下降;pH值为3.0–5.0时,随着pH增大,multi-FAE的稳定性随之增大,pH值为5.0时,multi-FAE的pH稳定性最高,pH值大于5.0时,multi-FAE的稳定性逐渐下降。multi-FAE对pH值较敏感,这可能是因为pH值影响了FAE的寡聚化程度,从而影响了酶的稳定性[23]。
|
| 图 9 重组FAE的最适pH Fig. 9 Effect of pH value on the activity of recombinant FAE. |
| |

|
| 图 10 重组FAE的pH稳定性 Fig. 10 Effect of pH value on the stability of recombinant FAE. |
| |
金属离子对重组酶活力的影响见表 2,对于mon-FAE,Mn2+、Mg2+有促进作用,K+、Ca2+、Fe3+、Cu2+对其有一定的抑制作用,而Zn2+对其有显著的抑制作用。对于multi-FAE,Mn2+、Zn2+、Ca2+、Mg2+、Fe3+、Cu2+对其有促进作用,K+对其有一定的抑制作用。
| Metal ion (10 mmol/L) | Mon-FAE relatively activity (%) | Multi-FAE relatively activity (%) |
| Control | 100±1.73 | 100±0.91 |
| Mn2+ | 103±1.22 | 105±0.51 |
| Mg2+ | 103±3.27 | 107±2.96 |
| Zn2+ | 77.6±1.76 | 107±4.21 |
| K+ | 95.1±1.47 | 94.5±0.42 |
| Ca2+ | 91.7±2.31 | 103±1.98 |
| Fe3+ | 92.4±5.13 | 116±4.21 |
| Cu2+ | 90.1±5.14 | 105±1.71 |
由于olig-FAE随温度升高会逐渐解聚为pre-olig-FAE,故在25 ℃测定动力学常数,见表 3。其中multi-FAE的Km (1.03±0.02)与mon-FAE (4.55±0.07)相比较小,说明multi-FAE的底物亲和力较mon-FAE有所提高,且提高倍数为3.42倍;multi-FAE的kcat/Km (3.94±1.12)与mon-FAE (0.46±0.063)相比较大,说明multi-FAE的催化效率较mon-FAE有明显提高,且提高倍数为7.57倍;同时,multi-FAE的kcat、Vmax较mon-FAE均有所提高。其他研究人员的报道也存在类似的结果,例如,Yang等[24]将寡肽与碱性淀粉酶的N-末端融合,AmyK-p1的比活性和催化常数(kcat)分别增加4.1倍和3.5倍,他们认为提高催化效率和比活性的主要原因是由肽的活性部位周围诱导更大的灵活性,同时寡聚化酶与底物的“邻近效应”,使酶的活性中心部位更集中,其底物浓度也大大高于溶液中的底物浓度,从而提高其催化效率。
| Mon-FAE | Multi-FAE | |
| Vmax (mmol/(mL∙s)) | 3.45±0.14 | 5.79±0.37 |
| Km (mg/mL) | 4.55±0.07 | 1.03±0.02 |
| kcat (s–1) | 2.28±2.14 | 4.35±0.97 |
| kcat/Km (mg/(mL∙s)) | 0.46±0.63 | 3.94±1.12 |
阿魏酸酯酶可在工农业、食品制造、医药等领域都发挥巨大的作用。为了商业应用,已有很多提高FAE酶催化性能的报道,并与本文研究结果相比,结果见表 4。
| Molecular engineering methods | Km | kcat/Km | References |
| DNA shuffling and site-directed mutagenesis | − | Increase 12.8-folds | [25] |
| Site-directed mutagenesis | Decrease 0.62-folds | Increase 10-folds | [26] |
| Directed evolution | Decrease 0.39-folds | Increase 1.9-folds | [27] |
| Random mutagenesis | Decrease 0.73-folds | Increase 1.05-folds | [28] |
| Introduce an extra disulfide bridge | Increase 1.65-folds | Increase 0.58-folds | [29] |
| Immobilization | Similarly | Decrease 10-folds (kcat) (SiO2) | [30-31] |
| Increase 1.23-folds (kcat) (Fe3O4) | |||
| Protein oligomerization | Decrease 3.42-folds | Increase 7.57-folds | This paper |
文献[25]报道应用DNA改组策略和定向突变后,催化效率提高12.8倍。文献[26]报道应用PoPMuSiC算法预测氨基酸取代的折叠自由能变化,通过定点诱变取代4个氨基酸,双突变体D93G/S187F显示出kcat/Km增加了9倍。文献[28]报道进行两轮随机诱变获得热稳定突变体M6,M6的kcat/Km值提高1.9倍。文献[29]报道使用MODIP和DbD两种计算工具来预测蛋白质中可能存在的用于热稳定性改善的二硫键,引入额外的二硫桥对FAE实施改造,并使用分子动力学模拟来设计额外的二硫桥,选择一个残基对A126-N152突变为半胱氨酸。突变型AuFaeA的最适温度提高了6 ℃,半衰期在55 ℃和60 ℃分别提高12.5倍和10倍,催化效率(kcat/Km)与野生型AuFaeA相似。文献[30]报道利用介孔二氧化硅材料作为固定支持物,通过物理吸附将FAE固定在介孔二氧化硅上,并通过改变支持孔径、固定缓冲液和pH来优化固定条件,实现最大负载和最大活性,在10次酯交换反应后,保留了其20%以上的活性,Km不受固定化的影响,但是kcat减少了10倍。值得一提的是本文报道的基于foldon引导的寡聚化FAE 25 ℃半衰期提高55.5%,Km减小了3.42倍,催化效率提高了7.57倍。文献中这些方法虽在不同程度上提高了酶活性,但是定点诱变需要对酶结构和功能之间的关系有清楚的了解[32],定向进化需要一种简单而有效的高通量筛选方法[33],化学修饰可能通过改变活性位点中的活性构象或必需残基而引起酶活性的意外丧失[34]。
本文的方法条件温和,操作简单,并不需要从突变文库筛选目标酶。与mon-FAE相比,multi-FAE底物亲和力以及催化效率显著提高,这可能是由于寡聚化结构域foldon具有高固定浓度从而减少焓相互作用,加速了催化效率[16],且寡聚化酶分子的局部浓度增加,也加速了活性位点和底物的接触[35]。但是很遗憾,因为空间位阻作用,pre-olig-FAE未完全形成寡聚化,pre-olig-FAE与olig-FAE中都含有His标签,且两者的相对分子质量相差仅仅66 kDa,表达量也有限,本实验室目前还无法将olig-FAE纯化,但multi-FAE因含有寡聚化的olig-FAE,与mon-FAE相比,酶催化效率已有很大的提高。本课题组前期也将foldon结构域与分子量较小且结构简单的木聚糖酶和地衣多糖酶分别在原核表达系统进行融合表达,酶的kcat/Km分别提高了4.2倍和3.0倍[36]。此次,本课题组将foldon结构域与分子量较大且结构相对复杂的FAE在真核表达系统融合表达,结果表明,目标酶催化效率有很大提高,进一步表明该方法有望成为一个提高酶催化性能的通用且高效的方法,其操作简单,无需事先详细了解酶的3D结构,具有良好的应用前景,值得深入研究。
4 结论mon-FAE以及pre-olig-FAE在P. pastoris GS115中成功表达,且含有由foldon引发寡聚化FAE的multi-FAE较mon-FAE的比活、底物亲和力以及催化效率显著提高。与mon-FAE相比,multi-FAE的最适温度及温度稳定性未受到太大影响。综上所述,foldon结构域可提高FAE催化性能,是一种改造酶的简单高效方法。
| [1] | Faulds CB, Williamson G. Purification and characterization of a ferulic acid esterase (FAE-Ⅲ) from Aspergillus niger: specificity for the phenolic moiety and binding to microcrystalline cellulose. Microbiology, 1994, 140(4): 779–787. DOI: 10.1099/00221287-140-4-779 |
| [2] | de Vries R, Visser J. Aspergillus enzymes involved in degradation of plant cell wall polysaccharides. Microbiol Mol Biol Rev, 2001, 65(4): 497–522. DOI: 10.1128/MMBR.65.4.497-522.2001 |
| [3] | Faulds CB, Mandalari G, LoCurto R, et al. Arabinoxylan and mono- and dimeric ferulic acid release from brewer's grain and wheat bran by feruloyl esterases and glycosyl hydrolases from Humicola insolens. Appl Microbiol Biotechnol, 2004, 64(5): 644–650. DOI: 10.1007/s00253-003-1520-3 |
| [4] | Kroon PA, Faulds CB, Williamson G. Purification and characterization of a novel esterase induced by growth of Aspergillus niger on sugar-beet pulp. Biotechnol Appl Biochem, 2011, 23(3): 255–262. |
| [5] |
Chen YH, Li H, Zhang GY, et al. Expression of feruloyl esterase O42807 in Pichia pastoris GS115.
J Huaqiao Univ: Nat Sci, 2016, 37(2): 224–229.
(in Chinese). 陈云华, 李慧, 张光亚, 等. 阿魏酸酯酶O42807在毕赤酵母GS115中的表达. 华侨大学学报:自然科学版, 2016, 37(2): 224-229. |
| [6] |
Li B, Cai GL, Zhu DW, et al. Codon optimization of feruloyl esterase gene of Aspergillus niger and its high expression in Pichia pastoris.
Microbiology China, 2017, 44(5): 1065–1073.
(in Chinese). 李兵, 蔡国林, 朱德伟, 等. 黑曲霉阿魏酸酯酶基因密码子优化及在毕赤酵母中的高效表达. 微生物学通报, 2017, 44(5): 1065-1073. |
| [7] | Zhang SB, Zhai HC, Wang L, et al. Expression, purification and characterization of a feruloyl esterase A from Aspergillus flavus. Protein Expr Purif, 2013, 92(1): 36–40. DOI: 10.1016/j.pep.2013.08.009 |
| [8] |
Sun JT. Construction of Thermostable β-1, 3-1, 4-glucanase and its Enzymatic Properties[D]. Wuxi: Jiangnan University, 2012 (in Chinese). 孙军涛.耐热β-1, 3-1, 4-葡聚糖酶的构建及其酶学性质研究[D].无锡: 江南大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10295-1013129783.htm |
| [9] |
Liu T. Improving stability of enzyme through the use of extremophiles.
J Chem Ind Eng, 2009, 30(6): 26–29.
(in Chinese). 刘涛. 利用嗜极微生物提高酶的稳定性. 能源化工, 2009, 30(6): 26-29. DOI:10.3969/j.issn.1006-7906.2009.06.008 |
| [10] | Lutz S. Beyond directed evolution-semi-rational protein engineering and design. Curr Opin Biotechnol, 2010, 21(6): 734–743. DOI: 10.1016/j.copbio.2010.08.011 |
| [11] | Papanikolopoulou K, Forge V, Goeltz P, et al. Formation of highly stable chimeric trimers by fusion of an adenovirus fiber shaft fragment with the foldon domain of bacteriophage t4 fibritin. J Biol Chem, 2004, 279(10): 8991–8998. DOI: 10.1074/jbc.M311791200 |
| [12] | Yang HQ, Li JH, Shin HD, et al. Molecular engineering of industrial enzymes: recent advances and future prospects. Appl Microbiol Biotechnol, 2014, 98(1): 23–29. |
| [13] | Can ö, Holland NB. Utilizing avidity to improve antifreeze protein activity: a type Ⅲ antifreeze protein trimer exhibits increased thermal hysteresis activity. Biochemistry, 2013, 52(48): 8745–8752. DOI: 10.1021/bi401345b |
| [14] | Engel J, Kammerer RA. What are oligomerization domains good for?. Matrix Biol, 2000, 19(4): 283–288. DOI: 10.1016/S0945-053X(00)00075-5 |
| [15] | Tao YZ, Strelkov SV, Mesyanzhinov VV, et al. Structure of bacteriophage T4 fibritin: a segmented coiled coil and the role of the C-terminal domain. Structure, 1997, 5(6): 789–798. DOI: 10.1016/S0969-2126(97)00233-5 |
| [16] | Stetefeld J, Frank S, Jenny M, et al. Collagen stabilization at atomic level: crystal structure of designed (GlyProPro)10 foldon. Structure, 2003, 11(3): 339–346. DOI: 10.1016/S0969-2126(03)00025-X |
| [17] | Sissoëff L, Mousli M, England P, et al. Stable trimerization of recombinant rabies virus glycoprotein ectodomain is required for interaction with the p75NTR receptor. J General Virol, 2005, 86: 2543–2552. DOI: 10.1099/vir.0.81063-0 |
| [18] | Malashkevich VN, Kammerer RA, Efimov VP, et al. The crystal structure of a five-stranded coiled coil in COMP: a prototype ion channel. Science, 1996, 274(5288): 761–765. DOI: 10.1126/science.274.5288.761 |
| [19] |
Li XL, Fan YM, Fang BS. Purification and characterization of ferulic acid esterase from Penicillium citrinum.
Microbiology China, 2010, 50(8): 1058–1064.
(in Chinese). 李夏兰, 范韵敏, 方柏山. 来自桔青霉的阿魏酸酯酶的分离纯化、理化性质. 微生物学报, 2010, 50(8): 1058-1064. |
| [20] | Li CC, Zhang GY. The fusions of elastin-like polypeptides and xylanase self-assembled into insoluble active xylanase particles. J Biotechnol, 2014, 177: 60–66. DOI: 10.1016/j.jbiotec.2014.02.020 |
| [21] | Juge N, Williamson G, Puigserver A, et al. High-level production of recombinant Aspergillus niger cinnamoyl esterase (FAEA) in the methylotrophic yeast Pichia pastoris. FEMS Yeast Res, 2002, 1(2): 127–132. |
| [22] | Bhardwaj A, Walker-Kopp N, Wilkens S, et al. Foldon-guided self-assembly of ultra-stable protein fibers. Protein Sci, 2010, 17(9): 1475–1485. |
| [23] |
Fang Y, Ou SY, Zhang N. Preparation and enzymatic properties of ferulic acid esterase.
J Jinan Univ: Nat Scie Med Ed, 2012, 33(5): 481–485.
(in Chinese). 方园, 欧仕益, 张宁. 阿魏酸酯酶的制备及其酶学性质. 暨南大学学报:自然科学与医学版, 2012, 33(5): 481-485. |
| [24] | Yang HQ, Lu XY, Liu L, et al. Fusion of an oligopeptide to the N terminus of an alkaline α-Amylase from Alkalimonas amylolytica simultaneously improves the enzyme's catalytic efficiency, thermal stability, and resistance to oxidation. Appl Environ Microbiol, 2013, 79(9): 3049–3058. DOI: 10.1128/AEM.03785-12 |
| [25] | Li JJ, Pei XQ, Zhang SB, et al. Improving the thermostability of feruloyl esterase by DNA shuffling and site-directed mutagenesis. Process Biochem, 2015, 50(11): 1783–1787. DOI: 10.1016/j.procbio.2015.08.009 |
| [26] | Zhang SB, Wu ZL. Identification of amino acid residues responsible for increased thermostability of feruloyl esterase A from Aspergillus niger using the PoPMuSiC algorithm. Bioresour Technol, 2011, 102(2): 2093–2096. DOI: 10.1016/j.biortech.2010.08.019 |
| [27] | Cao LC, Chen R, Xie W, et al. Enhancing the thermostability of feruloyl esterase EstF27 by directed evolution and the underlying structural basis. J Agric Food Chem, 2015, 63(37): 8225–8233. DOI: 10.1021/acs.jafc.5b03424 |
| [28] | Zhang SB, Pei XQ, Wu ZL. Multiple amino acid substitutions significantly improve the thermostability of feruloyl esterase A from Aspergillus niger. Bioresource Technology, 2012, 117: 140–147. DOI: 10.1016/j.biortech.2012.04.042 |
| [29] | Yin X, Hu D, Li JF, et al. Contribution of disulfide bridges to the thermostability of a Type A feruloyl esterase from Aspergillus usamii. PLoS ONE, 2015, 10(5): e0126864. DOI: 10.1371/journal.pone.0126864 |
| [30] | Bonzom C, Schild L, Gustafsson H, et al. Feruloyl esterase immobilization in mesoporous silica particles and characterization in hydrolysis and transesterification. BMC Biochem, 2018, 19(1): 1. DOI: 10.1186/s12858-018-0091-y |
| [31] | He FM, Zhang S, Liu XL. Immobilization of feruloyl esterases on magnetic nanoparticles and its potential in production of ferulic acid. J Biosci Bioeng, 2015, 120(3): 330–334. DOI: 10.1016/j.jbiosc.2015.01.006 |
| [32] | Illanes A, Cauerhff A, Wilson L, et al. Recent trends in biocatalysis engineering. Bioresour Technol, 2012, 115: 48–57. DOI: 10.1016/j.biortech.2011.12.050 |
| [33] | Dalby PA. Strategy and success for the directed evolution of enzymes. Curr Opin Struct Biol, 2011, 21(4): 473–480. DOI: 10.1016/j.sbi.2011.05.003 |
| [34] | Pešić M, Božić N, López C, et al. Chemical modification of chloroperoxidase for enhanced stability and activity. Process Biochem, 2014, 49(9): 1472–1479. DOI: 10.1016/j.procbio.2014.05.025 |
| [35] | Luo TZ, Kiick K. Noncovalent modulation of the inverse temperature transition and self-assembly of elastin-b-collagen-like peptide bioconjugates. J Am Chem Soc, 2015, 137(49): 15362–15365. DOI: 10.1021/jacs.5b09941 |
| [36] | Wang XZ, Ge HH, Zhang DD, et al. Oligomerization triggered by foldon: a simple method to enhance the catalytic efficiency of lichenase and xylanase. BMC Biotechnol, 2017, 17(1): 57. DOI: 10.1186/s12896-017-0380-3 |
 2019, Vol. 35
2019, Vol. 35