中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王超玄, 孙航
- Wang Chaoxuan, Sun Hang
- 基因敲除小鼠技术的研究进展
- Progress in gene knockout mice
- 生物工程学报, 2019, 35(5): 784-794
- Chinese Journal of Biotechnology, 2019, 35(5): 784-794
- 10.13345/j.cjb.180417
-
文章历史
- Received: October 11, 2018
- Accepted: January 18, 2019
- Published: March 13, 2019
近年来人们的关注点逐渐转向阐明基因功能并寻找可能具有治疗价值的基因产物,实验动物在人类疾病机制的研究中发挥了重要作用。在模型生物中,小鼠模型在人类生物学和疾病的研究中有着特别的优势:1)小鼠是哺乳动物,其发育、生理、行为和疾病与人类有很多共同之处;2)几乎所有(99%)小鼠基因在人类中都有同源物;3)小鼠基因组通过胚胎干细胞(Embryonic stem cell,ESC)中的同源重组支持特定基因的定向诱变,从而允许基因有效且精确地改变。
20世纪80年代后期开发的敲除ESC和小鼠中特定基因的能力使得人们对某些基因与疾病之间的关系有了更加全面的认识。目前的敲除技术还可以在时间和空间上对敲除的基因进行有效的控制,借助生物遗传学的力量,为哺乳动物的生理和疾病研究提供支持。文中就目前已有的基因敲除小鼠的技术进行小结。
1 常规基因敲除小鼠技术基因敲除小鼠技术是指通过基因工程的方法使小鼠体内某种基因功能缺失的生物技术。它以基因靶向技术、小鼠ESC的分离和培养技术为基础。制作基因敲除小鼠主要有两种方法:基因打靶和基因捕获。基因打靶技术基于成功的ESC培养和体外同源重组,在研究基因敲除引起小鼠基因组中功能丧失及突变方面较为常用。第一次小鼠基因打靶实验于1989年完成,30年来基因打靶加速了对于基因功能的研究,并为生物学研究提供了重要生物资源。2007年,三位科学家Mario R. Capecchi、Oliver Smithies和Martin J. Evens因对基因打靶技术的重要研究而获得诺贝尔奖[1]。基因捕获是作为基因靶向技术的替代方法而开发出来,它是一种高通量的和随机突变的技术,尽管不像基因靶向技术那样特异,但是通过诱捕可以在短时间内制作大量基因敲除小鼠[2]。
1.1 基因打靶技术通过基因打靶进行遗传修饰有助于推动生物医学的研究,基因打靶的本质是通过同源重组(Homologous ecombination,HR)替换内源基因。HR定义为在DNA上的两个同源区域之间发生的基因组重排[3]。HR的生物学特征是修复双链DNA断裂(DSZBs),重新启动停滞的DNA复制叉和减数分裂重组[4],通过这种方式可以将基因定向破坏,称为敲除(Knockout,KO);或用作转运基因插入的停靠位点,称为敲入(Knockin,KI)。通过同源重组将位点特异性修饰引入ESC的基因组中称为基因靶向的过程,这一过程由电穿孔法将靶向载体引入ESC中实现[5]。
标准基因打靶载体含有两个与靶基因座位点同源的区域,称为“靶向臂”,位于选择性标记基因侧翼[6]。靶向载体也称之为DNA构建体,通常由3个基本单元组成:1) 5′端同源臂;2)阳性可选择基因标记,如新霉素抗性基因(Neo)或潮霉素抗性基因(Hyg)等;3) 3′端同源臂。转染的靶向载体可以随机插入基因组中,或通过5′端和3′端同源臂确定的同源重组整合。通过在含有新霉素或其他适当的抗生素的培养基中培养ESC,可以正确选择成功转染的细胞。如果阳性选择标记基因侧翼为LoxP或重组翻转酶(Lippase,FLP)位点,则可以通过在重组ESC中表达Cre酶或FLP从ESC或转基因小鼠中的靶向基因座中去除它,或通过嵌合体小鼠与表达Cre或FLP的转基因小鼠杂交去除[7-8]。
影响HR效率的主要因素有两个。第一,在培养皿中使用ESC代替整个生物进行基因靶向选择。第二,基因靶向选择包括两个主要步骤:1)鉴定基因组中包含有共定位靶向载体的ESC,这是通过设计新霉素抗性实现的,新霉素抗性基因包括靶向载体的同源区域;2)鉴定通过同源重组发生靶向载体掺入的ESC,即通过替换内源基因,插入随机基因座,这一步通过构建同源区域下游的胸苷激酶盒实现[9]。因此,这种双重选择方案有助于研究者快速有效地鉴定内源基因被靶向载体替换的ESC。因为靶向载体含有非阳性序列,例如新霉素盒,位于目的基因的重要外显子中间,当靶向载体取代了内源基因,或者在编码区上游或上下游都具有终止密码子时,蛋白质不会发生表达,或由于蛋白质结构发生改变导致丧失其生物学功能。因此,用这种技术诱导的突变被称为失效突变,并且由于转基因小鼠携带这种突变,称为敲除或失效突变小鼠[10]。
人们通常使用基因打靶技术将目的基因敲除,根据其作用方式不同可分为条件基因敲除法、诱导基因敲除法等,这些方法都是建立在DNA同源重组技术原理之上的,下文将对其进行阐述。
1.2 基因捕获技术1989年Gossler等发表了第一篇基因捕获论文,目的是识别小鼠中控制胚胎发育的基因[11]。在这篇文章中他们构建了一个可以监测整合位点附近内源基因表达的载体,该载体被称为“捕获载体”。1991年,Friedrich和Soriano报道了一种改进的启动子捕获系统[12],他们使用具有新型报告基因的质粒和逆转录病毒捕获载体β-geo,β-geo的作用是编码具有β-半乳糖苷酶和新霉素磷酸转移酶活性的融合蛋白,来自β-geo基因的产物能够在ESC培养物中进行药物选择并使被捕获基因的组织定位可视化[13]。随后Niwa和Wurst等使用多聚腺苷酸化(polyA)捕获载体的方法制作了一种用于筛选小鼠发育调控基因的大规模基因捕获技术[14]。此后大规模的基因捕获技术迅速扩展到世界范围。
诱捕基因的技术大致分为两种:启动子捕获和polyA捕获[14]。在启动子捕获中,捕获载体使用具有连接到剪接受体下游启动子的抗药性基因,当捕获载体插入ESC的表达基因中时抗性基因将随着启动子活性表达,抗药性克隆通常反映基因捕获,使得该方法对于筛选发生基因捕获的ESC非常有效。有研究显示可以将捕获载体插入ESC的非表达基因中,然而这些ESC不会具有耐药性,导致无法分离这些ESC。在polyA捕获中抗药性基因具有启动子但不具有polyA序列,因此只有当捕获载体可以表达小鼠内源基因的polyA添加序列时ESC才具有耐药性。该方法的优点是可以捕获ESC中的非表达基因,缺点是polyA捕获载体的整合位点集中在捕获基因的末端内含子中。Shigeoka等提出的研究证据表明,这种显著的滞后效应是由于polyA捕获过程中引起的mRNA降解,又被称为无义介导的mRNA衰变(Nonsense-mediated mRNA decay,NMD)[15],为了克服这个缺点他们开发了一种新的polyA捕获策略,即尿激酶型纤溶酶原激活物(Urokinase plasminogen activator,UPA)捕获,UPA捕获可以抑制选择标记mRNA的NMD并能够捕获转录沉默基因而没有载体整合位点的偏移。由于产生了捕获基因和药物抗性基因之间的融合mRNA,因此在两种方法中捕获的基因均容易被鉴定。在启动子捕获中,位于载体插入位点上游的外显子信息可以通过5′-RACE获得。在polyA捕获中,位于载体插入位点下游的外显子信息可通过3′-RACE获得[16]。
尽管质粒载体可以通过电穿孔等方法递送到ESC中,但经常会出现基因捕获载体的多拷贝插入、多联体、缺失以及整合位点的重排。相反,逆转录病毒载体整合到小鼠基因组中不会引起宿主侧翼序列的重排。逆转录病毒基因捕获的另一个优点是逆转录病毒介导的整合多发生在基因5′末端的DNaseⅠ超敏性位点附近,这增加了产生无效等位基因的频率。Friedrich和Soriano展示了用于基因捕获的两种载体,分别是质粒载体(SAβ-geo)和逆转录病毒载体(ROSAβ-geo)[17]。因为包含多腺苷酸化信号序列以终止基因转录,所以基因捕获元件以反向病毒转录的方式插入长末端重复序列(Long terminal repeat,LTR)之间,以避免干扰病毒包装。ROSAβgeo26 (RASA26)捕获小鼠系在胚胎发育过程中显示出报告基因的普遍表达,因此它可用作嵌合体实验中的标记物[18]。
基因捕获技术敲除基因的本质是以报告基因为诱饵来捕获基因,其基本过程是在基因组中插入一段含有报告基因的DNA载体以产生内源基因的失活突变,通过观察报告基因的表达提示已产生插入突变。通过筛选得到的插入突变的ESC克隆经囊胚注射转化为基因突变动物模型,每一种ESC克隆中含有不同的突变基因,在短期内可建立大量含不同基因突变的ESC克隆库。突变基因序列可通过基于PCR的一些方法鉴定,同时还可能发现一些在体内表达或不表达的新基因。
2 条件基因敲除小鼠技术条件基因敲除技术是指在小鼠发育的特定阶段或特定细胞组织类型中将目的基因进行敲除的技术。其本质是基于常规基因敲除技术,通过重组酶Cre对靶位点进行特异性重组,可以对小鼠的基因组修饰进行时间和空间上的调控[19]。自1994年GU等研制的第一例基于Cre/loxP系统的组织特异性基因敲除小鼠问世以来,条件基因敲除技术迅速取代传统的完全基因敲除技术成为主流[20]。
2.1 Cre/loxP系统Cre重组酶来自噬菌体P1,大小约为38 kDa。它可以作用于两个LoxP位点之间使其发生特异性的同源重组。LoxP序列长约34 bp,由两个13 bp的反向重复序列组成,中间被8 bp的非回文序列分隔开,这段序列决定了整个LoxP位点的方向[21]。Cre重组酶无法切除相反方向的LoxP位点之间的DNA序列,而位于同一方向的LoxP位点之间的DNA序列可以被切除。
Cre/loxP系统已成为生物遗传操作的有力工具,几乎所有感兴趣的DNA序列都可以使用两端插入LoxP位点的方式来删除[22]。此外Cre重组酶的条件性(时间或空间控制)表达使得能够保证在特定部位(例如在特定细胞类型或组织中)以及特定时间(在小鼠细胞及组织发育的特定阶段)删除DNA序列。对于条件位点特异性的基因组修饰通常需要两种小鼠:第一种是将具有目的DNA序列的小鼠侧翼插入LoxP位点即Floxed小鼠。第二种是Cre重组酶转基因小鼠,Cre重组酶在启动子的控制下瞬时表达,这些启动子仅在特定细胞类型或组织中表达活性,或在细胞及组织的特定发育阶段具有活性。当使用Cre转基因小鼠与Floxed小鼠交配时,插入floxed的DNA序列在表达Cre的特定细胞类型或组织中被删除[23]。
Cre/loxP系统不仅可以删除目的基因,还可以特异性地激活目的基因表达。例如通过在目的基因两端插入介导转录停滞的多聚腺苷酸化信号序列即LoxP序列使目的基因呈现出静止状态[24],再与Cre转基因小鼠交配或将Cre递送到floxed转基因小鼠中,通过Cre介导的切除效应除去LoxP序列,激活特定细胞类型或组织中的基因表达。这种Cre/loxP介导的基因活化可以避免该基因在小鼠胚胎发生过程中的有害影响或诱导针对转基因小鼠的免疫耐受性,例如通过静脉内注射Cre腺病毒产生的条件性表达人丙型肝炎病毒(HCV)的转基因小鼠,用以研究HCV感染的免疫应答和发病机制[25]。
另一种重组系统是酵母FLP/FLP识别靶标(FLP recombination target,FRT),其机制与Cre/ loxP重组系统相同,可以作为Cre/loxP的替代工具[26]。此外,两种重组系统的组合可以显着增加小鼠条件基因敲除的操作潜力[27]。
2.2 CRISPR/Cas9系统CRISPR/Cas9系统由CRISPR基因和Cas9核酸内切酶组成,CRISPR首次发现于1987年,是一种包含短重复序列(20–50 bp)的DNA片段[28],CRISPR基因存在于超过40%的细菌和90%的古细菌的基因组中。最近发现一组名为Cas (或称CRISPR相关基因)的基因与CRISPR密切相关[29-30]。Cas基因编码一种核酸酶及解旋酶蛋白,它可以解开和切割DNA序列。目前发现CRISPR/Cas有6种(Ⅰ–Ⅵ)类型,其中Ⅱ型由3种成分组成:靶特异性CRISPR衍生RNA (crRNA)、靶独立反式激活RNA (tracrRNA)和Cas9核酸酶,它们广泛地应用于基因工程中[31]。
CRISPR/Cas9系统的基本原理是:首先crRNA将Cas9复合物引导至靶序列,tracrRNA与crRNA结合并与Cas9核酸酶形成核糖核蛋白复合物,然后Cas9以靶向方式切割染色体DNA,产生位点特异性DNA双链断裂(Double-strand breaks,DSB)。通过HR或非同源末端连接(Non-homologous end joining,NHEJ)的内源性DNA修复系统可以在细胞中有效修复这些DSB[32]。crRNA和tracrRNA的基础部分可以连接形成单链指导RNA (sgRNA),sgRNA的碱基可以与靶标DNA配对编程靶向目的序列[33],唯一的限制是sgRNA结合位点必须与短DNA基序相邻,称之为原型间隔相邻基序(Protospacer adjacent motif,PAM)[34]。在PAM处人工设计sgRNA,sgRNA可以与目的基因的靶序列特异性结合;Cas9核酸酶在sgRNA引导下对靶序列进行切割,导致DNA双链断裂。通过HR、NHEJ等机制对切断的双链进行修复,在断裂处插入异常碱基或使碱基缺失,导致移码突变,最终实现目的基因敲除(图 1)[35-36]。

|
| 图 1 CRISPR/Cas9介导的基因修饰流程图 Fig. 1 Genetic modification flow chart by CRISPR/Cas9. |
| |
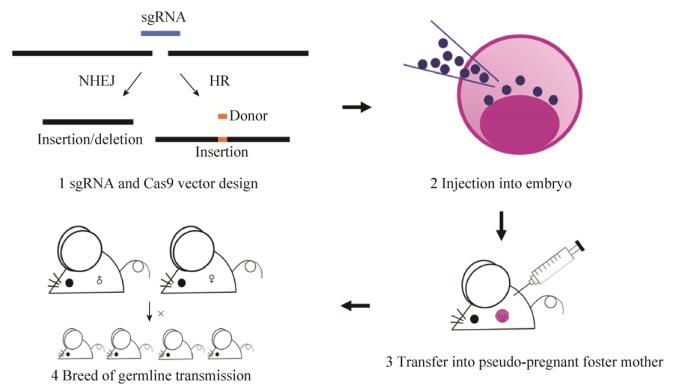
CRISPR/Cas9技术编辑胚胎基因组主要包括3个步骤:1)从超排卵的雌性中分离受精卵;2)将sgRNA和Cas9 mRNA递送到受精卵中;3)随后将胚胎转移到假孕动物中以产生F0代(图 2)[37]。2013年Jaenisch实验室应用CRISPR/Cas9技术成功构建了第一个基因敲除小鼠,研究者将Tet1和Tet2 sgRNA与Cas9 mRNA一起注入受精卵,产生的基因敲除小鼠中这两种基因的突变率达到80%[38]。此项研究表明CRISPR/Cas9系统是一种快速、便捷和有效的生产敲除小鼠的方法,为转基因动物的产生提供了新的技术平台[39]。迄今为止,该技术已成功应用于多种生物中,包括大鼠[40]、猪[41]、山羊[42]、兔子[43]、狗[44]、猴子[45]和人类胚胎[46]。Cas9/sgRNA指导的基因编辑通过在单链寡核苷酸模板的存在下将Cas9 mRNA和sgRNA共注射到受精卵中可以实现特异和精确的基因组编辑,包括敲除、敲入和基因修复。例如Yang等通过共注射Cas9、sgRNA和相应的基因DNA载体至受精卵中,在nanog、sox2和oct4基因中产生携带标签或荧光报告构建体的小鼠[47]。通常情况下,基因组编辑中涉及大片段的DNA插入、缺失或倒位时都存在效率低下的问题,片段越大重组效率越低[48],而Cas9系统由于具有同一染色体中两个不同靶向基因座的sgRNA可以显著提高重组效率,已被用于高效处理基因组中大片段的DNA。例如Fujii等通过将同一染色体中不同基因座的两个sgRNA注射到受精卵中,产生大片段(约10 kb)基因组修饰的小鼠,达到了33%的敲除效率[49]。此外Zhang等的研究表明Cas9系统可以实现大片段的DNA缺失和插入,他们通过将两个环状质粒共注射到受精卵中实现了大小为65 kb的Dip2a基因缺失和大小为5 kb的lacZ报告基因插入[50]。因此,CRISPR/ Cas9系统与两种sgRNA一起使用,是用于大基因组DNA片段操作的简单且有效的方法,可以加速动物模型的产生,便于研究疾病的机制和寻找治疗靶点。
|
| 图 2 CRISPR/Cas9技术构建基因敲除小鼠 Fig. 2 Gene knockout mice by CRISPR/Cas9 technology. |
| |
CRISPR介导的基因组编辑可以用于纠正与遗传性疾病相关的基因突变。Wu等首次报道了使用CRISPR/Cas9系统进行基因修复[51],他们选择了一种由Crygc基因突变引起的显性白内障疾病的小鼠模型,在3号外显子中造成1 bp的缺失,引起第76位氨基酸的终止密码子和截短的γC-晶状体蛋白的产生,导致纯合子和杂合子小鼠白内障。为了纠正Crygc基因的突变,研究者将突变等位基因的Cas9 mRNA和sgRNA共注射到受精卵中,内源等位基因或外源提供的模板通过HR进行校正以达到基因修复的目的[51]。杜氏肌营养不良症(Duchenne muscular dystrophy,DMD)是一种遗传性疾病,其特征是进行性肌肉无力和寿命缩短,目前尚无有效的治疗方法。DMD的分子基础是肌营养不良蛋白基因的23号外显子中的C至T点突变,其导致骨骼肌蛋白的完全缺失,而将Cas9 mRNA、sgRNA-DMD和外源单链寡核苷酸模板共注射到受精卵中,可以通过mdx小鼠种系中的HR修复来校正DMD的点突变[52]。这些进展表明我们距离CRISPR/Cas9最终应用于人类基因治疗并不遥远,然而Cas9/sgRNA是否可用于治疗多基因疾病或染色体结构变异需要进一步研究。
3 其他基因敲除方法 3.1 ZFN技术锌指核酸酶(Zinc finger nucleases,ZFN)是与DNA的多聚体锌指蛋白结构域相结合的工程化蛋白质,其中能够特异性结合三联体DNA序列的单个锌指基序连接在一起,形成具有核酸酶活性的限制性内切酶FokⅠ[53]。锌指结构域的可塑性允许研究者对ZFN进行设计,特异性则可以与目的序列特异性结合[54-55]。核酸酶结构域也可以用于切割靶序列,但是核酸酶结构域必须形成二聚体才可以切割双链DNA,所以需要两个ZFN来定位特定序列。当引入细胞时,两个ZFN与它们各自的结合位点结合,使它们的核酸酶结构域相互作用并在染色体中形成位点特异性双链断裂(Double-strand break,DSB),随后使用高度保守的同源依赖性修复(Homology- dependent repair,HDR)或非同源末端连接(Non-homologous end joining,NHEJ) DNA修复途径修复该断裂[53]。在HDR的情况下,序列可以被精确修复并且ZFN异二聚体可以在修复的靶序列上重新形成并重新切割。由于NHEJ介导的修复不如HDR准确,它偶尔会导致核苷酸的丢失或添加从而导致突变[56],这种突变通常导致基因编码序列中发生移码现象从而形成截短的或无义的肽链。从果蝇的开拓性实验开始[57],工程化的ZFN已被用于在来自多个物种的多种细胞和胚胎中产生位点特异性突变[58-59]。将ZFN技术初步应用于大鼠实验后研究者发现在基因中产生特异性突变位点时体现出惊人的高效率,通过将体外转录的ZFN编码核酸与单细胞胚胎组合,ZFN可用于在大鼠中产生可遗传的、位点特异性的靶向突变[60]。
ZFN基因敲除策略主要有3个优点。首先该技术非常迅速,整个过程仅需要几个月。其次它在所有菌株中都有效果[61-62]。第三,它不会导致外源DNA的掺入。ZFN当前的一个主要限制是需要从现有文库组装ZFN的序列,这可能妨碍设计的选择性。另一个限制是潜在的脱靶效应,其中ZFN在非靶向的基因座处引起DSB和突变。在ZFNs应用于斑马鱼中时出现了这种效应[63],通过回交可以减轻罕见脱靶事件的潜在影响。
ZFN可以与目的基因位点进行特异性结合,在核酸酶的作用下切割靶位点形成DSB,随后通过HDR或NHEJ进行自我修复来实现靶基因的删除与插入。由于ZFN技术可以通过多种重组方法进行切除后的DNA修复,与传统的基因敲除技术相比ZFN技术的敲除效率大大提升,但是由于ZFN技术存在脱靶这一亟待解决的问题,ZFN技术还不能完全取代传统的基因敲除小鼠技术。
3.2 TALEN技术与ZFN类似,另一种称为转录激活因子样效应核酸酶(Transcription activator-like effector nucleases,TALENs)的嵌合核酸酶系统已成为基因工程的另一个优秀工具。TAL (Transcription activator-like,TAL)的DNA结合域的结构特征非常典型,包括15.5–19.5个重复单元串联排列,每个重复单元包含34个高度保守的氨基酸,最后一个单元通常仅有20个氨基酸被认为是半个复合单元[64]。与ZFN一样,TAL存在与FokⅠ核酸内切酶结合的DNA结合基序[65-66],由特定的TAL (Transcription activator-like,TAL)与FokⅠ内切酶结合形成TALENs,Fok Ⅰ必须以二聚体的形式存在才具有活性[67],所以TALENs由两个活跃的单体组成,分别结合在两个靶位点,形成二聚体进行位点特异性切割。被TALENs切割产生的DSB可以通过NHEJ和HD两种方式修复。但是,TALENs与ZFN的不同之处在于如何组装DNA结合域(DNA-binding domain,DBD),与识别3个碱基对的ZFN不同,每个TAL的效应子模块与单个碱基对结合[65-66],TAL效应子DNA结合基序天然存在于植物病原细菌分泌的蛋白质中,每个效应子模块由34个氨基酸组成,残基12和13可以被DNA识别,也称为重复序列可变的双氨基酸残基(Repeat-variable di-residues,RVDs),它决定相应重复单元识别单个核苷酸的特异性。通过基于目标序列选择RVD,可以更容易地组装TALENs集合[68-69]。研究者可以很容易地从TALENs组文库收集TAL组合,这个文库目前已组装了18 700个人蛋白质编码基因[70]。易于组装、易于基因打靶的特点使TALENs技术成为基因工程应用的有力选择[68, 71]。到目前为止,已有研究者成功地将mRNA直接注射到猪和牛的一个细胞胚胎的细胞质中,证明了基因的靶向性。将TALENs递送到成纤维细胞中,在将近10%的细胞中产生双等位修饰并且靶向定位于同一染色体的两对TALENs的共转染能够产生大量的染色体缺失或倒位[72]。TALENs的一个主要问题是在脱靶位点的潜在分裂,使用特异性异二聚体FokⅠ核酸酶与TALENs的合理设计相结合可以缓解这些问题[73]。
TALENs技术由于操作的便捷性及较高的基因靶向特异性使得它成为基因工程的有力选择,迄今为止TALENs技术已被应用于不同物种的基因组编辑中,例如小鼠、酵母、水稻和玉米等[74]。与ZFN技术相比,TALENs技术可以更加特异的识别目的基因并且不受其上下游序列的影响,但是与ZFN技术一样由于存在脱靶效应使得TALENs技术还不能完全取代传统的基因敲除技术。
4 结语小鼠基因敲除技术提供了一种阐明生物体内基因功能的有力手段。目前为止,由基因敲除小鼠构建的疾病模型已有接近100种,通过对模型的建立和分析研究使得人们对部分疾病有了更加系统的认识并由此寻找新的治疗靶点。基因敲除小鼠技术的建立是生命科学领域内一项伟大的发明。
| [1] | Mak TW. Gene targeting in embryonic stem cells scores a knockout in Stockholm. Cell, 2007, 131(6): 1027–1031. DOI: 10.1016/j.cell.2007.11.033 |
| [2] | Araki M, Araki K, Yamamura KI. International Gene Trap Project: towards gene-driven saturation mutagenesis in mice. Curr Pharm Biotechnol, 2009, 10(2): 221–229. DOI: 10.2174/138920109787315006 |
| [3] | Capecchi MR. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet, 2005, 6(6): 507–512. DOI: 10.1038/nrg1619 |
| [4] | Ruff P, Koh KD, Keskin H, et al. Aptamer-guided gene targeting in yeast and human cells. Nucleic Acids Res, 2014, 42(7): e61. DOI: 10.1093/nar/gku101 |
| [5] | Qin WN, Dion SL, Kutny PM, et al. Efficient CRISPR/Cas9-mediated genome editing in mice by zygote electroporation of nuclease. Genetics, 2015, 200(2): 423–430. DOI: 10.1534/genetics.115.176594 |
| [6] | Yamazoe M, Sonoda E, Hochegger H, et al. Reverse genetic studies of the DNA damage response in the chicken B lymphocyte line DT40. DNA Repair, 2004, 3(8/9): 1175–1185. |
| [7] | Meier ID, Bernreuther C, Tilling T, et al. Short DNA sequences inserted for gene targeting can accidentally interfere with off-target gene expression. FASEB J, 2010, 24(6): 1714–1724. DOI: 10.1096/fj.09-140749 |
| [8] | Revell PA, Grossman WJ, Thomas DA, et al. Granzyme B and the downstream granzymes C and/or F are important for cytotoxic lymphocyte functions. J Immunol, 2005, 174(4): 2124–2131. DOI: 10.4049/jimmunol.174.4.2124 |
| [9] | Melton DW. Gene-targeting strategies//Clarke AR. Transgenesis Techniques: Principles and Protocols. Totowa, NJ: Springer, 2002: 151-173. |
| [10] | Skarnes WC, Rosen B, West AP, et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nat, 2011, 474(7351): 337. DOI: 10.1038/nature10163 |
| [11] | Gossler A, Joyner AL, Rossant J, et al. Mouse embryonic stem cells and reporter constructs to detect developmentally regulated genes. Science, 1989, 244(4903): 463–465. DOI: 10.1126/science.2497519 |
| [12] | Friedrich G, Soriano P. Promoter traps in embryonic stem cells: a genetic screen to identify and mutate developmental genes in mice. Genes Dev, 1991, 5(9): 1513–1523. DOI: 10.1101/gad.5.9.1513 |
| [13] | Skarnes WC, Rosen B, West AP, et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature, 2011, 474(7351): 337–342. DOI: 10.1038/nature10163 |
| [14] | Stanford WL, Cohn JB, Cordes SP. Gene-trap mutagenesis: past, present and beyond. Nat Rev Genet, 2001, 2(10): 756–768. DOI: 10.1038/35093548 |
| [15] | Shigeoka T, Kawaichi M, Ishida Y. Suppression of nonsense-mediated mRNA decay permits unbiased gene trapping in mouse embryonic stem cells. Nucleic Acids Res, 2005, 33(2): e20. DOI: 10.1093/nar/gni022 |
| [16] | Freeman LA. Cloning full-length transcripts and transcript variants using 5′ and 3′ RACE//Freeman LA. Lipoproteins and Cardiovascular Disease: Methods and Protocols. Totowa, NJ: Humana Press, 2013: 3-17. |
| [17] | Friedrich G, Soriano P. Promoter traps in embryonic stem cells: a genetic screen to identify and mutate developmental genes in mice. Genes Dev, 1991, 5(9): 1513–1523. DOI: 10.1101/gad.5.9.1513 |
| [18] | Srinivas S, Watanabe T, Lin CS, et al. Cre reporter strains produced by targeted insertion of EYFP and ECFP into the ROSA26 locus. BMC Dev Biol, 2001, 1: 4. DOI: 10.1186/1471-213X-1-4 |
| [19] | Schmidt-Supprian M, Rajewsky K. Vagaries of conditional gene targeting. Nat Immunol, 2007, 8(7): 665–668. DOI: 10.1038/ni0707-665 |
| [20] | Gu H, Marth JD, Orban PC, et al. Deletion of a DNA polymerase beta gene segment in T cells using cell type-specific gene targeting. Science, 1994, 265(5168): 103–106. DOI: 10.1126/science.8016642 |
| [21] | Kühn R, Torres RM. Cre/loxP recombination system and gene targeting. Methods Mol Biol, 2002, 180: 175–204. |
| [22] | McLellan MA, Rosenthal NA, Pinto AR. Cre-loxP-Mediated recombination: general principles and experimental considerations. Curr Protoc Mouse Biol, 2017, 7(1): 1–12. DOI: 10.1002/9780470942390.2017.7.issue-1 |
| [23] | Kawano F, Okazaki R, Yazawa M, et al. A photoactivatable Cre-loxP recombination system for optogenetic genome engineering. Nat Chem Biol, 2016, 12(12): 1059. DOI: 10.1038/nchembio.2205 |
| [24] | Nagy A. Cre recombinase: the universal reagent for genome tailoring. Genesis, 2000, 26(2): 99–109. DOI: 10.1002/(ISSN)1526-968X |
| [25] | Wakita T, Taya C, Katsume A, et al. Efficient conditional transgene expression in hepatitis C virus cDNA transgenic mice mediated by the Cre/loxP system. J Biol Chem, 1998, 273(15): 9001–9006. DOI: 10.1074/jbc.273.15.9001 |
| [26] | Chen ZY, Ren XD, Xu XJ, et al. Genetic engineering of human embryonic stem cells for precise cell fate tracing during human lineage development. Stem Cell Reports, 2018, 11(5): 1257–1271. DOI: 10.1016/j.stemcr.2018.09.014 |
| [27] | Rodríguez CI, Buchholz F, Galloway J, et al. High-efficiency deleter mice show that FLPe is an alternative to Cre-loxP. Nat Genet, 2000, 25(2): 139–140. DOI: 10.1038/75973 |
| [28] | Mojica FJM, Ferrer C, Juez G, et al. Long stretches of short tandem repeats are present in the largest replicons of the Archaea Haloferax mediterranei and Haloferax volcanii and could be involved in replicon partitioning. Mol Microbiol, 1995, 17(1): 85–93. DOI: 10.1111/mmi.1995.17.issue-1 |
| [29] | Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science, 2010, 327(5962): 167–170. DOI: 10.1126/science.1179555 |
| [30] | Al-Attar S, Westra ER, van der Oost J, et al. Clustered regularly interspaced short palindromic repeats (CRISPRs): the hallmark of an ingenious antiviral defense mechanism in prokaryotes. Biol Chem, 2011, 392(4): 277–289. |
| [31] | Mougiakos I, Bosma EF, de Vos WM, et al. Next generation prokaryotic engineering: the CRISPR-Cas toolkit. Trends Biotechnol, 2016, 34(7): 575–587. DOI: 10.1016/j.tibtech.2016.02.004 |
| [32] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262–1278. DOI: 10.1016/j.cell.2014.05.010 |
| [33] | Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823–826. DOI: 10.1126/science.1232033 |
| [34] | Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(3): 233–239. DOI: 10.1038/nbt.2508 |
| [35] |
Chang LX, Sun CC, Chen XJ, et al. Knocking out of human DNAH2 gene in U2OS cells by CRISPR/Cas9n double nick system.
Chin J Biotech, 2017, 33(2): 284–293.
(in Chinese). 常丽贤, 孙聪聪, 陈晓娟, 等. 利用CRISPR/Cas9n double nick系统构建人DNAH2基因敲除的U2OS细胞株. 生物工程学报, 2017, 33(2): 284-293. |
| [36] |
Zhan CS, Xia XY. Research progress of CRISPR-Cas9 system for gene therapy.
Chin J Biotech, 2016, 32(7): 861–869.
(in Chinese). 詹长生, 夏小雨. 基于CRISPR-Cas9技术的基因治疗研究进展. 生物工程学报, 2016, 32(7): 861-869. |
| [37] | Horii T, Hatada I. Genome editing using mammalian haploid cells. Int J Mol Sci, 2015, 16(10): 23604–23614. DOI: 10.3390/ijms161023604 |
| [38] | Wang HY, Yang H, Shivalila CS, et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 2013, 153(4): 910–918. DOI: 10.1016/j.cell.2013.04.025 |
| [39] | Shen B, Zhang J, Wu HY, et al. Generation of gene-modified mice via Cas9/RNA-mediated gene targeting. Cell Res, 2013, 23(5): 720–723. DOI: 10.1038/cr.2013.46 |
| [40] | Li DL, Qiu ZW, Shao YJ, et al. Heritable gene targeting in the mouse and rat using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 681–683. DOI: 10.1038/nbt.2661 |
| [41] | Hai T, Teng F, Guo RF, et al. One-step generation of knockout pigs by zygote injection of CRISPR/Cas system. Cell Res, 2014, 24(3): 372–375. DOI: 10.1038/cr.2014.11 |
| [42] | Ni W, Qiao J, Hu SW, et al. Efficient gene knockout in goats using CRISPR/Cas9 system. PLoS ONE, 2014, 9(9): e106718. DOI: 10.1371/journal.pone.0106718 |
| [43] | Honda A, Hirose M, Sankai T, et al. Single-step generation of rabbits carrying a targeted allele of the tyrosinase gene using CRISPR/Cas9. Exp Anim, 2015, 64(1): 31–37. |
| [44] | Zou QJ, Wang XM, Liu YZ, et al. Generation of gene-target dogs using CRISPR/Cas9 system. J Mol Cell Biol, 2015, 7(6): 580–583. DOI: 10.1093/jmcb/mjv061 |
| [45] | Chen YC, Zheng YH, Kang Y, et al. Functional disruption of the dystrophin gene in rhesus monkey using CRISPR/Cas9. Hum Mol Genet, 2015, 24(13): 3764–3774. DOI: 10.1093/hmg/ddv120 |
| [46] | Liang PP, Xu YW, Zhang XY, et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell, 2015, 6(5): 363–372. DOI: 10.1007/s13238-015-0153-5 |
| [47] | Yang H, Wang HY, Shivalila CS, et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell, 2013, 154(6): 1370–1379. DOI: 10.1016/j.cell.2013.08.022 |
| [48] | Stankiewicz P, Lupski JR. Structural variation in the human genome and its role in disease. Annu Rev Med, 2010, 61: 437–455. DOI: 10.1146/annurev-med-100708-204735 |
| [49] | Fujii W, Kawasaki K, Sugiura K, et al. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucleic Acids Res, 2013, 41(20): e187. DOI: 10.1093/nar/gkt772 |
| [50] | Zhang LQ, Jia RR, Palange NJ, et al. Large genomic fragment deletions and insertions in mouse using CRISPR/Cas9. PLoS ONE, 2015, 10(3): e0120396. DOI: 10.1371/journal.pone.0120396 |
| [51] | Wu YX, Liang D, Wang YH, et al. Correction of a genetic disease in mouse via use of CRISPR-Cas9. Cell Stem Cell, 2013, 13(6): 659–662. DOI: 10.1016/j.stem.2013.10.016 |
| [52] | Long CZ, Mcanally JR, Shelton JM, et al. Prevention of muscular dystrophy in mice by CRISPR/Cas9-mediated editing of germline DNA. Science, 2014, 345(6201): 1184–1188. DOI: 10.1126/science.1254445 |
| [53] | Porteus MH, Carroll D. Gene targeting using zinc finger nucleases. Nat Biotechnol, 2005, 23(8): 967–973. DOI: 10.1038/nbt1125 |
| [54] | Klug A. Towards therapeutic applications of engineered zinc finger proteins. FEBS Lett, 2005, 579(4): 892–894. DOI: 10.1016/j.febslet.2004.10.104 |
| [55] | Pabo CO, Peisach E, Grant RA. Design and selection of novel Cys2His2 zinc finger proteins. Annu Rev Biochem, 2001, 70: 313–340. DOI: 10.1146/annurev.biochem.70.1.313 |
| [56] | Lieber MR. The mechanism of human nonhomologous DNA end joining. J Biol Chem, 2008, 283(1): 1–5. |
| [57] | Bibikova M, Golic M, Golic KG, et al. Targeted chromosomal cleavage and mutagenesis in Drosophila using zinc-finger nucleases. Genetics, 2002, 161(3): 1169–1175. |
| [58] | Geurts AM, Moreno C. Zinc-finger nucleases: new strategies to target the rat genome. Clin Sci, 2010, 119(8): 303–311. DOI: 10.1042/CS20100201 |
| [59] | Carroll D. Progress and prospects: zinc-finger nucleases as gene therapy agents. Gene Ther, 2008, 15(22): 1463–1468. DOI: 10.1038/gt.2008.145 |
| [60] | Geurts AM, Cost GJ, Freyvert Y, et al. Knockout rats via embryo microinjection of zinc-finger nucleases. Science, 2009, 325(5939): 433. DOI: 10.1126/science.1172447 |
| [61] | Geurts AM, Cost GJ, Rémy S, et al. Generation of gene-specific mutated rats using zinc-finger nucleases. Methods Mol Biol, 2010, 597: 211–225. DOI: 10.1007/978-1-60327-389-3 |
| [62] | Rémy S, Tesson L, Ménoret S, et al. Zinc-finger nucleases: a powerful tool for genetic engineering of animals. Transgenic Res, 2010, 19(3): 363–371. DOI: 10.1007/s11248-009-9323-7 |
| [63] | Doyon Y, McCammon JM, Miller JC, et al. Heritable targeted gene disruption in zebrafish using designed zinc-finger nucleases. Nat Biotechnol, 2008, 26(6): 702–708. DOI: 10.1038/nbt1409 |
| [64] | Li T, Huang S, Jiang WZ, et al. TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and FokI DNA-cleavage domain. Nucleic Acids Res, 2011, 39(1): 359–372. DOI: 10.1093/nar/gkq704 |
| [65] | Boch J, Scholze H, Schornack S, et al. Breaking the code of DNA binding specificity of TAL-type Ⅲ effectors. Science, 2009, 326(5959): 1509–1512. DOI: 10.1126/science.1178811 |
| [66] | Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors. Science, 2009, 326(5959): 1501. DOI: 10.1126/science.1178817 |
| [67] | Bitinaite J, Wah DA, Aggarwal AK, et al. FokI dimerization is required for DNA cleavage. Proc Natl Acad Sci USA, 1998, 95(18): 10570–10575. DOI: 10.1073/pnas.95.18.10570 |
| [68] | Cermak T, Doyle EL, Christian M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res, 2011, 39(12): e82. DOI: 10.1093/nar/gkr218 |
| [69] | Miller JC, Tan SY, Qiao GJ, et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol, 2011, 29(2): 143–148. DOI: 10.1038/nbt.1755 |
| [70] | Kim Y, Kweon J, Kim A, et al. A library of TAL effector nucleases spanning the human genome. Nat Biotechnol, 2013, 31(3): 251–258. DOI: 10.1038/nbt.2517 |
| [71] | Morbitzer R, Elsaesser J, Hausner J, et al. Assembly of custom TALE-type DNA binding domains by modular cloning. Nucleic Acids Res, 2011, 39(13): 5790–5799. DOI: 10.1093/nar/gkr151 |
| [72] | Tan WF, Carlson DF, Walton MW, et al. Precision editing of large animal genomes. Adv Genet, 2012, 80: 37–97. DOI: 10.1016/B978-0-12-404742-6.00002-8 |
| [73] | Dahlem TJ, Hoshijima K, Jurynec MJ, et al. Simple methods for generating and detecting locus-specific mutations induced with TALENs in the zebrafish genome. PLoS Genet, 2012, 8(8): e1002861. DOI: 10.1371/journal.pgen.1002861 |
| [74] | Mussolino C, Cathomen T. TALE nucleases: tailored genome engineering made easy. Curr Opin Biotechnol, 2012, 23(5): 644–650. DOI: 10.1016/j.copbio.2012.01.013 |
 2019, Vol. 35
2019, Vol. 35