中国科学院微生物研究所、中国微生物学会主办
文章信息
- 甘海丽, 洪岭, 杨凤莲, 柳定凤, 金莉萍, 郑青亮
- Gan Haili, Hong Ling, Yang Fenglian, Liu Dingfeng, Jin Liping, Zheng Qingliang
- mRNA表观修饰方式及m6A功能研究进展
- Progress in epigenetic modification of mRNA and the function of m6A modification
- 生物工程学报, 2019, 35(5): 775-783
- Chinese Journal of Biotechnology, 2019, 35(5): 775-783
- 10.13345/j.cjb.180416
-
文章历史
- Received: October 11, 2018
- Accepted: January 24, 2019
以往认为中心法则中DNA主要发挥模板的作用而mRNA则扮演着信息传递的角色。mRNA表观修饰的发现超越了mRNA仅作为信息传递者这一观点。近期发现前体mRNA在外显子剪接、5′-加帽和3′-加尾等加工过程中碱基也会发生m6A、m1A、假尿嘧啶、5′-胞嘧啶甲基化等多种化学修饰[1-3]。这些修饰将影响mRNA的剪接、出核、稳定和翻译等mRNA代谢过程从而调控基因的表达。随后也发现mRNA表观修饰是动态可逆的,m6A修饰的可逆调控以及发挥生物学功能都需要特异的修饰酶(Writer)、去修饰酶(Eraser)以及特异性的识别修饰位点的蛋白(Reader)的参与,这些发现为在真核生物中研究转录后基因表达调控机制开辟了一个新领域。在这些修饰类型中,m6A修饰是目前研究最为深入的表观修饰,研究发现mRNA的Writer蛋白和Eraser蛋白将决定标靶mRNA的m6A修饰水平,而Reader蛋白将决定m6A修饰mRNA的翻译[4]或影响mRNA的稳定性。由于许多mRNA在稳定性和翻译效率方面存在显著差异,m6A甲基化修饰可以促使其同步表达从而在短时间内产生大量效应蛋白来应对自身和外界的调控,通过加速整个mRNA的代谢过程进而快速调控细胞增殖和分化。
1 mRNA表观修饰方式 1.1 m6A修饰N6-腺嘌呤(m6A)是真核生物中最丰富的一种化学修饰。m6A修饰广泛存在于各种细胞中,运用m6A特异性抗体的免疫沉淀结合高通量测序技术,发现在人类25%的转录子中存在约7 000个m6A修饰位点,并且主要在RRm6ACH([G/A/U] [G > A]m6AC[U > A > C])保守序列上进行修饰。m6A修饰主要位于外显子的终止密码子附近和3′-UTRs (3′-untranslated region)区域[5-6]。研究显示,m6A修饰参与mRNA代谢、mRNA与蛋白相互作用等各种生理学过程。
1.2 m5C修饰N5-胞嘧啶甲基化(m5C)是一种DNA的表观遗传修饰方式。后来发现mRNA上也存在这种修饰方式,但mRNA上的m5C修饰比m6A要少。通过对人细胞的mRNA测序发现在线粒体中富含m5C修饰,有别于m6A的是,m5C存在于mRNA的翻译起始点下游区域。m5C修饰后的mRNA由一种特定的m5C识别蛋白识别并在其协助下出核,因此m5C在某种程度上可以调节mRNA的出核并促进mRNA的表达[7]。线粒体中的mRNA的甲基化水平明显高于核基因转录组的RNA甲基化水平,因此m5C可能在调控线粒体生理功能方面扮演着重要角色[8]。然而m5C修饰的更多生理功能及其机制还有待进一步研究。
1.3 m1A修饰最初发现N1-腺苷酸甲基化(m1A)是一种广泛存在于tRNA、rRNA上的化学修饰,对RNA的生物学功能具有重要的调控作用。运用特异性m1A抗体免疫共沉淀结合高通量测序发现人细胞mRNA上存在1 000多个m1A的修饰位点[3]。有别于m6A的是,m1A在mRNA上特异分布于翻译区域的起始位点附近以及第一个剪切拼接位点附近。m1A甲基化修饰能改变mRNA的二级结构,例如位于5′-UTRs (5′-untranslated region)处的m1A能够通过降低mRNA的二级结构的稳定性来增加翻译效率[9]。目前m1A具体的生理功能尚不明确,需要进一步研究。
1.4 假尿嘧啶修饰假尿嘧啶修饰(Pseudouridine)广泛存在于非编码RNA,其可稳定tRNA和rRNA的结构。通过假尿嘧啶测序发现在酵母菌和人类的非编码RNA中存在数百个假尿嘧啶修饰位点。进一步分析发现mRNA中也存在高度保守的假尿嘧啶修饰位点,并且饥饿等环境因素可以调控mRNA上的假尿嘧啶修饰[10]。假尿嘧啶修饰可改变密码子与反密码子碱基配对的相互作用,因此假尿嘧啶不仅影响RNA的结构而且影响mRNA的编码能力[11]。综上,假尿嘧啶修饰和其他甲基化修饰一样能调控mRNA的表达,但其具体功能仍需进一步研究。
2 m6A修饰相关蛋白m6A功能的调节需要相关蛋白的参与,其中主要包括甲基化酶、去甲基化酶以及m6A阅读蛋白。
首先m6A的一个重要相关蛋白就是甲基化酶,其对应的编码基因称为Writers,主要包括甲基转移酶3 (Methyltransferase-like 3,METTL3)、甲基转移酶14 (Methyltransferase-like 14,METTL14)、Wilms’瘤相关蛋白(Wilms’ tumor1- associating protein,WTAP)、甲基转移酶16 (Methyltransferase-like 16,METTL16)、RNA结合模体蛋白15 (RNA binding motif protein 15,RBM15)、锌指结构域包含蛋白13 (Zinc finger CCCH domain-containing protein 13,ZC3H13)等,实际上甲基化酶主要是由METTL3和METTL14组成的异源二聚体结构并且与WTAP、ZC3H13等调节因子附着在二聚体上构成甲基化酶复合体。其中METTL3具有一个SAM结合区域[12]并且能够识别特定潜在的m6A修饰位点并将SAM的甲基转移该位点上。METTL14与METTL3结合后,在一定程度上能够促进METTL3识别m6A。WTAP是一种剪切体相关蛋白,通过与METTL3-METTL14二聚体结合能够使甲基化酶复合体迅速识别m6A潜在修饰位点并且活化METTL3/METTL14甲基化酶复合体[13]。此外,最近研究发现,锌指结构(ZC3H13)能够结合WTAP从而使WTAP固定存在于核内而不转运到核外,实现强化甲基化酶复合体的目的[14],以保证足够的甲基化酶复合体在核内对转录合成的mRNA进行甲基化。
其次,去甲基化酶主要包括肥胖相关基因Fat mass and obesity-associated (FTO)和AlkB homolog 5 (ALKBH5),其对应的编码基因称为Erasers。FTO起初是作为一种与肥胖相关的基因出现在人们的视线中,FTO是第一个被发现的m6A去甲基化酶,该发现证实了m6A是一个可逆的mRNA修饰过程。FTO和ALKBH5分布具有差异性,FTO广泛存在于成人和胚胎的组织中,在大脑中表达尤其高,而ALKBH5存在于大部分组织的细胞核中。FTO能够有效地去除mRNA上的m6A修饰并且FTO受抑制能够增高整体mRNA的m6A修饰水平[15]。ALKBH5主要作用是使已经m6A修饰的mRNA发生去甲基化,通过参与调控mRNA的出核等代谢过程发挥其功能,其与精子的发育密切相关。
最后,m6A相关蛋白是m6A结合蛋白,其编码基因被称为Readers,主要包括YTH同源结构域蛋白家族:YTH结构域家族1 (YTH domain family 1,YTHDF1)、YTH结构域家族2 (YTH domain family 2,YTHDF2)、YTH结构域家族3 (YTH domain family 3,YTHDF3)、YTH结构域包含蛋白1 (YTH domain-containing proteins 1,YTHDC1)、YTH结构域包含蛋白2 (YTH domain-containing proteins 2,YTHDC2)和核不均一核糖核蛋白(Heterogeneous nuclear ribonucleoproteins C,HNRNPC)等,YTHDC1主要能够通过结构域与特定的m6A位点结合或者与剪接调控因子结合影响m6A修饰的mRNA的出核和剪接[16]。HNPNPC家族的蛋白同样也可以直接或间接地调控mRNA的选择性剪接和mRNA结构的改变[17]。YTHDF1与mRNA上的m6A结合提高相应的mRNA翻译效率[18]。YTHDF2主要作用则是促进mRNA的降解[19]。有趣的是,YTHDF3与YTHDF1结合时能够增强其翻译能力,而YTHDF3与YTHDF2结合时又能促进其促降解能力。YTHDC2是一种具有螺旋酶结构域和蛋白重复结构域的reader,由于这种特殊的结构使其具有调节RNA结合效率、调控RNA结构以及同时结合多种蛋白的特点,其能够促进mRNA的翻译[20]。
这些m6A相关蛋白以及影响因子相互作用影响m6A水平,进而影响mRNA的剪接、出核、翻译、降解和表达等各个方面,从而影响细胞的分化、凋亡等生命过程。
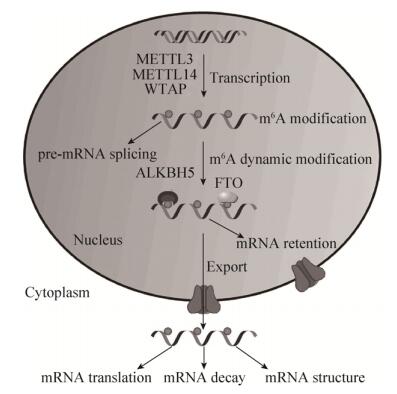
3 m6A调控mRNA的代谢功能m6A广泛存在于各种细胞中,m6A相关蛋白和甲基化调节因子以及m6A的识别蛋白相互作用构建了一个调节m6A基因表达的网络,m6A在mRNA的剪接、出核、翻译、降解和结构变化等方面发挥着重要的调控作用(图 1)。
|
| 图 1 m6A修饰参与调控mRNA代谢的模式图 Fig. 1 Function of m6A in mRNA metabolism. |
| |
前体mRNA成熟为mRNA的过程主要包括5′端加帽、3′端加尾以及内含子剪接3个部分。研究表明m6A主要集中存在于前体mRNA的内含子并且m6A的甲基化酶和Reader蛋白主要存在于核内[16],因此可以推测甲基化过程主要发生在核内并且在核内调控mRNA的选择性剪接。研究发现敲除METTL3后,m6A水平下降,精子来源的相关基因发生错误剪接进而表达下调,说明经METTL3修饰的m6A能够调控多功能基因的选择性剪接和睾丸中基因的表达从而调控精原细胞分化和精母细胞形成[21]。WTAP作为甲基化酶复合体的一部分,敲除后的甲基化酶复合体对mRNA的甲基化效率降低,m6A水平下降也会导致mRNA的选择性剪接异常。m6A去甲基化酶FTO通过去除RNA上的m6A以及阻止SR蛋白与mRNA结合来调控RNA的选择性剪接,SR蛋白是一种参与调控可变剪接的重要调控因子,例如FTO能够阻止SRSF2与mRNA结合从而调控mRNA的剪接以及转录。FTO倾向于结合靠近外显子选择性剪接区域的内含子区域,结合了FTO的内含子处m6A水平下降并且抑制了SRSF2与mRNA的结合,因此该处内含子后面的外显子不能进行选择性的剪接以及转录表达,这一过程称之为外显子跳跃[22]。
此外一些阅读蛋白也可以通过抑制或促进SR蛋白家族的成员与mRNA结合来调控mRNA的剪接。例如YTHDC1促进SRSF3和抑制SRSF10与mRNA结合来影响mRNA的剪接,通过识别XIST (X染色体失活基因)上的m6A来使X染色体沉默[23]。m6A不仅仅通过Reader蛋白等来影响mRNA的选择性剪接,还能够直接调控mRNA选择性剪接。
总之,m6A是一个动态的调控系统,通过快速改变m6A修饰位点甲基化水平,影响其与甲基化酶、去甲基化酶和Reader蛋白等相互作用来影响mRNA的剪接。
3.2 m6A促进mRNA的出核mRNA的出核是连接mRNA转录和翻译的关键步骤同时能选择性地调控基因表达。据报道m6A甲基化能加快mRNA的出核,YTHDC1结合SRF3和NXF1 (核ENA输出因子)会促进m6A修饰mRNA的出核。敲除METTL3,m6A水平下调,mRNA的出核受阻,表达受抑制,而敲除ALKBH5对m6A去甲基化作用减弱,m6A的水平提高,mRNA结合readers和调控出核的影响因子促进mRNA的出核[24]。本课题组发现过表达ALKBH5后能引起抗病毒转录子mRNA的核滞留,从而抑制抗病毒转录子的表达[25]。综上表明m6A促进mRNA的出核可能是其调控基因表达的重要方式,但其具体机制仍有待研究。
3.3 m6A促进mRNA的翻译据报道YTHDF1结合m6A修饰的mRNA并且招募翻译起始因子复合体eIF3促进mRNA的翻译。该过程是依赖METTLE3甲基化酶活性以及YTHDF1-eIF3通路[26]。研究发现YTHDC2的YTH和R3H结构域能够促进其与细胞的RNA的结合并且与小核糖体相互作用从而促进mRNA的翻译[27],表明m6A可以通过不同的分子机制来调控mRNA的翻译。
3.4 m6A影响mRNA的降解降解是mRNA代谢调控的重要步骤,m6A能够降低mRNA的稳定性,mRNA经过m6A修饰后可能被迅速降解。METTL3或METTLE14敲除后m6A水平下降,mRNA降解效率降低,表达水平增高[13]。研究表明,METTL3缺失的幼稚T细胞中的细胞因子诱导性SH2结构域包含蛋白(Cytokine inducible SH2 containing protein,SOCS或CISH)家族基因的mRNA降解速度较慢,从而使mRNA和蛋白表达水平升高,SOCS基因家族主要负责编码STAT信号抑制蛋白,敲除METTL3后SOCS高表达,STAT信号抑制蛋白增加从而抑制IL-7介导的STAT5活化以及T细胞的稳态增殖和分化[28]。
另外YTHDF2的羧基末端YTH结构域能选择性地结合甲基化mRNA,而YTHDF2的氨基末端的结构域能将结合的mRNA转运到RNA降解器中进一步降解[29]。RNA降解器是由细胞内许多因子组成且能够降解mRNA的复合体,在RNA降解器上存在一些降解mRNA的酶,能够对已经翻译或错误翻译、不能翻译的mRNA进行不同途径的降解。敲除YTHDF2后,mRNA的稳定性提高并且标靶mRNA表达水平提高,提示m6A能够影响mRNA的降解。总之,m6A可以降低mRNA的稳定性并且促进含有m6A的mRNA的降解从而调控基因的表达。
3.5 m6A改变RNA结构m6A可以改变mRNA的结构使其更易与核不均一核糖核蛋白C (HNRNPC)和核不均一核糖核蛋白G (HNRNPG)结合,例如肺腺癌转移相关转录子1 (Metastasis associated lung adenocarcinoma transcript 1,MALAT1)和CDP二酰基甘油合成酶2 (CDP-diacylglycerol synthase 2,CDS2),这些mRNA发生m6A修饰后双链结构容易被打开并暴露单链的U核苷酸位点,该位点被HNRNPC识别后调控这些基因的剪接[17]。这种由于m6A修饰所引起的结构重塑而影响mRNA与蛋白结合的现象称为m6A开关(m6A switch)。m6A开关广泛存在于转录组中,通过调控mRNA与结合蛋白之间的结合从而发挥相应的生物学功能。
4 m6A调控细胞的生物学功能 4.1 m6A调控细胞的分化能力干细胞是一种具有向不同类型细胞分化能力的多能细胞,正常细胞分化是生物体发育的重要环节,分化障碍会导致各种严重疾病的发生。多种编码发育调控因子转录本的稳定性和表达水平与m6A的水平呈负相关。METTL3或METTL14下调后,m6A水平下降,维持细胞多能性的转录因子高表达,胚胎干细胞的自我更新能力显著降低[30-31]。据报道小鼠胚胎干细胞NANOG (细胞多能性编码因子) mRNA上存在多个METTL3的结合位点,敲除METTL3,多能性细胞多能性编码因子的m6A修饰水平下降,胚胎干细胞自我更新以及分化能力受到抑制,胚胎干细胞还保留着一部分原始细胞的状态,在种植后由于外界状态的改变以及部分基因程序的启动,导致胚胎死亡[32]。
另外,低氧环境下乳腺癌肿瘤细胞中高表达ALKBH5,使得关键因子NANOG发生去甲基化,m6A水平下降,从而提高mRNA的稳定性以及表达水平,进一步促进了癌细胞的增殖,调控乳腺癌肿瘤细胞的分化[33]。另一种RNA去甲基化酶FTO在急性白血病患者中呈高表达,实验证明FTO通过降低m6A水平增强了白血病细胞的增殖和分化,抑制了细胞的凋亡,显著增强了白血病致癌基因介导的正常造血干细胞向病态细胞的转化[34]。脂肪细胞中FTO能通过调控m6A的水平进而影响可变剪接调控因子SRSF2与FTO的结合,影响脂肪形成关键基因的选择性剪接从而调控前脂肪细胞的分化[35]。综上表明m6A表观相关酶在调控干细胞或肿瘤细胞的分化发育方面发挥重要的作用。
4.2 m6A调控应激反应在低氧、营养不良等极端环境中,机体需要快速应对外界环境变化而需在mRNA转录后水平上进行调控,而不是重新合成新的mRNA。在热休克状态下,YTHDF2定位于核内并且可以抑制FTO与mRNA的结合,阻止mRNA的去甲基化,从而确保5′-UTRs端有足够的m6A,5′-UTRs端的m6A能推动帽子端的独立翻译过程,提高相关蛋白的形成。
除此之外,YTHDF1能使应激颗粒局限于核内,应激颗粒是由一些蛋白包裹mRNA和蛋白颗粒形成的,在YTHDF1的作用下,应激颗粒向胞内聚集导致蛋白不释放和mRNA不表达,等应激源消失后,应激颗粒重新解聚释放,被包裹的mRNA重新表达[36],这种机制可以避免应激源诱发的炎症反应的发生并且对损伤后的修复有一定的帮助。此外,在遗传上定义的永生化和致癌转化的人乳腺上皮细胞中,过表达MTTLL3和METTL14或敲除ALKBH5能增加m6A的水平并促进细胞增殖和迁移。永生化和致癌转化细胞中的m6A水平随着缺氧而增加,提示在应激状态下细胞可以调节m6A水平从而应对应激源的攻击[37]。本课题组在机体应对病原体入侵这一应激环境中作了一些工作,发现ALKBH5在巨噬细胞的固有免疫反应中发挥重要作用,在病毒感染后,核蛋白DDX46可以招募ALKBH5,使得抗病毒转录子发生去m6A甲基化修饰而被滞留在核内并抑制其表达,从而抑制巨噬细胞的抗病毒天然反应。我们目前在m6A修饰如何参与胚胎滋养细胞应对病原体入侵方面也进行了深入研究,并取得了一些重要进展。另有研究发现,在感染KHSV病毒的细胞中,细胞的整体m6A水平下调而病毒本身转录子的m6A水平却上调,而当m6A修饰酶被敲除之后KSHV病毒裂解的催化因子ORF50表达会受到抑制,而YTHDF2和METTL3敲除后会增加ORF50的水平而起到抗病毒的作用[37]。综上所述,m6A参与调控在热刺激或低氧等环境下细胞的生理功能状态。
4.3 m6A调控细胞生理节律据报道在哺乳动物生理节律的研究中首次发现了m6A对细胞功能的影响。生理节律的维持涉及到基因表达的负反馈环路,然而研究表明只有五分之一的节律基因是需要重新转录合成[38],表明转录后水平的基因调控在生物节律调控中发挥重要作用。节律基因和时钟输出基因的转录本均可以发生m6A甲基化修饰,METTL3敲除后导致两个关键时钟基因(PER3和芳基烃类受体核易位体样蛋白1,ARNTL) mRNA的m6A水平下降使出核受阻,导致周期蛋白表达减少、细胞生理周期延长,细胞凋亡能力下降。这表明mRNA的m6A修饰改变将对细胞的生命周期产生深远的影响[39]。以上报道表明m6A在调控细胞节律方面发挥重要作用,但其具体机制还有待进一步的研究。
5 总结与展望mRNA上的m6A修饰主要特征是其广泛存在性、分布独特以及动态可逆性,研究发现m6A修饰通过调控mRNA代谢过程的各个方面,影响mRNA的蛋白表达,参与调控细胞分化、应激反应、生物节律等细胞生物学过程,调控失常将导致各种疾病的发生。因此研究清楚m6A的动态修饰规律及其如何影响细胞生物学功能的具体分子机制尤为重要。
我们认为以下几方面可能是未来研究的重要方向:1)随着研究的深入,发现人类基因中尽管有超过7 000个m6A修饰位点,但是还有很多符合保守序列(RRACH motifs)的潜在m6A位点并没有被修饰,表明m6A甲基化修饰作为转录后调控机制还存在着更大的潜力。m6A在转录本上的分布是呈不均匀以及具有组织特异性。这些特征意味着转录本上每一个m6A修饰位点均处于甲基化和未甲基化的动态平衡状态,从而使机体对外界的刺激能够作出迅速的反应,那么机体是如何特异性地维持这种甲基化的动态修饰状态呢?是依赖于修饰位点附近特异的碱基序列还是特异性的Reader蛋白呢?2) mRNA的m6A甲基化修饰能选择性地调控特异mRNA的代谢以及调控在细胞分化过程中细胞状态的改变。那么如何实现选择性地与特异mRNA结合,以及甲基化酶、去甲基化酶和Reader蛋白在应对不同的信号通路调控时是如何协调的机制仍然不清楚,需要深入研究,因为任何一个环节的缺失都可能会诱发疾病的发生。3) m6A仅是mRNA转录后修饰的一种方式。除了m6A外,还有m1A、m5C、假尿苷pseudouridine等。每一种化学修饰均可能拥有相关的特异修饰酶、去修饰酶和Reader蛋白,或许这些蛋白能共同作用于多种修饰。mRNA经过化学修饰后,也需要通过相关蛋白共同作用来调控基因的表达,那么寻找这些特异的修饰相关蛋白将是一个非常重要的方面。4)随着mRNA的化学修饰领域不断的扩展,如何能够直接、及时地检测到这些化学修饰是否存在及其作用位点并及时进行干预,对于生物的发育、疾病的控制有重要的意义。mRNA测序技术的进一步发展将会是认识各种动态修饰的关键一步。
| [1] | Spenkuch F, Motorin Y, Helm M. Pseudouridine: still mysterious, but never a fake (uridine)!. RNA Biol, 2014, 11(12): 1540–1554. DOI: 10.4161/15476286.2014.992278 |
| [2] | Hussain S, Aleksic J, Blanco S, et al. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biol, 2013, 14(11): 215. DOI: 10.1186/gb4143 |
| [3] | Dominissini D, Nachtergaele S, Moshitch-Moshkovitz S, et al. The dynamic N1-methyladenosine methylome in eukaryotic messenger RNA. Nature, 2016, 530(7591): 441–446. DOI: 10.1038/nature16998 |
| [4] | Wang X, Zhao BS, Roundtree IA, et al. N6-methyladenosine modulates messenger RNA translation efficiency. Cell, 2015, 161(6): 1388–1399. DOI: 10.1016/j.cell.2015.05.014 |
| [5] | Dominissini D, Moshitch-Moshkovitz S, Schwartz S, et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature, 2012, 485(7397): 201–206. DOI: 10.1038/nature11112 |
| [6] | Meyer KD, Saletore Y, Zumbo P, et al. Comprehensive analysis of mRNA methylation reveals enrichment in 3′ UTRs and near stop codons. Cell, 2012, 149(7): 1635–1646. DOI: 10.1016/j.cell.2012.05.003 |
| [7] | Yang X, Yang Y, Sun BF, et al. 5-methylcytosine promotes mRNA export-NSUN2 as the methyltransferase and ALYREF as an m5C reader. Cell Res, 2017, 27(5): 606–625. DOI: 10.1038/cr.2017.55 |
| [8] | Wei Z, Panneerdoss S, Timilsina S, et al. Topological characterization of human and mouse m5C epitranscriptome revealed by bisulfite sequencing. Int J Genom, 2018, 2018: 1351964. |
| [9] | Zhou HQ, Kimsey IJ, Nikolova EN, et al. m1A and m1G disrupt A-RNA structure through the intrinsic instability of Hoogsteen base pairs. Nat Struct Mol Biol, 2016, 23(9): 803–810. DOI: 10.1038/nsmb.3270 |
| [10] | Carlile TM, Rojas-Duran MF, Zinshteyn B, et al. Pseudouridine profiling reveals regulated mRNA pseudouridylation in yeast and human cells. Nature, 2014, 515(7525): 143–146. DOI: 10.1038/nature13802 |
| [11] | Fernández IS, Ng CL, Kelley AC, et al. Unusual base pairing during the decoding of a stop codon by the ribosome. Nature, 2013, 500(7460): 107–110. DOI: 10.1038/nature12302 |
| [12] | Bokar JA, Shambaugh ME, Polayes D, et al. Purification and cDNA cloning of the AdoMet-binding subunit of the human mRNA (N6-adenosine)-methyltransferase. RNA, 1997, 3(11): 1233–1247. |
| [13] | Liu JZ, Yue YN, Han DL, et al. A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation. Nat Chem Biol, 2014, 10(2): 93–95. |
| [14] | Wen J, Lv RT, Ma HH, et al. Zc3h13 regulates nuclear RNA m6A methylation and mouse embryonic stem cell self-renewal. Mol Cell, 2018, 69(6): 1028–1038. DOI: 10.1016/j.molcel.2018.02.015 |
| [15] | Su R, Dong L, Li CY, et al. R-2HG exhibits anti-tumor activity by targeting FTO/m6A/MYC/ CEBPA signaling. Cell, 2018, 172(1/2): 90–105. |
| [16] | Xiao W, Adhikari S, Dahal U, et al. Nuclear m6A reader YTHDC1 regulates mRNA splicing. Mol Cell, 2016, 61(4): 507–519. DOI: 10.1016/j.molcel.2016.01.012 |
| [17] | Liu N, Dai Q, Zheng GQ, et al. N6-methyladenosine-dependent RNA structural switches regulate RNA-protein interactions. Nature, 2015, 518(7540): 560–564. DOI: 10.1038/nature14234 |
| [18] | Shi HL, Zhang XL, Weng YL, et al. m6A facilitates hippocampus-dependent learning and memory through YTHDF1. Nature, 2018, 563(7730): 249–253. DOI: 10.1038/s41586-018-0666-1 |
| [19] | Li ZR, Qian PX, Shao WQ, et al. Suppression of m6A reader Ythdf2 promotes hematopoietic stem cell expansion. Cell Res, 2018, 28(9): 904–917. DOI: 10.1038/s41422-018-0072-0 |
| [20] | Wojtas MN, Pandey RR, Mendel M, et al. Regulation of m6A transcripts by the 3'→5' RNA helicase YTHDC2 is essential for a successful meiotic program in the mammalian germline. Mol Cell, 2017, 68(2): 374–387. DOI: 10.1016/j.molcel.2017.09.021 |
| [21] | Xu K, Yang Y, Feng GH, et al. Mettl3-mediated m6A regulates spermatogonial differentiation and meiosis initiation. Cell Res, 2017, 27(9): 1100–1114. DOI: 10.1038/cr.2017.100 |
| [22] | Bartosovic M, Molares HC, Gregorova P, et al. N6-methyladenosine demethylase FTO targets pre-mRNAs and regulates alternative splicing and 3'-end processing. Nucleic Acids Res, 2017, 45(19): 11356–11370. DOI: 10.1093/nar/gkx778 |
| [23] | Patil DP, Chen CK, Pickering BF, et al. m6A RNA methylation promotes XIST-mediated transcriptional repression. Nature, 2016, 537(7620): 369–373. DOI: 10.1038/nature19342 |
| [24] | Zheng GQ, Dahl JA, Niu YM, et al. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility. Mol Cell, 2013, 49(1): 18–29. |
| [25] | Zheng QL, Hou J, Zhou Y, et al. The RNA helicase DDX46 inhibits innate immunity by entrapping m6A-demethylated antiviral transcripts in the nucleus. Nat Immunol, 2017, 18(10): 1094–1103. DOI: 10.1038/ni.3830 |
| [26] | Taketo K, Konno M, Asai A, et al. The epitranscriptome m6A writer METTL3 promotes chemo- and radioresistance in pancreatic cancer cells. Int J Oncol, 2018, 52(2): 621–629. |
| [27] | Kretschmer J, Rao H, Hackert P, et al. The m6A reader protein YTHDC2 interacts with the small ribosomal subunit and the 5'-3' exoribonuclease XRN1. RNA, 2018, 24(10): 1339–1350. DOI: 10.1261/rna.064238.117 |
| [28] | Li HB, Tong J, Zhu S, et al. m6A mRNA methylation controls T cell homeostasis by targeting the IL-7/STAT5/SOCS pathways. Nature, 2017, 548(7667): 338–342. DOI: 10.1038/nature23450 |
| [29] | Wang X, Lu ZK, Gomez A, et al. N6-methyladenosine-dependent regulation of messenger RNA stability. Nature, 2014, 505(7481): 117–120. DOI: 10.1038/nature12730 |
| [30] | Lin Z, Hsu PJ, Xing XD, et al. Mettl3-/Mettl14-mediated mRNA N6-methyladenosine modulates murine spermatogenesis. Cell Res, 2017, 27(10): 1216–1230. DOI: 10.1038/cr.2017.117 |
| [31] | Wang Y, Li Y, Toth JI, et al. N6-methyladenosine modification destabilizes developmental regulators in embryonic stem cells. Nat Cell Biol, 2014, 16(2): 191–198. DOI: 10.1038/ncb2902 |
| [32] | Geula S, Moshitch-Moshkovitz S, Dominissini D, et al. Stem cells. m6A mRNA methylation facilitates resolution of naïve pluripotency toward differentiation. Science, 2015, 347(6225): 1002–1006. DOI: 10.1126/science.1261417 |
| [33] | Zhang CZ, Samanta D, Lu HQ, et al. Hypoxia induces the breast cancer stem cell phenotype by HIF-dependent and ALKBH5-mediated m6A-demethylation of NANOG mRNA. Proc Natl Acad Sci USA, 2016, 113(14): E2047–E2056. DOI: 10.1073/pnas.1602883113 |
| [34] | Li ZJ, Weng HYY, Su R, et al. FTO plays an oncogenic role in acute myeloid leukemia as a N6-methyladenosine RNA demethylase. Cancer Cell, 2017, 31(1): 127–141. DOI: 10.1016/j.ccell.2016.11.017 |
| [35] | Zhao X, Yang Y, Sun BF, et al. FTO-dependent demethylation of N6-methyladenosine regulates mRNA splicing and is required for adipogenesis. Cell Res, 2014, 24(12): 1403–1419. DOI: 10.1038/cr.2014.151 |
| [36] | Zhou J, Wan J, Gao XW, et al. Dynamic m6A mRNA methylation directs translational control of heat shock response. Nature, 2015, 526(7574): 591–594. DOI: 10.1038/nature15377 |
| [37] | Fry NJ, Law BA, Ilkayeva OR, et al. N6-methyladenosine contributes to cellular phenotype in a genetically-defined model of breast cancer progression. Oncotarget, 2018, 9(58): 31231–31243. |
| [38] | Koike N, Yoo SH, Huang HC, et al. Transcriptional architecture and chromatin landscape of the core circadian clock in mammals. Science, 2012, 338(6105): 349–354. DOI: 10.1126/science.1226339 |
| [39] | Fustin JM, Doi M, Yamaguchi Y, et al. RNA-methylation-dependent RNA processing controls the speed of the circadian clock. Cell, 2013, 155(4): 793–806. DOI: 10.1016/j.cell.2013.10.026 |
 2019, Vol. 35
2019, Vol. 35