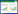中国科学院微生物研究所、中国微生物学会主办
文章信息
- 方卉, 吕常江, 花雨娇, 胡升, 赵伟睿, 方文姬, 宋奎, 黄俊, 梅乐和
- Fang Hui, Lü Changjiang, Hua Yujiao, Hu Sheng, Zhao Weirui, Fang Wenji, Song Kui, Huang Jun, Mei Lehe
- 利用脯氨酸效应提高短乳杆菌谷氨酸脱羧酶的热稳定性
- Increasing the thermostability of glutamate decarboxylase from Lactobacillus brevis by introducing proline
- 生物工程学报, 2019, 35(4): 636-646
- Chinese Journal of Biotechnology, 2019, 35(4): 636-646
- 10.13345/j.cjb.180390
-
文章历史
- Received: September 20, 2018
- Accepted: December 4, 2018
2. 浙江大学 宁波理工学院 生物与化学工程学院,浙江 宁波 315100
2. Department of Biological and Pharmaceutical Engineering, Ningbo Institute of Technology, Zhejiang University, Ningbo 315100, Zhejiang, China
γ-氨基丁酸(γ-aminobutyric acid,GABA)是一种天然存在的非蛋白质氨基酸,具有降血压、抗抑郁、抗惊厥、改善睡眠等多种重要的生理功能,在食品、医药、化工等领域具有广阔的应用前景[1-5]。谷氨酸脱羧酶(Glutamate decarboxylase,GAD;EC 4.1.1.15)能专一、不可逆地催化L-谷氨酸(L-glutamic acid,L-Glu)脱去α-羧基生成GABA和CO2,通过蛋白质工程手段获得催化效率更高、稳定性更好的GAD突变酶,对于拓展其工业应用具有重要的意义[6-7]。
GAD广泛存在于细菌、酵母和丝状真菌等各种微生物中,介导胞内GABA的合成,并在细胞抵御外部酸性环境的过程中发挥重要的作用[8-9]。值得关注的是,在这些所报道的菌株中,作为发酵食品最主要的有益微生物——乳酸菌通常具有较高的GABA合成能力,乳酸菌源GAD也因此受到更为广泛的关注[10]。迄今为止,人们已从鲜奶、泡菜、清酒及各种传统的发酵乳制品和肉制品中分离并鉴定了数百株具有GABA合成能力的乳酸菌,分布于8个属45个种和亚种[10-11]。
课题组前期分离并鉴定了多株具有GABA合成能力的乳酸菌[12]。其中,分离于未灭菌生牛奶中的短乳杆菌Lactobacillus brevis CGMCC No. 1306具有较高的GABA发酵性能[13]。随后,基于分子生物学手段实现了该菌株GAD在大肠杆菌Escherichia coli中的异源重组表达,该重组酶在pH为3–6时具有催化L-Glu生成GABA的能力,但当pH高于5.6时,催化活性降至10%以下[14]。此外,本课题组解析了该蛋白的晶体结构(PDB ID:5GP4),并通过定向进化及计算机辅助的蛋白质分子理性设计等方法有效地拓宽了其pH催化范围,同时实现了其催化效率的显著提升[7,15]。尽管该蛋白呈现出较高的工业应用潜力,但其稳定性相对较差,当温度高于50 ℃时,GAD活力显著下降[12]。因此,如何提高该GAD的热稳定性,对于实现其工业化应用具有重要的价值。
与传统的定向进化相比,利用理性设计来提高蛋白质的热稳定性具有目的性强、效率高等优点,对阐明蛋白质结构和功能的关系亦具有重要意义[16-18]。目前,提高蛋白质热稳定性的理性方法主要有同源序列比对[19]、预测折叠自由能[20]、温度因子(B-factor)设计[21]、引入二硫键[22]、优化蛋白质表面电荷[23]及脯氨酸效应[24]等。大量研究表明,蛋白质分子中引入脯氨酸(Pro)可以降低蛋白质去折叠的骨架熵,增加蛋白质的刚性,显著提高蛋白质的热稳定性。目前关于利用脯氨酸效应提高GAD热稳定性的研究鲜有报道。
基于以上分析,本研究首先将L. brevis CGMCC No. 1306与嗜热古细菌Thermococcus kodakarensis的GAD氨基酸序列进行比对,选择来自T. kodakarensis GAD中Pro所对应的L. brevis GAD氨基酸位点作为突变位点,然后采用定点突变技术在L. brevis GAD对应的氨基酸位点引入Pro,并通过比色法快速筛选具有高热稳定性的GAD突变体,以期提高实验效率。此外,分别利用YASARA和PyMOL软件进行分子动力学模拟及可视化剖析,探讨蛋白质热稳定性提高的机制,并为进一步深入研究GAD家族的热稳定性提供理论依据。
1 材料与方法 1.1 菌种与试剂L. brevis CGMCC 1306、E. coli BL21 (DE3)及E. coli DH5α为本实验室保藏。磷酸吡哆醛(Pyrodoxal-5′-phosphate,PLP)、硫酸卡那霉素(Kan)、丹磺酰氯(DNS-Cl)、改良型Bradford蛋白浓度测定试剂盒购自生工生物工程(上海)股份有限公司;DpnⅠ购自Thermo Scientific公司;PrimeSTAR Max DNA聚合酶购自TaKaRa公司;Ni-NTA层析介质购自北京全式金生物技术有限公司;溴甲酚紫(Bromocresol purple,BCP)购自上海三爱思试剂有限公司;PCR引物由通用生物系统(安徽)有限公司合成。
1.2 GABA及L-Glu含量的测定将100 μL待测样品、900 μL 0.2 mol/L NaHCO3溶液(pH 9.8)及500 μL 4 g/L DNS-Cl丙酮溶液混合均匀后置于40 ℃下避光衍生1 h,然后采用HPLC法测定样品中的GABA及L-Glu含量,具体参考文献[25]。
1.3 突变文库的构建以来源于T. kodakarensis GAD的氨基酸序列为参照,使用DNAMAN软件进行序列比对获取目标蛋白中可引入Pro的氨基酸残基位点,并设计相应定点突变PCR引物。以pET28a(+)-GAD质粒为模板,采用表 1中的引物,PCR克隆获取目标突变体质粒基因;使用DpnⅠ对PCR反应产物进行消化处理(37 ℃,2 h),以消除父本模板;采用热击转化法将克隆产物转入E. coli DH5α感受态细胞中。突变体质粒经通用生物系统(安徽)有限公司测序验证后进一步转化至E. coli BL21(DE3)感受态细胞中,获取目标重组菌株。
| Primer name | Primer sequence (5′–3′) |
| GAD-M70P-F | TTCTGTCAGACTTACCCGGAACCGGAAGCGGTTG |
| GAD-M70P-R | AACCGCTTCCGGTTCCGGGTAAGTCTGACAGAAC |
| GAD-K89P-F | AAAAACGCCATCGATCCGTCCGAGTATCCTCGG |
| GAD-K89P-R | CGAGGATACTCGGACGGATCGATGGCGTTTTTC |
| GAD-S162P-F | TCAACCTAATATTGTCATCCCGGCCGGTTATCAAGTTTGTTG |
| GAD-S162P-R | ACAAACTTGATAACCGGCCGGGATGACAATATTAGGTTGATG |
| GAD-A224P-F | GACAATACGACGATTTACCGCGATTAGATGCCGTTGTAG |
| GAD-A224P-R | TACAACGGCATCTAATCGCGGTAAATCGTCGTATTGTCC |
| GAD-Y242P-F | ACTAAGTTCCCGGTACCGATCCATGTCGATGCC |
| GAD-Y242P-R | GCATCGACATGGATCGGTACCGGGAACTTAGTC |
| GAD-L261P-F | CGTTTATTGAACCCGAGCCGAAGTGGGACTTCCGTTTAAAC |
| GAD-L261P-R | TTAAACGGAAGTCCCACTTCGGCTCGGGTTCAATAAACGGC |
| GAD-G277P-F | CCATCAATGCCTCCCCGCACAAATATGGCTTG |
| GAD-G277P-R | CAAGCCATATTTGTGCGGGGAGGCATTGATGG |
| GAD-Y284P-F | CAAATATGGCTTGGTTCCTCCCGGAGTCGGCTG |
| GAD-Y284P-R | CAGCCGACTCCGGGAGGAACCAAGCCATATTTG |
| GAD-V306P-F | AGCTGGTCTTTAAGCCGAGCTACTTGGGTGGTG |
| GAD-V306P-R | ACCACCCAAGTAGCTCGGCTTAAAGACCAGCTC |
| GAD-G364P-F | ATCGCTCACTAAATTACCGGGCTTTTCCCTCATTAATG |
| GAD-G364P-R | TAATGAGGGAAAAGCCCGGTAATTTAGTGAGCGATTTC |
| GAD-S323P-F | CATCAACTTCTCCCACCCGGCCTCCCAATTAATCGG |
| GAD-S323P-R | CGATTAATTGGGAGGCCGGGTGGGAGAAGTTGATGG |
| GAD-G372P-F | CGCTCACTAAATTACCGGGCTTTTCCCTCATTAATG |
| GAD-G372P-R | ATGAGGGAAAAGCCCGGTAATTTAGTGAGCGATTTC |
| GAD-A427P-F | GGATCGTGGTTCGGCCGGACTTTGGTATGAG |
| GAD-A427P-R | TCATACCAAAGTCCGGCCGAACCACGATCCG |
| Underlined letters are mutation sites. | |
为快速筛选出具有较高热稳定性的GAD突变体,建立了基于比色法的GAD突变体筛选流程:突变体接种于96深孔板中,每个孔装有1 mL含50 μg/mL Kan的LB培养基,37 ℃、200 r/min摇床培养12 h后,取100 μL培养液转接至上述相同培养体系中;37 ℃、200 r/min再次培养OD600至0.6–0.8后,加入IPTG至终浓度为0.5 mmol/L;25 ℃、150 r/min诱导8–12 h后离心收集菌体细胞,存放至–80 ℃超低温冰箱;冻融后用150 μL的PBS溶液(磷酸钠缓冲液,pH 7.5)将菌体重悬,加入终浓度为0.5 mg/mL的溶菌酶,于37 ℃静置1 h以破碎细胞;离心取上清液,并于55 ℃水浴中处理15 min,随后立即于冰浴中放置5 min;取处理后的酶液20 μL于96微量孔板中,加入200 μL含有20 mmol/L L-Glu的20 mmol/L醋酸钠缓冲液(50 μmol/L BCP,0.01 mmol/L PLP,pH 4.8),48 ℃、200 r/min振荡2 h后观察颜色变化。
1.5 野生酶和突变酶的表达与纯化挑取含重组表达质粒的单菌落并接种至5 mL含50 μg/mL Kan的LB培养基中,37 ℃、200 r/min条件下培养过夜。随后,以2% (V/V)的接种量转接至200 mL相同培养基中,继续培养至OD600值为0.6–0.8时,加入IPTG至终浓度为0.5 mmol/L,并于25 ℃、150 r/min条件下诱导8 h后离心收集菌体。
将收集的菌体细胞经20 mmol/L PBS (pH 7.5)离心洗涤2次后重悬于20 mL破胞缓冲液(20 mmol/L PBS,300 mmol/L氯化钠,20 mmol/L咪唑,pH 7.5),在冰浴条件下超声处理破碎菌体(超声功率为300 W,工作3 s,间歇6 s,超声15 min)。超声处理完毕后,细胞破碎液于8 000 r/min、4 ℃下离心30 min,收集上清液,即得含有GAD的粗酶液。粗酶液经0.45 μm滤膜过滤后采用Ni-NTA亲和层析获取目标重组蛋白,并分别采用SDS-PAGE和Bradford法测定纯化后的蛋白纯度及浓度。
1.6 酶活力测定GAD酶活力的测定参考Ueno等[26]所述方法:400 μL底物溶液(0.2 mol/L醋酸钠缓冲液,0.01 mmol/L PLP,100 mmol/L L-Glu,pH 4.8)与20 μL纯酶在37 ℃条件下反应15 min,然后加900 μL 0.2 mol/L NaHCO3溶液(pH 9.8)终止反应,采用HPLC法测定反应生成的GABA含量。
1.7 野生酶和突变酶酶学参数及热稳定性测定分别将纯化后的野生酶和突变酶在0–70 ℃水浴中孵育15 min,孵育结束后迅速放置在冰上冷却5 min,采用上述1.6方法测定酶活力。设未经温浴条件下所测得的酶活力为100%,将经过不同温度热处理后所测得的酶活力折合为相对剩余酶活力;并以温度为横坐标,以热处理后与处理前酶活力的比值为纵坐标作图,采用Origin 8.0软件进行Boltzmann S型函数拟合,并计算相对剩余酶活力降为50%时所对应的温度,即半失活温度(T5015)。
将野生酶和突变酶在55 ℃条件下分别孵育10–70 min,孵育结束后迅速放置在冰上冷却5 min,采用上述1.6方法测定酶活力。以时间为横坐标,以经过不同时间热处理后与处理前的酶活力的比值为纵坐标作图,通过Origin8.0软件拟合非线性方程y = exp(–kd·t),一阶速率常数(kd)经非线性回归确定,并计算相对剩余酶活力降低为50%时所对应的孵育时间,即半衰期(t1/2)。
采用0.2 mol/L醋酸钠缓冲液(含0.01 mmol/L PLP,pH 4.8)配制不同浓度(1–100 mmol/L)的底物(L-Glu)溶液,利用上述1.6方法测定酶活力。以底物浓度[S]对反应速率[V]作图,通过Origin 8.0软件非线性拟合计算相应的Km和Vmax值,根据kcat = Vmax/[E0] ([E0]为酶初始浓度,单位mmol/L)计算求得的kcat和催化效率kcat/Km。
1.8 野生酶和突变酶分子动力学模拟为进一步解释突变位点对蛋白构象的影响,通过计算野生酶及突变酶的分子动力学模拟轨迹中均方根偏差(Root mean square deviation,RMSD)和均方根涨落(Root mean square fluctuation,RMSF),来比较野生酶及突变酶整个系统势能的涨落情况和蛋白结构局部区域的灵活性。本研究以L. brevis CGMCC 1306的GAD晶体结构(PDB ID: 5GP4)为模板,利用FoldX软件构建突变酶的三维结构,并采用YASARA软件的Amber14力场分别对野生酶和突变酶在313 K下进行时长为10 ns的动力学模拟。模拟过程为:以PDB格式的野生酶和突变酶的三维结构作为分子动力学模拟的初始结构,经加氢处理后将晶体结构放置于10 Å×10 Å×10 Å的立方体盒子中,以0.98 g/L的密度填充水分子,并向体系中添加合适的抗衡离子(Na+/Cl–),使模拟系统呈电中性。范德华力相互作用的短程截断距离为7.86 Å。采用Particle Mesh Ewald方法计算长程静电相互作用。模拟结果数据的可视化及分析采用PyMOL软件分析。
2 结果与分析 2.1 突变文库的构建来源于超嗜热古细菌的T. kodakarensis是一种仅存于高温地热环境的严格厌氧菌,具有超高的最适生长温度(95 ℃)。Tomita等[27]报道来源于T. kodakarensis GAD的最适反应温度为85 ℃,在80 ℃和90 ℃的半衰期分别为10 h和5.5 h。L. brevis的GAD氨基酸序列与T. kodakarensis的GAD氨基酸序列具有44.74%的相似性,应用同源比对策略,选择来自T. kodakarensis GAD中Pro所对应的L. brevis GAD氨基酸位点作为突变位点,通过定点突变技术在L. brevis CGMCC 1306的GAD中引入Pro的位点分别为M70、K89、S162、A224、Y242、L261、G277、Y284、V306、S323、G364、G372、A427 (图 1),突变位点在L. brevis GAD (PDB ID: 5GP4)三维结构的位置如图 2所示。

|
| 图 1 L. brevis CGMCC 1306中GAD氨基酸序列与T. kodakarensis中GAD氨基酸序列对比结果图 Fig. 1 Sequence alignment of glutamate decarboxylase from L. brevis and T. kodakarensis. The amino acid sequences of GADs from L. brevis and T. kodakarensis were aligned with the DNAMAN program using default parameters. The proline residues which are in the amino acid sequences of T. kodakarensis are indicated with red square |
| |
|
| 图 2 L. brevis CGMCC 1306 GAD三维结构及突变位点 Fig. 2 Mutation sites in L. brevis CGMCC 1306 GAD. Loop, helix and sheet are colored in green, red and yellow, respectively. Mutation sites include: Met70, Lys89, Ser162, Ala224, Tyr 242, Leu261, Gly277, Tyr 284, Val306, Gly364, Gly372, Ala427 and Ser323. The coenzyme PLP is covalently attached to the catalytic Lys279 |
| |
为快速获取具有较高热稳定性的GAD突变体,本研究采用比色法对进行热处理后的GAD酶活力进行初筛[28]。BCP是一种酸碱指示剂,其在低于pH 5.0的条件下显示黄色,高于pH 6.8的条件中呈现紫色,在pH 5.0–6.8时为浅紫色或橙色。而L-Glu在GAD催化生成GABA的过程中,由于质子的消耗和碱性物质GABA的产生,使其在弱缓冲体系中的pH随着反应而发生变化。因此,可通过向缓冲液中添加一定浓度的BCP作为指示剂用于反应体系中GAD活性高低的定性分析。采用上述比色分析方法,利用96深孔板对13个突变体进行初步筛选。在所考察的体系中,与96微孔板中11号所在位置的GAD-WT相比(图 3),4号呈现出更深的紫色反应,其所对应的突变体为G364P。另外,质子化和去质子化形式的BCP的吸收光谱见图 4A。两种形式之间的吸光度的最大差异发生在590 nm,而进一步的OD590处吸收值也验证了颜色区别所体现的GAD活性变化(图 4B)。与GAD-WT相比,G364P在OD590处的吸收值明显高于GAD-WT。为此,选择G364P进行进一步热稳定性分析。

|
| 图 3 具有较高热稳定性的GAD突变体的初步筛选 Fig. 3 Colour changes of reaction system. Reaction mixtures contained 0.01 mmol/L PLP, 50 μmol/L BCP, 20 mmol/L L-Glu and 20 μL crude extract in 20 mmol/L acetate buffer (pH 4.8). Cell extracts from the same plasmid construct without insertion served as a negative control. 1: V306P; 2: Y284P; 3: A427P; 4: G364P; 5: G277P; 6: S323P; 7: G372P; 8: A261P; 9: K89P; 10: A242P; 11: GAD-WT; 12: A224P; 13: S162P; 14: M70P; NC: negative control |
| |

|
| 图 4 不同质子化状态的溴甲酚紫吸收光谱 Fig. 4 Absorption spectra of protonated and deprotonated forms of BCP. BCP concentration is 50 μmol/L. (A) Absorption spectra (350–650 nm) of protonated and deprotonated forms of BCP at different pH. (B) Increased absorbance at 590 nm is due to the deprotonation of indicator BCP via the glutamate decarboxylation reaction, and the mutants G364P exhibited higher absorbance than the GAD-WT at 590 nm |
| |
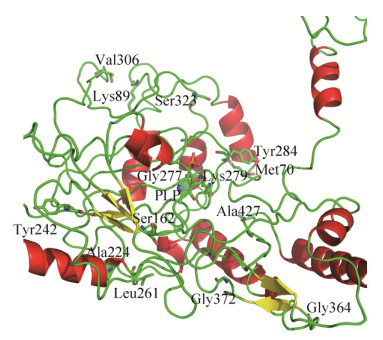
经Ni-NTA亲和层析纯化后的野生酶及突变酶的SDS-PAGE检测结果如图 5所示。纯化后的突变酶G364P及GAD-WT电泳条带清晰且单一,其分子量约为56 kDa,与理论分子量相一致。随后,按照1.6所述方法测定突变酶G364P与GAD-WT的t1/2和T5015。由图 6A可见,GAD-WT在55 ℃下的t1/2为23.6 min,而突变酶G364P则达到了43.0 min,是GAD-WT的1.82倍;在55 ℃条件下处理60 min后,GAD-WT的残余酶活力仅为24.5%,而突变酶G364P的残余酶活力仍有45.2%。这表明364位点发生的Pro替换增强了蛋白质的稳定性,对热失活有了更强的抗性,减缓了酶的热失活速率。由图 6B可见,GAD-WT的T5015为56.2 ℃,而突变酶G364P则提高到了61.5 ℃,比GAD-WT提高了5.3 ℃;在60 ℃条件下处理15 min后,GAD-WT的残余酶活力降为25.3%,而突变酶G364P仍保留65.9%的残余酶活力。

|
| 图 5 野生酶和突变酶的SDS-PAGE检测结果图 Fig. 5 SDS-PAGE analysis of GAD-WT and G364P. 1: protein marker; 2: GAD-WT; 3: G364P |
| |
|
| 图 6 野生酶和突变酶G364P的热稳定性 Fig. 6 Stability analysis of GAD-WT and G364P. (A) Thermal inactivation half-life (t1/2) of G364P and GAD-WT at 40 ℃. (B) Stability analysis of GAD-WT and G364P. Thermal inactivation of G364P and GAD-WT at different temperatures over 10 min (T5010). The error bars show standard deviation calculated for three replicated experiments |
| |
以L-Glu为底物,在pH 4.8、37 ℃的条件下,分别测定GAD-WT及突变酶G364P的米氏动力学常数,采用Hyperbola函数进行非线性拟合得到动力学参数,结果如表 2所示。由表 2可知,突变酶G364P的米氏常数Km值(45.718 mmol/L)比GAD-WT的Km值(41.283 mmol/L)高4.435 mmol/L,突变酶G364P对底物的亲和力略微下降。而突变酶G364P的kcat值(31.904 s–1)与GAD-WT的kcat值(30.461 s–1)较为接近,说明突变酶G364P的催化速率与GAD-WT相比相差不大。此外,突变酶G364P的kcat/Km为GAD-WT的0.86倍,即保留了原始GAD催化活性的86%。由此表明,突变酶G364P热稳定性得到提高的同时,并未显著降低酶的催化效率。
| Vm (U/mg) | Km (mmol/L) | kcat (s–1) | kcat/Km (L/(mmol·s)) | |
| GAD-WT | 0.597±0.015 | 41.283±2.279 | 31.904±0.015 | 0.773 |
| G364P | 0.570±0.021 | 45.718±3.602 | 30.461±0.021 | 0.666 |
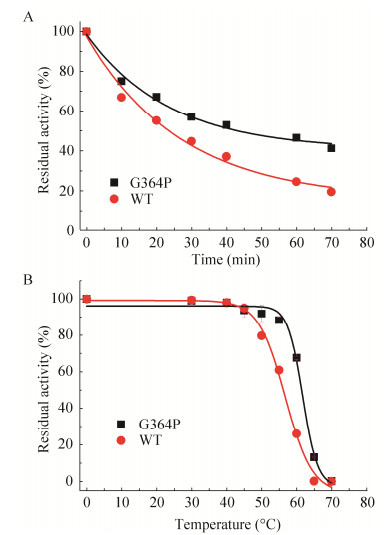
本研究采用YASARA软件进行分子动力学模拟,通过结合RMSD值(图 7A)与RMSF值(图 7B),分析引入的Pro突变位点对GAD热稳定性的贡献。由图 7A可知,随着模拟不断推进,GAD-WT和突变酶G364P的RMSD值起初有个明显上升的阶段,突变酶G364P的RMSD在1 ns处达到系统平衡,而野生酶的RMSD在4 ns处才达到系统平衡。平衡后野生酶的RMSD平均为(5.81±0.41) nm,突变酶G364P的RMSD平均为(3.80±0.69) nm。RMSD越低则分子构象越稳定。由此说明,突变酶G364P的蛋白质构象位移较小,酶整体柔性有所降低。
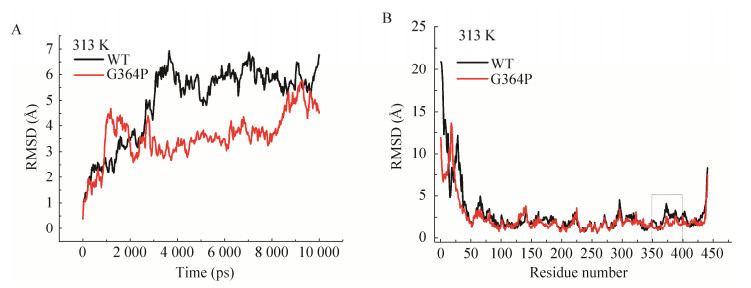
|
| 图 7 野生酶与突变酶G364P的分子动力学模拟分析 Fig. 7 MD simulation analysis of G364P and GAD-WT for 10 ns at 313 K using YASARA |
| |
对GAD-WT和突变酶G364P进行时长10 ns的分子动力学模拟,计算GAD-WT和突变酶G364P每个氨基酸的RMSF值,可反映模拟过程中氨基酸的涨落变化状况。GAD-WT和突变酶G364P的RMSF曲线如图 7B所示。由图 7B可知,突变酶G364P和野生酶柔性最大的区域均为蛋白质的N端,突变酶G364P在蛋白质N端以及突变位点的Loop区域RMSF值小于野生型。由图 2可知,G364位于GAD的Loop区域并且远离活性中心,该Loop区域在热运动作用下易于形变,显著影响蛋白质结构的稳定性。在该区域引入Pro后,能增加该区域的刚性,降低蛋白质去折叠的骨架熵。
3 讨论本研究基于比色法可快速筛选经热处理后仍具有高活力的GAD突变酶,可在相对短的时间内得到性质改善的突变酶,节省时间成本,对指导实验设计具有积极作用。影响酶的热稳定性因素众多,酶分子热稳定的内在机制主要包括静电相互作用[23]、疏水相互作用[29]、氢键[30]、二硫键[22]、芳香环的相互作用[31]、氨基酸组成等[24,32]。为深入探讨突变酶稳定性提高的机制,本研究利用在线服务器(http://pic.mbu.iisc.ernet.in/)进一步对野生酶及突变酶G364P的364位氨基酸残基分别进行了分子间相互作用的模拟分析。选取疏水相互作用(距离设为5 Å)和氢键选项,具体分析方法参照参考文献[33],模拟结果如图 8所示。GAD野生酶的364位氨基酸为Gly,其O原子与Phe366侧链的N原子形成空间距离为2.9 Å的氢键;突变酶G364P的364位氨基酸是Pro,其O原子与Phe366侧链的N原子同样形成空间距离仍为2.9 Å的氢键。突变酶G364P与野生酶相比,364位氨基酸的氢键数量没有发生变化,说明对于突变酶G364P热稳定性的提高,氢键贡献不大。从图 8 C、D中所知,野生酶364位点的Gly并不与其相邻的氨基酸残基形成疏水相互作用,但在该位点引入Pro后,突变酶G364P的364位点的Pro与363位点的Leu之间形成疏水相互作用。Pace等[34]研究了疏水相互作用和氢键对22个不同分子量大小的蛋白质(氨基酸残基介于36–534之间)稳定性的影响,疏水相互作用对这22个蛋白质的稳定性平均贡献为60%±4%,氢键对这22个蛋白质的稳定性平均贡献为40%±4%。因此,在364位点引入Pro后,突变酶G364P增加了氨基酸残基之间的疏水相互作用,进而使突变酶G364P的热稳定性得到一定的提高。

|
| 图 8 突变酶G364P与GAD-WT的364位点附近分子间相互作用模拟 Fig. 8 Molecular interaction simulation between the mutant enzyme G364P and GAD-WT near the 364 site. (A) In the GAD-WT, Glu 364 was involved in hydrogen bond with the neighbouring Phe366. (B) In the G364P, Pro 364 was involved in hydrogen bond with the neighbouring Phe366. (C) Leu 363 of wild type was involved in hydrophobic interaction with the neighbouring hydrophobic amino acids Leu360, Phe366, Ile438, Leu441 and Ile445. (D) Leu 363 of mutant G364P was involved in hydrophobic interaction with the neighbouring hydrophobic amino acids Leu360, Phe366, Ile438, Leu441 and Pro 364 |
| |
| [1] | Leventhal AG, Wang YC, Pu ML, et al. GABA and its agonists improved visual cortical function in senescent monkeys. Science, 2003, 300(5620): 812–815. |
| [2] | Inoue K, Shirai T, Ochiai H, et al. Blood-pressure-lowering effect of a novel fermented milk containing γ-aminobutyric acid (GABA) in mild hypertensives. Eur J Clin Nutr, 2003, 57(3): 490–495. DOI: 10.1038/sj.ejcn.1601555 |
| [3] | Kai KL, Ruusuvuori E, Seja P, et al. GABA actions and ionic plasticity in epilepsy. Curr Opin Neurobiol, 2014, 26: 34–41. |
| [4] | Diana M, Quílez J, Rafecas M. Gamma-aminobutyric acid as a bioactive compound in foods: a review. J Funct Foods, 2014, 10: 407–420. DOI: 10.1016/j.jff.2014.07.004 |
| [5] | Lammens TM, Franssen MCR, Scott EL, et al. Synthesis of biobased N-methylpyrrolidone by one-pot cyclization and methylation of γ- aminobutyric acid. Green Chem, 2010, 12(8): 1430–1436. DOI: 10.1039/c0gc00061b |
| [6] | Ueno H. Enzymatic and structural aspects on glutamate decarboxylase. J Mol Catal B: Enzym, 2000, 10(1/3): 67–79. |
| [7] | Huang J, Fang H, Gai ZC, et al. Lactobacillus brevis CGMCC 1306 glutamate decarboxylase: crystal structure and functional analysis. Biochem Biophys Res Commun, 2018, 503(3): 1703–1709. |
| [8] | Lyu CJ, Zhao WR, Hu S, et al. Physiology-oriented engineering strategy to improve gamma-aminobutyrate production in Lactobacillus brevis. J Agric Food Chem, 2017, 65(4): 858–866. DOI: 10.1021/acs.jafc.6b04442 |
| [9] | Foster JW. Escherichia coli acid resistance: tales of an amateur acidophile. Nat Rev Microbiol, 2004, 2(11): 898–907. |
| [10] | Li H, Cao Y. Lactic acid bacterial cell factories for gamma-aminobutyric acid. Amino Acids, 2010, 39(5): 1107–1116. DOI: 10.1007/s00726-010-0582-7 |
| [11] | Dhakal R, Bajpai VK, Baek KH. Production of gaba (γ-Aminobutyric acid) by microorganisms: a review. Braz J Microbiol, 2012, 43(4): 1230–1241. DOI: 10.1590/S1517-83822012000400001 |
| [12] | Huang J, Mei LH, Sheng Q, et al. Purification and characterization of glutamate decarboxylase of Lactobacillus brevis CGMCC 1306 isolated from fresh milk. Chin J Chem Eng, 2007, 15(2): 157–161. DOI: 10.1016/S1004-9541(07)60051-2 |
| [13] |
Xia J. Breeding of γ-aminobutyric acid-producing Lactobacillus and optimization of fermentation conditions[D]. Hangzhou: Zhejiang University, 2016 (in Chinese). 夏江.产γ-氨基丁酸的乳酸菌菌株选育及其发酵条件优化[D].杭州: 浙江大学, 2006. |
| [14] | Fan EY, Huang J, Hu S, et al. Cloning, sequencing and expression of a glutamate decarboxylase gene from the GABA-producing strain Lactobacillus brevis CGMCC 1306. Ann Microbiol, 2012, 62(2): 689–698. |
| [15] | Yu K, Lin L, Hu S, et al. C-terminal truncation of glutamate decarboxylase from Lactobacillus brevis CGMCC 1306 extends its activity toward near-neutral pH. Enzyme Microb Technol, 2012, 50(4/5): 263–269. |
| [16] | Yang HQ, Liu L, Li JH, et al. Rational design to improve protein thermostability: recent advances and prospects. ChemBioEng Rev, 2015, 2(2): 87–94. DOI: 10.1002/cben.v2.2 |
| [17] | Durrenberger M, Ward TR. Recent achievments in the design and engineering of artificial metalloenzymes. Curr Opin Chem Biol, 2014, 19: 99–106. |
| [18] | Jones BJ, Lim HY, Huang J, et al. Comparison of five protein engineering strategies for stabilizing an α/β-hydrolase. Biochemistry, 2017, 56(50): 6521–6532. |
| [19] | Max KEA, Wunderlich M, Roske Y, et al. Optimized variants of the cold shock protein from in vitro selection: structural basis of their high thermostability. J Mol Biol, 2007, 369(4): 1087–1097. DOI: 10.1016/j.jmb.2007.04.016 |
| [20] | Benedix A, Becker CM, de Groot BL, et al. Predicting free energy changes using structural ensembles. Nat Methods, 2009, 6(1): 3–4. |
| [21] | Reetz MT, Carballeira JD, Vogel A. Iterative saturation mutagenesis on the basis of B factors as a strategy for increasing protein thermostability. Angew Chem Int Ed, 2006, 118(46): 7909–7915. DOI: 10.1002/(ISSN)1521-3757 |
| [22] | Xie DF, Fang H, Mei JQ, et al. Improving thermostability of (R)-selective amine transaminase from Aspergillus terreus through introduction of disulfide bonds. Biotechnol Appl Biochem, 2018, 65(2): 255–262. |
| [23] | Strickler SS, Gribenko AV, Gribenko AV, et al. Protein stability and surface electrostatics: a charged relationship. Biochemistry, 2006, 45(9): 2761–2766. |
| [24] | Huang J, Jones BJ, Kazlauskas RJ. Stabilization of an α/β-hydrolase by introducing proline residues: salicylic acid binding protein 2 from tobacco. Biochemistry, 2015, 54(28): 4330–4341. |
| [25] |
Chen XX, Li D, Lü JX, et al. Determination of γ-aminobutyric acid and glutamic acid in human cerebrospinal fluid by high performance liquid chromatography.
Chin J Chromatogr, 1997, 15(3): 237–239.
(in Chinese). 陈希贤, 李东, 吕建新, 等. 高效液相色谱法测定人脑脊液中γ-氨基丁酸和谷氨酸. 色谱, 1997, 15(3): 237-239. |
| [26] | Ueno Y, Hayakawa K, Takahashi S, et al. Purification and characterization of glutamate decarboxylase from Lactobadllus brevis IFO 12005. Biosci Biotechnol Biochem, 1997, 61(7): 1168–1171. DOI: 10.1271/bbb.61.1168 |
| [27] | Tomita H, Yokooji Y, Ishibashi T, et al. An archaeal glutamate decarboxylase homolog functions as an aspartate decarboxylase and is involved in β-alanine and coenzyme a biosynthesis. J Bacteriol, 2014, 196(6): 1222–1230. DOI: 10.1128/JB.01327-13 |
| [28] | Yu K, Hu S, Huang J, et al. A high-throughput colorimetric assay to measure the activity of glutamate decarboxylase. Enzyme Microb Technol, 2011, 49(3): 272–276. DOI: 10.1016/j.enzmictec.2011.06.007 |
| [29] | Gromiha MM, Pathak MC, Saraboji K, et al. Hydrophobic environment is a key factor for the stability of thermophilic proteins. Proteins, 2013, 81(4): 715–721. DOI: 10.1002/prot.v81.4 |
| [30] | Shirley BA, Stanssens P, Hahn U, et al. Contribution of hydrogen bonding to the conformational stability of ribonuclease T1. Biochemistry, 1992, 31(3): 725–732. DOI: 10.1021/bi00118a013 |
| [31] | Burley SK, Petsko GA. Aromatic-aromatic interaction: a mechanism of protein structure stabilization. Science, 1985, 229(4708): 23–28. DOI: 10.1126/science.3892686 |
| [32] | Deckert G, Warren PV, Gaasterland T, et al. The complete genome of the hyperthermophilic bacterium Aquifex aeolicus. Nature, 1998, 392(6674): 353–358. DOI: 10.1038/32831 |
| [33] | Tina KG, Bhadra R, Srinivasan N. PIC: protein interactions calculator. Nucleic Acids Res, 2007, 35(S2): W473–W476. |
| [34] | Pace CN, Fu HL, Fryar KL, et al. Contribution of hydrophobic interactions to protein stability. J Mol Biol, 2011, 408(3): 514–528. DOI: 10.1016/j.jmb.2011.02.053 |
 2019, Vol. 35
2019, Vol. 35