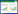中国科学院微生物研究所、中国微生物学会主办
文章信息
- 高德英, 曹佳雯, 孙小宝, 周可鑫, 张铁涛, 王谦
- Gao Deying, Cao Jiawen, Sun Xiaobao, Zhou Kexin, Zhang Tietao, Wang Qian
- 基于异肽键连接的分子粘合剂研究进展
- Advances in isopeptide bond-mediated molecular superglue
- 生物工程学报, 2019, 35(4): 607-615
- Chinese Journal of Biotechnology, 2019, 35(4): 607-615
- 10.13345/j.cjb.180303
-
文章历史
- Received: July 20, 2018
- Accepted: September 14, 2018
2. 中国农业科学院 特产研究所,吉林 长春 130112;
3. 浙江大学 动物科学学院,浙江 杭州 310012
2. Institute of Special Wild Economic Animals and Plants, Chinese Academy of Agricultural Sciences, Changchun 130112, Jilin, China;
3. College of Animal Sciences, Zhejiang University, Hangzhou 310012, Zhejiang, China
蛋白质因其高效性和特异性在工业催化、食品制造、医药等领域应用广泛。然而,由于蛋白质对热、抑制剂等耐受能力较差,导致易降解、稳定性较差,限制了其在工业加工和特定环节中的应用。因此,如何提高蛋白质的稳定性成为近年来的研究热点。传统提升酶稳定性的方法主要包括筛选耐热突变体、化学修饰、添加稳定剂、改变离子浓度以及包被技术等。这些方法虽然能够提高蛋白质的热稳定性,但往往盲目性较大,收效甚微[1]。早前,有研究人员在细菌、植物、真菌和动物上发现一类环状蛋白质。与常规线性蛋白质相比,环状蛋白质具有更高的热稳定性和结构稳定性[2-3]。
通常,蛋白质稳定性是由蛋白质分子中氨基酸主链及侧链之间的相互作用和蛋白质分子与周围溶液环境作用的综合效应共同决定,包括疏水作用力、盐键、氢键、范德华力以及在三级结构中形成的亲水表面及内部疏水核心等。因此,蛋白质分子的稳定性与其氨基酸组成及空间构象有着重要关联[4-5]。
异肽键(Isopeptide bond)是指蛋白质序列中氨基酸之间至少有一个非α位的氨基或羧基参与形成的酰胺键,广泛存在于革兰氏阳性菌的菌毛蛋白中。牛津大学的Mark Howarth团队围绕异肽键和蛋白质分子环化开展了系统的研究工作。他们重点分析了环化蛋白的热稳定性及其稳定性机制,进一步优化了异肽键的形成条件,发现异肽键在极短的时间(小于10 min)即可形成,且不需要特定的理化环境和催化剂[6-8]。此外,他们还对利用热变性纯化环化蛋白进行了探索[9]。之后,研究人员进一步采用SpyTag/SpyCatcher系统设计了多种大分子的拓扑结构,并研究其分子稳定性机制[10]。由于异肽键具备良好的热稳定性和抗逆能力,近年来,人们探索了异肽键的形成机制,并利用异肽键作为分子粘合剂在合成疫苗、细菌纳米生物反应器、人工代谢通路及蛋白质水凝胶等领域的应用开展了一系列研究工作。
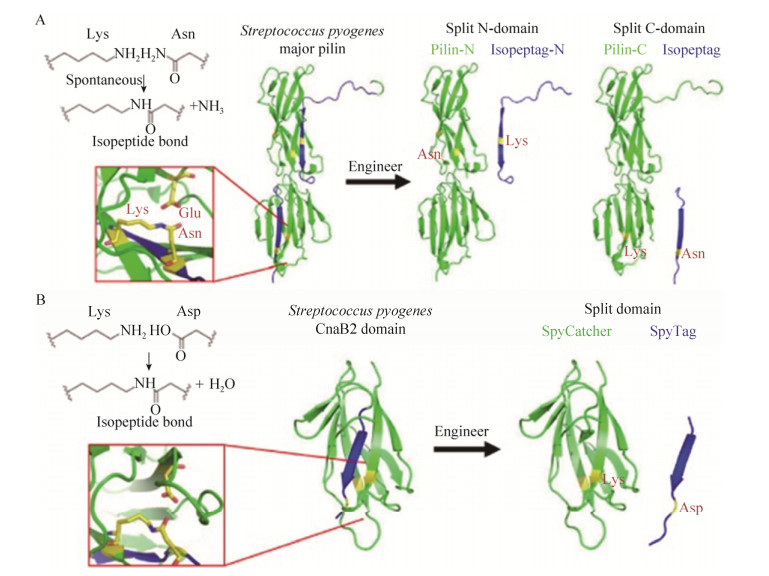
1 异肽键分子粘合剂的类型自然界中发现的异肽键通常是通过谷氨酰胺转移酶[11]、泛素连接酶[12]或转肽酶[13]催化形成。通过Lys侧链氨基与Asn侧链酰胺基或Asp侧链羧基自发结合形成的共价键,具有较高的特异性和稳定性[6-7]。人们最早在HK97噬菌体衣壳蛋白组装过程中发现可以自发形成的异肽键[14]。2007年,Kang等[15]首次在革兰氏阳性菌酿脓链球菌Streptococcus pyogenes菌毛蛋白(Pilin) Spy0128中发现分子内可自发形成的异肽键。根据物种来源和肽段性质的差异,目前常见的异肽键分子粘合剂主要分为isopeptag-N/pilin-N (PilinRing-N)、isopeptag/pilin-C (PilinRing-C)、SnoopTag/ SnoopCatcher (SnoopRing)和SpyTag/SpyCatcher (SpyRing)。
PilinRing作为第一代分子粘合剂最初在革兰氏阳性菌酿脓链球菌菌毛蛋白(Pilin) Spy0128 (PDB登录号:3B2M)中观察并分离得到[16]。Spy0128包含两个异肽键结构域,即携带Asn残基的Pilin-N和携带Lys残基的N端结构域以及携带Lys残基的Pilin-C和携带Asn残基的C端结构域(图 1)。之后,研究人员进一步发现来自于肺炎链球菌S. pneumoniae粘着物RrgA (PDB登录号:2WW8)的SnoopRing,在SnoopTag和SnoopCatcher肽段上分别携带Lys残基和Asn残基[17]。最近,研究人员从酿脓链球菌S. pyogenes纤连蛋白CnaB2 (PDB登录号:2X5P)中发现第二代分子粘合剂SpyRing[7, 18]。其中,SpyTag和SpyCatcher肽段分别携带形成异肽键的Asp残基和Lys残基。由于SpyTag/SpyCatcher形成的异肽键结合稳定、肽链较短且反应迅速,引起研究人员的广泛关注。
2 异肽键的形成机制从异肽键的形成机制来看,异肽键分子粘合剂的反应可以分为脱氨和脱水两种形式。PilinRing、SnoopRing等第一代分子粘合剂的形成主要包括两个过程:首先未质子化的Lys氨基组以N为基础,亲和攻击Asn的Cγ,Glu作为反应催化剂协同两个质子转移;之后,自发形成异肽键并释放氨分子[19]。第二代分子粘合剂SpyRing形成异肽键的机制与PilinRing相似,首先是Lys亲核性攻击Asp,随后Glu协同质子转移,最后形成酰胺键并释放出水分子[20]。
异肽键分子粘合剂的自发反应十分高效。以SpyTag和SpyCatcher为例,两个肽段分别以10 μmol/L的浓度进行混合,反应半衰期约为1 min (k=1.4×103/(mol/L·s))[20]。为了探究异肽键在复杂环境中的形成情况,研究人员测试了环境因素(不同pH、温度、时间、缓冲液)及两个短肽的比例对异肽键形成的影响。结果显示,异肽键的形成不需要特殊的理化环境,SpyTag和SpyCatcher两个肽段在25 ℃、pH 7.0的条件下反应15 min的结合率达到80%以上[7],且增加SpyTag肽段的摩尔浓度有助于反应的彻底进行[8]。
为了验证Lys、Asn/Asp和Glu是形成异肽键的关键残基,Howarth团队对这几个氨基酸依次进行点突变(KA/NA/DA/EQ),发现当其中的任意一个氨基酸发生突变,均不能形成异肽键[7, 9, 21-22]。该突变结果从侧面验证了Lys、Asn/Asp和Glu是异肽键形成机制中的关键氨基酸。
此外,若将几种不同的肽段混合,它们相互之间是否会产生交叉反应呢?研究表明,SpyRing与SnoopRing相互之间并无交叉反应[8]。虽然SnoopTag含有Lys残基,SpyTag带有Asp残基,但两个肽段并不包含发挥质子转移作用的Glu残基,因此无法形成异肽键。近期,本课题组通过实验测试了isopeptag-N/pilin-N (PilinRing)和Spying及isopeptag-N/pilin-N (PilinRing)和SnoopRing之间同样没有发生交叉反应。关于isopeptag/pilin-C (PilinRing)与其他肽段是否产生交叉反应仍有待验证。
3 异肽键介导的分子环化环肽(Cyclic peptide)是一类天然存在的具有环状结构的多肽分子[1-2]。由于环化结构具有良好的分子稳定性,人们采用基因工程、蛋白质工程等方法研发出多种分子环化技术,如蛋白质反式剪接(Protein trans-splicing,PTS)[23-24]、内含肽介导的蛋白表达连接(Expressed protein ligation,EPL)[25]、转肽酶(Sortase)介导的转肽作用(Sortagging)[26-27]等。虽然这些环化方式可以提高蛋白的稳定性,但实现上述连接方式通常需要复杂的反应条件或特定催化剂的参与,且反应效率较低,甚至存在剪接的不可控性、缺乏通用性、产物复杂、得率较低等问题[28-29]。
超强分子粘合剂由于其特异性、稳定性且反应快速的优势,为蛋白质环化开辟了新思路。近年来,利用异肽键介导的分子粘合剂对蛋白质进行分子修饰,取得了较大进展(表 1)。2015年,Schoene等[9]将SnoopTag/SnoopCatcher和isopeptag/pilin-C系统应用于β-内酰胺酶的分子环化,发现野生酶在55 ℃条件下发生聚沉、可溶性蛋白显著减少,导致活性快速丧失,而环化蛋白活性则几乎不受影响。差示扫描量热法(Differential scanning calorimetry)分析线性和环化蛋白的溶解温度,与线性蛋白相比,环化蛋白的Tm值显著上升。野生植酸酶在55 ℃以上的环境中加热10 min可溶性蛋白活性开始降低,而SpyTag/SpyCatcher环化的植酸酶在90 ℃才开始略微下降,且活性影响较小。圆二色谱(Circular dichroism)检测结果显示,与野生型植酸酶相比,环化后的植酸酶二级结构稳定性显著增加。Wang等[30]将SpyTag/ SpyCatcher用于介导地衣聚糖酶的环化,经热处理后,环化蛋白的活性显著高于线性蛋白。此外,有研究发现SpyTag/SpyCatcher环化增强了萤火虫荧光素酶的热稳定性,同时其生物活性不受影响[32]。以上各研究团队的结果可以得出同样的结论,即环化后蛋白的热稳定性、抗逆能力显著提高。
| Applications | Targets/platforms | Molecular glues | Effects | References |
| Enhanced thermostability | Lichenase | SpyTag/SpyCatcher | Retained 80% of the initial activity after treatment at 100 ℃ for 10 min | [30] |
| Phytase | SpyTag/SpyCatcher | Retained 80% of the initial activity after treatment at 100 ℃ for 10 min | [9] | |
| β-lactamase | Pilin-C/Isopeptag SnoopTag/SnoopCatcher | Retained 60% of the initial activity after treatment at 100 ℃ for 10 min Retained 70% of the initial activity after treatment at 100 ℃ for 10 min | [9] | |
| Xylanase | SpyTag/SpyCatcher | Retained 67% of the initial activity after treatment at 100 ℃ for 10 min | [31] | |
| Luciferase | SpyTag/SpyCatcher | Thermal inactivation value (T50) increased by 10.1 ℃ | [32] | |
| Protein topology engineering | Elastin-like protein (ELP) | SpyTag/SpyCatcher | Circular, tadpole, star and H-shaped | [10] |
| ELP/p53dim | SpyTag/SpyCatcher | Cyclic monomers, catenanes and olypseudorotaxanes | [33-34] | |
| Protein purification | eGFP | SpyTag/SpyCatcher and Sortase A | 80% protein were captured within 10 min | [35] |
| Xylanase-lichenase-ELP chimera | SpyTag/SpyCatcher | 95% protein were purified after 2-cycles of inverse transition cycling (ITC) | [36] | |
| Phytase/β-lactamase | SpyTag/SpyCatcher | Heat-induced, simple and fast | [32] | |
| Synthetic vaccine | Virus-like particles/ CIDR and Pfs25 | SpyTag/SpyCatcher | Antibody response after only a single Immunization without adjuvant | [21] |
| αDEC205-Sc-OVA8-ED3 | SpyTag/SpyCatcher | Efficient in inducing both T and B cell responses | [37] | |
| Synthetic hydrogel | Folded globular domains (GB1 and FnIII domain) | SpyTag/SpyCatcher | Soft, stable and biologically compatible | [38] |
| B12-denpendent Light-sensing hydrogel | SpyTag/SpyCatcher | Self-assemble in the dark and disassemble rapidly on light exposure | [39] | |
| Bacterial nanobiological reactor | MS2 phage-based nanoreactor | SpyTag/SpyCatcher | 60% increase of indigo production in vivo | [40] |
| OmpA-based outer Membrane vesicles (OMVs) | SpyTag/SpyCatcher | Enhanced long-term enzymatic stability by 90% after 7 d in vitro Phosphotriesterase-loaded OMVs improved the enzyme production and stability against freezing and thawing | [41] | |
| Biofilm integrated nanofiber display (BIND) | SpyTag/SpyCatcher | Versatile platform for stable and highly efficient immobilization | [42] |
基于环化蛋白的耐热性,环化方式可以用于鉴定目标蛋白的存在及纯化目标蛋白。与一般蛋白纯化方式相比,该方法虽然会损失部分酶活性,但操作简便快速,检测及纯化效果依然可观[9]。本实验室长期致力于工业酶的分子改良,已成功构建了多种不同构象的酶分子,并对环化蛋白的热稳定性及结构稳定性进行研究,为后续研发多功能复合酶打下了良好的基础。然而,利用异肽键连接的分子粘合剂并非“无痕” (Not traceless)[7],对蛋白质进行分子环化时需要在其末端加上SpyTag/SpyCatcher肽段。虽然实际操作时通常在各功能结构域之间加入连接肽来减少空间构象造成的相互干扰,但目前关于SpyTag/SpyCatcher肽段的插入是否会影响蛋白催化性质并没有一致的结论。有研究表明,环化前后荧光素酶和β-内酰胺酶的Km值没有显著变化[9, 32],而环化前后的木聚糖酶和地衣聚糖酶的动力学参数则表现出显著性差异[30, 36]。此外,异肽键并未达到接近扩散极限(Diffusion limit)的反应速率(如生物素-链霉亲和素)[43]。SpyTag/SpyCatcher仅在微摩尔级和更高浓度肽段间发生反应,从而限制了其应用范围。
4 基于异肽键分子粘合剂的拓扑结构21世纪以来,生物技术和高分子化学均取得了很大发展。然而,要实现对高分子的序列、尺寸、组装过程和拓扑结构的全面精确控制仍是人们面临的一大挑战。近期,利用可基因编码的SpyTag/SpyCatcher合成类弹性蛋白(Elastin-like proteins,ELPs),为蛋白质拓扑结构的开发提供了新思路。
北京大学的张文彬研究员将SpyTag和SpyCatcher基因插入类弹性蛋白基因中,以便在原位进行转录后的拓扑修饰。利用带有SpyTag和SpyCatcher不同标签的弹性蛋白在体外组合,获得蝌蚪型、三臂星型和H型等多种蛋白拓扑结构[10]。研究人员同时探讨了不同表达速度对环化蛋白大小的影响,表达速度较慢时只形成单环,而快速表达时,既有单环也有多环的形成,这一结果为构建目标环化蛋白提供了依据。随后张文彬团队将SpyTag/SpyCatcher用于蛋白质索烃的细胞合成,其研究结果表明,较之线性突变及循环单体蛋白,蛋白质索烃结构具有更好的稳定性[34]。最近,该团队进一步开发出高效的、基因编码的、机械联锁的SpyX模块(AXB和BXA),用于体内工程蛋白质的拓扑结构[33]。包含SpyX模块的融合蛋白的表达导致了多种机械联锁蛋白拓扑的形成,包括蛋白质链状结构、专性二聚体和恒星蛋白。在蛋白质工程中使用SpyX模块有很多优势:形成的专性二聚体在较低浓度下也不会电离;可以简单地通过直接表达来形成星型蛋白质;可以提高蛋白质的稳定性,如增强抗胰蛋白酶酶解的能力。SpyX模块扩大了蛋白质拓扑结构的范围,成为进一步设计蛋白质功能特性的通用平台。该团队基于SpyTag/ SpyCatcher和固有无序蛋白(Intrinsically disordered proteins,IDPs)高电荷密度的属性,进一步改造天然SpyCatcher短肽,使其带有大量电荷,发现带有大量电荷的SpyCatcher (-)短肽仍然可以与SpyTag形成异肽键[44]。此外,SpyCatcher (-)短肽还能络合相反电荷的聚合物,实现展开和折叠态双态的转变等功能,大大扩宽了生物材料体内外的合成。
5 异肽键分子粘合剂的应用 5.1 合成疫苗合成疫苗是疫苗开发历史上的一个里程碑,与传统减毒或灭活疫苗相比,合成疫苗更加安全。第一代合成疫苗主要是基于DNA重组技术和基因操纵,不仅耗费大量时间,而且成本高昂[37]。
病毒样颗粒(Virus-like particle,VLPs)是非传染性的自组装纳米粒子,在医学和纳米技术中发挥重要作用,特别是在疫苗接种方面。然而,通过基因融合或化学修饰,对目标抗原进行修饰,往往会导致衣壳的错误组装或抗原错误折叠,从而阻碍保护性免疫的产生。近年来,Howarth团队将分子粘合剂SpyTag/SpyCatcher用于疫苗接种,通过将SpyCatcher融合了噬菌体蛋白AP205 (AP205 CP3)、SpyTag与疟疾抗原(CIDR和Pfs25)混合后与SpyCatcher-AP205共价耦合,可以显著提高抗原免疫原性[21]。Singh等[45]利用SpyTag- Pfs48/45与SpyCatcher-AP205共价结合,同样使抗原免疫原性得到提高。该团队进一步验证了表皮生长因子受体和端粒酶中与癌症相关的肽链的耦合。注射用疟疾抗原修饰的SpyCatcher-VLPs,在一次免疫后就能有效地诱导抗体反应[21]。此外,通过多聚体支架或类病毒颗粒增强细胞信号也可以增强免疫反应不佳的抗原的免疫应答[27]。SpyTag/SpyCatcher蛋白质组装技术产生的新疫苗在发挥个体功能的同时,还可以有效地诱导T细胞和B细胞反应。这种蛋白质组装策略可能对高通量抗原筛选或疫苗的快速产生具有重要理论与实践意义[37, 46]。另有研究人员将SpyTag/SpyCatcher用于设计抗体标记,成功实现在亚纳米级范围内展示细胞毒性位点的抗体药物复合体,从而扩展了特定抗体的结合位点[47]。
5.2 细菌纳米生物反应器迄今为止,研究涉及的所有革兰氏阳性或阴性细菌,都是从其表面产生外膜囊泡(Outer membrane vesicles,OMVs)[48],致病细菌菌株毒性因子通过外膜囊泡感染宿主细胞。基于这种感染途径,研究人员结合分子粘合剂SpyTag/SpyCatcher系统开发了一种使细菌能够同时生产、包装和分泌活性酶的新方法。OmpA是一种高度表达的细菌重组蛋白,与细菌膜上小分子的运输有关[49]。来自缺陷短波单胞菌Brevundimonas diminuta的磷酸三酯酶(Phosphotriesterase,PTE)含有Zn/Zn活性位点,能通过水解反应将芳基磷酸酯转化为无毒的二基磷酸和芳醇,从而分解有机磷酸酯。然而,磷酸三酯酶存在水解效率低、易失活等缺陷,制约了其在有机磷降解中的应用。针对这一问题,研究人员设计了OmpA-SpyTag和SpyCatcher-PTE两种构象蛋白,通过异肽键实现共价结合。这种共价表达的途径不仅提高了PTE的生产水平,也提高了其稳定性,为有机磷污染区域的环境修复提供了一个很好的模型[44]。这项研究的结果还可应用于设计类似的蛋白质包装策略,用于不同的药物输送、医学诊断等。Giessen等[40]将SpyTag/SpyCatcher用于构建MS2噬菌体衣壳的体内封装系统,为生成高度稳定的多酶纳米反应器提供了一种简单而有效的方法。
研究人员将SpyTag/SpyCatcher系统用于工程催化生物膜,利用大肠杆菌的纤维系统来创建一个功能性的纳米纤维网络,进行酶的共价固定。将一个α-淀粉酶与SpyCatcher进行重组,并将其固定在大肠杆菌的纤维上与SpyTag进行共价结合。这种酶化的改良生物膜可以在广泛的pH值和有机溶剂条件下维持其活性。此外,与其他依赖于高细胞代谢的基于生物膜的催化剂相比,改良后的生物膜在细胞死亡后仍然保持活跃状态。这一研究将在绿色生物催化领域发挥广泛作用,如药物合成、废水中的药物分解、地下水中污染物的去除和生物能源的生产等[37]。
5.3 水凝胶由于蛋白质水凝胶的生物友好性、潜在的生物医学应用以及精确控制蛋白质结构和功能的能力,以蛋白质为基础的水凝胶已经引起了研究人员的极大关注。由合成聚合物或天然生物分子组成的传统的水凝胶,机械性质较弱,制约其在不同领域的广泛应用。近年来,基于分子粘合剂SpyTag/ SpyCatcher良好的特异性和稳定性,研究人员将其用于蛋白质水凝胶的研究。Gao等[38]设计了SpyTag/SpyCatcher介导的串联模块化蛋白质水凝胶。结果表明,带有SpyTag/SpyCatcher模块化的蛋白质可以迅速相互反应,从而形成柔软的、化学交联的蛋白质水凝胶。该团队成功地使用这些水凝胶来封装和培养3D的人类肺纤维细胞并将其作为药物输送的容器。Wang等[39]利用SpyTag/ SpyCatcher研发出腺苷钴胺素(B12)依赖的光反应蛋白水凝胶,并将其应用于控制干细胞蛋白质的释放,将刺激反应的蛋白质直接组装到水凝胶中,提供了一种设计可动态调节材料的通用策略。
5.4 人工代谢通路随着生物工程及酶工程的迅速发展,酶在工业生产中的应用越来越广泛。工业酶普及的同时,酶稳定性和催化活性低的问题不容忽视。针对这一问题,研究人员利用分子粘合剂SpyTag/SpyCatcher来设计人工代谢通路,提高工业酶的应用价值。蛋白质支架可以实现多酶复合体的超分子结构,但是蛋白质支架需要特定的结合域,存在不同结合域的交叉反应及不同物种亲和力不高的问题[50],SpyTag/ SpyCatcher的特异性结合及异肽键的稳定性可以改善蛋白质支架的亲和力和稳定性。此外,在还原条件下共价二硫键很容易分离,是可逆反应[16]。而分子粘合剂SpyTag/SpyCatcher是基因编码的不可逆的共价结合,更有利于构建稳定的多酶复合体。
6 结语随着生物技术的快速发展,有关分子粘合剂的研究成果丰硕。近年来,研究人员相继发现了PilinRing、SnoopRing及SpyRing几种类型的分子粘合剂,并针对异肽键的形成机理开展了深入研究,为后续分子粘合剂的应用提供了理论依据。绝大多数蛋白质的稳定性较差,制约了其在工业催化、食品制造、医药领域的应用。长期以来,如何有效提升蛋白质的稳定性一直是科研工作者关注的焦点。基于异肽键连接的分子粘合剂具备良好的稳定性及特异性,使其在多个领域具有广阔的应用前景。此外,可基因编码、修饰的SpyTag/ SpyCatcher系统,为人工多价疫苗、纳米生物反应器、蛋白质拓扑结构的开发等提供了新思路。
| [1] | Aboye TL, Camarero JA. Biological synthesis of circular polypeptides. J Biol Chem, 2012, 287(32): 27026–27032. DOI: 10.1074/jbc.R111.305508 |
| [2] | Cascales L, Craik DJ. Naturally occurring circular proteins: distribution, biosynthesis and evolution. Org Biomol Chem, 2010, 8(22): 5035–5047. DOI: 10.1039/c0ob00139b |
| [3] | Jagadish K, Borra R, Lacey V, et al. Expression of fluorescent cyclotides using protein trans-splicing for easy monitoring of cyclotide-protein interactions. Angew Chem Int Ed, 2013, 52(11): 3126–3131. DOI: 10.1002/anie.201209219 |
| [4] | Turner P, Mamo G, Karlsson EN. Potential and utilization of thermophiles and thermostable enzymes in biorefining. Microb Cell Fact, 2007, 6: 9. DOI: 10.1186/1475-2859-6-9 |
| [5] | Volkmann G, Murphy PW, Rowland EE, et al. Intein-mediated cyclization of bacterial acyl carrier protein stabilizes its folded conformation but does not abolish function. J Biol Chem, 2010, 285(12): 8605–8614. DOI: 10.1074/jbc.M109.060863 |
| [6] | Zakeri B, Howarth M. Spontaneous intermolecular amide bond formation between side chains for irreversible peptide targeting. J Am Chem Soc, 2010, 132(13): 4526–4527. DOI: 10.1021/ja910795a |
| [7] | Zakeri B, Fierer JO, Celik E, et al. Peptide tag forming a rapid covalent bond to a protein, through engineering a bacterial adhesin. Proc Natl Acad Sci USA, 2012, 109(12): E690–E697. DOI: 10.1073/pnas.1115485109 |
| [8] | Veggiani G, Nakamura T, Brenner MD, et al. Programmable polyproteams built using twin peptide superglues. Proc Natl Acad Sci USA, 2016, 113(5): 1202–1207. DOI: 10.1073/pnas.1519214113 |
| [9] | Schoene C, Bennett SP, Howarth M. SpyRing interrogation: analyzing how enzyme resilience can be achieved with phytase and distinct cyclization chemistries. Sci Rep, 2016, 6: 21151. DOI: 10.1038/srep21151 |
| [10] | Zhang WB, Sun F, Tirrel DA, et al. Controlling macromolecular topology with genetically encoded SpyTag-SpyCatcher chemistry. J Am Chem Soc, 2013, 135(37): 13988–13997. DOI: 10.1021/ja4076452 |
| [11] | Bakker ENTP, Pistea A, VanBavel E. Transglutaminases in vascular biology: relevance for vascular remodeling and atherosclerosis. J Vasc Res, 2008, 45(4): 271–278. DOI: 10.1159/000113599 |
| [12] | Komander D, Rape M. The ubiquitin code. Annu Rev Biochem, 2012, 81: 203–229. DOI: 10.1146/annurev-biochem-060310-170328 |
| [13] | Shah NH, Muir TW. Inteins: nature's gift to protein chemists. Chem Sci, 2014, 5(2): 446–461. DOI: 10.1039/C3SC52951G |
| [14] | Dhillon EKS, Dhillon TS, Lai ANC, et al. Host range, immunity and antigenic properties of lambdoid coliphage Hk97. J Gen Virol, 1980, 50(1): 217–220. DOI: 10.1099/0022-1317-50-1-217 |
| [15] | Kang HJ, Coulibaly F, Clow F, et al. Stabilizing isopeptide bonds revealed in gram-positive bacterial pilus structure. Science, 2007, 318(5856): 1625–1628. DOI: 10.1126/science.1145806 |
| [16] | Veggiani G, Zakeri B, Howarth M. Superglue from bacteria: unbreakable bridges for protein nanotechnology. Trends Biotechnol, 2014, 32(10): 506–512. DOI: 10.1016/j.tibtech.2014.08.001 |
| [17] | Izoré T, Contreras-Martel C, El Mortaji L, et al. Structural basis of host cell recognition by the pilus adhesin from Streptococcus pneumoniae. Structure, 2010, 18(1): 106–115. DOI: 10.1016/j.str.2009.10.019 |
| [18] | Amelung S, Nerlich A, Rohde M, et al. The FbaB-type fibronectin-binding protein of Streptococcus pyogenes promotes specific invasion into endothelial cells. Cell Microbiol, 2011, 13(8): 1200–1211. DOI: 10.1111/cmi.2011.13.issue-8 |
| [19] | Hu XQ, Hu H, Melvin JA, et al. Autocatalytic intramolecular isopeptide bond formation in gram-positive bacterial pili: a QM/MM simulation. J Am Chem Soc, 2011, 133(3): 478–485. DOI: 10.1021/ja107513t |
| [20] | Reddington SC, Howarth M. Secrets of a covalent interaction for biomaterials and biotechnology: SpyTag and SpyCatcher. Curr Opin Chem Biol, 2015, 29: 94–99. DOI: 10.1016/j.cbpa.2015.10.002 |
| [21] | Brune KD, Leneghan DB, Brian IJ, et al. Plug-and-display: decoration of virus-like particles via isopeptide bonds for modular immunization. Sci Rep, 2016, 6: 19234. DOI: 10.1038/srep19234 |
| [22] | Fierer JO, Veggiani G, Howarth M. SpyLigase peptide-peptide ligation polymerizes affibodies to enhance magnetic cancer cell capture. Proc Natl Acad Sci USA, 2014, 111(13): E1176–E1181. DOI: 10.1073/pnas.1315776111 |
| [23] | Mujika JI, Lopez X, Mulholland AJ. Mechanism of C-terminal intein cleavage in protein splicing from QM/MM molecular dynamics simulations. Org Biomol Chem, 2012, 10(6): 1207–1218. DOI: 10.1039/C1OB06444D |
| [24] | Böcker JK, Friedel K, Matern JCJ, et al. Generation of a genetically encoded, photoactivatable intein for the controlled production of cyclic peptides. Angew Chem Int Ed, 2015, 127(7): 2144–2148. DOI: 10.1002/ange.201409848 |
| [25] | de Rosa L, Cortajarena AL, Romanelli A, et al. Site-specific protein double labeling by expressed protein ligation: applications to repeat proteins. Org Biomol Chem, 2012, 10(2): 273–280. |
| [26] | Jia XY, Kwon S, Wang CIA, et al. Semienzymatic cyclization of disulfide-rich peptides using sortase A. J Biol Chem, 2014, 289(10): 6627–6638. DOI: 10.1074/jbc.M113.539262 |
| [27] | Wu ZM, Guo XQ, Gao J, et al. Sortase A-mediated chemoenzymatic synthesis of complex glycosylphosphatidylinositol-Anchored protein. Chem Commun, 2013, 49(99): 11689–11691. DOI: 10.1039/c3cc47229a |
| [28] | Minato Y, Ueda T, Machiyama A, et al. Segmental isotopic labeling of a 140 kDa dimeric multi-domain protein CheA from Escherichia coli by expressed protein ligation and protein trans-splicing. J Biomol NMR, 2012, 53(3): 191–207. DOI: 10.1007/s10858-012-9628-3 |
| [29] | Cui CX, Zhao WT, Chen JL, et al. Elimination of in vivo cleavage between target protein and intein in the intein-mediated protein purification systems. Protein Expr Purif, 2006, 50(1): 74–81. DOI: 10.1016/j.pep.2006.05.019 |
| [30] | Wang JD, Wang YL, Wang XZ, et al. Enhanced thermal stability of lichenase from Bacillus subtilis 168 by SpyTag/SpyCatcher-mediated spontaneous cyclization. Biotechnol Biofuels, 2016, 9: 79. DOI: 10.1186/s13068-016-0490-5 |
| [31] | Gilbert C, Howarth M, Harwood CR, et al. Extracellular self-assembly of functional and tunable protein conjugates from Bacillus subtilis. ACS Synth Biol, 2017, 6(6): 957–967. DOI: 10.1021/acssynbio.6b00292 |
| [32] | Si M, Xu Q, Jiang L, et al. SpyTag/SpyCatcher cyclization enhances the thermostability of firefly luciferase. PLoS ONE, 2016, 11(9): e0162318. DOI: 10.1371/journal.pone.0162318 |
| [33] | Liu D, Wu WH, Liu YJ, et al. Topology engineering of proteins in vivo using genetically encoded, mechanically interlocking SpyX modules for enhanced stability. ACS Cent Sci, 2017, 3(5): 473–481. DOI: 10.1021/acscentsci.7b00104 |
| [34] | Wang XW, Zhang WB. Cellular synthesis of protein catenanes. Angew Chem, 2016, 55(10): 3442–3446. DOI: 10.1002/anie.201511640 |
| [35] | Wang HH, Altun B, Nwe K, et al. Proximity-based sortase-mediated ligation. Angew Chem, 2017, 56(19): 5349–5352. DOI: 10.1002/anie.201701419 |
| [36] | Lin YQ, Jin WH, Wang JD, et al. A novel method for simultaneous purification and immobilization of a xylanase-lichenase chimera via SpyTag/SpyCatcher spontaneous reaction. Enzyme Microb Technol, 2018, 115: 29–36. DOI: 10.1016/j.enzmictec.2018.04.007 |
| [37] | Liu ZD, Zhou H, Wang WJ, et al. A novel method for synthetic vaccine construction based on protein assembly. Sci Rep, 2014, 4: 7266. |
| [38] | Gao XY, Fang J, Xue B, et al. Engineering protein hydrogels using SpyCatcher-SpyTag chemistry. Biomacromolecules, 2016, 17(9): 2812–2819. DOI: 10.1021/acs.biomac.6b00566 |
| [39] | Wang R, Yang ZG, Luo JR, et al. B12-dependent photoresponsive protein hydrogels for controlled stem cell/protein release. Proc Natl Acad Sci USA, 2017, 114(23): 5912–5917. DOI: 10.1073/pnas.1621350114 |
| [40] | Giessen TW, Silver PA. A catalytic nanoreactor based on in vivo encapsulation of multiple enzymes in an engineered protein nanocompartment. ChemBioChem, 2016, 17(20): 1931–1935. DOI: 10.1002/cbic.v17.20 |
| [41] | Alves NJ, Turner KB, Daniele MA, et al. Bacterial nanobioreactors-directing enzyme packaging into bacterial outer membrane vesicles. ACS Appl Mater Interfaces, 2015, 7(44): 24963–24972. DOI: 10.1021/acsami.5b08811 |
| [42] | Botyanszki Z, Tay PKR, Nguyen PQ, et al. Engineered catalytic biofilms: site-specific enzyme immobilization onto E. coli curli nanofibers. Biotechnol Bioeng, 2015, 112(10): 2016–2024. DOI: 10.1002/bit.v112.10 |
| [43] | Hyre DE, Le Trong I, Merritt EA, et al. Cooperative hydrogen bond interactions in the streptavidin-biotin system. Protein Sci, 2006, 15(3): 459–467. DOI: 10.1110/ps.051970306 |
| [44] | Cao Y, Liu D, Zhang WB. Supercharging SpyCatcher toward an intrinsically disordered protein with stimuli-responsive chemical reactivity. Chem Commun, 2017, 53(63): 8830–8833. DOI: 10.1039/C7CC04507G |
| [45] | Singh SK, Thrane S, Janitzek CM, et al. Improving the malaria transmission-blocking activity of a Plasmodium falciparum 48/45 based vaccine antigen by SpyTag/SpyCatcher mediated virus-like display. Vaccine, 2017, 35(30): 3726–3732. DOI: 10.1016/j.vaccine.2017.05.054 |
| [46] | Leneghan DB, Miura K, Taylor IJ, et al. Nanoassembly routes stimulate conflicting antibody quantity and quality for transmission-blocking malaria vaccines. Sci Rep, 2017, 7: 3811. DOI: 10.1038/s41598-017-03798-3 |
| [47] | Siegmund V, Piater B, Zakeri B, et al. Spontaneous isopeptide bond formation as a powerful tool for engineering site-specific antibody-drug conjugates. Sci Rep, 2016, 6: 39291. DOI: 10.1038/srep39291 |
| [48] | Avila-Calderon ED, Araiza-Villanueva MG, Cancino-Diaz JC, et al. Roles of bacterial membrane vesicles. Arch Microbiol, 2015, 197(1): 1–10. DOI: 10.1007/s00203-014-1042-7 |
| [49] | Wang Y. The function of OmpA in Escherichia coli. Biochem Biophys Res Commun, 2002, 292(2): 396–401. DOI: 10.1006/bbrc.2002.6657 |
| [50] | Pröschel M, Detsch R, Boccaccini AR, et al. Engineering of metabolic pathways by artificial enzyme channels. Front Bioeng Biotechnol, 2015, 3: 168. |
 2019, Vol. 35
2019, Vol. 35