中国科学院微生物研究所、中国微生物学会主办
文章信息
- 孙中贯, 周波, 王孟祺, 王亚平, 邢爽, 郭学武, 肖冬光
- Sun Zhongguan, Zhou Bo, Wang Mengqi, Wang Yaping, Xing Shuang, Guo Xuewu, Xiao Dongguang
- 高温高浓发酵工业啤酒酵母菌种的构建
- Construction of industrial brewing yeast for fermentation under high temperature and high gravity condition
- 生物工程学报, 2019, 35(3): 522-534
- Chinese Journal of Biotechnology, 2019, 35(3): 522-534
- 10.13345/j.cjb.180310
-
文章历史
- Received: July 29, 2018
- Accepted: October 8, 2018
- Published: February 22, 2019
2. 天津科技大学 工业发酵微生物教育部重点实验室,天津 300457
2. Key Laboratory of Industrial Fermentation Microbiology, Ministry of Education, Tianjin University of Science and Technology, Tianjin 300457, China
啤酒高浓高温发酵是指采用较高浓度(14 °P以上)的麦芽汁在较高的温度条件下进行发酵,在啤酒成熟后过滤前,用经处理的含饱和CO2的脱氧水将啤酒稀释成常规浓度(10-12 °P)的技术[1-2]。高浓高温发酵技术在不增加糖化、发酵等生产设备的基础上,能够大幅提高企业的生产效率,减少能源消耗,降低生产成本,获得更高的经济效益。但是,高温高浓发酵技术也会给啤酒发酵带来一系列的问题,如啤酒风味不协调、泡沫稳定性差、发酵结束后酵母絮凝性下降等[3-4]。
酵母菌絮凝是指酵母细胞之间相互聚集形成絮状或颗粒状细胞团,并迅速沉降到发酵液底部的一种生理特性,絮凝的发生是一种无性的、钙依赖的、可逆的过程[5]。絮凝的发生依赖于絮凝蛋白与邻近细胞表面寡聚甘露糖链间的结合[6],而工业酵母菌株絮凝特性的差异主要是由絮凝蛋白的浓度所决定的[7]。高浓度麦芽汁发酵结束后,发酵液中过量存在的甘露糖等糖类物质可抑制酵母菌株的絮凝性[8]。发酵温度较低时,对酵母菌株絮凝性的影响较小,酵母细胞均具有较好的絮凝性[9-10]。有关学者认为较低的发酵温度下,酵母的新陈代谢速度减缓,进而缓解了CO2对酵母菌株的扰动作用,故温度较低时酵母细胞趋于絮凝[11];本课题组前期研究结果表明,麦芽汁浓度对工业啤酒酵母S6的絮凝性影响较显著,而高温发酵对S6菌株的絮凝性影响较小[12]。
Dietvorst等研究发现在高浓度麦芽糖或葡萄糖的发酵条件下,与甲基转移酶相关的复合蛋白(COMPASS)能够使FLO1、FLO5以及FLO9基因表达沉默,从而导致酵母菌株的絮凝能力下降[13]。酵母菌细胞壁上的絮凝蛋白表现出丰富的遗传多样性,其中FLO5基因编码的絮凝蛋白与糖链的结合能力最强[14]。
啤酒中的高级醇是酵母在发酵过程中的主要副产物之一,是构成啤酒风味的重要物质,适宜的高级醇含量能够增加啤酒的醇厚感,但含量过高反而会造成酒体风味物质失衡,饮用后产生头痛、口渴等症状[15-16]。研究表明较高的发酵温度和麦芽汁浓度均有助于高级醇的生成[16-17]。酵母细胞内高级醇的合成分为氨基酸分解代谢途径(Ehrlich pathway)和糖酵解合成代谢途径(Harris pathway)[18],如图 1所示。酿酒酵母菌株的BAT基因编码的支链氨基酸转氨酶催化支链氨基酸转氨生成高级醇的前体物α-酮酸,因而BAT基因的缺失将有助于抑制高级醇的合成[19-20]。
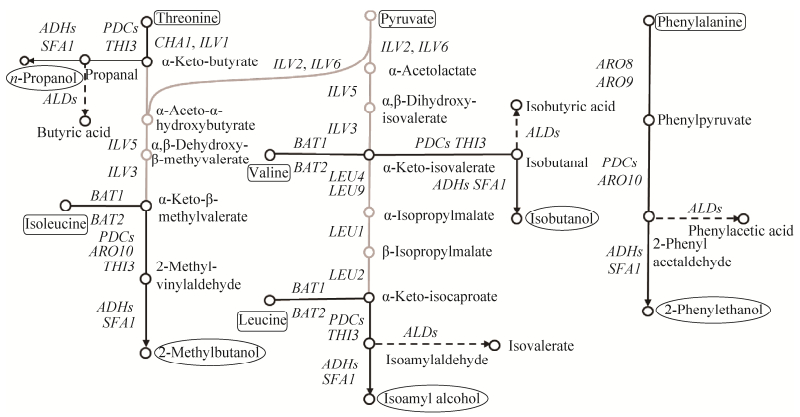
|
| 图 1 酿酒酵母高级醇合成代谢网络图 Fig. 1 Biosynthetic pathways for higher alcohols formation in Saccharomyces cerevisiae. Black solid lines represent that the higher alcohols are derived from the Ehrlich pathway. Gray solid lines represent that the higher alcohols are derived from the Harris pathway. Black dotted lines represent the synthesis of corresponding carboxylic acids. |
| |
目前有关利用代谢工程提高高浓高温发酵条件下工业啤酒酵母絮凝性同时降低高级醇合成能力的研究还未有报道。本研究将在敲除BAT基因的基础上,实现FLO5基因的过量表达,以构建高温高浓发酵条件下高絮凝性、低高级醇合成能力的工业啤酒酵母优质菌株。
1 材料与方法 1.1 材料 1.1.1 菌种与质粒本研究所有菌株和质粒均为作者所在实验室保存,详见表 1。重组菌株的构建均采用单片段基因重组法[21]。
| Name | Description | Source |
| Strains | ||
| E. coli DH5α | Φ80 lacZΔM15 ΔlacU169 recA1 endA1 hsdR17 supE44 thi-1 gyrA relA1 | Lab collection |
| S6 | Industrial diploid brewer’s yeast; Saccharomyces pastorianus | Lab collection |
| S6-BF | S6 BAT2/Δbat2::PGK1P-FLO5-PGK1T-loxP-KanMX-loxP | This study |
| S6-BFK | S6 BAT2/Δbat2::PGK1P-FLO5-PGK1T-loxP | This study |
| S6-BF2 | S6 Δbat2::PGK1P-FLO5-PGK1T-loxP/Δbat2::PGK1P-FLO5-PGK1T-loxP- KanMX-loxP | This study |
| S6-BF2K | S6 Δbat2::PGK1P-FLO5-PGK1T-loxP/Δbat2::PGK1P-FLO5-PGK1T-loxP | This study |
| S6-BF2B1 | S6Δbat2::PGK1P-FLO5-PGK1T-loxP/Δbat2::PGK1P-FLO5-PGK1T-loxP/ BAT1/Δbat1::loxP-KanMX-loxP | This study |
| Plasmids | ||
| pUG6 | Kanr, containing loxP-KanMX-loxP disruption cassette | Lab collection |
| pSH-Zeocin | Zeor, Cre recombinant enzyme expression vector | Lab collection |
| pUC19 | Apr, cloning vector | Invitrogen |
| Yep-PGK | Apr, containing PGK1P-PGK1T expression cassette | Lab collection |
| Yep352 | URA3+, Ampr ori control vector | Invitrogen |
| Yp-AFKB | Apr, Kanr, containing the BA-PGK1P-FLO5-PGK1T-loxP-KanMX-loxP-BB gene expression cassette | This study |
| Yp-A2FKB2 | Apr, Kanr, containing the BA2-PGK1P-FLO5-PGK1T-loxP-KanMX-loxP-BB2 gene expression cassette | This study |
| pUC-A1KB1 | Apr, Kanr, containing the A1-loxP-KanMX-loxP-B1 gene expression cassette | This study |
实验中所用的引物列于表 2。
| Primer name | Primer sequence (5′-3′) |
| For plasmid construction | |
| FLO5-U | TCCAGATCTCCTCGAGATGACAATTGCACACCACTGCATATT |
| FLO5-D | TCGCAGATCCCTCGAGTTAAATAATTGCCAGCAATAAGGACG |
| BA-U | TCCCCCGGGAAGAGACTGAGAAACCTACTCCCAA |
| BA-D | TCCCCCGGGAAACTCGTGGAGATGCTTTCCCTTA |
| BB-U | CATGCATGCAGTATCGCTATTGCTACGTAAAG |
| BB-D | CATGCATGCAGTTAGCTGTTTGAAGATCGGTC |
| BA2-U | TCCCCCGGGTAAGAACGATATGACCTTGG |
| BA2-D | TCCCCCGGGTTCTGTAAGCCTTCATCC |
| BB2-U | CATGCATGCTGGACCATTAGTGAACGC |
| BB2-D | CATGCATGCCAGTAACAACCCTTGACCA |
| A1-U | CGGAATTCACGGCAAGGAAAGGGTTT |
| A1-D | CGGGATCCCATTTACGCTTTACAGTTCA |
| B1-U | CGGGATCCATCATCGTGGCATACAGG |
| B1-D | AACTGCAGCAATCGAAAGCATAGCAG |
| K-U | CGGGATCCCAGCTGAAGCTTCGTACGCT |
| K-D | CGGGATCCGCATAGGCCACTAGTGGATCTG |
| For PCR verification | |
| B-U | ATGTCGCCGCCGTCAATA |
| K-X | CAAGACTGTCAAGGAGGGTA |
| K-D1 | ACTAACGCCGCCATCCAG |
| P-U | GTTCGGGTTCAGCGTATT |
| B-D | TGACAAAGGGAGTAGCAT |
| A1-S | ATGCTGAATCGTAGCGTCTT |
| A1-X | ATCGCGTATTTCGTCTCG |
| B1-S | TCAGTGGCAAATCCTAAC |
| B1-X | CTCTAGCCCAAGAAGTGT |
| For real-time qPCR | |
| ACT1-F | GGTAACGAAAGATTCAGAGC |
| ACT1-R | AGGTAGTCAAAGAAGCCAAG |
| FLO5-F | TCAATCACGCTGAAACAC |
| FLO5-R | AGGACGCAATGAAGACAC |
| BAT2-F | GTCATTTGCTGCCCTGTG |
| BAT2-R | GTTCCTTCTTGCCCGTTT |
| BAT1-F | GCCGCTAGAATTTGTTTG |
| BAT1-R | AAGGACCGACTGGAGAAG |
| The underlined characters indicated restriction sites for plasmid construction. | |
LB培养基(g/L):胰蛋白胨10,氯化钠10,酵母浸粉5,pH 7.0,115 ℃灭菌20 min。需要时使用前加入氨苄青霉素至100 μg/mL,固体培养基添加20 g琼脂,用于大肠杆菌培养。
YEPD培养基(g/L):葡萄糖20,蛋白胨20,酵母浸粉10,自然pH,115 ℃灭菌20 min。制备平板时添加20 g琼脂,用于酵母培养。筛选转化子时需添加G418至500 μg/mL。
YPG半乳糖诱导培养基(g/L):半乳糖20,蛋白胨20,酵母浸粉10,自然pH,115 ℃灭菌20 min。
麦芽汁培养基:采用二段式浸出糖化法。粉碎后的大麦芽,按1:4的料水比于62 ℃糖化30 min,随后升温至70 ℃糖化至碘检完毕,过滤煮沸后冷却至室温,离心去沉淀后,用自来水调整外观糖度至18 °P,115 ℃灭菌20 min。
引物委托北京鼎国昌盛生物技术有限责任公司合成。高保真性DNA扩增酶采用TransTaq HiFi,购自北京全式金生物技术有限公司。限制性内切酶、DNA连接酶、去磷酸化酶购自大连宝生物工程有限公司。质粒提取试剂盒和酵母菌总DNA提取试剂盒购自大连宝生物公司。硫酸盐遗传霉素(G418)、氨苄青霉素、卡那霉素、博来霉素(Zeocin)购自北京鼎国昌盛生物技术有限责任公司。鲑鱼精DNA购自北京索莱宝科技有限公司。
1.2 啤酒发酵实验 1.2.1 菌种活化将甘油管中保存菌种转接至YEPD斜面试管30 ℃活化培养2 d。
1.2.2 一级种子培养取活化后的斜面菌种一环,接种于装有5 mL 12 °P麦芽汁培养基的试管中,在28 ℃、180 r/min条件下培养24 h。
1.2.3 二级种子培养一级种子液按10% (V/V)的接种量接入装有50 mL 12 °P麦芽汁培养基的150 mL三角瓶内,16 ℃静置培养72 h。
1.2.4 啤酒发酵二级种子液经离心无菌水洗涤2次后得到的酵母泥,按0.5% (W/V)的接种量接入盛有150 mL 18 °P麦芽汁培养基的250 mL三角瓶内,16 ℃下静置发酵。
1.3 方法 1.3.1 目的片段的扩增与质粒构建以酿酒酵母S6菌株的基因组DNA为模板,利用引物对FLO5-U/FLO5-D、BA-U/BA-D、BB-U/BB-D、BA2-U/BA2-D、BB2-U/BB2-D、A1-U/A1-D、B1-U/B1-D扩增FLO5基因、BAT2基因的2条上游同源序列BA和BA2、2条下游同源序列BB和BB2以及BAT1基因的上下游同源序列A1和B1。以质粒pUG6为模板,K-U和K-D为引物对扩增KanMX片段。
将目的基因FLO5经XhoⅠ酶切处理并去磷酸化后,用In-fusion酶连接至相应切口的质粒Yep-PGK中,转化验证后依次将经BamHⅠ酶切并磷酸化后的KanMX片段,经SmaⅠ酶切并磷酸化后的BA片段,经SphⅠ酶切并磷酸化后的BB片段,连接到表达载体Yep-PGK相应的位点上并进行转化验证,得到质粒Yp-AFKB。
将经SmaⅠ酶切并磷酸化后的BA2片段,经SphⅠ酶切并磷酸化后的BB2片段,依次连接到含有KanMX片段的表达载体Yep-PGK相应的位点上,转化验证后得到质粒Yp-A2FKB2。
将BAT1基因的上游同源序列A1片段用EcoRⅠ和BamHⅠ进行双酶切,连接到相应切口的质粒pUC19中,转化验证后再将质粒用BamHⅠ和PstⅠ进行双酶切,与经同样酶切后的BAT1基因的下游同源序列B1片段连接,转化验证后将KanMX基因片段用BamHⅠ进行单酶切,连接到重组质粒pUC19相应的位点上,转化验证后得到质粒pUC-A1KB1。
以质粒YP-AFKB为模板,BA-U和BB-D为引物对,PCR扩增目的片段,经纯化回收、测序后,得到敲除BAT2基因一个等位基因同时过表达FLO5基因的重组片段BA+KanMX+PGKp+FLO5+ PGKt+BB;以质粒YP-A2FKB2为模板,BA2-U和BB2-D为引物对,PCR扩增目的片段,经纯化回收、测序后,得到敲除BAT2基因的另一个等位基因同时过表达FLO5基因的重组片段BA2+KanMX+ PGKp+FLO5+PGKt+BB2;以质粒pUC-A1KB1为模板,A1-U和B1-D为引物对,PCR扩增目的片段,经纯化回收、测序后,得到敲除BAT1基因一个等位基因的重组片段A1+KanMX+B1。
1.3.2 酵母的转化和重组子的筛选酵母的转化采用LiAc/SS carrier DNA/PEG法[22]。利用G418筛选转化子,提取转化子基因组进行PCR定点验证。
1.3.3 KanMX抗性基因的去除采用Cre/loxP报告基因挽救系统[23],剔除阳性转化子中的筛选标记基因KanMX。利用影印平板法筛选转化子,以K-U/K-D为引物对进行PCR验证。
1.3.4 Real-Time qPCR测定基因转录水平酵母RNA的提取及RNA的反转录均按照产品说明书进行操作,使用SYBR®Premix Ex TaqTM Ⅱ (TaKaRa)试剂盒进行实时荧光定量PCR,通过2-ΔΔCt值法对目的基因及内参基因ACT1进行基因表达量的分析。
1.3.5 CO2排放量的测定发酵前预先称量发酵体系总重,发酵过程中每隔12 h称重一次,称重前应先摇晃三角瓶,以去除发酵液中的CO2,当失重小于0.1 g时,表示发酵已经结束。
1.3.6 酵母菌絮凝能力的测定采用光密度改良法对酵母菌絮凝能力进行测定[24]。
1.3.7 啤酒中高级醇含量的测定啤酒发酵液经蒸馏后得到的样品使用气相色谱法(Agilent,USA)测定。色谱条件为:色谱柱LAP-930,50 m×0.32 mm×1.0 μm;检测器为氢火焰离子化检测器。初始柱温为50 ℃,保持8 min,以5 ℃/min的升温速度升至200 ℃,保持5 min,进样量为1.0 μL,分流比为10:1。
1.3.8 其他发酵参数的测定利用手持糖度计测定原麦汁的表观糖度;利用斐林试剂法测定发酵液中还原糖的含量;酒精度、发酵度及双乙酰的测定依据啤酒分析方法进行测定。
2 结果与分析 2.1 重组菌株S6-BF的构建与性能分析为了提高高温高浓发酵条件下工业啤酒酵母S6的絮凝能力,同时降低高级醇的生成量;选择BAT2基因为重组片段的整合位点,将重组片段BA+KanMX+PGKp+FLO5+PGKt+BB整合到出发菌株S6的基因组中。对在G418浓度为500 mg/L的YEPD平板上生长的转化子进行PCR验证。转化子的PCR验证结果表明,重组片段成功整合到酵母S6基因组中相应的位点,将得到的阳性转化子命名为S6-BF (图 2)。
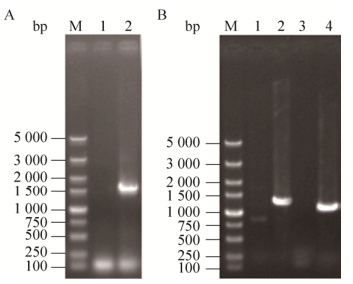
|
| 图 2 重组菌株S6-BF的PCR验证 Fig. 2 The PCR verification of the mutant strain S6-BF. M: DL5000 DNA marker. (A) Primers: K-U and K-D. 1: S6; 2: S6-BF. (B) 1-2: primers: B-U and K-D1; 1: S6; 2: S6-BF. 3-4: primers: P-U and B-D; 3: S6; 4: S6-BF. |
| |
以出发菌株S6为对照菌株,分析重组菌株S6-BF、BAT2基因单敲除重组菌株S6-SB2及FLO5基因单过表达重组菌株S6-SF5的BAT2基因与FLO5基因转录水平、絮凝能力、高级醇合成能力以及发酵性能。基因转录水平分析结果表明,FLO5基因在重组菌株S6-BF中的转录水平较出发菌株得到了非常显著的提高,约为出发菌株的12倍;与重组菌株S6-SF5相比,FLO5基因在重组菌株S6-BF中的转录水平无显著变化,表明BAT2基因的单敲除对FLO5基因的转录水平没有明显的影响(图 3A);同时,絮凝能力分析结果显示,重组菌株S6-SF5与S6-BF的絮凝能力均得到非常显著的提升,是出发菌株S6絮凝能力的1.3倍(图 3B)。与出发菌株相比,重组菌株S6-BF的BAT2基因转录水平显著降低,约为出发菌株的0.6倍,且FLO5基因的过量表达没有对BAT2基因的转录水平产生影响(图 3A);在高级醇类物质中,仅有异戊醇的合成量得到非常显著的降低,但降低量仅有7.3%,其他高级醇的合成水平均没有显著的变化,同时重组菌株S6-BF絮凝能力的提高对高级醇的合成没有产生影响(图 3C)。由表 3可知,BAT2基因的单敲除、FLO5基因的单过表达以及BAT2基因与FLO5基因的组合敲除过表达对菌株S6的发酵能力都没有产生显著的影响,因而可以通过再次降低BAT2基因表达量同时过量表达FLO5基因的方式,以期提高出发菌株S6的絮凝能力同时降低高级醇的合成水平。
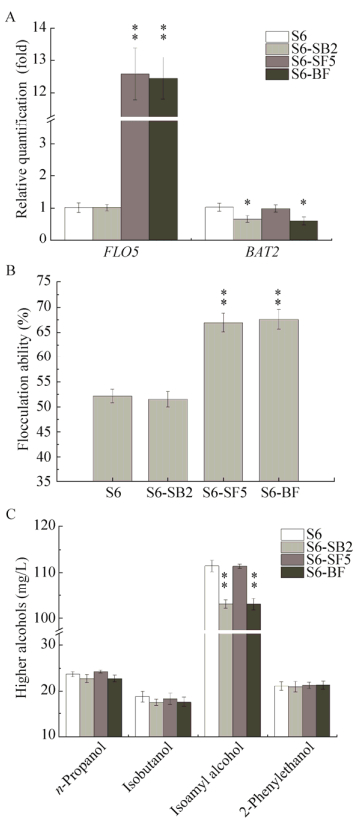
|
| 图 3 重组菌株S6-SB2、S6-SF5及S6-BF基因转录水平及性能分析 Fig. 3 Transcriptional levels and characteristics of S6-SB2, S6-SF5, S6-BF and parent S6. (A) Transcriptional levels of FLO5, BAT2 genes in mutants and the parental strain. (B) Flocculation ability of mutants and the parental strain. The flocculation ability was analyzed using the standard method. (C) The final levels of higher alcohols in mutants and the parental strain. Data are presented as the means of the results of three independent experiments. Error bars indicate standard deviations. Note: ** P < 0.01, * P < 0.05. |
| |
| Strains | Fermentation time (d) | CO2 release (g) | End extract (g/L) | Ethanol (%, V/V) | Fermentation degree (%) |
| S6 | 10 | 9.43±0.058 | 9.3±0.08 | 6.64±0.07 | 70.42±1.21 |
| S6-SB2 | 10 | 9.43±0.110 | 9.4±0.07 | 6.72±0.11 | 70.01±1.01 |
| S6-SF5 | 10 | 9.40±0.058 | 9.3±0.06 | 6.68±0.10 | 69.85±0.88 |
| S6-BF | 10 | 9.33±0.058 | 9.4±0.11 | 6.50±0.12 | 69.81±1.54 |
| S6-DB2 | 10 | 9.40±0.000 | 9.4±0.08 | 6.52±0.07 | 70.23±1.37 |
| S6-DF5 | 10 | 9.43±0.058 | 9.3±0.07 | 6.60±0.08 | 70.65±1.21 |
| S6-BF2 | 10 | 9.43±0.000 | 9.4±0.05 | 6.57±0.11 | 70.21±0.91 |
| S6-SB1 | 10 | 9.33±0.058 | 9.3±0.05 | 6.67±0.07 | 70.55±1.41 |
| S6-BF2B1 | 10 | 9.50±0.100 | 9.5±0.06 | 6.71±0.09 | 69.68±1.58 |
| Data are presented as the means of the results of three independent experiments. ** P < 0.01; * P < 0.05. | |||||
以重组菌株S6-BF为出发菌株,构建FLO5基因过量表达的重组菌株,需要再次利用KanMX抗性基因作为筛选标记,因而必须剔除存在于重组菌株S6-BF基因组上的KanMX抗性基因。利用Cre/loxP报告基因挽救系统可以实现KanMX抗性标记的剔除和反复利用。
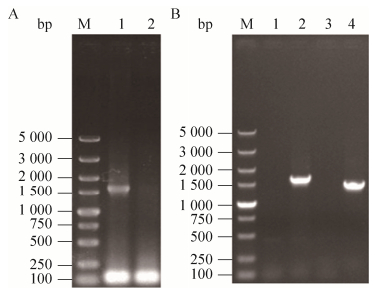
重组菌株S6-BF基因组中KanMX抗性基因剔除的PCR验证结果如图 4A所示,将得到的阳性转化子命名为S6-BFK。选择重组菌株S6-BFK的BAT2基因为整合位点,采用缩进式基因整合的方式,即同源序列BA2位于同源序列BA的下游区域,同源序列BB2位于同源序列BB的上游区域,且同源序列彼此之间均无重复区域,将重组片段BA2+KanMX+PGKp+FLO5+PGKt+BB2整合到重组菌株S6-BFK的基因组中。对在G418浓度为500 mg/L的YEPD平板上生长的转化子进行PCR验证。转化子的PCR验证结果表明,重组片段成功整合到酵母S6基因组中相应的位点,将得到的阳性转化子命名为S6-BF2 (图 4B)。
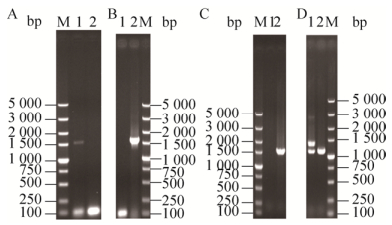
|
| 图 4 重组菌株S6-BF2的PCR验证 Fig. 4 The PCR verification of the mutant strain S6-BF2. M: DL5000 DNA marker. (A) Primers: K-U and K-D. 1: S6-BF; 2: S6-BFK. (B) Primers: K-U and K-D. 1: S6-BFK; 2: S6-BF2. (C) Primers: B-U and K-D1. 1: S6-BFK; 2: S6-BF2. (D) Primers: P-U and B-D. 1: S6-BFK; 2: S6-BF2. |
| |
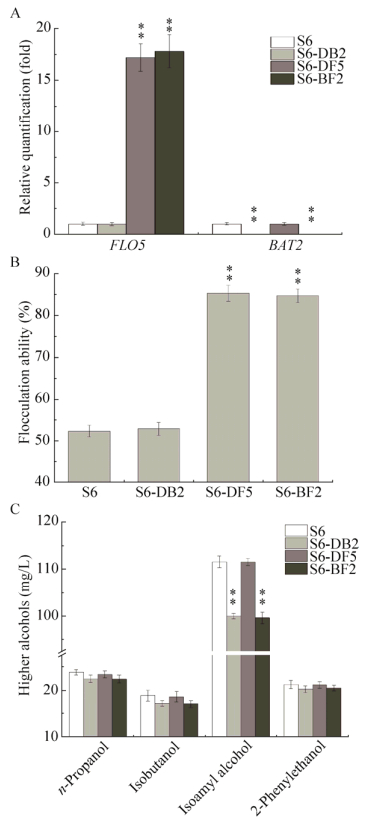
以原始菌株S6为对照菌株,测定BAT2基因双敲重组菌株S6-DB2、FLO5基因两次过表达重组菌株S6-DF5以及重组菌株S6-BF2的BAT2基因与FLO5基因转录水平、絮凝能力、高级醇合成能力及发酵性能。FLO5基因在重组菌株S6-BF2中的转录水平得到了进一步的提高,较原始菌株S6提高了17.8倍,与重组菌株S6-DF5中FLO5基因转录水平无显著差异(图 5A);同时,重组菌株S6-BF2的絮凝能力与重组菌株S6-DF5的絮凝能力没有明显的差异,均较原始菌株S6提高了62.8%,絮凝能力达到了85.44% (图 5B)。同时实验结果也表明BAT2基因功能的缺失没有对重组菌株的絮凝性产生显著的影响。在重组菌株S6-DB2和S6-BF2中没有检测到BAT2基因的转录(图 5A),表明重组菌株S6-DB2和S6-BF2基因组上的BAT2基因已被全部破坏无法正常表达。重组菌株S6-BF2合成高级醇的能力也有显著降低,其中异戊醇的合成量下降最为明显;与原始菌株S6相比,重组菌株S6-BF2合成高级醇的能力降低了9.15%,与重组菌株S6-DB2的高级醇合成能力保持一致,实验结果表明重组菌株絮凝能力的提高没有对高级醇的合成能力产生影响(图 5C)。由表 3可知,重组菌株S6-BF2的发酵能力与原始菌株S6相比没有显著变化,表明FLO5基因的过量表达及BAT2基因功能的缺失没有对工业酿酒酵母S6的发酵能力产生影响,同时也表明在BAT2基因和FLO5基因的改造过程中不存在相互协同或相互干扰的现象,且基因BAT2是酿酒酵母S6的非必需基因。
|
| 图 5 重组菌株S6-DB2、S6-DF5及S6-BF2基因转录水平及性能分析 Fig. 5 Transcriptional levels and characteristics of S6-DB2, S6-DF5, S6-BF2 and parent S6. (A) Transcriptional levels of FLO5, BAT2 genes in mutants and the parental strain. (B) Flocculation ability of mutants and the parental strain. The flocculation ability was analyzed using the standard method. (C) The final levels of higher alcohols in mutants and the parental strain. Data are presented as the means of the results of three independent experiments. Error bars indicate standard deviations. Note: ** P < 0.01; * P < 0.05. |
| |
虽然重组菌株高级醇的合成能力下降较为显著,但将高浓啤酒稀释50%至常规浓度(12 °P)后高级醇的含量仍然高于优质拉格啤酒所要求的高级醇含量的最适浓度[15]。酿酒酵母基因组中存在着BAT2基因的同源基因BAT1,而BAT1基因所编码的蛋白同样具有支链氨基酸转氨酶的作用。以重组菌株S6-BF2为出发菌株敲除BAT1基因,考察BAT1基因的缺失对重组菌株高级醇合成能力的影响。
2.3 重组菌株S6-BF2B1的构建与性能分析以重组菌株S6-BF2为出发菌株实现对BAT1基因的敲除仍然需要以KanMX抗性基因为筛选标记,因此在构建菌株之前需要剔除重组菌株S6-BF2基因组中的KanMX抗性基因。试验方法及验证同重组菌株S6-BFK的构建过程,将剔除KanMX抗性基因的重组菌株命名为S6-BF2K。PCR验证结果如图 6A所示。将重组片段A1+KanMX+B1转化至重组菌株S6-BF2K基因组中并进行PCR验证,同时测定BAT1基因的转录水平、阳性转化子及BAT1基因单敲除重组菌株S6-SB1的发酵性能、絮凝能力及高级醇合成水平。转化子的PCR验证结果表明,重组片段A1+KanMX+B1已整合到重组菌株S6-BF2K基因组中相应的位点,将得到的阳性转化子命名为S6-BF2B1 (图 6B)。
|
| 图 6 重组菌株S6-BF2B1的PCR验证 Fig. 6 The PCR verification of the mutant strain S6-BF2B1. M: DL5000 DNA marker. (A) Primers: K-U and K-D. 1: S6-BF2; 2: S6-BF2K. (B) 1-2: primers: A1-S and A1-X. 1: S6-BF2K; 2: S6-BF2B1. 3-4: primers: B1-S and B1-X. 3: S6-BF2K; 4: S6-BF2B1. |
| |
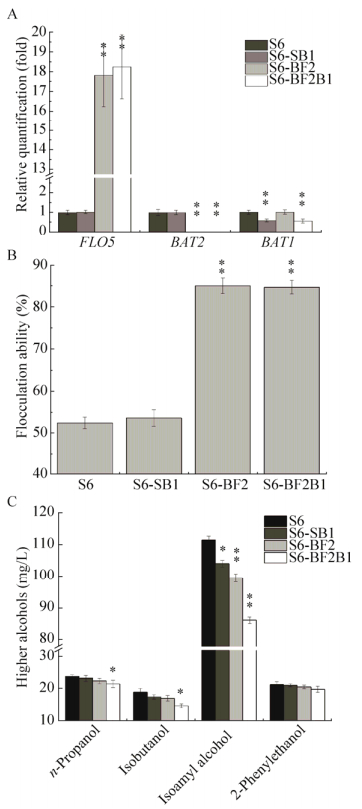
基因转录水平分析结果表明,与原始菌株S6相比,重组菌株S6-BF2B1的BAT1基因转录水平下降显著,是原始菌株BAT1基因转录水平的56.33%;重组菌株S6-BF2B1中FLO5、BAT2基因的转录水平与重组菌株S6-BF2相比没有发生明显的变化,且重组菌株S6-SB1中FLO5、BAT2基因的转录水平与原始菌株S6保持一致,证明BAT1基因的单次敲除没有对FLO5、BAT2基因的转录水平造成任何影响(图 7A)。酵母絮凝性分析试验结果显示,重组菌株S6-BF2B1的絮凝性是原始菌株絮凝性的1.62倍,这一水平与重组菌株S6-BF2的絮凝能力相当,同时BAT1基因的单敲除没有对菌株S6的絮凝能力产生影响,表明BAT1基因的单敲除没有影响菌株的絮凝能力(图 7B)。重组菌株S6-SB1、S6-BF2B1的高级醇合成水平与原始菌株相比均有显著降低,重组菌株S6-BF2B1高级醇的合成量降低了19.05%,由原始菌株S6的175.58 mg/L下降至142.13 mg/L (图 7C)。将重组菌株S6-BF2B1高温高浓发酵液稀释50%至常规浓度(12 °P)时,高级醇含量与原始菌株S6在10 ℃发酵条件下的高级醇生成量基本相当[12]。试验结果表明,BAT1基因功能的部分缺失有助于降低工业酿酒酵母S6的高级醇合成能力,且对出发菌株的发酵性能没有产生显著的影响(表 3);获得的重组菌株S6-BF2B1能够在高温高浓的酿造条件下有效提高酿酒酵母的絮凝性同时显著降低高级醇的生成量。
|
| 图 7 重组菌株S6-SB1、S6-BF2及S6-BF2B1基因转录水平及性能分析 Fig. 7 Transcriptional levels and characteristics of S6-SB1, S6-BF2, S6-BF2B1 and parent S6. (A) Transcriptional levels of FLO5, BAT2, BAT1 genes in mutants and the parental strain. (B) Flocculation ability of mutants and the parental strain. The flocculation ability was analyzed using the standard method. (C) The final levels of higher alcohols in mutants and the parental strain. Data are presented as the means of the results of three independent experiments. Error bars indicate standard deviations. Note: ** P < 0.01; * P < 0.05. |
| |
在高温高浓的发酵条件下进行啤酒发酵实验,待主发酵结束后,对重组菌株S6-BF2B1及原始菌株S6代谢产生的主要风味物质进行了测定,结果如表 4所示。
| Compound | S6 (mg/L) | S6-BF2B1 (mg/L) |
| n-Propanol | 23.83±0.58 | 21.45±1.12* |
| Isobutanol | 18.91±1.15 | 14.70±0.54* |
| Isoamyl alcohol | 111.61±1.24 | 86.24±1.02** |
| 2-Phenyethanol | 21.23±0.90 | 19.74±0.89 |
| Ethyl acetate | 42.55±1.18 | 30.55±0.96** |
| Isoamyl acetate | 8.71±0.52 | 4.25±0.29** |
| Diacetyl | 0.53±0.041 | 0.50±0.055 |
| Data are presented as the means of the results of three independent experiments. ** P < 0.01; * P < 0.05. | ||
由表 4可以看出,与原始菌株S6相比,重组菌株S6-BF2B1的高级醇合成能力及酯类物质的合成水平均有显著的降低,醇酯比例由原始菌株S6的3.42上升至4.08,达到拉格啤酒要求的较为适宜的醇酯比例(1:4-1:5);双乙酰的含量没有发生较为显著的变化。高级醇中异戊醇的含量降低最为明显,从而导致乙酸异戊酯的含量显著降低;此外,重组菌株S6-BF2B1合成乙酸乙酯的能力也明显下降,这是否与BAT基因功能的减弱有关,还有待于进一步的研究。由于本研究中的啤酒发酵是实验室锥形瓶小试发酵,只是完成了啤酒主发酵阶段的实验没有进行后发酵和低温贮藏,因而啤酒中双乙酰的含量未能达到成熟啤酒的要求(双乙酰阈值范围为0.10-0.15 mg/L)。
3 讨论近年来在啤酒行业中兴起的高温高浓发酵技术为啤酒生产企业降低了生产成本,提高了经济效益,但同时也为生产企业带来了一系列新的问题。高温高浓发酵导致的高渗透压、高乙醇含量等问题对酵母的生长繁殖和发酵造成的影响是不可避免的,进而会对啤酒的口感、风味及酵母的絮凝性等方面带来一些不良的影响。发酵结束时啤酒酵母能够迅速地絮凝沉降,不但可以减少分离酵母所产生的能源消耗,也可以避免酵母长时间悬浮于发酵液中发生自溶现象,而超过5%的酵母发生自溶,将对啤酒质量造成难以挽回的影响[25]。针对高温高浓发酵技术的研究,国内学者多从啤酒的风味物质出发,而酵母絮凝性方面的研究相对缺乏[4, 16];国外学者的研究多集中在絮凝基因的调控机制方面,有关絮凝基因应用方面的研究较少[13, 26]。因此探索絮凝基因和高级醇代谢基因的协同改造是一条同时解决高温高浓发酵条件下酿酒酵母絮凝性问题和高级醇合成问题的有效途径。
本研究以工业啤酒酵母S6为研究对象,运用基因同源重组的方式过量表达酵母絮凝性关键基因FLO5的同时,敲除参与高级醇合成代谢的BAT基因,最终获得絮凝能力较强、高级醇合成水平适宜的工业啤酒酵母重组菌株S6-BF2B1。研究结果表明,重组菌株S6-BF2B1在高温高浓的发酵条件下,不仅极大地增强了发酵结束后酵母菌株的絮凝能力,而且啤酒风味物质中高级醇的含量也得到了显著的降低,达到了适宜的醇酯比例。实验结果对发酵结束后酵母菌株的沉降分离以及改善啤酒中的醇酯比例具有积极的意义,同时也表明BAT基因和FLO5基因在改造过程中不存在相互协同或相互干扰的现象。酵母细胞质中编码支链氨基酸转氨酶的BAT2基因以及线粒体中编码支链氨基酸转氨酶的BAT1基因能够将支链氨基酸转化为高级醇合成的重要前提物质α-酮酸,因而具有促进高级醇合成的作用;文中对BAT基因的敲除也达到了降低工业啤酒酵母S6高级醇合成水平的效果。
通过对FLO5基因、BAT2基因以及BAT1基因的遗传改造增强了酵母细胞在高温高浓发酵条件下的絮凝性同时降低了高级醇的合成水平,这为研究酿酒酵母的絮凝性和高级醇的代谢提供了一定的理论依据,同时对指导啤酒的高温高浓发酵技术具有重要的实践价值。
| [1] |
Wan CY, Zhao HF, Zhao MM. Effects of very high gravity brewing on the metabolism of Saccharomyces cerevisiae.
Food Ferment Ind, 2010, 36(12): 55–58.
(in Chinese). 万春艳, 赵海锋, 赵谋明. 超高麦汁浓度酿造对啤酒酵母代谢的影响. 食品与发酵工业, 2010, 36(12): 55-58. |
| [2] |
Yu ZM. The metabolic analysis and regulation of brewer's yeast under very high gravity brewing[D]. Guangzhou: South China University of Technology, 2011 (in Chinese). 俞志敏.啤酒超高浓酿造中酵母代谢的分析与调控[D].广州: 华南理工大学, 2011. |
| [3] | Lei HJ, Zhao HF, Yu ZM, et al. Effects of wort gravity and nitrogen level on fermentation performance of brewer's yeast and the formation of flavor volatiles. Appl Biochem Biotechnol, 2012, 166(6): 1562–1574. DOI: 10.1007/s12010-012-9560-8 |
| [4] |
Liu ML, Chang ZM, Yin H, et al. Research of improving the ratio of alcohol ester of high gravity brewing.
Sci Technol Food Ind, 2015, 36(19): 190–194, 279.
(in Chinese). 刘明丽, 常宗明, 尹花, 等. 高浓啤酒醇酯调控技术的研究与应用. 食品工业科技, 2015, 36(19): 190-194, 279. |
| [5] |
Du ZL, Cheng YF, Zhu H, et al. Improvement of acetic acid tolerance and fermentation performance of industrial Saccharomyces cerevisiae by overexpression of flocculent gene FLO1 and FLO1c.
Chin J Biotech, 2015, 31(2): 231–241.
(in Chinese). 杜昭励, 程艳飞, 朱卉, 等. 絮凝基因FLO1及FLO1c高表达提高工业酿酒酵母乙酸耐受性及发酵性能. 生物工程学报, 2015, 31(2): 231-241. |
| [6] | Govender P, Domingo JL, Bester MC, et al. Controlled expression of the dominant flocculation genes FLO1, FLO5, and FLO11 in Saccharomyces cerevisiae. Appl Environ Microbiol, 2008, 74(19): 6041–6052. DOI: 10.1128/AEM.00394-08 |
| [7] | Nayyar A, Walker G, Wardrop F, et al. Flocculation in industrial strains of Saccharomyces cerevisiae: role of cell wall polysaccharides and lectin-like receptors. J Inst Brew, 2017, 123(2): 211–218. DOI: 10.1002/jib.v123.2 |
| [8] | van Holle A, Machado MD, Soares EV. Flocculation in ale brewing strains of Saccharomyces cerevisiae: re-evaluation of the role of cell surface charge and hydrophobicity. Appl Microbiol Biotechnol, 2012, 93(3): 1221–1229. DOI: 10.1007/s00253-011-3502-1 |
| [9] | Soares EV, Teixeira JA, Mota M. Effect of cultural and nutritional conditions on the control of flocculation expression in Saccharomyces cerevisiae. Can J Microbiol, 1994, 40(10): 851–857. DOI: 10.1139/m94-135 |
| [10] | Jin YL, Speers RA. Effect of environmental conditions on the flocculation of Saccharomyces cerevisiae. J Am Soc Brew Chem, 2000, 58(3): 108–116. |
| [11] | Soares EV. Flocculation in Saccharomyces cerevisiae: a review. J Appl Microbiol, 2011, 110(1): 1–18. |
| [12] |
Zhou B, Zhang CY, Chen YF, et al. Effects of high gravity and high temperature on fermentation performance of beer yeast.
China Brew, 2016, 35(2): 9–12.
(in Chinese). 周波, 张翠英, 陈叶福, 等. 高浓高温对啤酒酵母发酵性能的影响. 中国酿造, 2016, 35(2): 9-12. |
| [13] | Dietvorst J, Brandt A. Flocculation in Saccharomyces cerevisiae is repressed by the COMPASS methylation complex during high-gravity fermentation. Yeast, 2008, 25(12): 891–901. DOI: 10.1002/yea.v25:12 |
| [14] | Tofalo R, Perpetuini G, Di Gianvito P, et al. Genetic diversity of FLO1 and FLO5 genes in wine flocculent Saccharomyces cerevisiae strains. Int J Food Microbiol, 2014, 191: 45–52. DOI: 10.1016/j.ijfoodmicro.2014.08.028 |
| [15] | Pires EJ, Teixeira JA, Brányik T, et al. Yeast: the soul of beer's aroma——a review of flavour-active esters and higher alcohols produced by the brewing yeast. Appl Microbiol Biotechnol, 2014, 98(5): 1937–1949. DOI: 10.1007/s00253-013-5470-0 |
| [16] |
Yang XL, Luo ZM, Hu SP, et al. Reducing total higher alcohol content in high gravity fermentation of beer.
Food Sci, 2011, 32(9): 188–192.
(in Chinese). 杨小兰, 罗正明, 胡仕屏, 等. 降低高浓啤酒发酵中高级醇含量的研究. 食品科学, 2011, 32(9): 188-192. |
| [17] | Saerens SMG, Verbelen PJ, Vanbeneden N, et al. Monitoring the influence of high-gravity brewing and fermentation temperature on flavour formation by analysis of gene expression levels in brewing yeast. Appl Microbiol Biotechnol, 2008, 80(6): 1039–1051. DOI: 10.1007/s00253-008-1645-5 |
| [18] | Eden A, van Nedervelde L, Drukker M, et al. Involvement of branched-chain amino acid aminotransferases in the production of fusel alcohols during fermentation in yeast. Appl Microbiol Biotechnol, 2001, 55(3): 296–300. |
| [19] | Styger G, Jacobson D, Prior BA, et al. Genetic analysis of the metabolic pathways responsible for aroma metabolite production by Saccharomyces cerevisiae. Appl Microbiol Biotechnol, 2013, 97(10): 4429–4442. DOI: 10.1007/s00253-012-4522-1 |
| [20] | Zhang CY, Qi YN, Ma HX, et al. Decreased production of higher alcohols by Saccharomyces cerevisiae for Chinese rice wine fermentation by deletion of Bat aminotransferases. J Ind Microbiol Biotechnol, 2015, 42(4): 617–625. |
| [21] | Wu DG, Guo XW, Lu J, et al. A rapid and efficient one-step site-directed deletion, insertion, and substitution mutagenesis protocol. Anal Biochem, 2013, 434(2): 254–258. |
| [22] | Gietz RD, Schiestl RH. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nat Protoc, 2007, 2(1): 31–34. DOI: 10.1038/nprot.2007.13 |
| [23] | Güldener U, Heck S, Fiedler T, et al. A new efficient gene disruption cassette for repeated use in budding yeast. Nucleic Acids Res, 1996, 24(13): 2519–2524. DOI: 10.1093/nar/24.13.2519 |
| [24] |
Zhang YY, Screening of brewer's yeast with low acetaldehyde[D]. Wuxi: Jiangnan University, 2011 (in Chinese). 张媛媛.低产乙醛啤酒酵母的选育与研究[D].无锡: 江南大学, 2011. |
| [25] |
Li J, Wang JJ, Li Q. Overexpression of FKS1 by 18S rDNA targeted influence on yeast autolysis.
Chin J Biotech, 2015, 31(9): 1344–1354.
(in Chinese). 李佳, 王金晶, 李崎. 18S rDNA介导的FKS1基因过表达对酵母自溶性能的影响. 生物工程学报, 2015, 31(9): 1344-1354. |
| [26] | Dietvorst J, Brandt A. Histone modifying proteins Gcn5 and Hda1 affect flocculation in Saccharomyces cerevisiae during high-gravity fermentation. Curr Genet, 2010, 56(1): 75–85. |
 2019, Vol. 35
2019, Vol. 35




