中国科学院微生物研究所、中国微生物学会主办
文章信息
- 潘飞翔, 汤定钦, 周明兵
- Pan Feixiang, Tang Dingqin, Zhou Mingbing
- 一个毛竹LTR反转录转座子结构鉴定及表达模式分析
- Identification and expression pattern analysis of a Moso Bamboo LTR retrotransposon
- 生物工程学报, 2019, 35(3): 445-457
- Chinese Journal of Biotechnology, 2019, 35(3): 445-457
- 10.13345/j.cjb.180318
-
文章历史
- Received: August 1, 2018
- Accepted: December 5, 2018
- Published: January 9, 2019
2. 浙江省竹资源与高效利用协同创新中心 浙江农林大学,浙江 杭州 311300
2. Zhejiang Provincial Collaborative Innovation Center for Bamboo Resources and High-Efficiency Utilization, Zhejiang A & F University, Hangzhou 311300, Zhejiang, China
转座子广泛存在于真核生物基因组中,是基因组的主要组成成分。转座子主要包括DNA转座子和RNA转座子,RNA转座子又称为反转录转座子。长末端重复序列(Long terminal repeat,LTR)转座子属于反转录转座子的一个大类,包含有Ty1-Copia、Ty3-Gypsy、Bel/Pao、Retroviridae、Caulimoviridae、LARD (Large retrotransposons derivatives)和TRIM (Terminal-repeat retrotransposons in miniature) 7类[1]。LTR反转录转座子有自主转座子和非自主转座子两种。自主转座子结构完整,具有自主转座功能;而非自主转座子结构不完整,无法在单独存在时转座,只有在自主转座子的协助下才能发生转座。植物LTR反转录转座子的长度通常在2–18 kb之间,两端各有一个长度约100–5 000 bp正向重复的长末端重复序列(LTRs),结构通常为5′-TG…CA-3′。在5′和3′末端两侧通常具有4–6 bp的末端靶位点重复序列(Target site duplications,TSDs)[2]。LTRs不编码蛋白质,但包含转录的起始信号、终止信号和一些顺式作用元件,而编码区有1–3个开放阅读框(ORF)编码转座所需的酶类,主要包括2个与转座有关的基因,即GAG (种属特异抗原)基因和POL (聚合酶)基因。GAG基因编码的蛋白质(Retrotransposon gag protein,GAG)参与反转录转座子RNA的成熟与包装,使反转录转座子的RNA整合到基因组。POL基因是反转录转座子复制和转座所必需的基因,包括编码蛋白酶基因(Pepsin-like aspartate proteases,AP)、整合酶基因(Integrase,INT)、反转录酶基因(Reverse transcriptases,RT)和RNA酶基因(Ribonuclease H,RH)[3-4]。
大量LTR反转录转座子稳定存在于真核生物基因组内,在受到逆境和甲基化水平的改变而激活转座。短期铁过量会导致日本晴幼苗内部分LTR反转录转座子表达上调[5],热处理可以激活粟酒裂殖酵母中一类LTR反转录转座子Tf1[6]和拟南芥中的LTR反转录转座子ONSEN[7],激光辐射诱导水稻多个位点发生MITE类转座子mPing转座[8],一些非生物因素(如机械作用、水杨酸(SA)、茉莉酮酸(JA)等)可刺激Tto1转座子发生转座[9],而活跃转座子又被甲基化而沉默[10]。
LTR反转录转座子在毛竹基因组含量也非常丰富,其中Ty3-Gypsy类LTR反转录转座子有24.6%,Ty1-Copia类LTR反转录转座子有12.3%[11],全部LTR反转录转座子占全部转座子序列的一半以上,LTR反转录转座子插入基因组的时间主要分布于200–500万年前,晚于毛竹基因组四倍化的时间[12]。目前,本课题组已从毛竹基因组中克隆到6个全长的LTR反转录转座子(Ph-LTR1、PHRE1、PHRE2、PHRE3、PHRE4和PHRE5) [13-14],其中PHRE1、PHRE2和PHRE5能在逆境下转录激活,PHRE1和PHRE2还能在拟南芥异源转座。
为了在毛竹中开发更多的活性LTR反转录转座子,应用于竹子诱变育种,本研究利用公布的毛竹基因组数据库,选取了一个结构完整、新近插入的LTR反转录转座子为研究对象,命名为Ph-LTR2 (Phyllostachys edulis LTR retrotransposon 2),并对Ph-LTR2转座子的结构及在毛竹基因组的分布进行了系统分析,通过实时荧光定量PCR详细调查Ph-LTR2在不同非生物胁迫下的表达模式,揭示不同胁迫与Ph-LTR2转座活性的关系,为开发基于Ph-LTR2转座子标签奠定了一定的理论基础。
1 材料与方法 1.1 材料毛竹幼叶、成熟叶和根以及笋均取自同一株毛竹(栽种于浙江农林大学亚热带森林培育国家重点实验培育基地翠竹园)。
毛竹Phyllostachys edulis种子来自广西灵川县,采自当年同一棵开花毛竹所结的种子,经培育获得毛竹实生苗。
1.2 方法 1.2.1 毛竹基因组DNA与RNA的提取实验中所有种子均采用同一棵毛竹当年所结的种子。将毛竹种子分成未处理种子、甲基化抑制剂处理种子、辐照处理种子3类。
未处理种子选择饱满以及大小相近的种子,用蒸馏水冲洗1遍,用70%酒精消毒种子表面30 s,再用蒸馏水冲洗3遍,无菌蒸馏水浸种24 h以恢复种子活力。置于灭菌的双层滤纸上萌发,25 ℃并全程黑暗,保持水分充足,2–3周后将幼苗转入温室培养。种子萌发所需的营养土配比为:珍珠岩:蛭石:泥炭土=1:1:1。萌发温度为25 ℃。当幼苗生长到4至5片叶时,全部转移到直径90 cm的花盆中继续生长。
选择5-氮杂胞苷作为甲基化抑制剂,浓度设置50、150、250 µmol/L,共3个梯度。用70%酒精消毒种子表面30 s,无菌水冲洗3遍,用不同浓度氮杂胞苷浸种24 h。置于双层滤纸床上发芽。种子萌发环境25 ℃,黑暗处理,使用不同浓度的氮杂胞苷代替灭菌水浸润种子,2–3周后将幼苗移入温室继续培养4个月,培养条件同未处理。每个梯度采5株作为重复。
辐照剂量设置30、50、70 GY,共3个梯度。辐射源为浙江省农业科学院作物与核技术利用研究所辐照中心提供的137Cs-γ。种子辐照后同未处理种子一样,萌发发芽。
选取萌发90 d、长势一致且健康的未处理毛竹种子幼苗(幼苗处于8叶1心期)分成对照、高盐处理、高温胁迫处理及低温胁迫处理4组。对照组培养条件不变,不做任何处理。将NaCl配置成0.1、0.2、0.3 mol/L的溶液对毛竹实生苗浇灌3 d[15],后续处理同对照组。将高温组与低温组实生苗分别置于42 ℃与4 ℃培养箱,分别处理4 h与16 h[16],后续处理同对照组。
取对照组、辐照组、甲基化抑制剂组、高盐处理组、高温胁迫处理组及低温胁迫处理组的叶片液氮速冻,−80 ℃保存备用。
以毛竹嫩叶为实验材料,通过CTAB法提取毛竹基因组DNA,检测浓度后−20 ℃储存。取对照组、辐照组、甲基化抑制剂组、高盐处理组、高温胁迫处理组及低温胁迫处理组实生苗叶片,与来自同一株野外毛竹的叶(成熟叶)、笋以及根,通过Trizol法提取总RNA,利用TaKaRa公司的PrimeScriptTM 1st Strand cDNA Synthesis Kit试剂盒反转录成cDNA,−20 ℃储存。
1.2.2 Ph-LTR2的生物信息学分析根据Zhou等建立的毛竹LTR反转录转座子数据库[17],选取了一个结构完整、新近插入的LTR反转录转座子为研究对象,命名为Ph-LTR2 (Phyllostachys edulis LTR retrotransposon 2)。为了分析Ph-LTR2转座子的序列特征,利用NetGene2 Server (http://www.cbs.dtu.dk/services/NetGene2/)工具查找Ph-LTR2转座子序列的外显子,然后通过DNAMAN软件将其翻译为氨基酸序列,通过NCBI上的blastp在线比对确认,对转座子的结构进行鉴定。
在Gypsy Database (http://www.gydb.org/index.php/Main_Page)下载Ty3-gypsy各个家族的代表性反转录转座子RT氨基酸序列(Del亚家族的Del、Retrosat2、Peabody和Tma,CRM亚家族的CRM和Beetle1,Reina亚家族的Reina、lfg7、Gimli和Gloin,REM1亚家族的REM1,Ty1-Copia家族的Olco1),与Ph-LTR2转座子的RT氨基酸序列比对,利用MEGA6软件中Neighbor-Joining方法构建进化树,分析其所在的转座子家族。根据Ph-LTR2转座子序列在BambooGDB数据库(http://www.bamboogdb.org/index.jsp)中搜索Ph-LTR2转座子在毛竹基因组的分布。利用PlantCARE在线软件分析了LTR序列中顺式作用元件的组成。
在反转录过程中,单一模板合成LTR反转录转座子两端的LTR,所以在整合至基因组时,DNA序列是相同的。如果已知宿主的DNA替换速率,就可通过计算同一转座子两端LTR序列的分化度来估算转座子的插入时间[18]。比对Ph-LTR2转座子两端LTR序列的同源性,求得分化度K,用公式T= K/2r (r代表LTR序列的平均替换率,约为1.3×10–8 bp/年[19])计算插入时间。
1.2.3 Ph-LTR2的基因序列克隆与cDNA序列克隆以提取的毛竹基因组DNA为模板扩增Ph-LTR2的基因序列(引物见表 1),PCR扩增反应体系:DNA,1.0 μL;10×LA PCR缓冲液,5.0 μL;dNTPs,6.0 μL;Ph-LTR2-F,2.0 μL;Ph-LTR2-R,2.0 μL;LA Taq,0.5 μL;ddH2O补充至50 μL。扩增参数:94 ℃ 5 min;94 ℃ 40 s,65 ℃ 40 s,72 ℃ 5 min,35个循环;72 ℃ 10 min;4 ℃保温。PCR产物经琼脂糖凝胶电泳后使用SIMGEN公司的凝胶DNA回收试剂盒回收,连接到pMD18-T载体,热击法转化到大肠杆菌菌株DH5α中,菌检后−80 ℃保存。
| Primer | Sequence (5′–3′) | Function |
| Ph-LTR2-F | CCCAAGAAGAGGATGTAAAG | Amplification of Ph-LTR2 transposon |
| Ph-LTR2-R | CAAAGGATCGCTGGTAGGAC | |
| PheACT2-2F | CAGCAACTGGGATGATATGGAGAA | RT-PCR primers for PheACT2 |
| PheACT2-2R | TGGCAACGTACATAGCAGGAGTGT | |
| Ph-LTR2-INT-F | GGTGACCTCTGTGGCCCCGTAA | INT domain primers for RT-PCR |
| Ph-LTR2-INT-R | TCCGTGCGTAGGACCCGAAGC | |
| Ph-LTR2-RT-F | ATCAAGGTTAGTGTGAGTCC | RT domain primers for RT-PCR |
| Ph-LTR2-RT-R | CCGAAGGTCCAACTTAGAG | |
| Ph-LTR2-RH-F | TAGAGACGGATGCTTGTG | RH domain primers for RT-PCR |
| Ph-LTR2-RH-R | CTCCAGAATTGCCATTGC | |
| Ph-LTR2C-F | ATGGTGGCAAAGACTAGATCCCA | Amplification of Ph-LTR2 cDNA |
| Ph-LTR2C-R | TCAAGGAGTAAAACTCGGGAAGGTC |
以毛竹笋cDNA为模板扩增Ph-LTR2的cDNA序列(引物见表 1),PCR扩增反应体系:cDNA,1.0 μL;10×LA PCR缓冲液,5.0 μL;dNTPs,6.0 μL;Ph-LTR2C-F,2.0 μL;Ph-LTR2C-R,2.0 μL;LA Taq,0.5 μL;dd H2O补充至50 μL。扩增参数:94 ℃ 5 min;94 ℃ 40 s,58 ℃ 40 s,72 ℃ 5 min,35个循环;72 ℃ 10 min;4 ℃保温。PCR产物经琼脂糖凝胶电泳后使用SIMGEN公司的凝胶DNA回收试剂盒回收,经杭州擎科梓熙生物技术有限公司测序。
1.2.4 Ph-LTR2转座子INT、RT和RH检测采用荧光定量PCR (qRT-PCR)方法检测Ph-LTR2转座子INT、RT和RH的表达水平。内参基因选取参考文献中的ACT2-2,命名为PheACT2[20]。分别在Ph-LTR2的RT序列、RH序列和INT序列设计引物(表 1),根据不同引物在不同样品中的值,按照2–ΔCt法(无对照)计算出Ph-LTR2转座子INT、RT和RH在不同部位的相对表达量[20],按照2–ΔΔCt法(有对照)计算出不同处理下Ph-LTR2转座子INT、RT和RH的相对表达量。每个处理3组重复,每个重复3个样本,最终结果的平均值绘成柱形图,在SPSS软件上采用LSD法分析各相对表达量与CK的显著性差异,极显著结果(误差水平小于1%)在柱形图上用星号表示。
2 结果与分析 2.1 Ph-LTR2转座子序列克隆与cDNA序列检测提取的毛竹基因组DNA经1%琼脂糖凝胶电泳检测;用紫外分光光度计测定浓度为1 000 ng/μL左右,OD260/OD280值在1.8–2.0之间。DNA质量和纯度都符合实验要求,可用于后续实验。
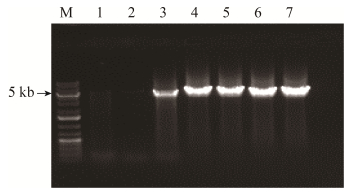
以毛竹基因组DNA为模板,通过PCR扩增Ph-LTR2转座子。Ph-LTR2转座子全长6 000 bp左右(图 1的泳道3)。PCR扩增产物在琼脂糖凝胶电泳确认条带后割胶回收目的片段。
|
| 图 1 pMD18-Ph-LTR2菌落PCR电泳图 Fig. 1 Colony PCR of pMD18-Ph-LTR2 in E. coil. M: marker; lane 1–2: negative control; lane 3: positive control; lane 4–7: positive colony. |
| |
将胶回收产物连接pMD18-T载体,转入大肠杆菌DH5α,菌落PCR (图 1)验证后将获得的阳性克隆送往Invitrogen公司测序,测序验证成功的质粒命名为pMD18-Ph-LTR2,同时保存pMD18-Ph-LTR2的大肠杆菌菌液。
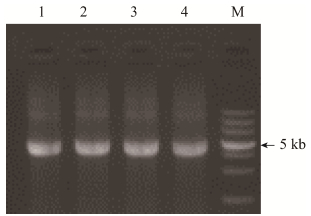
为验证该转座子是否能够转录,以毛竹笋cDNA为模板,通过PCR扩增Ph-LTR2转座子的cDNA序列。Ph-LTR2转座子ORF长度约为5 000 bp (图 2)。PCR扩增产物测序结果与基因组预测一致。
|
| 图 2 Ph-LTR2转座子cDNA的PCR扩增电泳图 Fig. 2 The PCR result of Ph-LTR2 transposon cDNA. M: marker; lane 1–4: the PCR result from the same P. edulis shoots cDNA. |
| |
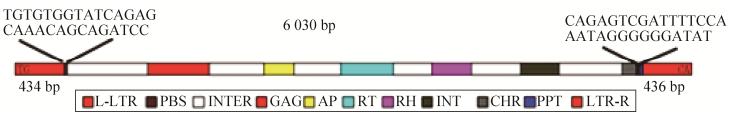
Ph-LTR2全长6 030 bp,右端LTR长度为478 bp,左端LTR长度为489 bp,开放阅读框含有转座所需6种酶的编码序列,结构顺序依次为5′-GAG-PR-RT-RH-INT-CHR-3′,根据结构属于Ty3-Gypsy家族成员。
Ph-LTR2全长序列如图 3所示,包括左端LTR (L-LTR)、6种酶和右端LTR (LTR-R)的序列及结构。核苷酸序列编码区含有4 971 bp的开放阅读框,共编码1 659个氨基酸。其中211–391位为反转录转座子GAG蛋白核心区,其编码的蛋白主要负责反转录转座子RNA的成熟和包装,其保守性较差;第553–642位为PR蛋白酶核心区,水解酶存在于很多反转录病毒、反转录转座子元件中,参与反转录完成后,负责将多聚蛋白前体切割为功能性多肽;第780–934位为RT核心区,编码反转录酶,能够催化单链RNA或DNA合成DNA,是转座子转座的必要条件;第1 049–1 163位为LTR反转录转座子中Ty3-Gypsy家族RH核心区,编码核糖核酸酶H,这是一种广泛分布于原核生物和真核生物的水解酶,负责原始RNA模板的水解,RNA模板是病毒生命周期中逆转录过程后产生的RNA/DNA杂交体的一部分;第1 311–1 421位为INT结构域,负责编码整合酶,催化反转录转座子插入宿主基因组;第1 608–1 651位为染色质组织修饰域(Chromatin organization modifier,CHR),对染色质的重构及基因的表达起到重要作用。PBS序列为5′-TGTGTGGTATCAGAGCAAAC AGCAGATCC-3′,PPT的序列为5′-CAGAGTCGA TTTTCCAAATAGGGGGGATAT-3′。
|
| 图 3 Ph-LTR2转座子的结构 Fig. 3 Structure of the Ph-LTR2. |
| |
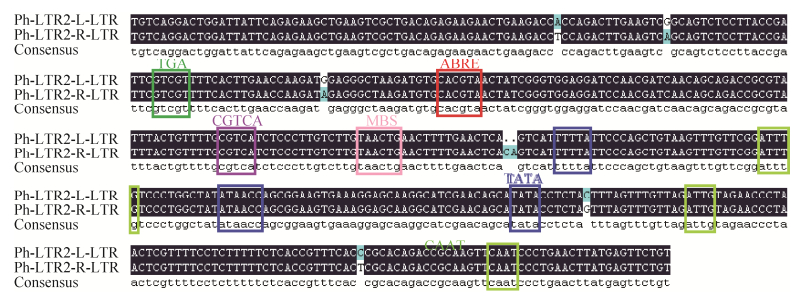
Ph-LTR2转座子的5′ LTR序列与3′ LTR序列非常相似(图 4),只有其中7个核苷酸序列不一致。LTR序列富含多种顺式作用元件,包括:3个核心启动元件TATA-box,3个启动元件CAAT-box,1个与生长素相关的调控元件TGA,1个与脱落酸相关的调控元件ABRE,1个茉莉酸甲酯调控元件CGTGCA-motif,1个干旱反应元件MBS (MYB binding site,MBS),以上所有顺式作用元件均已在图 4标出。
|
| 图 4 Ph-LTR2转座子LTR序列比对示意图 Fig. 4 The alignment of LTR sequences of Ph-LTR2. |
| |
通过公式T=K/2r推导插入时间。Ph-LTR2转座子两端LTR同源性为96.41%,计算得出插入时间均约为61.92万年。
根据Ph-LTR2的结构可以推测Ph-LTR2属于Ty3-Gypsy家族,在Gypsy Database下载其他Ty3-Gypsy各亚家族代表序列,根据进化树拓扑结构可知Ph-LTR2属于Ty3-Gypsy的Reina亚家族(图 5)。
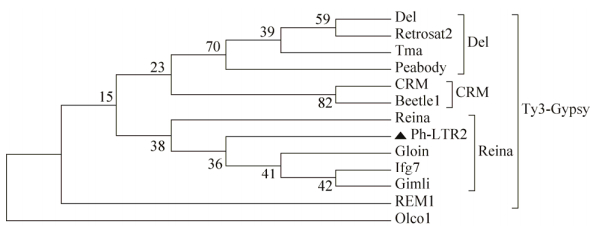
|
| 图 5 Ph-LTR2转座子进化树 Fig. 5 Ph-LTR2 phylogenetic tree. |
| |
通过Ph-LTR2转座子序列在毛竹数据库的比对,共鉴定5个拷贝,以它们在scaffold的位置命名。分别为:PH01001192_35073-40113、PH01007203_ 7355-11931、PH01000426_369740-375716、PH01001071_ 150832-145883和PH01001876_177292-170516。其中PH01001876_177292-170516缺失部分右侧LTR序列,PH01001192_35073-40113缺失GAG基因区,PH01007203_7355-11931缺失INT基因区,除PH01001876_177292-170516外所有拷贝均缺失全部或部分左右LTR序列(图 6)。PH01000426_ 369740-375716和Ph-LTR2完全一致,是同一个拷贝。
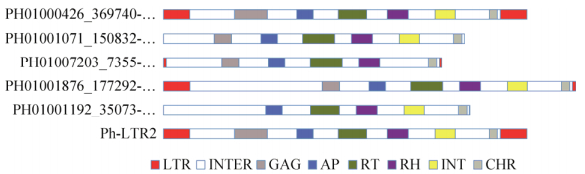
|
| 图 6 Ph-LTR2转座子在基因组中的各拷贝序列比对图 Fig. 6 The copy sequence alignment of Ph-LTR2 in P. edulis genome. |
| |
在Ph-LTR2转座子6个结构域中,选取了INT、RT和RH检测它们的表达模式。比对INT、RT和RH在野生毛竹不同部位中的相对表达量,INT在根中相对表达量最高,叶片中略低于根,笋的相对表达量最低;RH在根里相对表达丰度最高,其次是叶,笋中表达最低;而RT在叶片中表达丰度最高,其次才是根,笋中表达同样最低(图 7)。三个结构域在不同组织相对表达量差异极显著。

|
| 图 7 Ph-LTR2转座子INT、RT和RH在不同部位相对表达量 Fig. 7 Relative expression level of INT, RT and RH of Ph-LTR2 in bamboo shoots, leaves and roots. Statistical significance was calculated using LSD t-test (*P < 0.001). |
| |
在辐照处理过程中,INT、RT和RH的相对表达量呈现先上升后下降的趋势,在30 GY处理的时候大幅上升,50 GY辐照下开始下降,70 GY时INT、RT和RH的相对表达量继续下降,但幅度不大。其中RT的变化幅度最大,在30 GY的表达量上调最为明显,涨幅达到了44倍,随着辐射剂量增加,相对表达量下降的也最大,50 GY时下降为原来的3.03%,70 GY时降为30GY的2.18%;INT变化程度略小于RT,30 GY辐射处理组相对表达量上升明显,涨幅为31倍,50 GY和70 GY的相对表达量相近,下降到30 GY时的9.82%和9.20%,幅度小于RT;RH表达水平变化不大,其他结构域涨幅最大的30 GY只有对照组的1.3倍,50 GY和70 GY相对30 GY的下降程度也不大,只下降了22.96%和20.16% (图 8A)。

|
| 图 8 不同处理Ph-LTR2转座子INT、RT和RH相对表达量 Fig. 8 Relative expression level of INT, RT and RH of Ph-LTR2 in treatment bamboo seedlings. (A) Radiation treatment. (B) Methylation inhibitor treatment. (C) Different temperature stress. (D) Different concentrations of salt solution. Statistical significance was calculated using LSD t-test (*P < 0.001). |
| |
在甲基化抑制剂处理组中,INT、RT和RH的相对表达量大致呈现上升趋势,50 µmol/L浓度时相对表达量上升,150 µmol/L浓度时相对表达量较50 µmol/L浓度略微下降,在250 µmol/L的浓度处理下达到最高值。其中RT的相对表达量涨幅最大,尤其是250µmol/L时远高于其他结构域,相对于对照组涨了8.3倍,差异极显著;INT在50 µmol/L和150 µmol/L时与RT的相对表达量相近,250 µmol/L时上升,但程度低于RT,相对CK只涨了4.9倍,差异极显著;RH在3个浓度的甲基化抑制剂处理中略微上升,150 µmol/L上升最少,50µmol/L和250 µmol/L上升幅度相近,略大于150µmol/L,但相对于其他两个结构域来说,上升的幅度非常低,3个浓度相对于对照组分别上升了1.6倍、1.2倍和1.6倍,差异均不显著(图 8B)。
在低温处理组中,INT、RT和RH的相对表达量均升高,其中INT升高的幅度最大,相对于对照组上升了9.9倍,差异极显著,RT其次,但升高幅度不大,只有1.6倍,差异不显著,RH变化近似于RT,只上升1.2倍,同样差异不显著(图 8C)。而高温处理组中,INT、RT和RH的相对表达量均大幅升高,上升幅度高于低温处理组,INT升高的幅度最大,上升达11倍,差异极显著,但相较于低温处理结果变化不大,RT上升幅度虽然次于INT,上升达5倍,差异极显著,但与低温处理组相比,上升的幅度明显,RH相较其他两个结构域依旧变化不大,但也可以明显看出有所上调,涨幅达3倍,差异极显著(图 8C)。
在高盐处理组中,INT、RT和RH的相对表达量基本呈现先上升后下降的趋势。在0.1 mol/L处理组中,INT、RT和RH的相对表达量达到最高值,分别达到对照组的62倍、5倍和52倍,在0.2 mol/L和0.3 mol/L处理组中持续下降,INT分别下降了36%和57%,RT下降了45%和75%,RH分别下降了62%和68%。其中INT与RH相对表达量变化相近,远远大于RT (图 8D)。
3 讨论本研究通过生物信息学技术和分子生物学手段鉴定一个典型的LTR反转录转座子Ph-LTR2,该反转录转座子具有转座所需的6种酶和高度相似LTR的序列,是一个结构完整的反转录转座子,且在毛竹笋cDNA中检测到其表达,说明其具有表达活性。Ph-LTR2的LTR序列中富含顺式调控元件,可能会在一些外界胁迫下激活转座,可以作为毛竹基因标签开发的候选转座子。
3.1 Ph-LTR2转座子是一个结构完整、富含顺式调控元件的转座子毛竹Ph-LTR2反转录转座子具有完整的LTR序列及转座必需的所有结构域,包括左端LTR、6个酶(GAG、PR、RT、RH、INT、CHR)和右端LTR,结构完整,同时经过实验检测,Ph-LTR2具有转录活性,这些都表明其可能具备转座能力。
在植物基因组中,有报道过几百万年内插入的LTR转座子还存在活性,如小麦中的Wis_116F02-1、水稻中的Mtr6和Mtr76[21-23]。Ph-LTR2转座子5′ LTR长478 bp,3′ LTR长489 bp,存在7个碱基突变,LTR序列同源为96.41%,插入时间约为61.92万年。相较于插入时间为几百万年的活性LTR转座子来说,Ph-LTR2转座子还属于非常年轻的转座子,可能依然具有活性。
Ph-LTR2的LTR序列中富含顺式调控元件,包括:3个核心启动元件TATA-box,3个启动元件CAAT-box,1个茉莉酸甲酯调控元件CGTGCA-motif,1个与生长素相关的调控元件TGA,1个干旱反应元件MBS,1个与脱落酸相关的调控元件ABRE。而茉莉酸甲酯、脱落酸、生长素调控元件和干旱反应元件与外界环境变化有着重大关联,因此可能会在一些外界胁迫下激活转座。
3.2 Ph-LTR2转座子能够响应外界环境变化,改变INT、RT和RH的表达模式1) 细胞DNA甲基化与转座子转录活性关系
转座子转录活性与DNA甲基化密切相关。DNA甲基化是生物体应对转座子转座的一种手段,通过全基因甲基化分析,转座子序列更容易被甲基化[24-25]。转座子在植物内会被甲基化而沉默[10, 26],在DNA甲基转移酶或染色质重塑子DDM1缺失突变体中,一些转座子被去甲基化而解除阻遏被激活[27-28]。甲基化水平在植物不同部位与不同时期也不同,一些生长发育旺盛的部位DNA甲基化程度高,而这些部位的部分转座子活性往往受到抑制,如种子[29]、花药[30]和茎尖[31]。
本研究的结果显示甲基化抑制剂对Ph-LTR2反转录转座子的活性有促进作用。甲基化抑制剂在一定程度上可以降低DNA的甲基化[32],因此降低基因组的DNA的甲基化水平可能可以激活Ph-LTR2反转录转座子的转座行为。同时也发现笋期的Ph-LTR2反转录转座子的活性被明显抑制,这可能和笋期这个时期的甲基化水平变化有关。
2) 逆境与转座子转录活性的关系
逆境胁迫会导致植物体内甲基化水平、内源激素水平与离子浓度等各类生理生化水平的改变,这些改变会导致转座子活性的改变[5, 10, 33]。逆境胁迫过程中的生理生化水平改变的是一个整体,因此虽然部分实验与调控元件关联度不大,但是无法确定这些胁迫会不会以间接的途径影响转座子的活性,所以所有的逆境胁迫实验都是必要的。
辐射作为一种现今常用的育种手段,其对转座子的转座具有一定影响。有研究显示,γ射线会激活水稻mPing等内源转座子转座,以及导致DNA甲基化变异[34],本实验结果显示低剂量的辐射会上调Ph-LTR2转座子的活性。而随着剂量增加Ph-LTR2转座子的活性随之下降,推测其原因是高剂量的辐射对植物体的伤害太大,对植物造成的破坏已经超过植物本身应对能力[35]。
低温和高温胁迫都是植物常见的胁迫,这些胁迫会使植物生理发生变化[36-39]。实验结果显示Ph-LTR2转座子INT、RT和RH的转录水平上调(图 8C),Ph-LTR2转座子的LTR区域含有丰富的顺式调控元件,不适宜植物生长的温度可能导致毛竹的一些转录因子表达增强,如抗寒相关转录因子CBF1、DREB1、MYB和WRKY表达量上升[40],这些转录因子有一定几率与Ph-LTR2转座子顺式调控元件结合,调控Ph-LTR2转座子结构域转录水平。
植物在盐胁迫下,体内一氧化氮和茉莉酸积累[41],甲基化水平也会降低[42],这有一定概率是Ph-LTR2转座子在不同浓度盐胁迫下INT、RT和RH均有不同程度升高的原因(图 8D),这些变化在某种程度上导致Ph-LTR2转座子活性的改变。
4 结论本研究通过对Ph-LTR2的结构、进化及不同胁迫下的表达量进行研究,发现Ph-LTR2的活性具有组织特异性,且会被甲基化抑制剂激活,证明Ph-LTR2在植物体中会因甲基化而沉默,同时在高盐、高温、低温和辐射胁迫下活性均有上升,证明胁迫会促进Ph-LTR2转座,这可能与这些胁迫会导致甲基化水平改变有关。而数量众多的顺式作用元件也从另一个角度证明Ph-LTR2是一个对外界环境变化敏感的反转录转座子。
| [1] | Llorens C, Futami R, Covelli L, et al. The Gypsy Database (GyDB) of mobile genetic elements: release 2.0. Nucleic Acids Res, 2011, 39(D1): D70–D74. |
| [2] | Vitte C, Panaud O, Quesneville H. LTR retrotransposons in rice (Oryza sativa, L.): recent burst amplifications followed by rapid DNA loss. BMC Genomics, 2007, 8(1): 218. DOI: 10.1186/1471-2164-8-218 |
| [3] | Eickbush TH, Jamburuthugoda VK. The diversity of retrotransposons and the properties of their reverse transcriptases. Virus Res, 2008, 134(1/2): 221–234. |
| [4] |
Liang LL, Zhou MB. Plant active LTR retrotransposons: a review.
Chin J Biotech, 2016, 32(4): 409–429.
(in Chinese). 梁琳琳, 周明兵. 植物活性长末端重复序列反转录转座子研究进展. 生物工程学报, 2016, 32(4): 409-429. |
| [5] | Finatto T, de Oliveira AC, Chaparro C, et al. Abiotic stress and genome dynamics: specific genes and transposable elements response to iron excess in rice. Rice, 2015, 8(1): 13. |
| [6] | Feng G, Leem YE, Levin HL. Transposon integration enhances expression of stress response genes. Nucleic Acids Res, 2013, 41(2): 775–789. DOI: 10.1093/nar/gks1185 |
| [7] | Ito H, Gaubert H, Bucher E, et al. An siRNA pathway prevents transgenerational retrotransposition in plants subjected to stress. Nature, 2011, 472(7341): 115–119. DOI: 10.1038/nature09861 |
| [8] |
Wang D. Heritable alterations in DNA methylation pattern and mobilization of transposon induced by laser irradiation in Jijing88[D]. Changchun: Jilin Agricultural University, 2011 (in Chinese). 王丹.激光辐射诱导水稻吉粳88号可遗传DNA甲基化变异和转座子转座激活的研究[D].长春: 吉林农业大学, 2011. |
| [9] | Takeda S, Sugimoto K, Otsuki H, et al. A 13-bp cis-regulatory element in the LTR promoter of the tobacco retrotransposon Tto1 is involved in responsiveness to tissue culture, wounding, methyl jasmonate and fungal elicitors. Plant J, 1999, 18(4): 383–393. DOI: 10.1046/j.1365-313X.1999.00460.x |
| [10] | Saze H, Kakutani T. Differentiation of epigenetic modifications between transposons and genes. Curr Opin Plant Biol, 2011, 14(1): 81–87. DOI: 10.1016/j.pbi.2010.08.017 |
| [11] | Peng ZH, Lu Y, Li LB, et al. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla). Nat Genet, 2013, 45(4): 456–461. DOI: 10.1038/ng.2569 |
| [12] |
Hu T, Ma YJ, Li XP, et al. Identification and evolutionary study of full-length LTR-retrotransposons in moso bamboo genome.
Mol Plant Breed, 2014, 12(6): 1265–1274.
(in Chinese). 胡陶, 马艳军, 李雪平, 等. 毛竹全长LTR逆转座子的鉴定和进化分析. 分子植物育种, 2014, 12(6): 1265-1274. |
| [13] |
Zhou M, Tang DQ, Zhou MB. Cloning, characterization and phylogenetic analysis of a typical long terminal repeat retrotransposon in Phyllostachys heterocycla cv. pubescens.
J Bamboo Res, 2014, 33(3): 1–10.
(in Chinese). 周敏, 汤定钦, 周明兵. 一个毛竹典型LTR转座子的克隆、鉴定及进化分析. 竹子研究汇刊, 2014, 33(3): 1-10. DOI:10.3969/j.issn.1000-6567.2014.03.001 |
| [14] |
Zhou M. Cloning, identification and analysis characteristics of LINEs and Ty3-gypsy retrotransposons from bamboo[D]. Hangzhou: Zhejiang A & F University, 2014 (in Chinese). 周敏.竹子LINEs、Ty3-gypsy类转座子的克隆、鉴定及特性分析[D].杭州: 浙江农林大学, 2014. |
| [15] |
Yang Y, Huang YW, Luo SP, et al. Effects of NaCl stress on chlorophyll fluorescence and physiological characteristics of moso bamboo seedlings.
J Bamboo Res, 2010, 29(1): 29–32.
(in Chinese). 杨洋, 黄业伟, 罗淑萍, 等. NaCl胁迫对毛竹幼苗叶绿素荧光特性及生理指标的影响. 竹子学报, 2010, 29(1): 29-32. DOI:10.3969/j.issn.1000-6567.2010.01.006 |
| [16] |
Liu LL, Chen L, Zhang CY, et al. Characterization of two LEA genes and their response to abiotic stresses in wheat.
Sci Agric Sin, 2014, 47(19): 3736–3745.
(in Chinese). 刘露露, 陈雷, 张春艳, 等. 两个小麦LEA基因的特征及其对非生物胁迫的响应. 中国农业科学, 2014, 47(19): 3736-3745. DOI:10.3864/j.issn.0578-1752.2014.19.002 |
| [17] | Zhou MB, Hu BJ, Zhu YH. Genome-wide characterization and evolution analysis of long terminal repeat retroelements in moso bamboo (Phyllostachys edulis). Tree Genet Genom, 2017, 13(2): 43. DOI: 10.1007/s11295-017-1114-3 |
| [18] | Sanmiguel P, Gaut BS, Tikhonov A, et al. The paleontology of intergene retrotransposons of maize. Nat Genetics, 1998, 20(1): 43–45. DOI: 10.1038/1695 |
| [19] | Ma JX, Jackson SA. Retrotransposon accumulation and satellite amplification mediated by segmental duplication facilitate centromere expansion in rice. Genome Res, 2006, 16(2): 251–259. |
| [20] |
Qi FY, Hu T, Peng ZH, et al. Screening of reference genes used in qRT-PCR and expression analysis of PheTFL1 gene in Moso Bamboo.
Acta Bot Boreal-Occident Sin, 2013, 33(1): 48–52.
(in Chinese). 齐飞艳, 胡陶, 彭镇华, 等. 毛竹实时荧光定量PCR内参基因的筛选及成花基因PheTFL1表达分析. 西北植物学报, 2013, 33(1): 48-52. DOI:10.3969/j.issn.1000-4025.2013.01.010 |
| [21] | Ma J, Bennetzen JL. Rapid recent growth and divergence of rice nuclear genomes. Proc Natl Acad Sci USA, 2004, 101(34): 12404–12410. DOI: 10.1073/pnas.0403715101 |
| [22] | Sanmiguel PJ, Ramakrishna W, Bennetzen JL, et al. Transposable elements, genes and recombination in a 215-kb contig from wheat chromosome 5Am. Funct Integr Genomics, 2002, 2(1/2): 70–80. |
| [23] | Liu JS, Hao W. LTR retrotransposon landscape in Medicago truncatula: more rapid removal than in rice. BMC Genomics, 2008, 9(1): 382. DOI: 10.1186/1471-2164-9-382 |
| [24] | Lippman Z, Gendrel AV, Black M, et al. Role of transposable elements in heterochromatin and epigenetic control. Nature, 2004, 430(6998): 471–476. DOI: 10.1038/nature02651 |
| [25] | Zhang XY, Yazaki J, Sundaresan A, et al. Genome-wide high-resolution mapping and functional analysis of DNA methylation in Arabidopsis. Cell, 2006, 126(6): 1189–1201. DOI: 10.1016/j.cell.2006.08.003 |
| [26] | Willing EM, Rawat V, Mandáková T, et al. Genome expansion of Arabis alpina linked with retrotransposition and reduced symmetric DNA methylation. Nat Plants, 2015, 1(2): 14023. DOI: 10.1038/nplants.2014.23 |
| [27] | Miura A, Yonebayashi S, Watanabe K, et al. Mobilization of transposons by a mutation abolishing full DNA methylation in Arabidopsis. Nature, 2001, 411(6834): 212–214. DOI: 10.1038/35075612 |
| [28] | Singer T, Yordan C, Martienssen RA. Robertson's Mutator transposons in A. thaliana are regulated by the chromatin-remodeling gene Decrease in DNA Methylation (DDM1). Genes Dev, 2001, 15(5): 591–602. DOI: 10.1101/gad.193701 |
| [29] | Feng S, Jacobsen SE, Reik W. Epigenetic reprogramming in plant and animal development. Science, 2010, 330(6004): 622–627. DOI: 10.1126/science.1190614 |
| [30] | Slotkin RK, Vaughn M, Borges F, et al. Epigenetic reprogramming and small RNA silencing of transposable elements in polle. Cell, 2009, 136(3): 461–472. DOI: 10.1016/j.cell.2008.12.038 |
| [31] | Tamaki S, Tsuji H, Matsumoto A, et al. FT-like proteins induce transposon silencing in the shoot apex during floral induction in rice. Proc Natl Acad Sci USA, 2015, 112(8): E901–E910. DOI: 10.1073/pnas.1417623112 |
| [32] |
Liu QX. DNA methylation research of Oryza sativa (Nipponbare) after treated with 5-Aza-2'- deoxycytidine[D]. Hangzhou: Zhejiang University, 2014 (in Chinese). 刘秋香. DNA甲基化抑制剂5-Aza-2'-deoxycytidine处理后水稻的DNA甲基化研究[D].杭州: 浙江大学, 2014. |
| [33] |
Zhou ZB. Expressive activity of retrotransposon atrl by stresses and the effect in fanchangchang jujube by phytoplasma[D]. Hefei: Anhui Agricultural University, 2014 (in Chinese). 周张彬.逆转座子atrl胁迫诱导表达活性研究及植原体对繁昌长枣的影响[D].合肥: 安徽农业大学, 2014. |
| [34] |
Jiang LL. Mobilization of transposons mPing and pong and alterations in DNA methylation induced by γ irradiation in rice[D]. Harbin: Northeast Normal University, 2008 (in Chinese). 姜丽丽. γ射线诱导水稻转座子mPing及Pong的激活和DNA甲基化的变异[D].哈尔滨: 东北师范大学, 2008. |
| [35] |
Wang XX. Biological effects induced on moso seed by 60Coγ-irradiation[D]. Hefei: Anhui Agricultural University, 2010 (in Chinese). 王新新. 60Coγ射线辐照毛竹种子的生物学诱变效应[D].合肥: 安徽农业大学, 2010. |
| [36] |
Rao YC, Yang YL, Huang LC, et al. Research progress on cold stress in rice.
Mol Plant Breed, 2013, 11(3): 443–450.
(in Chinese). 饶玉春, 杨窑龙, 黄李超, 等. 水稻耐冷胁迫的研究进展. 分子植物育种, 2013, 11(3): 443-450. |
| [37] |
Yu XC, Xing YX, Ma H, et al. Changes of hormone in grafted and non grafted cncumber seedlings under low temperature stress.
Acta Hortict Sin, 1999, 26(6): 406–407.
(in Chinese). 于贤昌, 邢禹贤, 马红, 等. 低温胁迫下黄瓜嫁接苗和自根苗内源激素的变化. 园艺学报, 1999, 26(6): 406-407. DOI:10.3321/j.issn:0513-353X.1999.06.014 |
| [38] |
Shen Man. Preliminary study on the relations between membrane permeability, endogenous hormones and cold resistance of ivy.
Acta Hortict Sin, 2005, 32(1): 141–144.
(in Chinese). 沈漫. 常春藤质膜透性和内源激素与抗寒性关系初探. 园艺学报, 2005, 32(1): 141-144. DOI:10.3321/j.issn:0513-353X.2005.01.035 |
| [39] |
Song Y, Cui XS, Chen JJ, et al. The profiling of eleven phytohormones in Pyropia haitanensis under different high-temperature environments.
J Fish China, 2017, 41(10): 1578–1587.
(in Chinese). 宋悦, 崔晓山, 陈娟娟, 等. 不同高温胁迫条件下的坛紫菜中植物激素分析. 水产学报, 2017, 41(10): 1578-1587. |
| [40] |
Feng FM, Shi SJ, Cao Y, et al. Effects of chilling and freezing stress on the physiological metabolism and the expression of cold-related transcription factors in seedlings of Phyllostachys heterocycla cv. Pubescens.
J Bamboo Res, 2014, 33(2): 29–33.
(in Chinese). 冯芳敏, 史世京, 曹颖, 等. 冷冻胁迫对毛竹幼苗生理代谢及抗寒相关转录因子表达的影响. 竹子研究汇刊, 2014, 33(2): 29-33. DOI:10.3969/j.issn.1000-6567.2014.02.006 |
| [41] |
Li J, Chen K, Tang J, et al. Relationship between nitric oxide and JA accumulation in maize seedling under NaCl stress.
Acta Bot Boreal-Occident Sin, 2008, 28(8): 1629–1636.
(in Chinese). 李杰, 陈康, 唐静, 等. NaCl胁迫下玉米幼苗中一氧化氮与茉莉酸积累的关系. 西北植物学报, 2008, 28(8): 1629-1636. DOI:10.3321/j.issn:1000-4025.2008.08.021 |
| [42] |
Lu XK, Wang DL, Yin ZJ, et al. Genomic DNA methylation polymorphism analysis of cotton under NaCl and Na2CO3 stress.
Sci Agrict Sin, 2014, 47(16): 3132–3142.
(in Chinese). 陆许可, 王德龙, 阴祖军, 等. NaCl和Na2CO3对不同棉花基因组的DNA甲基化影响. 中国农业科学, 2014, 47(16): 3132-3142. DOI:10.3864/j.issn.0578-1752.2014.16.002 |
 2019, Vol. 35
2019, Vol. 35




