中国科学院微生物研究所、中国微生物学会主办
文章信息
- 左伟东, 康宁, 李春林, 栾悦, 童晓玲, 代方银, 鲁成
- Zuo Weidong, Kang Ning, Li Chunlin, Luan Yue, Tong Xiaoling, Dai Fangyin, Lu Cheng
- 家蚕超长链脂肪酸延长酶基因Bmelo424的克隆及功能分析
- Cloning and function analysis of elongase of very long chain fatty acid gene Bmelo424 in silkworm
- 生物工程学报, 2019, 35(3): 435-444
- Chinese Journal of Biotechnology, 2019, 35(3): 435-444
- 10.13345/j.cjb.180310
-
文章历史
- Received: October 6, 2018
- Accepted: December 4, 2018
2. 西南大学 生物技术学院,重庆 400715
2. College of Biotechnology, Southwest University, Chongqing 400715, China
超长链脂肪酸延长酶(Elongases of very long chain fatty acid,ELOVL)是定位于内质网中的微粒体酶,在催化合成长链脂肪酸(C16,C18)和超长链脂肪酸(≥C20)的过程中起到限速作用[1]。酵母中首次鉴定出的该酶的编码基因ELO1参与延伸C14脂肪酸到C16,而ELO2和ELO3则分别对延伸C24和C26脂肪酸必不可少[2-3]。目前,在哺乳动物中已经有7个家族成员被鉴定(ELOVL1–ELOVL7)。其中,ELOVL1负责延伸C20到C28的脂肪酸,并与C24鞘脂类的合成相关[4-5]。ELOVL2控制精巢中C28:5n-6和C30:5n-6的脂肪酸合成,进而影响小鼠的精子成熟和雄性生殖力[6]。ELOVL3参与C20–C24脂肪酸的合成,并与毛囊皮脂腺的发育以及饮食性肥胖抵抗(Diet induced obesity resistant,DIO-R)相关[7-8]。ELOVL4参与合成C28和C30饱和脂肪酸以及C28–C38多不饱和脂肪酸,且与黄斑营养不良、脊髓小脑性共济失调和可变性红斑角皮症相关[9-10]。ELOVL5能够延伸C18和C20的多不饱和脂肪酸底物,而且和脊髓小脑性共济失调以及开角型青光眼相关[11-13]。ELOVL6延伸稍短的C12–C16脂肪酸,其基因多态性和Ⅱ型糖尿病相关联[14-15]。ELOVL7延伸C16–C22脂肪酸,并参与前列腺癌细胞增殖和巨细胞病毒复制[16-18]。
与哺乳动物不同,昆虫ELOVL基因数量较多但功能验证的结果较少。果蝇中第一个被鉴定的ELOVL基因elo68α在雄性生殖系统中特异性表达,其产物能够延伸C14和C16单不饱和脂肪酸,参与雄性信息素的合成[19]。在雌性果蝇中专一表达的eloF基因,其产物能够延伸C19-C30脂肪酸,与雌性果蝇信息素合成和求偶行为相关[20]。另外,与ELOVL6基因相似度较高的noa基因,其RNA水平的减少导致果蝇运动障碍和生存能力显著下降,而针对精巢包囊细胞的RNAi会引起雄性不育[21]。过表达果蝇脂肪酸延长酶基因Bond能使酿酒酵母elo2/elo3双缺失致死突变菌株恢复活力,突变Bond基因影响精母细胞胞质分裂;而且Bond基因沉默能显著抑制雄性生育力[22-23]。此外,白纹伊蚊中脂酰辅酶A延长酶(ELO)的表达水平是其滞育卵抗干燥能力的重要影响因素[24]。与表皮碳氢化合物生物合成相关的ELOVL基因,在欧洲蜜蜂中也采用RT-qPCR进行了表达谱分析[25]。桔小实蝇被单增李斯特菌、金黄色葡萄球菌和大肠杆菌侵染后,noa基因的表达上调,参与调节Toll/Imd免疫信号传导途径[26]。在黄粉虫中,TmELO1主要参与C16和C20脂肪酸的合成,且能形成痕量的C22和C24脂肪酸,而TmELO2仅合成C16脂肪酸,这两个基因的RNAi都没有改变黄粉虫的脂肪酸组分,但TmELO1基因的RNAi会导致黄粉虫死亡率上升[27]。
ELOVL基因在昆虫中已有影响育性、抗干燥性、免疫信号传导和致死等方面的功能研究,而家蚕作为重要的经济昆虫和鳞翅目昆虫的典型代表,其ELOVL基因仅有信息分析方面的报道[28]。为了进一步探索ELOVL基因在家蚕中的具体功能,本研究从家蚕幼虫中克隆获得Bmelo424的基因序列,分析其组织表达特征及蛋白结构特点,并借助酿酒酵母真核表达系统和GC-MS探究其基因功能,同时对重组酵母细胞的温度胁迫响应也作了分析。
1 材料与方法 1.1 材料 1.1.1 实验昆虫家蚕品种Dazao取自西南大学家蚕基因资源库,幼虫在25 ℃室温的自然光照条件下以新鲜桑叶饲育。在五龄三天幼虫期取整蚕和各组织冻存于−80 ℃冰箱备用。
1.1.2 试剂耗材DNA胶回收试剂盒和小量质粒提取试剂盒购自Omega公司;感受态细胞Trans1-T1购自北京全式金公司;酿酒酵母INVSc1和表达载体pYES2购自Invitrogen公司;Taq DNA聚合酶、M-MLV反转录试剂盒、pMD19-T载体、DNA Marker、限制性内切酶NotⅠ、BamHⅠ和T4 DNA连接酶等购自TaKaRa公司;酵母抽提物、蛋白胨和葡萄糖等购自生工生物工程(上海)股份有限公司;酵母尿嘧啶缺陷型合成培养基(SC-Ura)购自上海哈灵公司,酵母化学感受态细胞制备试剂盒购自北京天恩泽公司;RNA提取试剂盒购自BioTeke公司;SYBR Green荧光定量PCR试剂盒购自Bio-Rad公司。
1.2 方法 1.2.1 RNA提取及cDNA合成将五龄三天家蚕各组织(头、体壁、中肠、脂肪体、前中丝、后丝、马氏管、精巢、卵巢和血液)及整蚕的冻存样品取出,分别放入液氮预冷后的研钵中,快速研磨至细滑粉末状,用无RNase枪头取适量粉末于Trizol中裂解。通过RNA提取试剂盒获得家蚕各组织及整蚕的总RNA,并使用分光光度计测定每个样品的RNA浓度,之后利用反转录试剂盒制备cDNA模板。
1.2.2 Bmelo424基因的克隆依据家蚕基因组数据库SilkDB中的超长链脂肪酸延长酶信息[28],利用Primer Premier 5.0设计Bmelo424基因(BGIBMGA000424)的克隆引物(表 1)。以五龄三天Dazao整蚕cDNA为模板进行PCR扩增,然后通过1%琼脂糖凝胶电泳检测产物大小。目的DNA片段经切胶回收后,与pMD19-T载体连接,将连接产物转化到大肠杆菌感受态细胞中,通过菌液PCR筛选阳性克隆并进行测序验证。
| Primer name | Primer sequence (5′–3′) | Use |
| Bmelo424-F | ATGCCATTACAGATCTTCTTTGTGC | ORF cloning |
| Bmelo424-R | TCAGTTGCCGGCCGGCAGAACGGTG | |
| Bmelo424-F(BamHⅠ) | GGATCCATGCCATTACAGATCTTCTTTGTGC | Vector |
| Bmelo424-R(NotⅠ) | GCGGCCGCTCAGTTGCCGGCCGGCAGAACGGTG | construction |
| Q-Bmelo424-F | GTTCTTCGGACTCCTCAATACG | qRT-PCR |
| Q-Bmelo424-R | AGAGTAAGATTGGTCGTAGAAGTCCT | |
| sw22934-F | TTCGTACTGGCTCTTCTCGT | qRT-PCR |
| sw22934-R | CAAAGTTGATAGCAATTCCCT | |
| Note: restriction sites are highlighted in italics. | ||
基于文献报道合成Bmelo424基因的荧光定量PCR引物Q-Bmelo424和内参引物sw22934[28],以家蚕五龄三天不同组织的cDNA为模板,检测其组织表达特征。反应总体系为10 μL (SYBR Green Ⅱ4.2 μL,上下游引物各0.3 μL,去离子水4.2 μL,cDNA模板1 μL)。反应程序:95 ℃,30 s;95 ℃,5 s,60 ℃,30 s,共40个循环。每个样品3个生物学重复,收集目的基因和内参基因的Cq值计算其相对表达量。
1.2.4 Bmelo424蛋白序列和结构分析利用在线工具对Bmelo424的跨膜结构域(http://www.cbs.dtu.dk/services/TMHMM/)和磷酸化位点(http://www.cbs.dtu.dk/services/NetPhos/)进行预测,采用Cell-PLoc 2.0[29] (http://www.csbio.sjtu.edu.cn/bioinf/euk-multi-2/)进行其亚细胞定位,通过PSIPRED v3.3 (http://bioinf.cs.ucl.ac.uk/psipred/)进行其二级结构预测。
1.2.5 Bmelo424表达载体的构建通过Bmelo424基因的克隆序列设计分别带有BamHⅠ和NotⅠ的上下游引物(表 1),采用TA克隆的方式首先构建pMD-Bmelo424重组质粒。将pMD-Bmelo424质粒和穿梭载体pYES2同时用BamHⅠ和NotⅠ双酶切,酶切产物经1%琼脂糖凝胶电泳后,分别切胶回收Bmelo424和pYES2目的DNA片段。回收产物用T4 DNA连接酶过夜连接,连接反应体系为:Bmelo424片段7 μL,pYES2片段1 μL,10×T4缓冲液1 μL,T4 DNA连接酶1 μL。连接产物转化大肠杆菌感受态细胞,采用菌液PCR筛选阳性克隆并进行酶切和测序双重验证。
1.2.6 pYES2-Bmelo424的诱导表达依据酵母化学感受态细胞制备试剂盒操作说明,将pYES2-Bmelo424和pYES2分别转化酿酒酵母INVSc1感受态细胞,把菌液涂布在含2%葡萄糖的SC-Ura固体选择培养基中,30 ℃恒温培养48 h,挑取单菌落做菌液PCR进一步验证阳性转化子。
分别挑取阳性转化单菌落接种于10 mL含2%葡萄糖的SC-Ura液体选择培养基中,30 ℃、200 r/min振荡培养并测定OD600吸光度。计算并取出相应体积菌液(参照酵母表达载体pYES2使用手册),3 000 r/min离心5 min,然后将菌体重悬于50 mL含2%半乳糖和1%棉籽糖的SC-Ura液体诱导培养基中,使得初始菌液OD600为0.4,30 ℃、200 r/min振荡培养30 h后,3 000 r/min离心5 min收集菌体,无菌水洗涤菌体3次,真空冷冻干燥48 h,置于−80 ℃冰箱备用,每个样品做3个重复。
1.2.7 重组产物的GC-MS分析脂肪酸提取及甲酯化依据文献略有调整[30]:称取50 mg酿酒酵母冻干粉于10 mL离心管,加入3 mL氢氧化钠甲醇溶液(1 mol/L),振荡混匀,80 ℃水浴20 min,水浴期间多次振荡混匀。甲酯化:往样品中加入3 mL盐酸甲醇溶液(2 mol/L)混匀,然后80 ℃水浴20 min,快速冷却。萃取净化:待上述溶液冷却,加入1 mL正己烷(含0.01% BHT),充分振荡萃取。离心,取1 μL正己烷层上机测定,使用仪器为安捷伦气相色谱质谱联用仪(Agilent7890-5975)。
色谱条件:所用色谱柱为安捷伦HP-5MS (60 m×0.25 mm×0.25μm)。进样口温度为280 ℃,进样量为1 μL,分流比为20:1,载气为氦气,恒线速度流速为1.5 mL/min;升温程序:初始柱温为120 ℃,保持1 min;以6 ℃/min的速率升到170 ℃;再以2 ℃/min的速率升到230 ℃,保持12 min;最后以10 ℃/min的速率升到285 ℃,保持10 min。
质谱条件:离子源温度为200 ℃,四级杆温度为150 ℃,传输线温度为260 ℃,电子轰击能量为70 ev,采用全扫描模式,扫描范围m/z 40–550,扫描速率为2.83次/s,溶剂切除时间为4.4 min。
通过检索NIST11谱图库并结合标准品图谱和各组分的保留时间来定性脂肪酸,再按照峰面积归一法计算其百分含量。
1.2.8 酿酒酵母温度胁迫处理将1.2.6中用诱导培养基振荡培养30 h后的菌液分别取出5 mL,调整pYES2-Bmelo424和pYES2菌液密度至OD600值相同,分别在4 ℃和40 ℃胁迫处理24 h,然后对处理后的菌液分别作10倍梯度稀释,最后各取2 μL接种在含2%葡萄糖的SC-Ura固体培养基上,30 ℃培养48 h,观察酿酒酵母受温度胁迫后的生长状况。
2 结果与分析 2.1 Bmelo424基因的克隆与分析以家蚕品种Dazao五龄三天幼虫整蚕的cDNA为模板,用Bmelo424基因的克隆引物进行PCR扩增,PCR产物的大小采用1%琼脂糖凝胶电泳检测,电泳结果显示有600 bp左右的特异性条带,和预期片段大小相同(图 1A)。对该PCR产物进行切胶回收,连接转化pMD19-T载体,筛选出阳性克隆进行测序,得到558 bp的开放阅读框(ORF)序列,编码185个氨基酸,具备高度保守的组氨酸簇HXXHH (图 1B),比对结果表明Bmelo424克隆序列和家蚕SilkDB数据库预测基因序列完全一致。
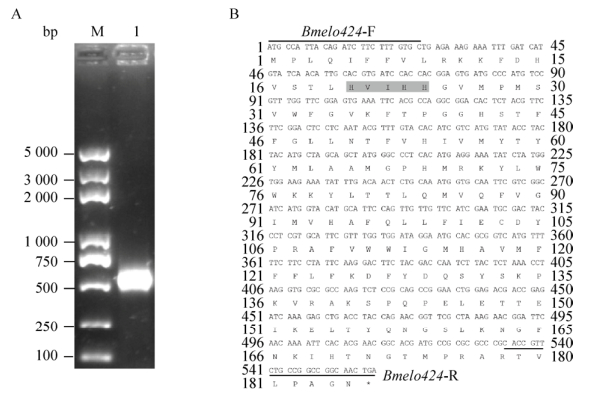
|
| 图 1 家蚕Bmelo424基因的克隆(A)及分析(B) Fig. 1 Cloning and analysis of Bmelo424 gene. (A) PCR amplification of the Bmelo424 gene. (B) The ORF sequence of Bmelo424 gene and its deduced amino acid, HXXHH motif was highlighted in grey. |
| |
通过TMHMM Server 2.0工具在线预测Bmelo424蛋白的跨膜区域,结果显示其有4个跨膜区,预测位置分别在13–35、45–67、79–101、105–124的氨基酸区域(图 2A)。利用NetPhos 3.1 Server预测Bmelo424的磷酸化位点有:6个丝氨酸(Serine)磷酸化位点(分别在第30、43、131、133、141和204位点)、8个苏氨酸(Threonine)磷酸化位点(第38、44、81、82、148、149、173和179位点)、4个酪氨酸(Tyrosine)磷酸化位点(第61、128、132和156位点) (图 2B)。通过PSIPRED v3.3工具在线分析其二级结构,其中,无规则卷曲所占比例最大,为53.3%,α-螺旋和β-折叠股分别占26.7%和20% (图 2C)。蛋白质亚细胞定位预测结果显示Bmelo424蛋白位于内质网中。

|
| 图 2 Bmelo424蛋白序列分析 Fig. 2 Protein sequence analysis of Bmelo424. (A) Transmembrane domain prediction of Bmelo424. (B) Phosphorylation sites prediction of Bmelo424. (C) Secondary structure prediction of Bmelo424. |
| |
采用荧光定量PCR的方法对Bmelo424基因在五龄三天家蚕幼虫组织的表达模式进行分析,结果显示,其在精巢、卵巢、头、体壁、脂肪体、中肠、血细胞、前中丝、后丝和马氏管中均有表达(图 3)。其中,在头和体壁中表达量较高,在精卵巢中表达量稍低,在血细胞中表达量最低。

|
| 图 3 Bmelo424基因的组织表达谱 Fig. 3 Expression profiles of Bmelo424 gene in different tissues. |
| |
用带有酶切位点的引物PCR扩增Bmelo424基因(表 1),通过TA克隆获得pMD-Bmelo424重组质粒,将该重组质粒与pYES2质粒用BamHⅠ和NotⅠ分别双酶切后进行连接和转化,筛选阳性质粒并进行双酶切鉴定(图 4),阳性质粒酶切后出现目的基因大小的片段,同时双酶切后的载体片段与空质粒pYES2的大小一致,而且阳性质粒测序结果也和克隆序列相同,说明成功构建pYES2-Bmelo424表达载体。

|
| 图 4 pYES2-Bmelo424重组质粒的双酶切鉴定 Fig. 4 Restriction enzyme digestion of the recombinant pYES2-Bmelo424. M: marker; 1: pYES2; 2: pYES2-Bmelo424; 3: pYES2 plasmid digested with BamHⅠand NotⅠ; 4: pYES2-Bmelo424 plasmid digested with BamHⅠand NotⅠ. |
| |
酿酒酵母INVSc1属于营养缺陷型菌株,只有在pYES2存在的情况下,才能在尿嘧啶缺陷型培养基(SC-Ura)上生长。提取构建好的pYES2-Bmelo424重组质粒和pYES2空质粒,转化酿酒酵母INVSc1感受态细胞,涂布培养后在SC-Ura培养基上长出单菌落。挑取单菌落进行菌液PCR反应,得到与目的基因大小相符的片段,进一步验证重组质粒成功转化至酿酒酵母INVSc1细胞中(图 5)。

|
| 图 5 酵母转化子的PCR鉴定 Fig. 5 Identification of yeast transformants by PCR. M: marker; 1: pYES2; 2: pYES2-Bmelo424. |
| |
将含有空质粒和重组质粒的酿酒酵母分别接种SC-Ura液体培养基,过夜培养后收集适量菌体,再把菌体重悬于诱导培养基中振荡培养30 h,离心收集菌体后冷冻干燥,并对培养物进行脂肪酸提取和甲酯化,利用GC-MS检测其脂肪酸组分。结果显示,携带空质粒pYES2的酿酒酵母细胞中主要含有棕榈酸(C16:0)、棕榈油酸(C16:1n-7)、硬脂酸(C18:0)和油酸(C18:1n-9) 4种脂肪酸。而携带pYES2-Bmelo424重组质粒的酵母细胞中,主要脂肪酸含量发生变化,除了棕榈油酸含量显著上升外,其余3种脂肪酸的含量都有所下降(图 6)。

|
| 图 6 pYES2酵母细胞和pYES2-Bmelo424重组酵母细胞的脂肪酸含量比较 Fig. 6 Comparison of fatty acids content between pYES2 and pYES2-Bmelo424 of yeast cells. *P < 0.05. |
| |
酿酒酵母在正常温度(30 ℃)培养时,携带pYES2-Bmelo424重组质粒和pYES2空质粒的细胞的生长速度和菌落形态没有明显差别。而不同温度胁迫处理后的两种酿酒酵母在细胞生长活性上表现出差异,其中,低温(4℃)处理后,携带pYES2- Bmelo424重组质粒的酿酒酵母细胞比携带pYES2空质粒的酿酒酵母细胞生长状况要好,但是高温(40℃)处理后,其生长状况却截然相反(图 7)。

|
| 图 7 两种酵母细胞在不同温度胁迫处理后的生长状况比较 Fig. 7 Comparison of the growth of two kinds of yeast cells after different temperature stress treatments. (A) 40 ℃. (B) 30 ℃ (Control). (C) 4 ℃. |
| |
ELOVL基因突变会引起诸如鱼鳞癣、黄斑退化、肌病、智力缺陷和髓鞘脱失等遗传性疾病[31],昆虫中ELOVL基因具有影响信息素合成、雄性生育力和Toll/Imd免疫信号传导调节等功能[19, 23-24]。家蚕是典型的鳞翅目经济昆虫,其ELOVL基因多达13个,但还没有功能鉴定报道[28]。本文以家蚕五龄三天全蚕cDNA为模板克隆获得Bmelo424基因558 bp的ORF序列,其推导氨基酸序列具有4个潜在的跨膜结构域,高度保守的组氨酸簇HXXHH,蛋白质亚细胞定位预测显示其位于内质网中,表明Bmelo424和其他延长酶一样属于膜结合蛋白,但其主要功能发挥区域同样局限于内质网中[32],另外,其具备发挥活性所必需的HXXHH基序,并且其活性位点和赖氨酸残基之间的距离决定超长链脂肪酸的碳链合成长度[33]。Bmelo424有6个丝氨酸磷酸化位点、8个苏氨酸磷酸化位点和4个酪氨酸磷酸化位点,这些磷酸化位点可能对其生物学功能行使有重要影响。蛋白质二级结构预测结果显示α-螺旋和β-折叠股占46.7%,二者常位于内部且不易变形,可稳定维持蛋白质的高级结构。Bmelo424基因在每个组织中均有表达,这与芯片数据基本一致[28]。在昆虫类超长链脂肪酸延长酶家族成员中,既有在各组织中广泛表达的成员[23, 26],也有组织特异性表达的成员[19],Bmelo424基因在家蚕各组织均有表达,而其表达水平的高低暗示该基因在不同组织中行使的功能可能有所差异。此外,鉴于昆虫表皮脂类在失水屏障、影响化学农药吸收、阻止病原入侵以及信息素组成等方面的作用[34],结合Bmelo424基因主要在头和体壁高表达的结果,我们推测其可能与家蚕的环境适应性相关。
Bmelo424基因的异源表达会引起酿酒酵母细胞的脂肪酸组分含量的变化,pYES2-Bmelo424重组酵母比pYES2酵母的C16:1n-7脂肪酸含量有显著提高,表明外源Bmelo424基因在酿酒酵母细胞中对催化生成C16:1n-7脂肪酸有积极作用。家蚕Bmelo424基因仅影响长链脂肪酸的合成,值得一提的是,ELOVL参与合成不同碳链长度脂肪酸的功能在其他物种中也有报道,诸如酵母中ELO1限于延伸C14脂肪酸到C16[2];锥虫中ELO1延伸C4脂肪酸到C10,ELO2延伸C10脂肪酸到C14,ELO3延伸C14脂肪酸到C18[35];小鼠中ELOVL6参与延伸C12–C16脂肪酸[14];果蝇中ELO68α可以合成C16和C18脂肪酸[19];黄粉虫中TmELO2可以合成C16脂肪酸[27]。这些研究结果都说明ELOVL除了主要负责超长链脂肪酸的延伸外,也参与其他碳链长度的脂肪酸延伸。同时,我们将昆虫中已涉及功能实验的ELOVL成员作了比较,发现Bmelo424与黄粉虫的TmELO2亲缘关系最近,且黄粉虫的TmELO2也仅影响C16脂肪酸合成[27],因此,家蚕Bmelo424与黄粉虫中已报道的同系物功能基本吻合。
在果蝇中,其耐冷性增强后会导致单烯酸比例增加而二烯酸比例降低,同时伴有C16脂肪酸增加而C18脂肪酸减少的现象[36],Bmelo424基因引起酵母C16:1n-7脂肪酸含量提高可以增加酵母细胞的冷胁迫后的适应能力,但对热胁迫后的适应能力却明显减弱,这也间接暗示Bmelo424基因可能影响家蚕的抗逆性。另外,白纹伊蚊中ELO表达水平的上调和滞育卵抗干燥能力的提高相关,也说明ELOVL家族的部分基因影响其抗逆性[24]。这也提示我们可以通过改变ELOVL抗逆相关基因的表达,以提高经济昆虫适应逆境的能力,或用类似方式降低害虫适应逆境的能力以达到控害目标。
| [1] | Guillou H, Zadravec D, Martin PGP, et al. The key roles of elongases and desaturases in mammalian fatty acid metabolism: Insights from transgenic mice. Prog Lipid Res, 2010, 49(2): 186–199. DOI: 10.1016/j.plipres.2009.12.002 |
| [2] | Toke DA, Martin CE. Isolation and characterization of a gene affecting fatty acid elongation in Saccharomyces cerevisiae. J Biol Chem, 1996, 271(31): 18413–18422. DOI: 10.1074/jbc.271.31.18413 |
| [3] | Oh CS, Toke DA, Mandala S, et al. ELO2 and ELO3, homologues of the Saccharomyces cerevisiae ELO1 gene, function in fatty acid elongation and are required for sphingolipid formation. J Biol Chem, 1997, 272(28): 17376–17384. DOI: 10.1074/jbc.272.28.17376 |
| [4] | Sassa T, Ohno Y, Suzuki S, et al. Impaired epidermal permeability barrier in mice lacking elovl1, the gene responsible for very-long-chain fatty acid production. Mol Cell Biol, 2013, 33(14): 2787–2796. DOI: 10.1128/MCB.00192-13 |
| [5] | Ohno Y, Suto S, Yamanaka M, et al. ELOVL1 production of C24 acyl-CoAs is linked to C24 sphingolipid synthesis. Proc Natl Acad Sci USA, 2010, 107(43): 18439–18444. DOI: 10.1073/pnas.1005572107 |
| [6] | Zadravec D, Tvrdik P, Guillou H, et al. ELOVL2 controls the level of n-6 28:5 and 30:5 fatty acids in testis, a prerequisite for male fertility and sperm maturation in mice. J Lipid Res, 2011, 52(2): 245–255. DOI: 10.1194/jlr.M011346 |
| [7] | Westerberg R, Tvrdik P, Undén AB, et al. Role for ELOVL3 and fatty acid chain length in development of hair and skin function. J Biol Chem, 2004, 279(7): 5621–5629. DOI: 10.1074/jbc.M310529200 |
| [8] | Zadravec D, Brolinson A, Fisher RM, et al. Ablation of the very-long-chain fatty acid elongase ELOVL3 in mice leads to constrained lipid storage and resistance to diet-induced obesity. FASEB J, 2010, 24(11): 4366–4377. DOI: 10.1096/fj.09-152298 |
| [9] | Agbaga MP, Brush RS, Mandal MNA, et al. Role of Stargardt-3 macular dystrophy protein (ELOVL4) in the biosynthesis of very long chain fatty acids. Proc Natl Acad Sci USA, 2008, 105(35): 12843–12848. DOI: 10.1073/pnas.0802607105 |
| [10] | Cadieux-Dion M, Turcotte-Gauthier M, Noreau A, et al. Expanding the clinical phenotype associated with ELOVL4 mutation: study of a large French-Canadian family with autosomal dominant spinocerebellar ataxia and erythrokeratodermia. JAMA Neurol, 2014, 71(4): 470–475. DOI: 10.1001/jamaneurol.2013.6337 |
| [11] | Leonard AE, Bobik EG, Dorado J, et al. Cloning of a human cDNA encoding a novel enzyme involved in the elongation of long-chain polyunsaturated fatty acids. Biochem J, 2000, 350(3): 765–770. DOI: 10.1042/bj3500765 |
| [12] | Mabuchi F, Sakurada Y, Kashiwagi K, et al. Association between SRBD1 and ELOVL5 gene polymorphisms and primary open-angle glaucoma. Invest Ophthalmol Vis Sci, 2011, 52(7): 4626–4629. DOI: 10.1167/iovs.11-7382 |
| [13] | di Gregorio E, Borroni B, Giorgio E, et al. ELOVL5 mutations cause spinocerebellar ataxia 38. Am J Hum Genet, 2014, 95(2): 209–217. DOI: 10.1016/j.ajhg.2014.07.001 |
| [14] | Moon YA, Shah NA, Mohapatra S, et al. Identification of a mammalian long chain fatty acyl elongase regulated by sterol regulatory element-binding proteins. J Biol Chem, 2001, 276(48): 45358–45366. DOI: 10.1074/jbc.M108413200 |
| [15] | Liu Y, Wang F, Yu XL, et al. Genetic analysis of the ELOVL6 gene polymorphism associated with type 2 diabetes mellitus. Braz J Med Biol Res, 2013, 46(7): 623–628. DOI: 10.1590/1414-431X20133103 |
| [16] | Tamura K, Makino A, Hullin-Matsuda F, et al. Novel lipogenic enzyme ELOVL7 is involved in prostate cancer growth through saturated long-chain fatty acid metabolism. Cancer Res, 2009, 69(20): 8133–8140. DOI: 10.1158/0008-5472.CAN-09-0775 |
| [17] | Naganuma T, Sato Y, Sassa T, et al. Biochemical characterization of the very long-chain fatty acid elongase ELOVL7. FEBS Lett, 2011, 585(20): 3337–3341. DOI: 10.1016/j.febslet.2011.09.024 |
| [18] | Purdy JG, Shenk T, Rabinowitz JD. Fatty acid elongase 7 catalyzes lipidome remodeling essential for human cytomegalovirus replication. Cell Rep, 2015, 10(8): 1375–1385. DOI: 10.1016/j.celrep.2015.02.003 |
| [19] | Chertemps T, Duportets L, Labeur C, et al. A new elongase selectively expressed in Drosophila male reproductive system. Biochem Biophys Res Commun, 2005, 333(4): 1066–1072. DOI: 10.1016/j.bbrc.2005.06.015 |
| [20] | Chertemps T, Duportets L, Labeur C, et al. A female-biased expressed elongase involved in long-chain hydrocarbon biosynthesis and courtship behavior in Drosophila melanogaster. Proc Natl Acad Sci USA, 2007, 104(11): 4273–4278. DOI: 10.1073/pnas.0608142104 |
| [21] | Jung A, Hollmann M, Schäfer MA. The fatty acid elongase NOA is necessary for viability and has a somatic role in Drosophila sperm development. J Cell Sci, 2007, 120(16): 2924–2934. DOI: 10.1242/jcs.006551 |
| [22] | Szafer-Glusman E, Giansanti MG, Nishihama R, et al. A role for very-long-chain fatty acids in furrow ingression during cytokinesis in Drosophila spermatocytes. Curr Biol, 2008, 18(18): 1426–1431. DOI: 10.1016/j.cub.2008.08.061 |
| [23] | Ng WC, Chin JS, Tan KJ, et al. The fatty acid elongase Bond is essential for Drosophila sex pheromone synthesis and male fertility. Nat Commun, 2015, 6: 8263. DOI: 10.1038/ncomms9263 |
| [24] | Urbanski JM, Benoit JB, Michaud MR, et al. The molecular physiology of increased egg desiccation resistance during diapause in the invasive mosquito, Aedes albopictus. Proc Biol Sci, 2010, 277(1694): 2683–2692. DOI: 10.1098/rspb.2010.0362 |
| [25] | Falcón T, Ferreira-Caliman MJ, Franco-Nunes FM, et al. Exoskeleton formation in Apis mellifera: cuticular hydrocarbons profiles and expression of desaturase and elongase genes during pupal and adult development. Insect Biochem Mol Biol, 2014, 50: 68–81. DOI: 10.1016/j.ibmb.2014.04.006 |
| [26] | Dong XL, Li QJ, Zhang HY. The noa gene is functionally linked to the activation of the Toll/Imd signaling pathways in Bactrocera dorsalis (Hendel). Dev Comp Immunol, 2016, 55: 233–240. DOI: 10.1016/j.dci.2015.09.009 |
| [27] | Zheng TX, Li HS, Han N, et al. Functional characterization of two elongases of very long-chain fatty acid from Tenebrio molitor L. (Coleoptera: Tenebrionidae). Sci Rep, 2017, 7(1): 10990. DOI: 10.1038/s41598-017-11134-y |
| [28] | Zuo WD, Li CL, Luan Y, et al. Genome-wide identification and analysis of elongase of very long chain fatty acid genes in the silkworm, Bombyx mori. Genome, 2018, 61(3): 167–176. DOI: 10.1139/gen-2017-0224 |
| [29] | Chou KC, Shen HB. A new method for predicting the subcellular localization of eukaryotic proteins with both single and multiple sites: Euk-mPLoc 2.0. PLoS ONE, 2010, 5(4): e9931. DOI: 10.1371/journal.pone.0009931 |
| [30] | Li MZ, Mai KS, He G, et al. Characterization of two Δ5 fatty acyl desaturases in abalone (Haliotis discus hannai Ino). Aquaculture, 2013, 416-417: 48–56. DOI: 10.1016/j.aquaculture.2013.08.030 |
| [31] | Sassa T, Kihara A. Metabolism of very long-chain Fatty acids: genes and pathophysiology. Biomol Ther (Seoul), 2014, 22(2): 83–92. DOI: 10.4062/biomolther.2014.017 |
| [32] | Jakobsson A, Westerberg R, Jacobsson A. Fatty acid elongases in mammals: their regulation and roles in metabolism. Prog Lipid Res, 2006, 45(3): 237–249. DOI: 10.1016/j.plipres.2006.01.004 |
| [33] | Denic V, Weissman JS. A molecular caliper mechanism for determining very long-chain fatty acid length. Cell, 2007, 130(4): 663–677. DOI: 10.1016/j.cell.2007.06.031 |
| [34] | Blomquist GJ, Nelson DR, de Renobales M. Chemistry, biochemistry, and physiology of insect cuticular lipids. Arch Insect Biochem Physiol, 1987, 6(4): 227–265. DOI: 10.1002/(ISSN)1520-6327 |
| [35] | Lee SH, Stephens JL, Paul KS, et al. Fatty acid synthesis by elongases in trypanosomes. Cell, 2006, 126(4): 691–699. DOI: 10.1016/j.cell.2006.06.045 |
| [36] | Ohtsu T, Kimura MT, Katagiri C. How Drosophila species acquire cold tolerance-qualitative changes of phospholipids. Eur J Biochem, 1998, 252(3): 608–611. DOI: 10.1046/j.1432-1327.1998.2520608.x |
 2019, Vol. 35
2019, Vol. 35




