中国科学院微生物研究所、中国微生物学会主办
文章信息
- 岳晓平, 陈朋, 朱玥明, 曾艳, 刘汉民, 刘红彦, 王敏, 孙媛霞
- Yue Xiaoping, Chen Peng, Zhu Yueming, Zeng Yan, Liu Hanmin, Liu Hongyan, Wang Min, Sun Yuanxia
- 米曲霉酸性蛋白酶基因在毕赤酵母中的异源表达及酶学性质
- Heterologous expression and characterization of Aspergillus oryzae acidic protease in Pichia pastoris
- 生物工程学报, 2019, 35(3): 415-424
- Chinese Journal of Biotechnology, 2019, 35(3): 415-424
- 10.13345/j.cjb.180321
-
文章历史
- Received: August 2, 2018
- Accepted: November 16, 2018
- Published: December 7, 2018
2. 中国科学院天津工业生物技术研究所,天津 300308;
3. 中恩 (天津) 医药科技有限公司,天津 300308
2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
3. Sinofn (Tianjin) Pharmacy Technology Co. Ltd., Tianjin 300308, China
酸性蛋白酶又称天冬氨酸蛋白酶,主要来源于动物脏器和微生物分泌物,是一种适合在酸性条件(pH 2.0–5.0)下水解蛋白质的酶类,其被广泛应用于食品[1-7]、畜牧[8-9]、医学[10]、皮革[11]以及水产加工[12]等领域。酸性蛋白酶主要通过微生物发酵获得。产酸性蛋白酶的菌种包括黑曲霉、红曲霉、宇佐美曲霉、米曲霉等。
米曲霉作为美国FDA公布的安全菌株,具有蛋白酶系丰富、酶活高、不产毒素等诸多优点,是我国传统酿造食品酱、酱油和酒类的主要生产菌种[13-14]。然而,米曲霉菌株普遍产中性蛋白酶和碱性蛋白酶的能力较强而产酸性蛋白酶的能力偏弱[15],目前对米曲霉来源酸性蛋白酶的研究主要集中于菌株的筛选与诱变、产酶条件优化、酶学特性研究等方面[16-19],有关米曲霉酸性蛋白酶基因异源表达与酶学性质研究尚未有报道。此外,米曲霉酸性蛋白酶主要通过固态发酵生产,存在发酵周期长、劳动强度大、质量不稳、纯化困难等缺点,限制了其规模化生产。
基于巴斯德毕赤酵母Pichia pastoris的蛋白表达系统具有杂蛋白分泌较少、培养成本低廉、发酵工艺成熟等优点[20-26],具有实现米曲霉酸性蛋白酶的工业化生产的潜力。本研究通过宏基因组测序,从发酵豆制品中获得米曲霉酸性蛋白酶的编码基因pepA,并在毕赤酵母中实现异源表达,同时对重组酸性蛋白酶的酶学特性展开研究,以期为米曲霉来源酸性蛋白酶的规模化生产奠定基础。
1 材料与方法 1.1 材料毕赤酵母GS115菌株、pPIC9K质粒均为本实验室保存;大肠杆菌Escherichia coli DH5α、TreliefTM SoSoo Cloning Kit购自北京擎科新业生物技术有限公司;Plasmid Mini KitⅠ、Cycel-pure Kit、Gel Extraction Kit购自美国OMEGA Bio-Tek公司;限制性内切酶BamHⅠ、限制性内切酶NotⅠ、dNTPs购自上海Fermentas公司;葡萄糖、蛋白胨、酵母粉、琼脂糖购自英国OXOID公司;甲醇购自美国Sigma公司;其余试剂均为国产分析纯。大肠杆菌培养基LB、毕赤酵母培养基YPD、选择培养基MD、诱导表达培养基BMGY和BMMY的培养基配方见Invitrodgen公司毕赤酵母操作手册。
1.2 方法 1.2.1 酸性蛋白酶基因的获取提取发酵豆制品微生物菌群宏基因组,利用二代测序技术进行测序。通过功能基因注释,挖掘获得酸性蛋白酶基因。
1.2.2 重组表达载体pPIC9K-pepA的构建以特异性引物AOX-F1和AOX-R1 (序列见表 1)扩增目的基因pepA,回收PCR产物。用限制性内切酶BamHⅠ和NotⅠ对表达质粒pPIC9K进行双酶切,纯化回收线性化质粒。在55 ℃条件下利用重组酶TreliefTM SoSoo Cloning Kit进行重组,将重组产物转化至E. coli DH5α感受态细胞中,筛选阳性克隆,提取质粒,用BamHⅠ和NotⅠ进行双酶切验证并测序。
| Primer name | Primer sequence (5'–3') |
| AOX1-F | TACTATTGCCAGCATTGCTGCT |
| AOX1-R | AGGATGTCAGAATGCCATTTGCC |
| AOX-F1 | GATCAAAAAACAACTAATTATTCGAAGGATCCATGGTTATCTTGAGCAAAGTCGCTG |
| AOX-R1 | CTAAGGCGAATTAATTCGCGGCCGCTTAAGCCTGGGCGGCGAAGCCGAGT |
将构建成功的重组表达质粒pPIC9K-pepA与毕赤酵母GS115感受态混合,混匀后,转入冰预冷的0.2 cm电转杯,冰上放置1 min后电击(1.5 kV、25 μF、200 Ω、6 ms)转化,电转结束后迅速加入1 mL 1 mol/L冰预冷的无菌山梨醇至杯中,于超净台内将所有溶液转至灭菌离心管中,30 ℃下静置培养1 h后,取300 μL涂布于MD平板,30 ℃下倒置培养3−4 d后,随机挑取若干单菌落,用YPD液体培养基振荡培养24 h,提取染色体DNA,以表达载体pPIC9K的通用引物AOX-F、AOX-R进行PCR鉴定,鉴定成功的菌株通过酶活力测定进一步筛选。
1.2.4 重组菌株的诱导表达毕赤酵母含有醇氧化酶基因启动子(PAOX1),在甲醇的诱导下,PAOX1可驱动外源基因大量表达[27]。将筛选成功的菌株接种于含5 mL YPD培养基的试管中,30 ℃、200 r/min振荡培养1 d,按2%体积将种子液接种于含50 mL BMGY培养基的250 mL三角瓶中,30 ℃、200 r/min振荡培养1 d,离心收集菌体,然后全部转入含50 mL的BMMY培养基的250 mL三角瓶中,30 ℃、200 r/min振荡培养,每隔24 h添加甲醇至终浓度为0.5%,每隔24 h取样检测菌液浓度及发酵上清液酸性蛋白酶活力,实验重复3次,上清液浓缩后进行SDS-PAGE分析[27]。
1.2.5 重组蛋白的酶活力测定酶活力测定参考GB 1886.174-2016食品安全国家标准,以1%的酪蛋白为底物,采用Folin-酚法进行酶活测定。以1 mL液体酶在40 ℃和pH 3.0条件下,每分钟水解酪蛋白产生1 μg酪氨酸为1个酶活力单位,用U/mL表示。
1.3 酶学性质分析 1.3.1 最适反应温度与温度稳定性按1.2.5的方法,在30–60 ℃,每间隔10 ℃取样测定酶活,每个反应设3个平行,以最高酶活为100%,计算其他不同温度下的相对酶活,确定酶的最适反应温度。
将酶液分别置于30 ℃、35 ℃、40 ℃、45 ℃、50 ℃、55 ℃、60 ℃、65 ℃、70 ℃水浴锅中,保温15 min后取出酶液,测定残余酶活力,每个反应设3个平行,以所测最高酶活为100%,计算各个温度条件下的相对酶活,考察不同温度下的热稳定性。
1.3.2 最适反应pH与pH稳定性在pH 2.0−7.0下测定酶活,以最高酶活力为100%,计算其他不同pH下的相对酶活,每个反应设3个平行,探讨不同pH值对酶活力的影响。
将酶液置于pH 1.0、2.0、3.0、3.5、4.0、5.0、6.0、7.0的反应体系中,2 h后取出酶液,测定残余酶活力,每个反应设3个平行,以所测最高酶活为100%,计算各个pH条件下的相对酶活,确定酶的pH值稳定性。
1.3.3 金属离子对酶活力的影响为分析金属离子对该酸性蛋白酶的影响,将不同的金属离子(Fe3+、Al3+、Mn2+、Cu2+、Mg2+、Fe2+、Co2+、Ca2+、Na+、K+、Ag+)分别添加到酸性蛋白酶酶反应体系中,使金属离子终浓度为5 mmol/L,以不加金属离子的样品酶活为100%,每个反应设3个平行,计算不同金属离子存在下的相对酶活,考察金属离子对酶活力的影响。
2 结果与分析 2.1 米曲霉酸性蛋白酶基因的克隆与重组载体的构建利用二代测序技术对发酵豆制品进行宏基因组测序,通过功能基因注释,挖掘得到一种酸性蛋白酶基因pepA,其长度为1 215 bp,共编码287个氨基酸。利用MEGA4.6软件对不同微生物来源的酸性蛋白酶氨基酸序列构建进化树(图 1),并进行同源性分析,发现该酸性蛋白酶与来自米曲霉RIB40的酸性蛋白酶(GenBank基因登录号: XM_001824123.3)的序列完全一致[28],与来自烟曲霉af293的酸性蛋白酶相似性为70.69%[29],与来自黑曲霉CBS 513.88的酸性蛋白酶相似性为66.58%[30]。

|
| 图 1 米曲霉酸性蛋白酶PepA与其他微生物来源酸性蛋白酶的氨基酸序列进化树 Fig. 1 The phylogenetic tree based on the protein sequences of PepA from Aspergillums oryzae with other microbial acidic protease. |
| |
以提取的宏基因组为模板,经引物AOX-F1和AOX-R1 PCR扩增,回收PCR产物,经PCR扩增,得到1 200 bp左右的片段(图 2)。在55 ℃条件下进行重组,将重组产物转化至E. coli DH5α感受态细胞中,获得重组菌,提取质粒,经BamHⅠ和NotⅠ双酶切后,如图 3所示,得到两条大小分别为9 000 bp和1 200 bp的两条带,表明目的基因已经插入到pPIC9K载体上。测序结果表明该转化菌的质粒中确实含有目的基因片段,且阅读框正确,表达质粒pPIC9K-pepA构建成功。

|
| 图 2 pepA基因的PCR扩增电泳图 Fig. 2 Agarose gel electrophoresis of PCR amplified. M: DNA marker; 1: PCR products with pepA. |
| |

|
| 图 3 毕赤酵母表达质粒酶切鉴定 Fig. 3 Identification of expression plasmids for P. pastoris by digestion. M: DNA marker; 1: product of double enzyme digestion. |
| |
利用电转化的方式将表达质粒pPIC9K-pepA转入毕赤酵母GS115,利用组氨酸缺陷型MD培养基筛选阳性转化子,提取重组酵母菌基因组DNA,以其为模板,利用毕赤酵母表达载体通用引物AOX-F、AOX-R和目的基因特异性引物AOX1-F、AOX1-R进行PCR鉴定[31]。从图 4可以看出,使用表达载体通用引物,阳性重组菌扩增出1 600 bp左右的片段,使用基因特异性引物,扩增出1 200 bp左右片段,条带大小与理论值相符,证明pPIC9K-pepA与毕赤酵母GS115染色体基因组重组成功。

|
| 图 4 转化子的PCR鉴定结果 Fig. 4 Identification of transformant by PCR. M: DNA marker; 1: PCR products with universal primer; 2: PCR products with specific primer. |
| |
将阳性菌株接种于以甘油为唯一碳源的培养基中,30 ℃培养1 d后,收集菌体进行甲醇诱导培养,每隔24 h取样,进行菌体浓度和酶活的测定(如图 5所示),随着发酵的不断进行,重组菌株浓度也逐渐增加,当发酵4 d后,菌体浓度达到最大,OD600达到45,此时酶活也达到最高,大约为50.62 U/mL。
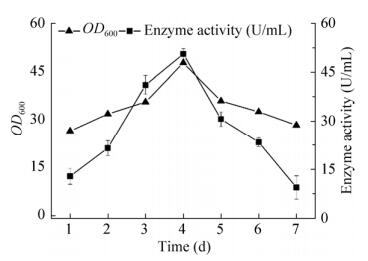
|
| 图 5 重组毕赤酵母菌株产酶时间曲线 Fig. 5 The time course of the enzyme activities produced by the engineered P. pastoris strain. |
| |
酸性蛋白酶经诱导表达后,取上清液进行离心、超滤浓缩,取浓缩后的上清蛋白进行SDS-PAGE分析,结果如图 6所示,分子量50 kDa处有明显的条带,无明显杂蛋白,可以开展后续酶学性质方面的研究。
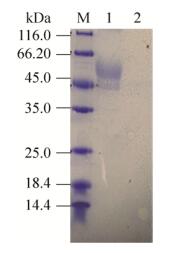
|
| 图 6 浓缩后酶的SDS-PAGE分析 Fig. 6 SDS-PAGE analysis of the concentrated enzyme. M: protein marker; 1: concentrated enzyme; 2: control. |
| |
如图 7A所示,该酶最适反应温度为50 ℃。当低于50 ℃时,随着反应温度的升高,酶活力逐渐增加。在50 ℃下,酶活力最高,为30 ℃下的2.5倍,但高于50 ℃时,酶活力随着反应温度的升高迅速下降。温度为60 ℃时,该酶基本失活。由图 7B可知,该酶在40 ℃以下放置15 min后仍然具有较高的活性。

|
| 图 7 温度对重组酸性蛋白酶PepA活性(A)和稳定性(B)的影响 Fig. 7 Effect of temperature on the activity (A) and stability (B) of recombinant PepA. |
| |
该酸性蛋白酶的最适pH为4.5 (图 8A)。当pH < 4.5时(2.0−4.5),随着pH值的增加,酸性蛋白酶的酶活力逐渐增大。酸性蛋白酶在pH 4.5下的酶活力是pH 2.0下酶活力的6倍。但当pH > 4.5时,随着pH值的增加,酸性蛋白酶的相对酶活力迅速降低。图 8B为酸性蛋白酶在pH 1.0–7.0下的稳定性。由图可知,该酸性蛋白酶在pH 3.0–4.0下放置120 min后,相对酶活仍可达70%以上。
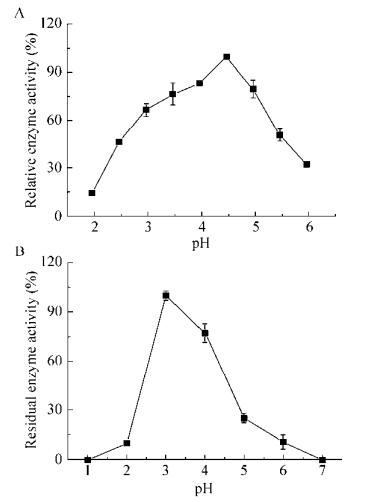
|
| 图 8 pH对重组酸性蛋白酶PepA的酶活(A)和稳定性(B)的影响 Fig. 8 Effects of pH on the activity (A) and stability (B) of recombinant PepA. |
| |
金属离子可以影响酶结构的正确折叠,进而影响酶活力。为分析金属离子对酸性蛋白酶酶活力的影响,以不添加金属离子的反应体系为对照,测定不同金属离子(Fe3+、Al3+、Mg2+、Fe2+、Co2+、Ca2+、Na+、K+和Ag+)对酶活力的影响。如图 9所示,该酸性蛋白酶可显著被Mn2+和Cu2+激活。添加5 mmol/L的Mn2+和Cu2+时,该酸性蛋白酶的酶活力分别为对照的2.6倍和1.3倍。而Fe3+、Fe2+与Ca2+对酸性蛋白酶具有显著的抑制作用。由此可见,不同的金属离子对酸性蛋白酶酶活力具有不同的激活或抑制作用。

|
| 图 9 金属离子对酸性蛋白酶活性的影响 Fig. 9 Effect of metal ions on the activity of acid protease. |
| |
酸性蛋白酶在制革工业、食品行业、畜牧业、医药等行业应用广泛,然而却受到酶不易获取、分离纯化困难、酶学性质不稳定等因素限制。由于毕赤酵母表达系统菌株稳定,发酵水平高,分泌能力强,自身蛋白分泌少[32],目前已有黑曲霉、宇佐美曲霉、牛胃、猪胃等来源的酸性蛋白酶实现了在毕赤酵母中的异源表达,却少有米曲霉酸性蛋白酶的相关报道。
宏基因组技术将环境中全部微生物的遗传信息看作一个整体,自上而下地研究微生物与自然环境或生物体之间的关系,可以从微生物的天然环境中直接提取基因组遗传物质和寻找新基因,大大地拓展了微生物学的研究思路与方法[33]。本研究利用宏基因组测序及注释技术从发酵豆制品中获得了编码来源于米曲霉的酸性蛋白酶基因pepA,尽管与来源于米曲霉RIB40的酸性蛋白酶基因序列一致,然而,到目前为止尚未见到由这一基因序列表达的酸性蛋白酶的酶学性质报道。此外,调研也没有获得其他米曲霉来源、可与pepA进行同源序列对比的酸性蛋白酶基因序列。
本研究基于分泌型表达载体pPIC9K携带的外源基因表达可抑制毕赤酵母自身蛋白表达的特点[34],利用pPIC9K载体,首次在毕赤酵母GS115中成功表达了米曲霉来源的酸性蛋白酶基因pepA。发酵所得上清液中蛋白组分单一,重组酸性蛋白酶活力约为50.62 U/mL。王鑫等[35]将来自黑曲霉CBS 513.88、与PepA相似性为66.58%的酸性蛋白酶在毕赤酵母中进行表达,测得重组黑曲霉酸性蛋白酶的酶活为8.76 U/mL。邱重晏[36]研究了粗糙脉孢霉酸性蛋白酶基因在毕赤酵母S115中的表达,测得重组粗糙脉孢霉酸性蛋白酶最高酶活为6.8 U/mL。与上述研究相比,本研究的重组酸性蛋白酶的酶活较高。而且,随着后续发酵条件优化以及密码子优化等分子生物学实验的开展,PepA的酶活力可进一步提高。
Tsujita和End [37]发现来自米曲霉365-U-64-1的酸性蛋白酶具有底物选择特异性。在以牛血清蛋白为底物时,最适pH值为4.2,在pH 5.0、50 ℃下加热10 min,仍可保持70%以上的酶活。Vishwanatha等[38]发现米曲霉MTCC 5341分泌的酸性蛋白酶最适温度和pH值分别为55 ℃和3.2,在40−57 ℃和pH 2.5−6.0下可保持一定的稳定性。Yin等[39]发现来自米曲霉BCRC 30118的酸性蛋白酶最适温度和pH值分别为60 ℃和3.0,Fe2+、Fe3+与Hg2+会抑制酶活。王鑫等[35]则发现由黑曲霉CBS 513.88分泌的酸性蛋白酶最适温度和pH分别为50 ℃和3.0,Fe2+、Ca2+和Mn2+可促进酶活,而Cu2+和Fe3+则可抑制酶活。本研究中的重组米曲酸性蛋白酶PepA的酶学性质与上述报道的曲霉酸性蛋白酶存在一定的差异,其最佳温度为50 ℃,40 ℃下较为稳定,最适pH为4.5,在pH 3.0−4.0酶活力较为稳定,金属离子Mn2+和Cu2+是该酶的激活剂,而Fe2+、Fe3+和Ca2+对酶活抑制程度较大。文献表明毕赤酵母表达蛋白的糖基化会影响其分泌及功能[40]。当糖基化修饰发生在表达蛋白的折叠有序区域时,会降低其稳定性[41]。如马清等[42]推断N-糖基化修饰导致毕赤酵母异源表达的β-甘露聚糖酶其局部结构改变,使之变为相对不耐热的结构,导致其热稳定性下降。王越等[43]认为毕赤酵母对蛋白的过度化糖基修饰,会造成产物的活性位点遮蔽。因此,虽然缺少天然野生型PepA的酶学性质信息,但考虑到PepA潜在多个N-糖基化修饰位点,推测PepA的酶学性质差异可能与其在毕赤酵母表达过程中的多肽链的错误折叠及蛋白糖基化作用有关[44]。
本研究实现了米曲霉酸性蛋白酶在毕赤酵母中的异源表达并对其酶学性质进行了表征,为酸性蛋白酶的异源表达研究及应用推广提供了借鉴参考,同时为食品、医药和皮革等工业提供了新型的米曲酸性蛋白酶来源。
| [1] |
Wei TY, Wang Z, Wei RF. Adding acid protease on the stability of Chinese rice wine fermentation mash.
Ferment Indust, 2015, 41(12): 148–152.
(in Chinese). 魏桃英, 汪钊, 魏瑞锋. 发酵醪中添加酸性蛋白酶对黄酒稳定性的影响. 食品与发酵工业, 2015, 41(12): 148-152. |
| [2] | Lagace LS, Bisson LF. Survey of yeast acid proteases for effectiveness of wine haze reduction. Am J Enol Viticult, 1990, 41(2): 147–155. |
| [3] |
Yu PB, Chen LL, Zhang B, et al. Study on improving the quality of rice wine Wheat Qu by making with two strains.
Ferment Indust, 2012, 38(9): 1–6.
(in Chinese). 余培斌, 陈亮亮, 张波, 等. 双菌种制曲改善黄酒麦曲品质的研究. 食品与发酵工业, 2012, 38(9): 1-6. |
| [4] |
Zhao H, Zhao SX, Zhang W, et al. The study on the application of acid proteinase to ethanol fermentation.
Ferment Indust, 1997, 23(2): 26–28.
(in Chinese). 赵华, 赵树欣, 张维, 等. 酒精发酵中应用酸性蛋白酶的研究. 食品与发酵工业, 1997, 23(2): 26-28. |
| [5] |
Xiao DG, Zhao H, Zhai JX, et al. Studies on application technology of acid protease in alcohol production Ⅲ. The application experiment in alcohol production.
Liquor-Making Sci Technol, 2000(4): 34–35, 37.
(in Chinese). 肖冬光, 赵华, 翟建新, 等. 酒用酸性蛋白酶在酒精生产中应用技术的研究Ⅲ.酒精生产应用试验. 酿酒科技, 2000(4): 34-35, 37. DOI:10.3969/j.issn.1001-9286.2000.04.008 |
| [6] | Arai S, Makoto A, Yamashita M, et al. Applying proteolytic enzymes on soybean. Agr Biol Chem, 1971, 35(4): 552–559. |
| [7] | Xu YS, Dai MJ, Zang JH, et al. Purification and characterization of an extracellular acidic protease of Pediococcus pentosaceus isolated from fermented fish. Food Sci Technol Res, 2015, 21(5): 739–744. DOI: 10.3136/fstr.21.739 |
| [8] |
Lu C, Bian LQ, Liu XJ, et al. Effects of neutral and acid protease on growth performance in weaned piglets.
Chin J Animal Nut, 2009, 21(6): 993–997.
(in Chinese). 卢晨, 边连全, 刘显军, 等. 中性和酸性蛋白酶对断奶仔猪生长性能的影响. 动物营养学报, 2009, 21(6): 993-997. DOI:10.3969/j.issn.1006-267x.2009.06.029 |
| [9] |
Wu YL, Huo SY. Research on the determination of acidic protease activity in feed premixed with enzyme additive.
J Agric Sci Technol, 2009, 11(1): 52–55.
(in Chinese). 武英利, 霍韶瑜. 饲用酶添加剂预混料中酸性蛋白酶活力测定方法研究. 中国农业科技导报, 2009, 11(1): 52-55. |
| [10] |
Chen Q. Anti-edema drug-swelling elimination.
Shanxi Medical J, 1985, 24(2): 112–113.
(in Chinese). 陈铨. 抗水肿新药——肿消净. 山西医药杂志, 1985, 24(2): 112-113. |
| [11] |
Li SP, Jiang QL, Bi CY. Studies and developments on the acid protease for leather processing.
Shandong Sci, 1996, 9(4): 62–65.
(in Chinese). 李树品, 蒋千里, 毕春元. 皮革用酸性蛋白酶的研究与开发. 山东科学, 1996, 9(4): 62-65. |
| [12] |
Lin SZ, Liu WC, Shan YZ. Application of enzyme mixtures in aquaculture feed.
Fisher Sci, 2009, 28(12): 798–800.
(in Chinese). 林少珍, 刘伟成, 单乐州. 酶制剂在水产养殖饲料中的应用. 水产科学, 2009, 28(12): 798-800. DOI:10.3969/j.issn.1003-1111.2009.12.022 |
| [13] |
Xu DF. Breeding of novel fusant with higher activity of acid protease from aspergillus and its preliminary application in soy sauce fermentation[D]. Guangzhou: South China University of Technology, 2010 (in Chinese). 徐德峰.高活力酸性蛋白酶曲霉融合子的选育及其在酱油发酵中的初步应用[D].广州: 华南理工大学, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10561-2010228721.htm |
| [14] |
Deng J, Xu J, Wu HC, et al. Screening of acid protease-overproducing stranins of Aspergillus oryzae.
China Cond, 2010, 35(1): 53–55, 59.
(in Chinese). 邓静, 徐静, 吴华昌, 等. 米曲霉高产酸性蛋白酶菌株的选育. 中国调味品, 2010, 35(1): 53-55, 59. DOI:10.3969/j.issn.1000-9973.2010.01.010 |
| [15] |
Cao XH, Zhang B, Lu MF, et al. A rapid method for screening excellent soy sauce producing Aspergillums oryza mutagenized by ion implanting.
China Cond, 2007, 5(5): 26–30.
(in Chinese). 曹小红, 张斌, 鲁梅芳, 等. 离子注入诱变米曲霉及酱油优良生产菌株的快速筛选. 中国调味品, 2007, 5(5): 26-30. DOI:10.3969/j.issn.1000-9973.2007.05.004 |
| [16] | de Castro RJS, Sato HH. Advantages of an acid protease from Aspergillus oryzae over commercial preparations for production of whey protein hydrolysates with antioxidant activities. Biocatal Agric Biotechnol, 2014, 3(3): 58–65. DOI: 10.1016/j.bcab.2013.11.012 |
| [17] | Srinubabu G, Lokeswari N, Jayaraju K. Screening of nutritional parameters for the production of protease from Aspergillus oryzae. E-J Chem, 2007, 4(2): 208–215. DOI: 10.1155/2007/915432 |
| [18] |
Guo JP, Ma G. Optimization of solid-state fermentation medium for acid protease of Aspergillus oryzae.
China Brewing, 2009(8): 125–128.
(in Chinese). 郭继平, 马光. 响应面法优化米曲霉酸性蛋白酶的固态发酵培养基. 中国酿造, 2009(8): 125-128. DOI:10.3969/j.issn.0254-5071.2009.08.037 |
| [19] | Lee SK, Wang JY, Choi SH, et al. Purification and characterization of Aspergillus oryzae LK-101 salt-tolerant acid protease isolated from soybean paste. Food Sci Biotechnol, 2010, 19(2): 327–334. |
| [20] |
Zhu TC, Li Y. Recent development of Pichia pastoris system: current status and future perspective.
Chin J Biotech, 2015, 31(6): 929–938.
(in Chinese). 朱泰承, 李寅. 毕赤酵母表达系统发展概况及趋势. 生物工程学报, 2015, 31(6): 929-938. |
| [21] | Ahmad M, Hirz M, Pichler H, et al. Protein expression in Pichia pastoris: recent achievements and perspectives for heterologous protein production. Appl Microbiol Biotechnol, 2014, 98(12): 5301–5317. DOI: 10.1007/s00253-014-5732-5 |
| [22] | Damasceno LM, Chung JH, Batt CA. Protein secretion in Pichia pastoris and advances in protein production. Appl Microbiol Biotechnol, 2012, 93(1): 31–39. |
| [23] | Cos O, Ramón R, Montesinos JL, et al. Operational strategies, monitoring and control of heterologous protein production in the methylotrophic yeast Pichia pastoris under different promoters: a review. Microb Cell Fact, 2006, 5: 17. DOI: 10.1186/1475-2859-5-17 |
| [24] | Daly R, Hearn MTW. Expression of heterologous proteins in Pichia pastoris: a useful experimental tool in protein engineering and production. J Mol Recognit, 2005, 18(2): 119–138. DOI: 10.1002/(ISSN)1099-1352 |
| [25] |
Xiong AS, Peng RH, Li X, et al. Influence of signal peptide sequences on the expression of heterogeneous proteins in Pichia pastoris.
Acta Bioch et Bioph Sin, 2003, 35(2): 154–160.
(in Chinese). 熊爱生, 彭日荷, 李贤, 等. 信号肽序列对毕赤酵母表达外源蛋白质的影响. 生物化学与生物物理学报, 2003, 35(2): 154-160. |
| [26] | Zhan HJ, Liu BS, Reid SW, et al. Engineering a soluble extracellular erythropoietin receptor (EPObp) in Pichia pastoris to eliminate microheterogeneity, and its complex with erythropoietin. Protein Eng, 1999, 12(6): 505–513. DOI: 10.1093/protein/12.6.505 |
| [27] |
Qin HB, Li MF, Yang HJ. Heteroespression of cellobiohydrolase A gene of Aspergillus niger in Pachia pastoris.
Biotechnology, 2017, 27(4): 330–336.
(in Chinese). 秦慧彬, 李梦菲, 杨洪江. 黑曲霉纤维二糖水解酶基因cbhA在毕赤酵母中异源表达. 生物技术, 2017, 27(4): 330-336. DOI:10.3969/j.issn.1673-713X.2017.04.007 |
| [28] | Gomi K, Arikawa K, Kamiya N, et al. Cloning and nucleotide sequence of the acid protease-encoding gene (pepA) from Aspergillus oryzae. Biosci Biotechnol Biochem, 1993, 57(7): 1095–1100. DOI: 10.1271/bbb.57.1095 |
| [29] | Nierman WC, Pain A, Anderson MJ, et al. Genomic sequence of the pathogenic and allergenic filamentous fungus Aspergillus fumigatus. Nature, 2005, 438(7071): 1151–1156. DOI: 10.1038/nature04332 |
| [30] |
Yang H. Expression and characterization of acid protease from Aspergillus usamil in Pichia pastoris[D]. Wuhan: Hubei University, 2014 (in Chinese). 杨琥.宇佐美曲霉酸性蛋白酶在毕赤酵母中的表达及其酶学性质的研究[D].武汉: 湖北大学, 2014. 宇佐美曲霉酸性蛋白酶在毕赤酵母中的表达及其酶学性质的研究 |
| [31] |
Zhang L, Jiang Y, Zhang J, et al. Recombinant expression of bovine chymosin in Pichia pastoris.
Chin J Biotech, 2009, 25(8): 1160–1165.
(in Chinese). 张莉, 姜媛媛, 张健, 等. 牛凝乳酶基因在毕赤酵母中的重组表. 生物工程学报, 2009, 25(8): 1160-1165. DOI:10.3321/j.issn:1000-3061.2009.08.006 |
| [32] | Cregg JM, Barringer KJ, Hessler AY, et al. Pichia pastoris as a host system for transformations. Mol Cell Biol, 1985, 5(12): 3376–3385. DOI: 10.1128/MCB.5.12.3376 |
| [33] | Handelsman J, Rondon MR, Brady SF, et al. Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products. Chem Biol, 1998, 5(10). |
| [34] | Huang P, Shi JL, Sun QW, et al. Engineering Pichia pastoris for efficient production of a novel bifunctional Strongylocentrotus purpuratus invertebrate-type lysozyme. Appl Biochem Biotechnol, 2018, 186(2): 459–475. DOI: 10.1007/s12010-018-2753-z |
| [35] |
Wang X, Jin P, Song P, et al. Cloning, expression and biochemical characterization of a novel acid protease EXPA from Aspergillus niger[EB/OL]. Ferment Indust, 2018. https://doi.org/10.13995/j.cnki.11-1802/ts.018387 (in Chinese). 王鑫, 金鹏, 宋鹏, 等.黑曲霉酸性蛋白酶EXPA的克隆表达与酶学性质解析.食品与发酵工业, 2018. doi: 10.13995/j.cnki.11-1802/ts.018387. |
| [36] |
Qiu CY, Wang ZX. Cloning and identification of a gene encoding an acidic protease from Neurospora crassa.
Microbiol China, 2006, 33(4): 91–94.
(in Chinese). 邱重晏, 王正祥. 粗糙脉孢霉酸性蛋白酶基因的克隆与鉴定. 微生物学通报, 2006, 33(4): 91-94. DOI:10.3969/j.issn.0253-2654.2006.04.018 |
| [37] | Tsujita Y, Endo A. Presence and partial characterization of internal acid protease of Aspergillus oryzae. Appl Environ Microbiol, 1978, 36(2): 237–242. |
| [38] | Vishwanatha KS, Rao AGA, Singh SA. Characterisation of acid protease expressed from Aspergillus oryzae MTCC 5341. Food Chem, 2009, 114(2): 402–407. DOI: 10.1016/j.foodchem.2008.09.070 |
| [39] | Yin LJ, Chou YH, Jiang ST. Purification and characterization of acidic protease from Aspergillus oryzae BCRC 30118. J Mar Sci Technol, 2013, 21(9): 105–110. |
| [40] |
Gu Y, Zhu XP, Wang SH. Glycosylation of proteins expressed in Pichia pastoris.
Chem Life, 2004, 24(4): 353–355.
(in Chinese). 顾园, 诸欣平, 王少华. 毕赤酵母表达蛋白质的糖基化. 生命的化学, 2004, 24(4): 353-355. DOI:10.3969/j.issn.1000-1336.2004.04.029 |
| [41] | Shental-Bechor D, Levy Y. Folding of glycoproteins: toward understanding the biophysics of the glycosylation code. Curr Opin Struc Biol, 2009, 19(5): 524–533. DOI: 10.1016/j.sbi.2009.07.002 |
| [42] |
Ma Q, Cai R, Jiang FC, et al. Effect of N-glycosylation on the heterologous expression of β-mannanase in Pichia pastoris.
Food Sci, 2017, 38(16): 86–91.
(in Chinese). 马清, 蔡瑞, 姜风超, 等. N-糖基化对β-甘露聚糖酶在毕赤酵母中异源表达的影响. 食品科学, 2017, 38(16): 86-91. DOI:10.7506/spkx1002-6630-201716013 |
| [43] |
Wang Y, Gong X, Chang SH, et al. A Pichia pastoris with α-1, 6-mannosyltransferases deletion and its use in the expression of HSA/GM-CSF chimera.
Chin J Biotech, 2007, 23(5): 907–914.
(in Chinese). 王越, 巩新, 唱韶红, 等. α-1, 6-甘露糖转移酶基因敲除的毕赤酵母菌株构建及其用于融合蛋白HSA/GM-CSF表达的研究. 生物工程学报, 2007, 23(5): 907-914. DOI:10.3321/j.issn:1000-3061.2007.05.026 |
| [44] | Wildt S, Gerngross TU. The humanization of N-glycosylation pathways in yeast. Nat Rev Microbiol, 2005, 3(2): 119–128. DOI: 10.1038/nrmicro1087 |
 2019, Vol. 35
2019, Vol. 35




