中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李阳丽, 杨小雨, 邓子新, 朱冬青
- Li Yangli, Yang Xiaoyu, Deng Zixin, Zhu Dongqing
- 微生物次生代谢产物生物合成中的拜耳-维立格单加氧酶
- Baeyer-Villiger monooxygenases in the biosynthesis of microbial secondary metabolites
- 生物工程学报, 2019, 35(3): 351-362
- Chinese Journal of Biotechnology, 2019, 35(3): 351-362
- 10.13345/j.cjb.180294
-
文章历史
- Received: July 14, 2018
- Accepted: September 11, 2018
- Published: February 27, 2019
1899年,德国化学家阿道夫·冯·拜耳和他的学生维克多·维立格报道了用卡洛酸作为氧化剂,催化环酮生成相应内酯的反应,即有机化学中著名的拜耳-维立格(Baeyer-Villiger,BV)氧化反应。反应机理如图 1所示,过氧酸亲核进攻羰基碳形成Criegee中间体,连接处两个碳中的一个迁移到氧上形成酯,同时释放一分子羧酸[1]。用过酸催化BV反应虽然成本相对较低,但在底物选择性、反应的区域选择性和立体选择性等方面并不能满足有机合成的需求,同时过酸的使用会引起生产过程中的安全隐患、废弃物处理以及环境污染等诸多问题。这些有机催化BV反应的缺点在下文将要提到的生物催化BV反应中是不存在的。
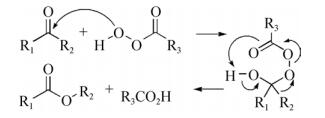
|
| 图 1 拜耳-维立格氧化反应 Fig. 1 Baeyer-Villiger oxidations. |
| |
1948年就已经有报道微生物可以催化BV反应[1]。1976年催化BV反应的拜耳-维立格单加氧酶(BVMO)第一次被分离纯化[2-3]。1988年第一个编码BVMO的基因从不动杆菌中被克隆[4],随后参与碳源分解代谢的环戊酮单加氧酶(CPMO)、环己酮单加氧酶(CHMO)、苯基丙酮单加氧酶(PAMO)等BVMO对应的基因陆续从各种微生物中被克隆并深入研究。目前所发现的BVMO都属于黄素依赖的单加氧酶,可以分为Ⅰ型、Ⅱ型和O型三类。Ⅰ型BVMO以FAD和NADPH为辅助因子,蛋白序列上具有一个保守的motif (FXGXXXHXXXWP),大部分BVMO属于这一类型;Ⅱ型BVMO以FMN和NAD(P)H为辅助因子,没有Ⅰ型BVMO所具有的保守motif,此类型相对较少;O型BVMO虽然需要FAD和NAD(P)H作为辅助因子,但是蛋白序列上不具备Ⅰ型BVMO的保守motif。
与过酸催化BV反应相比,BVMO在催化BV反应中表现出了高度的区域选择性和立体选择性,同时反应条件非常温和。分解代谢中的BVMO具有较广的底物选择范围,在有机合成领域具有更大的应用潜力,因此被深入研究和改造。已有不少文献对BVMO的研究进展进行了系统的整理,同样偏重于分解代谢中BVMO的相关工作[5-9]。本文仅仅针对微生物次生代谢产物生物合成中的BVMO研究进展进行概述。
1 微生物次生代谢产物生物合成途径中的BV反应依据蛋白一级结构所具有的保守结构域,将微生物次生代谢产物生物合成代谢途径中的BVMO分为以下4类:第1类具有CzcO结构域(NCBI的保守结构域数据库记录号COG2072,下同),即Ⅰ型BVMO;第2类具有SsuD结构域(COG2141),即Ⅱ型BVMO;第3类具有UbiH结构域(COG0654),即O型BVMO;第4类为具有ABM结构域(Pfam: PF03992)的BVMO。
1.1 具有CzcO结构域的BVMO 1.1.1 戊丙酯菌素生物合成途径中的PtlE、PntE和PenE戊丙酯菌素Pentalenolactone是链霉菌产生的倍半萜类抗生素。随着重要农用抗生素阿维菌素的产生菌阿维链霉菌基因组的测序完成,与戊丙酯菌素合成相关的基因簇被发现,即ptl基因簇。2009年美国布朗大学David E. Cane教授课题组确定了此基因簇中的BVMO基因ptlE所编码的蛋白催化1-deoxy-11-oxopentalenic acid生成新戊丙酯菌素D[10] (图 2A)。而脱叶链霉菌戊丙酯菌素生物合成基因簇pen中同源基因penE和沙场链霉菌戊丙酯菌素生物合成基因簇pnt中同源基因pntE所编码的酶催化BV反应生成的产物则是戊丙酯菌素D,与PtlE相比表现出相反的区域选择性[11]。

|
| 图 2 微生物次生代谢产物生物合成途径中具有CzcO结构域的单加氧酶催化的拜耳-维立格反应 Fig. 2 Baeyer-Villiger recations catalysed by BVMOs with CzcO domain in the biosynthesis of microbial secondary metabolites. (A) Pentalenolactone. (B) Cytochalasin. (C) Austinol and anditomin. (D) Pederin. (E) JBIR-76 and JBIR-77. |
| |
针对这一组同源蛋白进行点突变,实验数据显示PntE的185位亮氨酸突变成PtlE相应位置的丝氨酸后产生的突变体蛋白PntE (L185S)催化生成新戊丙酯菌素D,即与PtlE催化生成的产物相同[12]。说明此位置对这一组同源的BVMO在催化反应过程中的区域选择性有至关重要的作用。
1.1.2 细胞松弛素生物合成途径中的CcsB细胞松弛素Cytochalasin是真菌产生的具有抗血管增生活性的次生代谢产物。2011年美国唐奕教授课题组在棒曲霉中克隆并确定了大约30 kb细胞松弛素的生物合成基因簇,其中包括一个BVMO基因ccsB[13]。深入研究证明CcsB首先以Ketocytochalasin为底物,发生第一次氧的插入,生成Precytochalasin,紧接着以Precytochalasin为底物发生第二次氧的插入,生成Cytochalasin Z16,如图 2B所示[14]。
1.1.3 3, 5-二甲基苔色酸衍生的杂萜类化合物生物合成途径中的AusC、PrhK和AndJ3, 5-二甲基苔色酸衍生的杂萜类化合物是由真菌合成的一类天然产物。构巢曲霉产生的Austinol和Dehydroaustinol属于这类化合物,其生物合成基因簇于2012年确定[15],基因簇中的ausB基因所编码的具有CzcO结构域的蛋白被认为催化了生物合成途径中的一步BV反应[15]。但是紧接下来的实验数据推翻了这个结论,基因簇中另一个同源基因ausC所编码的蛋白才是真正负责此反应的BVMO[16],如图 2C所示。
青霉菌来源的同源蛋白PrhK和变色曲霉来源的同源蛋白AndJ分别在Paraherquonin和Anditomin生物合成途径中被认为催化一样或者类似的反应[17]。
1.1.4 毒隐翅虫素生物合成途径中的PedG毒隐翅虫共生菌能够产生毒隐翅虫素Pederin。2002年其生物合成基因簇被确定,其中含有一个BVMO基因pedG [18-19]。研究者对PedG如何参与毒隐翅虫素的生物合成给出了两种推测(图 2D):途径a,PedF最后一个模块的ACP结构域上连接的中间产物直接被PedG催化生成酯,紧接着水解生成毒隐翅虫素的主体结构;途径b,PedF催化产生的中间产物继续被PedH延伸和释放,生成Onnamide类型的中间体,在PedG催化下生成酯,然后水解生成毒隐翅虫素的主体结构。
1.1.5 异呋喃萘醌类化合物生物合成途径中的IfnQ和IfqQ异呋喃萘醌类化合物是植物、真菌和放线菌产生的具有抗氧化和抗疟原虫活性的天然产物[20]。2016年在链霉菌RI-77中确定了第一个此类化合物的生物合成基因簇[21]。JBIR-76和77是链霉菌RI-77中一个Ⅱ型聚酮合酶基因簇ifn负责产生的异呋喃萘醌类化合物[22],终产物并没有内酯结构,但是基因簇中包含一个BVMO基因ifnQ,暗示着生物合成途径中可能涉及到BV反应和含有内酯的中间产物。ifnQ基因敲除和IfnQ的生化反应等实验数据表明其可能负责二环中间体上支链的成酯反应,如图 2E所示,以便后期支链上碳链的缩短和第3个环的形成[21]。
2017年美国沈奔教授课题组在链霉菌CB01883中同样分离到此类化合物,确定了相关的生物合成基因簇ifq,与ifn基因簇同源[23]。其中IfqQ被认为负责与其同源蛋白IfnQ相同的反应。
1.2 具有SsuD结构域的BVMO潮霉素属于萘醌类安莎霉素,山东大学沈月毛教授课题组于2014年从链霉菌中克隆了潮霉素生物合成基因簇。其中hgc3基因编码的蛋白属于LLM家族FMN依赖的单加氧酶。依据基因hgc3敲除实验可以推测Hgc3可能以prohygrocin为底物,催化BV反应生成17-dehydroxyhygrocin (图 3)[24]。

|
| 图 3 微生物次生代谢产物生物合成途径中具有SsuD结构域的单加氧酶催化的拜耳-维立格反应 Fig. 3 Baeyer-Villiger recation catalysed by BVMOs with SsuD domain in the biosynthesis of microbial secondary metabolites. |
| |
临床抗癌药物光辉霉素生物合成途径中的MtmOIV是第一个被研究的已知天然底物的BVMO。光辉霉素由链霉菌产生,1996年其生物合成基因簇被克隆[25],随后生物合成途径被逐步研究[26-27]。光辉霉素的三环核心结构对活性来讲是必需的,因此生物合成过程中中间体前光辉霉素B上第4个环的清除显得尤为重要。mtmOIV编码的单加氧酶能够催化前光辉霉素B第4个环形成前光辉霉素B内酯,然后水解开环和脱羧生成光辉霉素DK[28-30],如图 4A所示。通过对MtmOIV蛋白三维结构的深入分析,逐步确认了其FAD的结合位点、底物的结合位点,以及部分NADPH的结合位点[31-33]。

|
| 图 4 微生物次生代谢产物生物合成途径中具有UbiH结构域的单加氧酶催化的拜耳-维立格反应 Fig. 4 Baeyer-Villiger recations catalysed by BVMOs with UbiH domain in the biosynthesis of microbial secondary metabolites. (A) Mithramycin. (B) FD-594. (C) BE-7585A. (D) Murayaquinone. (E) E-837, E-492 and aurafuron. (F) Legonmycin. (G) Pyrrolizixenamide and lipocyclocarbamate. |
| |
与光辉霉素同一类型的化合物Chromomycin的生物合成基因簇中cmmOIV与mtmOIV同源,被认为催化相同的反应[34]。
1.3.2 苯并夹氧杂蒽类抗生素FD-594生物合成途径中的PnxO4FD-594是链霉菌产生的具有抗肿瘤和抗菌活性的化合物,2011年其基因簇被克隆[35],推测其中含有一个FAD依赖的单加氧酶基因pnxO4所编码的酶催化BV反应产生内酯中间体,如图 4B所示。紧接着内酯水解、旋转、重新环化,环氧键打开,脱羧生成双键。
1.3.3 角环素类抗生素BE-7585A生物合成途径中的BexE和BexM角环素类化合物是一组由Ⅱ型聚酮合酶合成的天然产物。BE-7585A是从东方拟无枝酸菌中分离得到,具有抗癌活性[36]。基因簇中的两个基因bexE和bexM所编码的两个FAD依赖的酶被认为参与BV反应,如图 4C所示,其中BexE实现蒽环类骨架转角形成角环素类骨架[37]。而BexM则可能以BexE催化的产物为底物,通过BV反应产生内酯中间物,进而实现迁移碳的羟化。
1.3.4 9, 10-菲醌结构生物合成途径中的MrqO6Murayaquinone是链霉菌产生的含有9, 10-菲醌核心结构的化合物。与角环素类骨架形成类似,9, 10-菲醌中所具有的转角结构也是BVMO参与形成的。2017年上海交通大学的陶美凤教授课题组的实验数据显示,在3个氧化还原酶的催化下,线性的蒽烯发生氧化重排生成了具有转角结构的9, 10-菲醌[38],这其中处于中心的位置是催化BV反应的MrqO6,如图 4D所示。
1.3.5 呋喃酮类化合物生物合成途径中的Orf8_E837、Orf13_E492和AufJE-837、E-492和E-975是链霉菌中的Ⅰ型聚酮合酶催化合成的含有呋喃酮结构的天然产物[39]。两个生物合成基因簇分别含有一个BV单加氧酶基因orf8_E837和orf13_E492。研究者认为聚酮合酶合成的碳链经过环化和释放形成六元环,羟化后的中间物在Orf8_E837或者Orf13_E492催化下发生BV反应,如图 4E所示。紧接着开环脱羧,在其他酶催化下重新环化生成呋喃酮。
Aurafuron是从粘细菌中分离到的化合物[40]。2007年在橙色标桩粘细菌中克隆了生物合成基因簇,其中含有一个FAD依赖的单加氧酶基因aufJ[41]。研究者认为AufJ参与呋喃酮的形成,但与Orf8_E837和Orf13_E492不同,AufJ催化AufC最后一个模块的ACP结构域上连接的中间产物生成酯,水解释放出缩短了碳链的中间产物,然后环化生成呋喃酮。此推测的途径与毒隐翅虫素生物合成中PedG催化的反应途径a类似。
1.3.6 吡咯里西啶生物碱生物合成途径中的PxaB和LgnC吡咯里西啶生物碱是两个五元环形成的含氮杂环化合物,一般由植物产生,用于抵抗植食性昆虫。2015年武汉大学虞沂教授课题组从链霉菌中克隆了Legonmycin的生物合成基因簇[42]。基因簇中的基因lgnC编码的FAD依赖的氧化还原酶以吲哚里西啶中间物为底物,首先催化BV反应,实现扩环,随后水解、脱羧和重新环化,生成吡咯里西啶骨架结构,如图 4F所示。LgnC有可能催化随后羟化反应。与此同时德国Bode教授课题组从致病杆菌中克隆了Pyrrolizixenamide的生物合成基因簇[43]。基因簇中pxaB基因编码的蛋白催化与LgnC类似的反应,如图 4G所示,不做赘述。
1.3.7 Lipocyclocarbamate生物合成途径中的LpiC和BraC与吡咯里西啶生物碱类似,Lipocyclocarbamate (LCC)是一组具有吡咯并噁唑骨架结构的天然产物。2013年和2014年在荧光假单胞菌中克隆确定了这类化合物的生物合成基因簇[44-45]。两个基因簇中的同源基因lpiC和braC编码的蛋白不管是识别的底物还是催化的反应都与PxaB和LgnC非常类似(图 4F、4G)。
1.4 具有ABM结构域的BVMO新角蒽环类抗生素是一组由Ⅱ型PKS合成的具有新角蒽环骨架结构的天然产物,其中也包含了一些骨架结构在新角蒽环基础上发生了明显改变的化合物,例如Kinamycin、Jadomycin和Gilvocarcin。这些改变都与氧化还原酶参与的BV反应相关。2012年和2015年,中国科学院微生物研究所杨克迁教授课题组针对Jadomycin和Kinamycin生物合成途径中两个同源蛋白JadG和AlpJ(以及KinG)进行了研究,发现JadG和AlpJ负责新角蒽环骨架中B环的开环[46-47],2017年AlpJ的蛋白结构被解析[48]。在Gilvocarcin生物合成途径中的同源蛋白GilOII的研究过程中,一个含内酯的中间产物被分离和确定,如图 5所示,证明了B环的开环与BV反应有关[49]。与之同源的并且可能催化类似反应的还包括Lomaiviticin生物合成途径中的Lom28 (以及Strop_2200)[50-51]、Fluostatin生物合成途径中的FlsG (以及Flu_orf17)[52-53]、Chrysomycin生物合成途径中的ChryOII、Ravidomycin生物合成途径中的RavOII[54]。
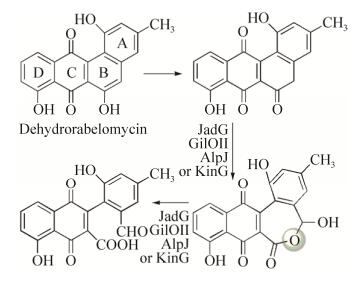
|
| 图 5 微生物次生代谢产物生物合成途径中具有ABM结构域的单加氧酶催化的拜耳-维立格反应 Fig. 5 Baeyer-Villiger recations catalysed by BVMOs with ABM domain in the biosynthesis of microbial secondary metabolites. |
| |
针对分解代谢中涉及的BVMO,研究者往往从生物催化应用的角度出发,选择各种结构简单的含酮化合物作为底物,测定酶的底物选择性和催化活性,以及反应的区域选择性和立体选择性。与分解代谢不同,一般情况下微生物次生代谢产物生物合成途径中的BVMO都有相对明确的天然底物,研究工作往往围绕天然底物展开。以上所列举的BVMO的天然底物存在巨大的差异,有的BVMO识别小分子化合物,也有的BVMO可以识别连接在蛋白(ACP)上的化合物。而作为底物的小分子也有明显的结构多样性,有相对简单的化合物,例如参与戊丙酯菌素生物合成的BVMO所识别的底物为简单的三环化合物(图 2A);也有相对复杂的化合物,例如参与光辉霉素生物合成的BVMO所识别的底物为发生糖基化修饰的稠环芳烃化合物(图 3A)。
就反应的区域选择性来讲,以上所列大部分BVMO遵循有机化学中BV反应区域选择性规律,即含氢少的碳发生迁移,生成正常的内酯,例如戊丙酯菌素生物合成中的PtlE、细胞松弛素生物合成中的CcsB、3, 5-二甲基苔色酸衍生的杂萜类化合物生物合成中的AusC、PrhK和AndJ、潮霉素生物合成中的Hgc3、角环素类抗生素生物合成中的BexE和BexM、9, 10-菲醌结构生物合成中的MrqO6、呋喃酮类化合物生物合成中的AufJ。少数BVMO并不遵循这一规律,即含氢多的碳发生了迁移,生成非正常内酯,例如戊丙酯菌素生物合成中的PntE和PenE。就反应的立体选择性来讲,戊丙酯菌素生物合成中的PtlE和细胞松弛素生物合成中的CcsB遵循了有机化学中BV反应立体选择性规律,即迁移的碳如果是手性碳,在迁移后都保留了迁移前的手性。其他BVMO或者不涉及此问题,或者研究者并没有给出明确的结论。
有的BVMO的天然底物中连接羰基碳的并不都是碳,例如CcsB第二次的BV反应底物(图 2B)、Orf8_E837或者Orf13_E492的底物(图 4E)以及LgnC、PxaB、LpiC和BraC的底物(图 4F,4G)。这些实验数据说明在BVMO在生物催化应用中不仅仅可以用来催化酮生成酯,也有可能催化酯类化合物生成碳酸酯类化合物,催化酰胺类化合物生成胺甲酸酯类化合物。
许多BVMO所催化的不仅仅是BV反应,往往还伴随着其他后续反应的发生,例如水解、脱羧、分子内重排、羟化等。对于酯来说,发生水解以及脱羧很容易理解。分子内重排的典型例子是MrqO6所参与的9, 10-菲醌的形成,这可能与内酯中间产物本身的结构有很大的关系。值得注意的是黄素依赖的蛋白催化碳原子或者杂原子的羟化有很多报道,但是LgnC和PxaB催化BV反应的同时也可以催化碳原子羟化的现象则较为少见。
目前并没有文献报道这些BVMO是否能够识别非天然的底物催化BV反应或者杂原子的氧化。我们小组在研究戊丙酯菌素生物合成时发现PntE能够以环戊酮和环己酮为底物催化BV反应生成相应的内酯,但是相对于天然底物,PntE对非天然底物的转化效率非常低(未发表)。
3 BVMO蛋白的特点利用以上列举的4类BVMO蛋白的氨基酸序列构建系统发育树,如图 6A所示,大部分的蛋白根据其具有的保守结构域聚成一群。

|
| 图 6 微生物次生代谢产物生物合成途径中的拜耳-维立格单加氧酶氨基酸序列分析 Fig. 6 Protein sequence analysis of BVMOs in the biosynthesis of microbial secondary metabolites. (A) Phylogenetic analysis. (B) Partial sequence alignment of BVMO proteins with CzcO domain. (C) Partial sequence alignment of BVMO proteins with UbiH domain. |
| |
1.1中列举的具有CzcO结构域的BVMO,即Ⅰ型BVMO,所催化的天然底物在结构上虽然相差很大,但这一组蛋白的氨基酸序列上依然表现出了相对较高的一致性和相似性。首先Ⅰ型BVMO具有的保守motif (FXGXXXHXXXWP),在BVMO都能找到,但略有出入,如图 6B箭头标示位置:第一,大部分蛋白此保守区域的序列是FXGXXXHXXXWD,即最后一个氨基酸并不是脯氨酸,而是天冬氨酸;第二,AusC、PrhK和AndJ此保守区域的序列为YXGXXXHXXXWD,即第一个氨基酸并不是苯丙氨酸,而是酪氨酸;第三,PedG与其他蛋白相比,在一级序列上的相似性和一致性较低,保守的motif序列为FXGXXXHXXXYK,即最后两个氨基酸不是色氨酸和脯氨酸,而是酪氨酸和赖氨酸。可以造成PntE区域选择性发生超过90%反转的突变L185S就位于此保守motif前15个氨基酸的位置,如图 6B左边三角标示位置。而另外一个对PntE区域选择性产生大约30%反转的突变是R484L,如图 6B右边三角标示位置。氨基酸序列同源比对表明此突变所在的区域是通过蛋白质工程对PAMO的区域选择性和立体选择性进行改造的重点区域。
与Ⅰ型BVMO相比,已发现的Ⅱ型BVMO并不多,研究深入的例子是假单胞菌中参与樟脑烷分解代谢的两个二酮樟脑烷单加氧酶:2, 5-二酮樟脑烷-1, 2-单加氧酶(2, 5-DKMO)和3, 6-二酮樟脑烷-1, 6-单加氧酶(3, 6-DKMO)。3, 6-DKMO是唯一的蛋白质结构被解析的Ⅱ型BVMO。但通过序列的同源比对,可以发现3, 6-DKMO活性中心与FMN结合相关的氨基酸残基在2, 5-DKMO中没有表现出保守性。氨基酸水平的序列比对可以发现3, 6-DKMO活性中心与FMN结合相关的氨基酸残基在Hgc3中同样没有表现出保守性,系统发育树中Hgc3也没有与3, 6-DKMO和2, 5-DKMO聚成一群。依据Hgc3蛋白具有SscD结构域和对hgc3基因敲除突变株的检测结果推测其催化BV反应是有其合理性的,但是缺乏可作为直接证据的体外生化反应,使得此结论的得出尚存疑虑。
与1.1所列举的Ⅰ型BVMO相比,1.3列举的O型BVMO在蛋白的氨基酸序列水平上的一致性和相似性明显降低。其中MtmOIV蛋白的三维结构被深入研究:MtmOIV有FAD结合结构域(氨基酸残基10–183和276–394组成)、中间结构域(氨基酸残基184–275)和C端结构域(氨基酸残基395–510) 3个结构域。O型BVMO蛋白序列上并不具备Ⅰ型BVMO所具有的保守motif序列,但从序列比对的结果可以看到保守的区域主要有3个(以MtmOIV为例说明所处位置),如图 6C所示:第一,MtmOIV的15–42氨基酸区域;第二,MtmOIV的159–182氨基酸区域;第三,MtmOIV的285–320氨基酸区域,即UbiH结构域。这3个氨基酸序列保守的区域均位于MtmOIV的FAD结合结构域。蛋白C端(即MtmOIV的477–485氨基酸区域)也存在一定的保守性。
到目前为止,具有合成代谢途径中具有ABM结构域的BVMO在分解代谢途径中未见报道,BVMO分类中也并不包括这一类。系统发育树暗示这一类BVMO在进化关系上可能与具有SsuD结构域的Ⅱ型BVMO相对较近。此类BVMO仅有不到250个氨基酸残基,明显小于其他三类。另外,此类BVMO虽然在催化反应时也需要FAD或者FMN参与,但蛋白本身与这些辅因子并没有很强的结合能力。已发现的这类蛋白的催化底物基本一致,所以其在氨基酸序列上具有较高的一致性和相似性。依据已被解析的AlpJ的三维结构,Asn60、Trp64和Trp181被认为可能与底物结合相关,His50和Tyr178被认为可能和三维结构的稳定相关[48]。在其他同源蛋白中,这5个氨基酸残基表现出高度的保守性。
需要注意的是当对某一个酶进行氨基酸序列比对时,如果发现其具有以上4种保守结构域中某一种,这并不能说明其必然催化BV反应。例如南京农业大学刘凤权教授课题组所报道的吩嗪类抗生素生物合成途径中的LaPhzNO1,其具有CzcO保守结构域,但催化N的氧化,并非BV反应[55]。
4 小结限制酶催化和生物催化在有机合成中应用的因素有很多,其中最重要的有两点:第一,酶作为蛋白质是不稳定的;第二,酶一般具有较窄的底物选择范围。一直以来微生物分解代谢中的CHMO和PAMO因其相对较宽的底物选择范围或相对较高的热稳定性而成为研究和改造的重点关注对象,例如通过蛋白质工程等手段拓宽底物范围,改变区域选择性,改善对映选择性,提高对硫的氧化能力,改变其辅因子,增加稳定性等[5-9]。从2002年开始出现利用BVMO实现公斤级以上规模生物催化的零星报道,也有固定化酶、固定化细胞、两相催化体系和大规模催化中氧的供给等问题的研究[6]。这些研究加深了我们对BVMO基础酶学和生物催化的认识,同时也对其工业应用作出了有力的推动。
微生物次生代谢产物是药物的重要来源之一,关注天然产物生物合成的课题组往往把研究重心放在生物合成基因簇的克隆,生物合成途径的推测和验证,酶催化机制的探索,在此基础上通过合成生物学手段获取新的活性化合物。其中相关酶在生物催化领域的潜在应用一般并不作为研究重点。文中针对这一点,总结了微生物次生代谢产物生物合成途径中所涉及的BV反应以及负责催化BV反应的酶,初步分析了反应的特点和相关酶在氨基酸序列上的保守性。希望通过以上总结和分析能够为BVMO的蛋白质工程改造提供更多的选择,推动其在生物催化领域的应用。
| [1] | Turfitt GE. The microbiological degradation of steroids. Biochem J, 1948, 42(3): 376–383. DOI: 10.1042/bj0420376 |
| [2] | Donoghue NA, Norris DB, Trudgill PW. The purification and properties of cyclohexanone oxygenase from Nocardia globerula CL1 and Acinetobacter NCIB 9871. Eur J Biochem, 1976, 63(1): 175–192. DOI: 10.1111/ejb.1976.63.issue-1 |
| [3] | Griffin M, Trudgill PW. Purification and properties of cyclopentanone oxygenase of Pseudomonas NCIB 9872. Eur J Biochem, 1976, 63(1): 199–209. DOI: 10.1111/ejb.1976.63.issue-1 |
| [4] | Chen YC, Peoples OP, Walsh CT. Acinetobacter cyclohexanone monooxygenase: gene cloning and sequence determination. J Bacteriol, 1988, 170(2): 781–789. DOI: 10.1128/jb.170.2.781-789.1988 |
| [5] | Balke K, Beier A, Bornscheuer UT. Hot spots for the protein engineering of Baeyer-Villiger monooxygenases. Biotechnol Adv, 2018, 36(1): 247–263. DOI: 10.1016/j.biotechadv.2017.11.007 |
| [6] | Bučko M, Gemeiner P, Schenkmayerová A, et al. Baeyer-Villiger oxidations: biotechnological approach. Appl Microbiol Biotechnol, 2016, 100(15): 6585–6599. DOI: 10.1007/s00253-016-7670-x |
| [7] | Leisch H, Morley K, Lau PCK. Baeyer-Villiger monooxygenases: more than just green chemistry. Chem Rev, 2011, 111(7): 4165–4222. DOI: 10.1021/cr1003437 |
| [8] | de Gonzalo G, Mihovilovic MD, Fraaije MW. Recent developments in the application of Baeyer-Villiger monooxygenases as biocatalysts. Chembiochem, 2010, 11(16): 2208–2231. DOI: 10.1002/cbic.v11:16 |
| [9] | Pazmiño DET, Dudek HM, Fraaije MW. Baeyer-Villiger monooxygenases: recent advances and future challenges. Curr Opin Chem Biol, 2010, 14(2): 138–144. |
| [10] | Jiang JY, Tetzlaff CN, Takamatsu S, et al. Genome mining in Streptomyces avermitilis: A biochemical Baeyer-Villiger reaction and discovery of a new branch of the pentalenolactone family tree. Biochemistry, 2009, 48(27): 6431–6440. DOI: 10.1021/bi900766w |
| [11] | Seo MJ, Zhu DQ, Endo S, et al. Genome mining in Streptomyces. Elucidation of the role of Baeyer-Villiger monooxygenases and non-heme iron-dependent dehydrogenase/oxygenases in the final steps of the biosynthesis of pentalenolactone and neopentalenolactone. Biochemistry, 2011, 50(10): 1739–1754. DOI: 10.1021/bi1019786 |
| [12] | Chen K, Wu SW, Zhu L, et al. Substitution of a single amino acid reverses the regiospecificity of the baeyer-villiger monooxygenase pnte in the biosynthesis of the antibiotic pentalenolactone. Biochemistry, 2016, 55(48): 6696–6704. DOI: 10.1021/acs.biochem.6b01040 |
| [13] | Qiao KJ, Chooi YH, Tang Y. Identification and engineering of the cytochalasin gene cluster from Aspergillus clavatus NRRL 1. Metabol Eng, 2011, 13(6): 723–732. DOI: 10.1016/j.ymben.2011.09.008 |
| [14] | Hu YC, Dietrich D, Xu W, et al. A carbonate-forming Baeyer-Villiger monooxygenase. Nat Chem Biol, 2014, 10(7): 552–554. DOI: 10.1038/nchembio.1527 |
| [15] | Lo HC, Entwistle R, Guo CJ, et al. Two separate gene clusters encode the biosynthetic pathway for the meroterpenoids austinol and dehydroaustinol in Aspergillus nidulans. J Am Chem Soc, 2012, 134(10): 4709–4720. DOI: 10.1021/ja209809t |
| [16] | Matsuda Y, Awakawa T, Wakimoto T, et al. Spiro-ring formation is catalyzed by a multifunctional dioxygenase in austinol biosynthesis. J Am Chem Soc, 2013, 135(30): 10962–10965. DOI: 10.1021/ja405518u |
| [17] | Matsuda Y, Wakimoto T, Mori T, et al. Complete biosynthetic pathway of anditomin: nature's sophisticated synthetic route to a complex fungal meroterpenoid. J Am Chem Soc, 2014, 136(43): 15326–15336. DOI: 10.1021/ja508127q |
| [18] | Piel J. A polyketide synthase-peptide synthetase gene cluster from an uncultured bacterial symbiont of Paederus beetles. Proc Natl Acad Sci USA, 2002, 99(22): 14002–14007. DOI: 10.1073/pnas.222481399 |
| [19] | Piel J, Höfer I, Hui DQ. Evidence for a symbiosis island involved in horizontal acquisition of pederin biosynthetic capabilities by the bacterial symbiont of Paederus fuscipes beetles. J Bacteriol, 2004, 186(5): 1280–1286. DOI: 10.1128/JB.186.5.1280-1286.2004 |
| [20] | Bezabih M, Abegaz BM, Dufall K, et al. Antiplasmodial and antioxidant isofuranonaphthoquinones from the roots of Bulbine capitata. Planta Med, 2001, 67(4): 340–344. |
| [21] | Katsuyama Y, Sone K, Satou R, et al. Involvement of the baeyer-villiger monooxygenase IfnQ in the biosynthesis of isofuranonaphthoquinone scaffold of JBIR-76 and -77. Chembiochem, 2016, 17(11): 1021–1028. DOI: 10.1002/cbic.v17.11 |
| [22] | Motohashi K, Izumikawa M, Kagaya N, et al. JBIR-76 and JBIR-77, modified naphthoquinones from Streptomyces sp. RI-77. J Antibiot, 2016, 69(9): 707–708. DOI: 10.1038/ja.2015.135 |
| [23] | Guo ZK, Pan GH, Xu ZR, et al. New isofuranonaphthoquinones and isoindolequinones from Streptomyces sp. CB01883. J Antibiot, 2017, 70(4): 414–422. DOI: 10.1038/ja.2016.122 |
| [24] | Li SR, Wang HX, Li YY, et al. Biosynthesis of hygrocins, antitumor naphthoquinone ansamycins produced by Streptomyces sp. LZ35. Chembiochem, 2014, 15(1): 94–102. DOI: 10.1002/cbic.201300599 |
| [25] | Lombó F, Blanco G, Fernández E, et al. Characterization of Streptomyces argillaceus genes encoding a polyketide synthase involved in the biosynthesis of the antitumor mithramycin. Gene, 1996, 172(1): 87–91. DOI: 10.1016/0378-1119(96)00029-7 |
| [26] | Lombó F, Brana AF, Méndez C, et al. The mithramycin gene cluster of Streptomyces argillaceus contains a positive regulatory gene and two repeated DNA sequences that are located at both ends of the cluster. J Bacteriol, 1999, 181: 642–647. |
| [27] | Remsing LL, Garcia-Bernardo J, Gonzalez A, et al. Ketopremithramycins and ketomithramycins, four new aureolic acid-type compounds obtained upon inactivation of two genes involved in the biosynthesis of the deoxysugar moieties of the antitumor drug mithramycin by Streptomyces argillaceus, reveal novel insights into post-PKS tailoring steps of the mithramycin biosynthetic pathway. J Am Chem Soc, 2002, 124(8): 1606–1614. DOI: 10.1021/ja0105156 |
| [28] | Prado L, Fernández E, Weiβbach U, et al. Oxidative cleavage of premithramycin B is one of the last steps in the biosynthesis of the antitumor drug mithramycin. Chem Biol, 1999, 6(1): 19–30. |
| [29] | Rodríguez D, Quirós LM, Braña AF, et al. Purification and characterization of a monooxygenase involved in the biosynthetic pathway of the antitumor drug mithramycin. J Bacteriol, 2003, 185(13): 3962–3965. DOI: 10.1128/JB.185.13.3962-3965.2003 |
| [30] | Gibson M, Nur-e-alam M, Lipata F, et al. Characterization of kinetics and products of the Baeyer-Villiger oxygenase MtmOIV, the key enzyme of the biosynthetic pathway toward the natural product anticancer drug mithramycin from Streptomyces argillaceus. J Am Chem Soc, 2005, 127(50): 17594–17595. DOI: 10.1021/ja055750t |
| [31] | Wang C, Gibson M, Rohr J, et al. Crystallization and X-ray diffraction properties of Baeyer-Villiger monooxygenase MtmOIV from the mithramycin biosynthetic pathway in Streptomyces argillaceus. Acta Cryst, 2005, 61: 1023–1026. |
| [32] | Beam MP, Bosserman MA, Noinaj N, et al. Crystal structure of Baeyer-Villiger monooxygenase MtmOIV, the key enzyme of the mithramycin biosynthetic pathway. Biochemistry, 2009, 48(21): 4476–4487. DOI: 10.1021/bi8023509 |
| [33] | Bosserman MA, Downey T, Noinaj N, et al. Molecular insight into substrate recognition and catalysis of Baeyer-Villiger monooxygenase MtmOIV, the key frame-modifying enzyme in the biosynthesis of anticancer agent mithramycin. ACS Chem Biol, 2013, 8(11): 2466–2477. DOI: 10.1021/cb400399b |
| [34] | Menéndez N, Nur-e-Alam M, Braña AF, et al. Biosynthesis of the antitumor chromomycin A3 in Streptomyces griseus: analysis of the gene cluster and rational design of novel chromomycin analogs. Chem Biol, 2004, 11(1): 21–32. |
| [35] | Kudo F, Yonezawa T, Komatsubara A, et al. Cloning of the biosynthetic gene cluster for naphthoxanthene antibiotic FD-594 from Streptomyces sp. TA-0256. J Antibiot, 2011, 64(1): 123–132. |
| [36] | Sasaki E, Ogasawara Y, Liu HW. A biosynthetic pathway for BE-7585A, a 2-thiosugar-containing angucycline-type natural product. J Am Chem Soc, 2010, 132(21): 7405–7417. DOI: 10.1021/ja1014037 |
| [37] | Jackson DR, Yu X, Wang G, et al. Insights into complex oxidation during BE-7585A biosynthesis: structural determination and analysis of the polyketide monooxygenase BexE. ACS Chem Biol, 2016, 11(4): 1137–1147. DOI: 10.1021/acschembio.5b00913 |
| [38] | Gao GX, Liu XY, Xu M, et al. Formation of an angular aromatic polyketide from a linear anthrene precursor via oxidative rearrangement. Cell Chem Biol, 2017, 24(7): 881–891. DOI: 10.1016/j.chembiol.2017.06.008 |
| [39] | Banskota AH, McAlpine JB, Sørensen D, et al. Isolation and identification of three new 5-alkenyl-3, 3(2H)-furanones from two Streptomyces species using a genomic screening approach. J Antibiot, 2006, 59(3): 168–176. |
| [40] | Kunze B, Reichenbach H, Müller R, et al. Aurafuron A and B, new bioactive polyketides from Stigmatella aurantiaca and Archangium gephyra (Myxobacteria). J Antibiot, 2005, 58(4): 244–251. DOI: 10.1038/ja.2005.28 |
| [41] | Frank B, Wenzel SC, Bode HB, et al. From genetic diversity to metabolic unity: studies on the biosynthesis of aurafurones and aurafuron-like structures in myxobacteria and streptomycetes. J Mol Biol, 2007, 374(1): 24–38. DOI: 10.1016/j.jmb.2007.09.015 |
| [42] | Huang S, Tabudravu J, Elsayed SS, et al. Discovery of a single monooxygenase that catalyzes carbamate formation and ring contraction in the biosynthesis of the legonmycins. Angewand Chem, 2015, 54(43): 12697–12701. DOI: 10.1002/anie.201502902 |
| [43] | Schimming O, Challinor VL, Tobias NJ, et al. Structure, biosynthesis, and occurrence of bacterial pyrrolizidine alkaloids. Angewand Chem, 2015, 54(43): 12702–12705. DOI: 10.1002/anie.201504877 |
| [44] | Johnston CW, Zvanych R, Khyzha N, et al. Nonribosomal assembly of natural lipocyclocarbamate lipoprotein-associated phospholipase inhibitors. Chembiochem, 2013, 14(4): 431–435. DOI: 10.1002/cbic.201200598 |
| [45] | Schmidt Y, van der Voort M, Crüsemann M, et al. Biosynthetic origin of the antibiotic cyclocarbamate brabantamide A (SB-253514) in plant-associated Pseudomonas. Chembiochem, 2014, 15(2): 259–266. DOI: 10.1002/cbic.201300527 |
| [46] | Fan KQ, Pan GH, Peng XJ, et al. Identification of JadG as the B ring opening oxygenase catalyzing the oxidative C-C bond cleavage reaction in jadomycin biosynthesis. Chem Biol, 2012, 19(11): 1381–1390. DOI: 10.1016/j.chembiol.2012.09.009 |
| [47] | Wang B, Ren JW, Li LY, et al. Kinamycin biosynthesis employs a conserved pair of oxidases for B-ring contraction. Chem Commun, 2015, 51(42): 8845–8848. DOI: 10.1039/C5CC01986A |
| [48] | Pan GH, Gao XQ, Fan KQ, et al. Structure and function of a C-C bond cleaving oxygenase in atypical angucycline biosynthesis. ACS Chem Biol, 2017, 12(1): 142–152. DOI: 10.1021/acschembio.6b00621 |
| [49] | Tibrewal N, Pahari P, Wang GJ, et al. Baeyer-Villiger C-C bond cleavage reaction in gilvocarcin and jadomycin biosynthesis. J Am Chem Soc, 2012, 134(44): 18181–18184. DOI: 10.1021/ja3081154 |
| [50] | Janso JE, Haltli BA, Eustáquio AS, et al. Discovery of the lomaiviticin biosynthetic gene cluster in Salinispora pacifica. Tetrahedron, 2014, 70(27/28): 4156–4164. |
| [51] | Kersten RD, Lane AL, Nett M, et al. Bioactivity-guided genome mining reveals the lomaiviticin biosynthetic gene cluster in Salinispora tropica. Chembiochem, 2013, 14(8): 955–962. DOI: 10.1002/cbic.v14.8 |
| [52] | Yang CF, Huang CS, Zhang WJ, et al. Heterologous expression of fluostatin gene cluster leads to a bioactive heterodimer. Org Lett, 2015, 17(21): 5324–5327. DOI: 10.1021/acs.orglett.5b02683 |
| [53] | Feng ZY, Kim JH, Brady SF. Fluostatins produced by the heterologous expression of a TAR reassembled environmental DNA derived type Ⅱ PKS gene cluster. J Am Chem Soc, 2010, 132(34): 11902–11903. DOI: 10.1021/ja104550p |
| [54] | Kharel MK, Nybo SE, Shepherd MD, et al. Cloning and characterization of the ravidomycin and chrysomycin biosynthetic gene clusters. Chembiochem, 2010, 11(4): 523–532. DOI: 10.1002/cbic.v11:4 |
| [55] | Zhao YY, Qian GL, Ye YH, et al. Heterocyclic aromatic N-Oxidation in the biosynthesis of phenazine antibiotics from Lysobacter antibioticus. Org Lett, 2016, 18(10): 2495–2498. DOI: 10.1021/acs.orglett.6b01089 |
 2019, Vol. 35
2019, Vol. 35




