中国科学院微生物研究所、中国微生物学会主办
文章信息
- 笪央央, 李微, 史理陇, 李正军
- Da Yangyang, Li Wei, Shi Lilong, Li Zhengjun
- 大肠杆菌利用葡萄糖和木糖合成乙醇酸、乳酸和3-羟基丁酸共聚酯
- Microbial production of poly (glycolate-co-lactate-co-3-hydroxybutyrate) from glucose and xylose by Escherichia coli
- 生物工程学报, 2019, 35(2): 254-262
- Chinese Journal of Biotechnology, 2019, 35(2): 254-262
- 10.13345/j.cjb.180199
-
文章历史
- Received: May 11, 2018
- Accepted: July 26, 2018
聚羟基脂肪酸酯(Polyhydroxyalkanoate,简称PHA)是一类由微生物在生长代谢不平衡条件下合成的作为碳源和能源储存物质的高分子聚酯[1-2]。与传统的石油来源的塑料材料相比,PHA具有生物可再生性和生物可降解性等优点,引起了国内外研究者的广泛关注。随着代谢工程和合成生物学技术的进步,越来越多的新型PHA合成菌株被构建出来,在提高聚合物产量、调控聚合物单体组成和比例、降低发酵成本等方面取得了良好的进展[3-4]。目前,PHA材料的生产成本仍然高于石油来源的塑料材料,成为PHA材料大规模工业化生产和应用的主要障碍。
葡萄糖是自然界中存在最为广泛的单糖,是微生物发酵的主要碳源[5]。氨基酸和有机酸等大多数发酵产品以葡萄糖为底物进行,存在“与人争粮、与粮争地”的难题。如果能以木质纤维素为原料来获得高附加值化学品,将会产生巨大的经济和社会效益。木糖是木质纤维素水解产物中含量仅次于葡萄糖的单糖[6],占木质纤维素原料的18%–30%[7]。然而,木糖作为微生物的发酵原料仍是难点,缺少有效转化木糖进行化学品生产的工程菌株[8]。例如,传统的乙醇发酵菌株,包括酿酒酵母Saccharomyces cerevisiae[9]和运动发酵单胞菌Zymomonas mobilis[10]等,都不能够有效利用木糖。因此,开发高效利用木糖获得生物基化学品和生物材料的工程菌株,实现木糖的高效转化,是促进木质纤维素资源大规模工业化应用的重要因素。
大肠杆菌是现代生物学中研究最多的细菌,将其作为宿主进行发酵具有许多优势,如遗传背景清楚、生长迅速、营养要求较低和底物利用范围广等。对大肠杆菌进行代谢工程改造,可以获得高级醇类[11-12]、多元醇[13]、短链烷烃[14]、琥珀酸[15]、葡糖二酸[16]和脂肪酸[17]等,部分产品已达较高发酵水平。在之前的研究工作中,我们利用大肠杆菌构建了葡萄糖合成乙醇酸、乳酸和3-羟基丁酸共聚酯poly(glycolate-co-lactate-co-3- hydroxybutyrate) [P(GA-LA-3HB)]的代谢途径[18]。在此基础上,文中构建了以葡萄糖和木糖为底物合成P(GA-LA-3HB)的生物合成途径,并通过混菌共培养的方法实现两种碳源的同时利用,为将来木质纤维素水解物的有效利用提供了参考。
1 材料与方法 1.1 菌种与质粒本研究使用的菌种和质粒见表 1。E. coli K12(DE3)及其木酮糖激酶基因xylB和乙醇酸氧化酶基因glcD双敲除突变体ΔxylB ΔglcD为实验室保存。质粒pKD13、pKD46和pCP20由美国耶鲁大学CGSC (The Coli Genetic Stock Center)惠赠。
| Name | Description | Reference |
| Strains | ||
| K12 (DE3) | Wild type | [20] |
| K12 ΔxylB ΔglcD | K12 (DE3) ΔxylB ΔglcD | [20] |
| K12 ΔxylB ΔglcD ΔldhA | K12 (DE3) ΔxylB ΔglcD ΔldhA | This study |
| K12 ΔxylB ΔglcD Δpts | K12 (DE3) ΔxylB ΔglcD Δpts | This study |
| K12 ΔxylB ΔglcD ΔxylA | K12 (DE3) ΔxylB ΔglcD ΔxylA | This study |
| PGA21 | K12 ΔxylB ΔglcD containing pET-pct-CAB and pGAx3 | This study |
| PGA22 | K12 ΔxylB ΔglcD containing pET-pct-PCAB and pGAx3 | This study |
| PGA23 | K12 ΔxylB ΔglcD ΔldhA containing pET-pct-PCAB and pGAx3 | This study |
| PGA24 | K12 ΔxylB ΔglcD Δpts containing pGAx3 | This study |
| PGA25 | K12 ΔxylB ΔglcD ΔxylA containing pET-pct-PCAB | This study |
| Plasmids | ||
| pKD13 | Template plasmid with KanR gene and FLP recognition target | Yale CGSC |
| pKD46 | λ-Red recombinase expression helper plasmid, AmpR | Yale CGSC |
| pCP20 | FLP recombinase helper plasmid, AmpR, CmR | Yale CGSC |
| pGAx3 | dte, fucA, fucK, and aldA expression plasmid, SpecR | [20] |
| pET-pct-CAB | pct, phaC, phaA, and phaB expression plasmid, AmpR | [18] |
| pET-pct-PCAB | pct, phaP, phaC, phaA, and phaB expression plasmid, AmpR | This study |
大肠杆菌的常规培养在LB培养基中进行,大肠杆菌合成聚合物的摇瓶实验在MMYE培养基中进行。含有质粒的重组菌株在培养时加入相应的抗生素,浓度分别为氨苄青霉素100 μg/mL,卡那霉素50 μg/mL,氯霉素34 μg/mL,盐酸大观霉素50 μg/mL。
LB培养基:5 g/L酵母粉,10 g/L蛋白胨,10 g/L NaCl,pH 7.0。LB固体培养基额外添加15 g/L的琼脂。
MMYE培养基:4 g/L酵母粉,2 g/L NH4Cl,5 g/L (NH4)2SO4,6 g/L KH2PO4,8.4 g/L 3-吗啉丙磺酸(MOPS, C7H15NO4S),0.5 g/L NaCl,0.24 g/L MgSO4,0.002 g/L Na2MoO4,1 mL/L微量元素溶液。微量元素溶液包括:3.6 g/L FeCl2·4H2O,5 g/L CaCl2·2H2O,1.3 g/L MnCl2·2H2O,0.38 g/L CuCl2·2H2O,0.5 g/L CoCl2·6H2O,0.94 g/L ZnCl2,0.03 g/L H3BO3,0.4 g/L Na2EDTA·2H2O,1.01 g/L thiamine-HCl。
1.3 主要试剂Pusion High-Fidelity DNA聚合酶、限制性内切酶和T4 DNA连接酶等购于New England Biolabs公司;质粒提取试剂盒和DNA回收试剂盒购于北京博迈德基因技术有限公司;聚-3-羟基丁酸酯和聚乳酸-乙醇酸共聚酯标准品购于Sigma-Aldrich公司。其他抗生素、异丙基硫代半乳糖苷(IPTG)、酵母粉和蛋白胨等试剂均为国产或进口分析纯。
1.4 pET-pct-PCAB的构建对罗氏真养菌Ralstonia eutropha H16的PHA颗粒结合蛋白基因phaP1的序列进行密码子优化,基因全序列合成由无锡青兰生物科技有限公司完成。人工合成的phaP1插入到质粒pET-pct- CAB[18]的PstⅠ和NotⅠ位点,构建质粒pET-pct- PCAB,含有T7启动子转录的pct、phaP1、phaC、phaA和phaB基因(图 1)。
|
| 图 1 pET-pct-PCAB的质粒图谱 Fig. 1 Schematic map of pET-pct-PCAB |
| |
采用Red同源重组的方法在大肠杆菌中构建基因缺失突变体[19],基因敲除所需的引物见表 2,引物合成和DNA测序由生工生物工程(上海)股份有限公司完成。
| Primer | Sequence (5'–3') |
| ptsF | TCGAACCGCCAGGCTAGACTTTAGTTCCACAACACTAAACCTATAAGTTGGGGAAATACAGTGTAGGCTGGAGCTGCTTCG |
| ptsR | AAAAATGGCGCCGATGGGCGCCATTTTTCACTGCGGCAAGAATTACTTCTTGATGCGGATATTCCGGGGATCCGTCGACC |
| xylAF | ATGCAAGCCTATTTTGACCAGCTCGATCGCGTTCGTTATGAAGGCTCAAAATCCTCAAACGTGTAGGCTGGAGCTGCTTCG |
| xylAR | CGGTGGTGAAACCGCCTGCTTTGAGAATTTCATACATCACCAGCGCATTCTCTTCCACACATTCCGGGGATCCGTCGACC |
| ldhAF | TATTTTTAGTAGCTTAAATGTGATTCAACATCACTGGAGAAAGTCTTATGGTGTAGGCTGGAGCTGCTTCG |
| ldhAR | CTCCCCTGGAATGCAGGGGAGCGGCAAGATTAAACCAGTTCGTTCGGGCAATTCCGGGGATCCGTCGACC |
以质粒pKD13为模板,ptsF和ptsR为引物,PCR扩增约1.5 kb的含有卡那霉素抗性基因的DNA片段,纯化之后,电击转化含有质粒pKD46的K12 ΔxylB ΔglcD感受态细胞,涂布含有卡那霉素的LB平板,30 ℃培养24 h,挑单克隆进行菌落PCR和DNA测序鉴定,得到含有卡那霉素抗性的丙酮酸-糖磷酸转移酶系统基因pts缺失突变体。
将质粒pCP20转化到上述突变体中,涂布含有氯霉素的LB平板,30 ℃培养24 h,消除卡那霉素抗性;挑单克隆转接到无抗的LB平板,42 ℃培养24 h,丢失pCP20质粒;最后验证菌株的抗性,选择没有氯霉素、卡那霉素和氨苄青霉素抗性的菌株进行菌落PCR和DNA测序鉴定,获得最终的pts缺失突变体K12 ΔxylB ΔglcD Δpts。
1.5.2 K12 ΔxylB ΔglcD ΔxylA的构建以K12 ΔxylB ΔglcD为初始菌株,木糖异构酶基因xylA的敲除与pts的敲除采用同样的方法进行。
1.5.3 K12 ΔxylB ΔglcD ΔldhA的构建以K12 ΔxylB ΔglcD为初始菌株,乳酸脱氢酶基因ldhA的敲除与pts的敲除采用同样的方法进行。
1.6 聚合物合成的摇瓶培养 1.6.1 单菌培养质粒pET-pct-CAB[18]和含有木糖合成乙醇酸相关基因的质粒pGAx3[20]共转化到E. coli K12 ΔxylB ΔglcD中,构建重组菌PGA21。质粒pET-pct-PCAB和pGAx3共转化到E. coli K12 ΔxylB ΔglcD中,构建重组菌PGA22。质粒pET-pct-PCAB和pGAx3共转化到E. coli K12 ΔxylB ΔglcD ΔldhA中,构建重组菌PGA23。
从冻存管中取出菌种PGA21、PGA22和PGA23,接种到LB培养基中37 ℃培养12 h作为种子液。3种菌分别以2% (V/V)的接种量转接到含有25 mL MMYE+5 g/L木糖培养基的250 mL摇瓶中,37 ℃培养18 h后,添加葡萄糖至终浓度20 g/L,添加IPTG诱导基因表达,继续培养54 h。
1.6.2 混菌培养质粒pGAx3电击转化到E. coli K12 ΔxylB ΔglcD Δpts中,构建重组菌PGA24。质粒pET-pct- PCAB电击转化到E. coli K12 ΔxylB ΔglcD ΔxlyA中,构建重组菌PGA25。
从冻存管中取出菌种PGA24和PGA25,接种到LB培养基中37 ℃培养12 h作为种子液。以1% (V/V)的接种量一起转接到含有25 mL MMYE+ 5 g/L木糖+20 g/L葡萄糖培养基的250 mL摇瓶中,37 ℃培养12 h后,添加IPTG诱导基因表达,继续培养60 h。
1.7 分析方法摇瓶培养结束后,8 000 r/min离心10 min收集菌体,然后用去离子水洗涤、离心,去掉菌体表面粘附的培养基成分。将得到的细胞在抽真空条件下冰冻干燥至恒重,测定细胞干重(Cell dry weight,CDW)。
取50 mg左右干细胞于酯化管中,加入2 mL酯化液(含有2 g/L苯甲酸、3% (V/V)浓硫酸的色谱纯甲醇溶液)和2 mL氯仿,加盖密闭。100 ℃酯化反应4 h。冷却至室温后,加入1 mL去离子水,充分振荡混匀,静置分层。待氯仿相与水相完全分离后,取氯仿相进行气相色谱(Gas chromatography,GC)分析。使用HP 6890型气相色谱仪和HP-5毛细管柱,进样口温度200 ℃,检测器温度220 ℃。柱温从80 ℃开始,停留1.5 min;以30 ℃/min的速率升温到140 ℃,停留0 min;再以40 ℃/min的速率升温到220 ℃,停留0.5 min。聚合物标品采用同样的方法进行,利用内标法计算干细胞中聚合物的组成和含量。
2 结果与分析 2.1 构建P(GA-LA-3HB)合成途径以葡萄糖和木糖为联合碳源,设计乙醇酸、乳酸和3-羟基丁酸共聚酯P(GA-LA-3HB)的生物合成途径。如图 2所示,葡萄糖经糖酵解途径生成丙酮酸,丙酮酸在乳酸脱氢酶LdhA催化下生成乳酸,表达辅酶A转移酶Pct,使乳酸转化为乳酰CoA,作为乳酸聚合的前体。表达β-酮硫解酶PhaA和乙酰乙酰辅酶A还原酶PhaB,使乙酰辅酶A生成3-羟基丁酰辅酶A,作为3-羟基丁酸聚合的前体。木糖在木糖异构酶XylA、塔格糖-3-差向异构酶Dte、核酮糖激酶FucK、醛缩酶FucA和醛脱氢酶AldA的催化下生成乙醇酸,再表达辅酶A转移酶Pct,使乙醇酸转化为乙醇酰CoA,作为乙醇酸聚合的前体。最后,表达PHA聚合酶PhaC,催化乙醇酰辅酶A、乳酰辅酶A和3-羟基丁酰辅酶A的聚合反应,获得含有乙醇酸、乳酸和3-羟基丁酸3种单体组分的聚合物。

|
| 图 2 葡萄糖和木糖合成P(GA-LA-3HB)的代谢途径 Fig. 2 P(GA-LA-3HB) biosynthetic pathway from glucose and xylose |
| |
构建大肠杆菌PGA21,过表达dte、fucK、fucA、aldA、pct、phaA、phaB和phaC基因。按照1.6.1所述的方法进行摇瓶实验,检测重组菌的细胞生长和聚合物合成情况。结果表明,PGA21的细胞干重达2.58 g/L,含有10.44 wt%的P(21.75 mol% GA-14.21 mol% LA-3HB)。这说明设计的共聚酯合成途径能够在大肠杆菌中成功表达。重组菌首先利用木糖合成乙醇酸单体,接下来利用葡萄糖合成P(GA-LA-3HB)聚合物,但共聚酯的含量较低,仅为细胞干重的10%左右。
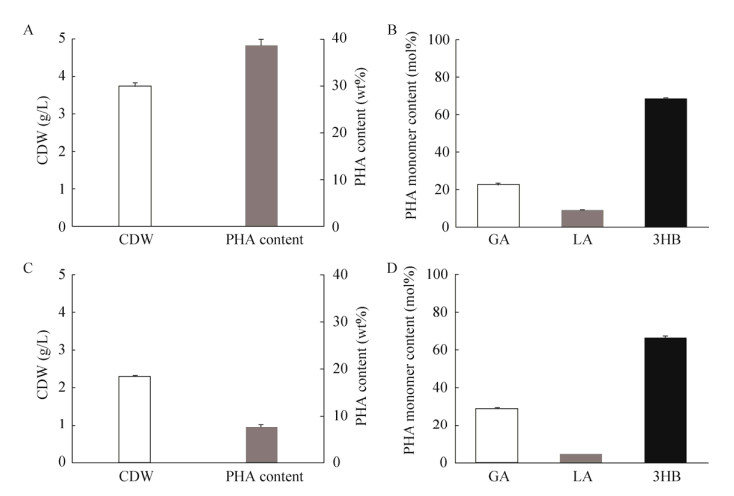
2.2 PHA颗粒结合蛋白表达对共聚酯合成的影响细胞内合成的聚羟基脂肪酸酯以不溶于水的颗粒形式存在,其表面除存在PHA聚合酶外,还有一类双亲性的PHA颗粒结合蛋白,能够阻止不同PHA颗粒之间的融合,调控细胞内PHA颗粒的大小和比表面积[21]。罗氏真养菌R. eutropha H16的PhaP1是研究最为清楚的PHA颗粒结合蛋白[22],其编码序列经密码子优化后,在大肠杆菌中异源表达。构建重组菌PGA22,研究PHA颗粒结合蛋白过表达对P(GA-LA-3HB)合成的影响。如图 3A和图 3B所示,PGA22的细胞干重达3.73 g/L,含有38.72 wt%的P(22.69 mol% GA-8.88 mol% LA-3HB)。过表达PhaP1明显提高了细胞干重和聚合物含量,与对照菌PGA21相比,PGA22中P(GA-LA-3HB)产量由0.27 g/L提高到1.44 g/L。de Almeida等[23]和Zhou等[24]分别报道了PHA颗粒结合蛋白对聚-3-羟基丁酸酯和聚4-羟基丁酸酯合成的促进作用,所使用的PhaP和聚合酶PhaC均来自于相同的菌株。本研究所使用的聚合酶PhaC来自于假单胞菌,这说明R. eutropha H16的PhaP1对异源聚合酶催化的PHA合成也有促进作用。
|
| 图 3 大肠杆菌PGA22和PGA23合成P(GA-LA-3HB)的摇瓶实验 Fig. 3 P(GA-LA-3HB) production by E. coli PGA22 or PGA23 grown in shake flasks. (A) CDW and PHA content of PGA22. (B) PHA monomer content of PGA22. (C) CDW and PHA content of PGA23. (D) PHA monomer content of PGA23 |
| |
乳酸脱氢酶基因ldhA在大肠杆菌中负责催化丙酮酸还原为乳酸。对ldhA基因进行敲除,研究其缺失对P(GA-LA-3HB)合成的影响。如图 3C和图 3D所示,与对照菌PGA22相比,ldhA基因敲除菌PGA23的细胞干重和聚合物含量都有明显降低,细胞干重从3.73 g/L下降到2.30 g/L,聚合物含量从38.72 wt%下降到7.49 wt%。虽然PGA23中的乳酸脱氢酶基因ldhA被敲除,但所得聚合物中仍然检测到少量乳酸单体。有研究表明,大肠杆菌细胞内存在甲基乙二醛循环,能以糖酵解中间体磷酸二羟丙酮为前体合成少量乳酸[25]。ldhA基因缺失导致了聚合物产量下降,提示乳酸单体的存在有利于P(GA-LA-3HB)的合成。
2.4 混菌共培养合成P(GA-LA-3HB)由于碳代谢物阻遏(Carbon catabolite repression,CCR)效应,大肠杆菌在含有葡萄糖和木糖的混合碳源中,首先利用葡萄糖进行生长,当葡萄糖耗尽后才能够代谢木糖[26-27]。因此,在单菌摇瓶培养中,首先向培养基中加入木糖,分解产生乙醇酸单体,再加入葡萄糖来合成聚合物。在木质纤维素水解液中,葡萄糖和木糖是同时存在的。为了达到两者的同时利用,采用了混菌共培养的方法。
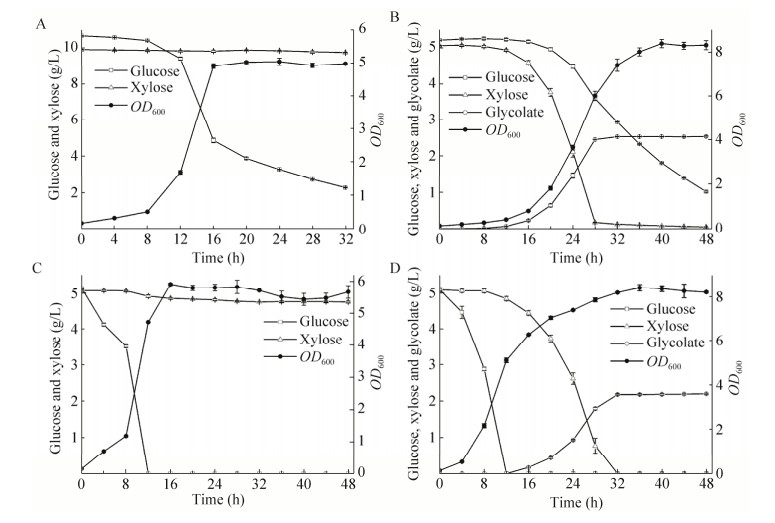
构建磷酸烯醇式丙酮酸-糖磷酸转移酶系统基因pts缺失的突变体K12 ΔxylB ΔglcD Δpts,该突变体在含有葡萄糖和木糖混合碳源的基本培养基中,只能利用葡萄糖进行生长,但葡萄糖代谢速度变慢(图 4A)。转入木糖合成乙醇酸相关基因dte、fucK、fucA、aldA,所得重组菌PGA24在葡萄糖和木糖同时存在的条件下,首先利用木糖生产乙醇酸,随后开始代谢葡萄糖(图 4B)。构建木糖异构酶基因xylA缺失的突变体K12 ΔxylB ΔglcD ΔxylA,该突变体在含有葡萄糖和木糖混合碳源的基本培养基中,只能利用葡萄糖进行生长(图 4C)。转入聚合物合成基因pct、phaA、phaB、phaC,构建重组菌PGA25。在葡萄糖和木糖混合物中,PGA24首先利用木糖,PGA25只能利用葡萄糖,将PGA24和PGA25混合共培养,两种碳源可以同时被消耗(图 4D)。
|
| 图 4 构建同时利用葡萄糖和木糖的混菌培养体系 Fig. 4 The construction of a coculture system to simultaneously utilize glucose and xylose (A) K12 ΔxylB ΔglcD Δpts. (B) PGA24. (C) K12 ΔxylB ΔglcD ΔxylA. (D) PGA24 and PGA25 coculture |
| |
进行混菌共培养合成P(GA-LA-3HB)的摇瓶实验,将PGA24和PGA25混合共培养在含有葡萄糖和木糖的MMYE培养基中,获得了4.01 g/L的细胞干重,含有21.54 wt%的P(2.81 mol% GA-2.82 mol% LA-3HB) (图 5A, 5B)。这说明,混菌共培养能够在含有葡萄糖和木糖混合物的培养基中获得共聚酯,但聚合物中乙醇酸和乳酸单体的比例较低,3-羟基丁酸单体的比例超过90 mol%。后续研究中,进一步调控PGA24和PGA25的接种比例,有可能获得单体含量多样的P(GA-LA-3HB)共聚酯材料。

|
| 图 5 大肠杆菌混菌共培养转化葡萄糖和木糖混合物合成P(GA-LA-3HB) Fig. 5 Conversion of glucose-xylose mixture to P(GA-LA-3HB) using an E. coli-E. coli coculture strategy. (A) CDW and PHA content. (B) PHA monomer content |
| |
本研究通过在大肠杆菌中表达塔格糖-3-差向异构酶、核酮糖激酶、醛缩酶、醛脱氢酶、丙酰辅酶A转移酶、β-酮硫解酶、乙酰乙酰辅酶A还原酶和聚合酶,成功构建了葡萄糖和木糖为碳源合成乙醇酸、乳酸和3-羟基丁酸共聚酯的生物合成途径。在此基础上,表达聚羟基脂肪酸酯颗粒结合蛋白,明显提高了聚合物的合成。
对大肠杆菌的糖代谢途径进行改造,获得在葡萄糖和木糖混合物中仅能利用葡萄糖或者木糖的突变体。将两种突变体混合培养,实现利用葡萄糖和木糖混合物合成聚合物,为将来纤维素水解物的有效利用提供了参考。
| [1] | Wang Y, Yin J, Chen GQ. Polyhydroxyalkanoates, challenges and opportunities. Curr Opin Biotechnol, 2014, 30: 59–65. DOI: 10.1016/j.copbio.2014.06.001 |
| [2] | Lenz RW, Marchessault RH. Bacterial polyesters: Biosynthesis, biodegradable plastics and biotechnology. Biomacromolecules, 2005, 6(1): 1–8. DOI: 10.1021/bm049700c |
| [3] | Chen GQ, Jiang XR. Engineering bacteria for enhanced polyhydroxyalkanoates (PHA) biosynthesis. Synth Syst Biotechnol, 2017, 2(3): 192–197. DOI: 10.1016/j.synbio.2017.09.001 |
| [4] | Chen GQ, Hajnal I, Wu H, et al. Engineering biosynthesis mechanisms for diversifying polyhydroxyalkanoates. Trends Biotechnol, 2015, 33(10): 565–574. DOI: 10.1016/j.tibtech.2015.07.007 |
| [5] | Becker J, Lange A, Fabarius J, et al. Top value platform chemicals: bio-based production of organic acids. Curr Opin Biotechnol, 2015, 36: 168–175. DOI: 10.1016/j.copbio.2015.08.022 |
| [6] | Saha BC. Hemicellulose bioconversion. J Ind Microbiol Biotechnol, 2003, 30(5): 279–291. DOI: 10.1007/s10295-003-0049-x |
| [7] |
Liu WX, Fu J, Zhang B, et al. Engineering of the xylose metabolic pathway for microbial production of bio-based chemicals.
Chin J Biotech, 2013, 29(8): 1161–1172.
(in Chinese). 刘维喜, 付晶, 章博, 等. 微生物木糖代谢途径改造制备生物基化学品. 生物工程学报, 2013, 29(8): 1161-1172. |
| [8] | Wang J, Shen XL, Lin YH, et al. Investigation of the synergetic effect of xylose metabolic pathways on the production of glutaric acid. ACS Synth Biol, 2018, 7(1): 24–29. |
| [9] | Jeffries TW. Engineering yeasts for xylose metabolism. Curr Opin Biotechnol, 2006, 17(3): 320–326. DOI: 10.1016/j.copbio.2006.05.008 |
| [10] | Zhang M, Eddy C, Deanda K, et al. Metabolic engineering of a pentose metabolism pathway in ethanologenic Zymomonas mobilis. Science, 1995, 267(5195): 240–243. DOI: 10.1126/science.267.5195.240 |
| [11] | Wen M, Bond-Watts BB, Chang MCY. Production of advanced biofuels in engineered E. coli. Curr Opin Chem Biol, 2013, 17(3): 472–479. |
| [12] | Peralta-Yahya PP, Zhang FZ, del Cardayre SB, et al. Microbial engineering for the production of advanced biofuels. Nature, 2012, 488(7411): 320–328. DOI: 10.1038/nature11478 |
| [13] | Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1, 4-butanediol. Nat Chem Biol, 2011, 7(7): 445–452. DOI: 10.1038/nchembio.580 |
| [14] | Choi YJ, Lee SY. Microbial production of short-chain alkanes. Nature, 2013, 502(7472): 571–574. DOI: 10.1038/nature12536 |
| [15] | Tan ZG, Chen J, Zhang XL. Systematic engineering of pentose phosphate pathway improves Escherichia coli succinate production. Biotechnol Biofuels, 2016, 9: 262. DOI: 10.1186/s13068-016-0675-y |
| [16] | Shiue E, Prather KLJ. Improving D-glucaric acid production from myo-inositol in E. coli by increasing MIOX stability and myo-inositol transport. Metab Eng, 2014, 22: 22–31. DOI: 10.1016/j.ymben.2013.12.002 |
| [17] | Xu P, Gu Q, Wang WY, et al. Modular optimization of multi-gene pathways for fatty acids production in E. coli. Nat Commun, 2013, 4: 1409. DOI: 10.1038/ncomms2425 |
| [18] | Li ZJ, Qiao KJ, Shi WC, et al. Biosynthesis of poly(glycolate-co-lactate-co-3-hydroxybutyrate) from glucose by metabolically engineered Escherichia coli. Metab Eng, 2016, 35: 1–8. DOI: 10.1016/j.ymben.2016.01.004 |
| [19] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci USA, 2000, 97(12): 6640–6645. DOI: 10.1073/pnas.120163297 |
| [20] | Pereira B, Li ZJ, de Mey M, et al. Efficient utilization of pentoses for bioproduction of the renewable two-carbon compounds ethylene glycol and glycolate. Metab Eng, 2016, 34: 80–87. DOI: 10.1016/j.ymben.2015.12.004 |
| [21] | Pötter M, Steinbüchel A. Poly(3-hydroxybutyrate) granule-associated proteins: impacts on poly(3-hydroxybutyrate) synthesis and degradation. Biomacromolecules, 2005, 6(2): 552–560. DOI: 10.1021/bm049401n |
| [22] | Pötter M, Müller H, Steinbüchel A. Influence of homologous phasins (PhaP) on PHA accumulation and regulation of their expression by the transcriptional repressor PhaR in Ralstonia eutropha H16. Microbiology, 2005, 151(3): 825–833. DOI: 10.1099/mic.0.27613-0 |
| [23] | de Almeida A, Nikel PI, Giordano AM, et al. Effects of granule-associated protein PhaP on glycerol-dependent growth and polymer production in poly(3-hydroxybutyrate)-producing Escherichia coli. Appl Environ Microbiol, 2007, 73(24): 7912–7916. DOI: 10.1128/AEM.01900-07 |
| [24] | Zhou XY, Yuan XX, Shi ZY, et al. Hyperproduction of poly(4-hydroxybutyrate) from glucose by recombinant Escherichia coli. Microb Cell Fact, 2012, 11: 54. DOI: 10.1186/1475-2859-11-54 |
| [25] | Grabar TB, Zhou S, Shanmugam KT, et al. Methylglyoxal bypass identified as source of chiral contamination in L(+) and D(-)-lactate fermentations by recombinant Escherichia coli. Biotechnol Lett, 2006, 28(19): 1527–1535. DOI: 10.1007/s10529-006-9122-7 |
| [26] | Kim Y, Ingram LO, Shanmugam KT. Construction of an Escherichia coli K-12 mutant for homoethanologenic fermentation of glucose or xylose without foreign genes. Appl Environ Microbiol, 2007, 73(6): 1766–1771. DOI: 10.1128/AEM.02456-06 |
| [27] | Deutscher J. The mechanisms of carbon catabolite repression in bacteria. Curr Opin Microbiol, 2008, 11(2): 87–93. DOI: 10.1016/j.mib.2008.02.007 |
 2019, Vol. 35
2019, Vol. 35




