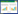中国科学院微生物研究所、中国微生物学会主办
文章信息
- 许楹, 殷超凡, 岳纹龙, 周宁一
- Xu Ying, Yin Chaofan, Yue Wenlong, Zhou Ning-Yi
- 石油基塑料的微生物降解
- Microbial degradation of petroleum-based plastics
- 生物工程学报, 2019, 35(11): 2092-2103
- Chinese Journal of Biotechnology, 2019, 35(11): 2092-2103
- 10.13345/j.cjb.190301
-
文章历史
- Received: July 7, 2019
- Accepted: September 6, 2019
塑料是一种以聚合物为主要成分的材料[1],可分为天然塑料和合成塑料两类。天然塑料是由天然的或微生物分泌的大分子物质组成的一类塑料,如多羟基丁酸(PHB)。这类塑料多具有可生物降解的特性,因而是一类环境友好型材料,但这类天然塑料在现实应用中仅占很小的一部分。合成塑料是由单体原料通过加聚或缩聚而成的高分子材料,其种类繁多、广泛应用于生产生活,包括聚苯乙烯(Polystyrene, PS)、聚乙烯(Polyethylene, PE)、聚丙烯(Polypropylene, PP)、聚对苯二甲酸乙二醇酯(Polyethylene terephthalate, PET)、聚氨酯(Polyurethane, PUR)、聚氯乙烯(Polyvinyl chloride, PVC)等石油基塑料(图 1)。随着塑料产业的发展及塑料制品的广泛使用和消耗,越来越多的废旧石油基塑料制品进入到了环境中。合成塑料作为一种非天然的石油基塑料,由于其分子量过大且疏水而难以通过生物膜,因此很难被微生物所利用;同时,塑料单体间往往通过C-C键等非常强的共价键结合,因此要破坏这种高分子聚合链非常困难。因此这些高分子聚合物进入环境后很难被微生物降解,带来了严重的环境污染,“白色污染”已经成为一个全球性问题。常规处理塑料废弃物的物理和化学的方法有具有成本高、副产品多和产生二次污染的局限性。

|
| 图 1 常见的石油基塑料 Fig. 1 Common petroleum-based plastics |
| |
生物处理技术是通过生物代谢过程将废物转化成二氧化碳、水和生物质等最终产物进入地球化学循环中。生物处理技术一般都具有高效降解和成本经济的特点,因此被认为是处理固体有机废物最具持续性的处理方式[2]。微生物作为自然界天然的分解者,生存环境丰富多样,它们具有非常高的进化能力和非常强的适应能力,天然存在的化合物几乎都可以被微生物完全利用和降解,而人工合成的化合物也会随着时间推移被微生物逐渐作为底物利用。通过微生物手段降解塑料不会造成污染,且不需要消耗大量的能量,是一种安全生态的降解方法。另一方面微生物具有可遗传改造性,微生物基因组较小,且有大量的适用于微生物的分子操作手段和工具可以被利用,这一方面有利于对代谢机制的研究,也有利于通过遗传改造提高微生物降解效率或改造代谢路径,达到实际生产应用需求。
1 微生物降解石油基塑料石油基塑料随着在环境中大量积累,与微生物之间进行广泛的接触,随着时间推移,微生物对难降解塑料化合物进行充分的适应。但是往往由于人工合成的塑料聚合物太大而不能通过细胞膜,它们必须首先解聚成较小的单体,然后在微生物细胞内被吸收和生物降解。目前已有报道揭示了微生物进化出完全矿化一些人工合成塑料的能力[3]。可见通过微生物降解塑料具有可行性。通过紫外线照射、高温、机械作用以及塑料添加剂如增塑剂等的破坏可导致塑料裂解并最终分解成微塑料。表面积的相对增加改善其生物可利用度。用微生物方法去处理含微塑料的环境变得具有很大的可行性。生物降解相较于物理化学降解来说,能够完全利用塑料作为碳源并将其彻底降解,但是微生物在塑料表面定殖困难,目前高效降解塑料的微生物还未发现,绝大部分微生物倾向于利用塑料表面光化学氧化产生的活性基团。
1.1 聚烯烃塑料的生物降解研究进展聚烯烃塑料是指聚乙烯(PE)、聚丙烯(PP)和聚苯乙烯(PS)等由烯烃类单体聚合而成的塑料。其消费量占到了众多种类的合成塑料总量的50%,是塑料废物的首要贡献者。导致聚烯烃难以生物降解的材料特性主要包括[4-5]:1)稳定的化学结构,主要由-C-C-和-C-H-共价键构成,没有易被氧化和水解的基团;2)大分子量长链结构形成的空间位阻,使其不能直接进入微生物细胞内进而被胞内酶降解;3)高度疏水性,细胞和酶难以接触发生反应。
1.1.1 聚苯乙烯塑料(PS)生物降解研究进展在聚苯乙烯塑料(PS)生物降解过程的研究历程中,国内外研究人员曾尝试通过提出假设以揭示PS降解过程的一系列研究。研究发现,对于微生物的攻击,PS产品具有极强的耐性和顽固性。对PS的生物降解研究始于20世纪70年代。1974年,为了准确定量测定土壤微生物降解聚苯乙烯的速率,加拿大多伦多大学化学系的Guillet等首次用α-和β-14C标记的苯乙烯单体合成了14C标记的聚苯乙烯单体(85%)和非标记的乙烯基甲酮(5%)实验合成共聚物的生物降解进行了研究[6]。Kaplan等研究的无菌培养真菌和混合微生物菌群对PS的生物降解也是非常缓慢[7]。Sielicki等通过测量释放的14CO2来研究[β-14C]PS在土壤和液体富集培养基中的生物降解,培养4个月后仅有1.5%–3.0%的PS被降解[8]。尽管此实验中PS去除有限,但却比Kaplan等研究报道[7]的结果高出15–30倍。Griffin将天然淀粉通过硅化、干燥等处理作为添加剂加入到PS中后,发现PS降解速率明显快于单独PS塑料培养[9]。
此后从1980–2000年的20年间,极少有关于PS生物降解研究的报道。直到2003年,东京Organo株式会社和东北大学的Eisaku首次报道了关于分离高效降解PS微生物的文章。文中采用重量损失法测试了从土壤中分离的5株细菌对苯乙烯单体和PS的降解能力,其中有3株菌黄单孢菌属Xanthomonas sp.和鞘氨醇杆菌属Sphingobacterium sp.能降解聚苯乙烯,芽孢杆菌属Bacillus sp. STR-Y-O可同时降解苯乙烯单体和PS[10]。然而从被埋藏于土壤8个月后的PS膜中分离出类芽孢杆菌属Paenibacillus urinalis NA26、绿脓假单胞菌Pseudomonas aeruginosa NB26和芽孢杆菌属Bacillus sp. NB6利用EPS作为唯一的碳源时,其FTIR光谱分析未观察到显著的表面变化,其降解能力尚未被验证[11]。2017年,南京大学季荣教授团队利用青霉菌属Penicillium variabile CCF3219有效地降解了14C标记的PS薄膜,并发现臭氧氧化预处理PS聚合物可有效强化其被真菌降解效率[12]。如上所述,先前关于PS生物降解的研究尽管降解能力有限,但已显示出令人鼓舞的研究前景。对于环境中是否存在微生物在其生存条件下能够快速降解PS还有待进一步研究。
近年来研究发现黄粉虫具有啮食和降解塑料的行为。2011年,沈叶红等对黄粉虫降解塑料的机制进行了初步探索,并从黄粉虫肠道中分离出8株具有降解特性的细菌[13]。2015年北京航空航天大学杨军教授团队对黄粉虫幼虫肠道中PS的生物降解和矿化进行相关研究,结果表明摄入的PS或EPS在黄粉虫肠中的保留时间小于24 h,且PS在经过黄粉虫肠道消化后发生了解聚;而在用庆大霉素喂养10 d后,黄粉虫肠道细菌活性减弱,进而发现PS解聚和矿化量减小,这表明黄粉虫对PS的降解可能依赖于其肠道微生物作用[28];并在黄粉虫肠道中分离出一株PS降解菌微杆菌属Exiguobacterium sp. YT2,将其在含有PS的液体培养基中单独培养,在60 d的培养周期中,PS降解率达到7.4%±0.4%。通过凝胶渗透色谱(Gel permeation chromatography, GPC)对残余物分析也证实了菌株YT2对PS的降解作用[14-15]。通过分析高通量测序的啮食PS泡沫塑料黄粉虫的肠道菌群结构,指出在黄粉虫生物降解塑料的过程中肠道细菌可能起主导作用[16]。近年来研究发现的昆虫具有特异的PS塑料降解能力,为今后PS塑料高效降解提供了一个崭新的思路。
不足之处及尚未解决的问题:迄今为止,尽管已经证实了PS可被微生物降解,并发现了PS降解微生物或生物酶,但是目前针对PS生物降解的研究仍存在以下问题:1)目前发现的PS生物降解效率均非常缓慢,还应进一步探索优化PS生物降解的手段,深入研究高效PS生物降解途径。2)实验证实有些细菌及真菌可以对PS进行降解作用,但是目前已发现的具备相关能力的菌株种类较少,因此寻找有效的生物降解PS微生物和酶系统、丰富PS降解菌株资源库方面工作亟待研究[17]。
1.1.2 聚乙烯塑料(PE)的生物降解研究进展关于微生物降解聚乙烯(PE)塑料污染物并将其无机矿化成H2O和CO2的研究最早可追溯到20世纪70年代。然而前期研究结果表明,高密度聚乙烯(HDPE)和低密度聚乙烯(LDPE)塑料降解的微生物降解进程及效率都极为缓慢[18],并主要分解其短链低聚物部分,对长链部分的水解比较有限,但具体可被土壤微生物降解的PE分子量上限以及分子量与降解速率之间的关系并不了解[19]。近年来,研究人员在从土壤中分离PE降解菌方面有了较为积极的成果[20]。2013年,印度Tribedi等从土壤中富集分离得到一种可降解LDPE的假单胞菌属细菌AKS2在45 d内达到4%–6%的PE降解效率[21],而在农田废弃地膜中富集分离出了红球菌C208可以每周0.86%的速率降解PE塑料薄膜[22-23],且研究表明C208所分泌的胞外漆酶在PE的生物降解中起到促进氧化的主要作用;当在降解过程中添加铜时,PE的生物降解增强75%[24]。2009年,韩秋霞等[25]从农田土壤、生活垃圾堆肥和污泥中分离到10株对改性PE具有潜在降解能力的霉菌,其中发现只有一株真菌可以以改性PE膜为唯一碳源,培养30 d PE膜重量损失可达20%以上,从而证实了改性PE膜的可生物降解性。之后,研究人员从垃圾堆埋场土壤中筛选分离的1株解淀粉芽孢杆菌Bacillus amyloliquefaciens菌株被证明具有高效降解淀粉填充PE的能力,且淀粉填充改性PE塑料可能强化PE塑料的降解效果[26]。近年来国内外研究人员也开展了一些海洋PE降解微生物筛选的研究工作。2008年,印度Doble等[27]从海洋浅水区分离到4种海洋细菌:球形芽孢杆菌Bacillu sphericus、蜡样芽孢杆菌Bacillus cereus、假单胞菌属Pseudomonas sp.和节杆菌属Arthrobacter sp.,且得到了较高的PE塑料失重率。2013年,印度Jha等[28]从阿拉伯海海水中分离出60种海洋细菌,以PE为唯一碳源,筛选得到可降解LDPE的3种细菌:沼泽考克氏菌M16、短小芽孢杆菌M27和枯草芽孢杆菌H1584,培养30 d后对LDPE的降解率分别达到1%、1.5%和1.75%。研究人员从印度马纳尔湾的塑料废物堆放场中分离出15种HDPE降解细菌(GMB1-GMB15),其中GMB5和GMB7在培养30 d后对PE膜的降解率分别达到12%和15%,且FTIR结果显示羰基键指数和乙烯基指数均增加[29]。2019年,希腊Kalogerakis团队成功利用人工构建的海洋微生物菌群实现了对PE和PS两种混合塑料的共降解[30]。同样,尽管部分真菌也被分离并研究证明其降解PE的能力[31],但是其降解能力均非常有限。
近年来,昆虫啮食并降解PE塑料的研究也有一些研究报道。2010年,苗少娟等在大麦虫的养殖过程中发现其同样具有取食塑料的能力,进而研究了大麦虫幼虫对不同塑料制品的喜食情况及降解作用[32-33]。2014年,北京航空航天大学杨军教授团队发现印度谷螟幼虫(Plodia interpunctella Hübner)可以咀嚼并进食PE塑料薄膜,进而从其肠道内分离出2株具有PE降解能力的细菌:阿氏肠杆菌YT1和芽孢杆菌YP1。研究表明YT1和YP1的悬浮培养物在60 d内对PE膜的降解率分别达到6.1%±0.3%和10.7%±0.2%[34]。2017年,张可等分别以塑料薄膜A (30%淀粉、70%的PE和助剂)和B (95%以上PE和少量助剂)为唯一食物饲养黄粉虫,黄粉虫对两种塑料薄膜均能实现完全降解[35]。上述研究表明,昆虫的肠道内存在PE降解细菌,为PE的生物降解提供了一种新途径。
不足之处及尚未解决的问题包括:PE类塑料制品相对分子质量大、表面疏水性强且可被微生物酶系统利用的官能团少,因而在自然环境中难以矿化[36]。目前对于PE微生物降解的具体机制尚无定论,尤其是从酶和基因水平上阐述PE塑料降解生物机制尚为空白,针对PE的微生物降解机制仍需深入探索。总体来说,PE高效生物降解有关报道仍然十分稀少,但从现有成果分析,PE生物降解具备的良好应用前景及环境优势已初现端倪。
1.1.3 聚丙烯(Polypropylene, PP)塑料聚丙烯(Polypropylene, PP)塑料是除聚乙烯(Polyethylene, PE)塑料之外生产和应用最广泛的一类塑料。其化学结构与聚乙烯塑料类似,是碳碳骨架塑料的典型代表,在化学结构上与聚乙烯塑料的不同之处是聚丙烯塑料侧链有甲基。聚丙烯塑料与其他碳碳骨架塑料类似,由于具有太高的分子量、太强的疏水性、太高的化学键能和太低的生物可及性,这些特性导致这类塑料在环境中很难被微生物降解。目前关于聚丙烯塑料生物降解的报道非常少。Cacciari等较早探究了微生物菌群对聚丙烯塑料的降解能力[37],其报道指出通过微生物菌群处理约6个月后,聚丙烯塑料重量有所下降,且有机试剂萃取得到的小分子产物有所上升,但微生物菌群处理后的降解效率非常低下。Arkatkar等发现对于未处理的聚丙烯塑料,在土壤混合培养物处理长达一年后,仅有0.4%的重量损失,而对于经过热处理的聚丙烯塑料在相同处理条件下,其重量损失提高到10%,这揭示了预处理对于微生物降解聚丙烯塑料的重要性[38]。从土壤中分离得到的微生物菌株对于经过紫外辐照后的聚丙烯塑料显示出一定的降解能力,而对于未经预处理的聚丙烯塑料没有降解能力[39]。这一方面说明获得的微生物降解效率低下,另一方面说明非生物因素的处理在提高聚丙烯塑料的生物可及性中发挥着重要功能。Jeyakumar等探究了不同预处理手段及混合淀粉等可降解材料对真菌降解聚丙烯塑料的辅助效果[40],结果发现紫外线处理、金属离子氧化剂处理或混合淀粉等可降解材料均能在一定程度上提高真菌对聚丙烯塑料的降解能力,而没有预处理的聚丙烯很难被真菌利用。
不足之处及尚未解决的问题包括:目前对于聚丙烯塑料微生物降解的研究还处在代谢菌株资源分离阶段,可研究和应用的聚丙烯塑料降解菌株严重缺乏。高效且高特异性的降解菌株未见报道;微生物代谢机理未知。
1.2 聚氨酯塑料的生物降解研究进展聚氨酯(Polyurethane, PUR),全称为聚氨基甲酸酯,是在大分子主链中含有氨基甲酸酯基的聚合物。合成聚氨酯的主要原料是有机多元异氰酸酯和端羟基化合物。多异氰酸酯基团作为硬段链构成结晶部分的程度是影响聚合物PUR的生物降解的重要因素[41]。PUR塑料可以通过微生物脲酶、酯酶和蛋白酶解聚,水解塑料的酯键和脲键[42]。Howard等从荧光假单胞菌Pseudomonas fluorescens中纯化的29 kDa胞外蛋白酶可以水解PUR[43]。Nomura等从丛毛单胞菌Comamonas acidovorans TB-35克隆到一个基因pudA,它编码的62 kDa酯酶可以水解PUR中的酯键[44-45]。Stern等从绿针假单胞菌Pseudomonas chlororaphis纯化出分子量为65 kDa和60 kDa的脂肪酶,分别由pueA和pueB基因编码[46-47]。Ruiz等从假单胞菌属Pseudomonas uorescens克隆到pulA基因,编码48 kDa的胞外水解PUR塑料的酯酶[48]。同时,在真菌中也纯化出降解PUR塑料的酶。Crabbe等从弯孢属真菌Curvularia senegalensis中纯化28 kDa具有酯酶特性的胞外水解酶[49]。Russel等从多毛孢属真菌Pestalotiopsis microspora E2712A中纯化21 kDa丝氨酸水解酶负责PUR的降解[50]。在酶水平,聚氨酯生物降解涉及两类主要的酶,属于酯酶和蛋白酶家族,大小范围从21 kDa到65 kDa。
从环境中分离一些PUR塑料降解的纯培养的微生物,通过添加到PUR塑料污染的场地,如何进一步提高PUR塑料的生物降解速率是一个实际的难题。Filip等将PUR塑料在垃圾填埋垃圾中孵育3个月后,大多数聚氨酯的测定均未发现明显的重量损失,这些初步研究表明聚氨酯对非生物和生物条件具有高水平的抗性[51]。在影响聚氨酯生物降解的非生物因素中,氧的存在似乎是至关重要的。Demirtas等发现在厌氧条件下,机械强度、重量损失、化学结构和微生物生长只会发生微小的变化;该观察结果表明,当限于厌氧状态时,微生物几乎不使用聚氨酯作为碳源或氮源;即使深埋在土壤中,聚氨酯泡沫的微生物降解也很少或不存在,这突显了这种聚合物对生物降解的顽固性[52]。PUR塑料降解是由多种风化因素引起的,包括热、阳光、水分、氧气和温度变化、微生物、放射性和化学污染物等。如何通过生物强化,生物刺激等条件的优化,进一步解决PUR塑料污染场地的修复,变得至关重要。
不足之处及尚未解决的问题包括:1) PUR塑料降解微生物资源匮乏。当前分离到的降解PUR塑料的微生物只能分解软段聚酯多元醇分子的酯键,而严重缺乏分解硬段链氨基甲酸酯重复结构单元的酯键或脲键的微生物。2) PUR塑料的代谢通路还不清晰。关于PUR降解酶的研究,仅有降解过程中第一步的长链软段分子的解聚酶的相关研究报道,尚无对解聚后的降解产物(如单体和寡聚物)的进一步代谢相关的酶的研究报道。
1.3 聚对苯二甲酸乙二醇塑料 (PET) 的生物降解研究进展聚对苯二甲酸乙二醇(Polyethylene terephthalate, PET)塑料由对苯二甲酸和乙二醇通过酯键连接[53],这类塑料广泛用作饮料瓶等包装材料,其年产量多达数千万吨。PET塑料之所以难以降解主要是由于其骨架上重复存在的芳香化合物对苯二甲酸[54-55]。PET作为一种半结晶化合物,在生产实践中,常根据生产需求生产不同结晶度的产品,例如,PET饮料瓶结晶度高达30%[56],而PET纤维结晶度可以高达40%[57]。PET材料的结晶部分较非结晶部分更难被微生物及酶降解,因此,通常认为,低结晶度PET较高结晶度PET具有更高的生物可降解性。
PET塑料的玻璃化转化温度(Tg)大约可以达到65 ℃[58],在玻璃化转化温度下,PET塑料的非晶态部分具有更高的柔性,因而更易被酶降解[59-60],但是,要在玻璃化转化温度下工作也意味着酶需要较强的耐热能力[60-63]。最近,日本科研工作者报道了在常温下可以水解非晶态PET酶[3],但此酶的效率并不是特别高。前人报道了一些来自真菌和放线菌的脂肪酶、酯酶[60, 64-66]或角质酶[60, 67-73]具有水解非晶态PET及修饰PET膜表面的功能。来自芽孢杆菌等的羧酸酯酶也能部分水解PET材料[74]。脂肪酶由于在结构上存在“眼睑”结构遮盖了疏水性活性中心,因而对PET表现出较低的催化能力[64, 75],相反,角质酶因为不存在这一“眼睑”结构,与底物空间结构上更容易靠近,因而表现出较高的催化活性[60-61, 76]。这类酶相对于其他酶而言,它们的活性中心在结构上更靠近表面。结构上,PET酶每次识别和结合4个对苯二甲酸单羟基乙酯(Monohydroxyethyl terephthalate, MHET)单元[77-78],其酶活性中心附近的裂隙影响着底物与酶的结合,研究人员通过对比PET酶及角质酶,将PET酶活性中心附近裂隙的2个残基突变为角质酶的残基,从而缩小了PET酶的裂隙大小,进而提升了PET酶的催化效率[79]。
来自嗜热真菌Thermomyces insolens的热稳定角质酶HiC可以在高温下(70 ℃)完全水解7%结晶度的PET[60],这意味着晶态部分也可以被水解。来自细菌的热稳性角质酶也可以在高温下水解低结晶度PET[61]。二价金属离子能够促进聚酯水解酶的热稳定性,并进而提高其在塑料玻璃化温度下的反应活性[61-62, 76, 80],通过盐桥或二硫键也能够提高这类酶的热稳定性[62, 80]。除此之外,磷酸阴离子也具有促进这类酶的稳定性的能力[71]。
PET塑料的强疏水性是限制其与水解酶接触的制约因素所在[81],对于天然高分子材料如多羟基脂肪酸[82]、纤维素[83]等,其对应的水解酶往往存在与底物相互结合的部分[69],而PET水解酶一般认为其通过围绕着活性中心的疏水部位与底物相互作用[70],通过定点突变改造PET水解酶的疏水部位,或者耦联其他疏水蛋白或两亲性分子[83-84],可以提高这类水解酶的水解效率。
不足之处及尚未解决的问题包括:1)尽管一些聚酯水解酶能够水解非晶态PET,但是对于晶态部分的降解效率非常低下[60, 64, 76],这也就意味着它们无法完全降解如饮料瓶等这种高结晶度的产品[56-57]。2)从PET塑料到高附加值产品高效转化的代谢路径还未构建。
1.4 PVC的生物降解研究进展聚氯乙烯(Polyvinyl chloride, PVC)作为另一种C-C骨架塑料,从结构上来看,它的特殊之处在于每隔一个C原子有一个氯原子,这个杂原子的引入很有可能进一步增加了其降解的难度。在生产实践中,聚氯乙烯塑料常作为管材使用,同时也经常添加一些塑料添加剂对其性能进行改变以应用于各个方面。Otake等通过长达32年之久的土地包埋实验也未发现PVC的降解[85]。Ali等通过土壤包埋或真菌处理均未发现PVC的明显降解现象[86]。通过微生物菌群处理PVC可能比单一菌株处理效果显著[87]。据我们所知,关于PVC的降解酶目前未见报道。
不足之处及尚未解决的问题包括:1)有效的PVC降解菌株资源未见报道;2) PVC降解酶未见报道。
2 讨论与展望迄今为止,尽管已经证实了多种塑料可被微生物降解,并发现了降解塑料的微生物或生物酶,但是目前针对塑料的微生物降解的研究仍存在以下问题:1)目前发现的塑料的微生物降解效率均非常缓慢,还应进一步探索优化微生物降解的手段,深入研究高效塑料的微生物降解途径。2)目前已发现的具备相关能力的菌株种类较少,因此寻找有效的生物降解塑料的微生物和酶系统,丰富降解菌株资源库方面工作亟待研究。
因此筛选多种塑料的降解菌和菌群,并对其降解特性、功能基因及关键酶进行遗传、物理、化学、分子和生化水平的研究,为储备塑料降解的微生物资源以及和塑料降解酶的资源,同时为构建高效降解微生物提供理论依据和可行性,也将作为酶改造工程的基础,为塑料制品的物质循环利用提供一定的理论基础。另外,利用传统的物理化学诱变手段来筛选具有特殊功能的塑料降解菌也是一个研究方向。
针对目前日益严重的“白色污染”,我国也开始资助微生物降解塑料的研究。国家自然科学基金委员会与欧盟委员会于2019年共同资助了中欧科学家在“Microorganism communities for plastics biodegradation”领域开展实质性合作研究项目。山东大学祁庆生教授(合成塑料降解转化微生物菌群)与南京工业大学姜岷教授(废塑料资源高效生物降解转化的关键科学问题与技术)的项目获得资助(http://bic.nsfc.gov.cn/Show. aspx?AI=1132)。同时,科技部2019年6月又在国家重点研发计划“变革性技术关键科学问题”中发布了“合成塑料降解酶的定向进化工程及应用”的项目指南,目前正在评审中。相信这些高强度资助项目的实施一定将使我国在基于微生物及其酶学的降解塑料研究方面得到令人瞩目的进展。
| [1] |
Quecholac-Piña X, García-Rivera MA, Espinosa-Valdemar RM, et al. Biodegradation of compostable and oxodegradable plastic films by backyard composting and bioaugmentation. Environ Sci Pollut Res, 2016, 24(33): 25725-25730. |
| [2] |
Lens PNL, Hamelers BVM, Hoitink H, et al. Resource recovery and reuse in organic solid waste management. IWA Publishing; Revised edition, 2007.
|
| [3] |
Yoshida S, Hiraga K, Takehana T, et al. A bacterium that degrades and assimilates poly (ethylene terephthalate). Science, 2016, 351(6278): 1196-1199. DOI:10.1126/science.aad6359 |
| [4] |
Gu JD. Microbiological deterioration and degradation of synthetic polymeric materials: recent research advances. Int Biodeter Biodegr, 2003, 52(2): 69-91. DOI:10.1016/S0964-8305(02)00177-4 |
| [5] |
Yang J, Song YL, Qin XY. Biodegradation of polyethylene. Environ Sci, 2007, 28(5): 1165-1168 (in Chinese). 杨军, 宋怡玲, 秦小燕. 聚乙烯塑料的生物降解研究. 环境科学, 2007, 28(5): 1165-1168. DOI:10.3321/j.issn:0250-3301.2007.05.041 |
| [6] |
Guillet JE, Regulski TW, Mcaneney TB. Biodegradability of photodegraded polymers. Ⅱ. Tracer studies of biooxidation of Ecolyte PS polystyrene. Environ Sci Technol, 2002, 8(10): 923-935. |
| [7] |
Kaplan DL, Hartenstein R, Sutter J. Biodegradation of polystyrene, poly (metnyl methacrylate), and phenol formaldehyde. Appl Environ Microbiol, 1979, 38(3): 551-553. |
| [8] |
Sielicki M, Focht DD, Martin JP. Microbial degradation of[14C]polystyrene and 1, 3-diphenylbutane. Can J Microbiol, 1978, 24(7): 798-803. DOI:10.1139/m78-134 |
| [9] |
Chen GQ. Introduction of bacterial plastics PHA, PLA, PBS, PE, PTT, and PPP//Chen GQ. Plastics from Bacteria. Berlin, Heidelberg: Springer, 2010, 14: 1-16.
|
| [10] |
Oikawa E, Linn KT, Endo T, et al. Isolation and characterization of polystyrene degrading microorganisms for zero emission treatment of expanded polystyrene. Doboku Gakkai Ronbunshuu G, 2011, 40: 373-379. |
| [11] |
Atiq N, Garba A, Ali MI, et al. Isolation and identification of polystyrene biodegrading bacteria from soil. African Journal of Microbiology Research, 2010, 4(14): 1537. |
| [12] |
Tian L, Kolvenbach B, Corvini N, et al. Mineralisation of 14C-labelled polystyrene plastics by Penicillium variabile after ozonation pre-treatment. New Biotechnol, 2017, 38: 101-105. DOI:10.1016/j.nbt.2016.07.008 |
| [13] |
Shen YH. Isolation of intestinal bacteria from T. molitor L and study on the phenomenon of plastic degradation[D]. Shanghai: East China Normal University, 2011. 沈叶红.黄粉虫肠道菌的分离和取食塑料现象的研究[D].上海: 华东师范大学, 2011. |
| [14] |
Yang Y, Yang J, Wu WM, et al. Biodegradation and Mineralization of polystyrene by plastic-eating mealworms: Part 1. Chemical and physical characterization and isotopic tests. Environ Sci Technol, 2015, 49(20): 12080-12086. DOI:10.1021/acs.est.5b02661 |
| [15] |
Yang Y, Yang J, Wu WM, et al. Biodegradation and Mineralization of polystyrene by plastic-eating mealworms: Part 2. Role of gut microorganisms. Environ Sci Technol, 2015, 49(20): 12087-12093. DOI:10.1021/acs.est.5b02663 |
| [16] |
Chen GZ, Zhang BL, Ji MM, et al. Gut microbiota of polystyrene-eating mealworms analyzed by high-throughput sequencing. Microbiol China, 2017, 44(9): 2011-2018 (in Chinese). 陈冠舟, 张白鹭, 纪梦梦, 等. 高通量测序探究啮食聚苯乙烯泡沫塑料黄粉虫的肠道菌群结构. 微生物学通报, 2017, 44(9): 2011-2018. |
| [17] |
Krueger MC, Seiwert B, Prager A, et al. Degradation of polystyrene and selected analogues by biological Fenton chemistry approaches: opportunities and limitations. Chemosphere, 2017, 173: 520-528. DOI:10.1016/j.chemosphere.2017.01.089 |
| [18] |
Albertsson AC. Biodegradation of synthetic polymers. Ⅱ. A limited microbial conversion of 14C in polyethylene to 14CO2 by some soil fungi. J Appl Polym Sci, 1978, 22: 3419-3433. DOI:10.1002/app.1978.070221207 |
| [19] |
Kawai F, Watanabe M, Shibata M, et al. Experimental analysis and numerical simulation for biodegradability of polyethylene. Polym Degradat Stabil, 2002, 76(1): 129-135. DOI:10.1016/S0141-3910(02)00006-X |
| [20] |
Zhong Y, Li YZ, Zhang RL, et al. Screening a polyethylene degrading strain and study on the degradation characteristics. Ecol Environmental Sciences, 2017, 26(4): 681-686 (in Chinese). 钟越, 李雨竹, 张榕麟, 等. 一株聚乙烯降解菌的筛选及其降解特性研究. 生态环境学报, 2017, 26(4): 681-686. |
| [21] |
Tribedi P, Sil AK. Low-density polyethylene degradation by Pseudomonas sp. AKS2 biofilm. Environ Sci Pollut Res, 2013, 20(6): 4146-4153. DOI:10.1007/s11356-012-1378-y |
| [22] |
Gilan I, Hadar Y, Sivan A. Colonization, biofilm formation and biodegradation of polyethylene by a strain of Rhodococcus ruber. Appl Microbiol Biotechnol, 2004, 65(1): 97-104. |
| [23] |
Sivan A, Szanto M, Pavlov V. Biofilm development of the polyethylene-degrading bacterium Rhodococcus ruber. Appl Microbiol Biotechnol, 2006, 72(2): 346-352. DOI:10.1007/s00253-005-0259-4 |
| [24] |
Santo M, Weitsman R, Sivan A. The role of the copper-binding enzyme-laccase-in the biodegradation of polyethylene by the actinomycete Rhodococcus ruber. Int Biodeter Biodegr, 2013, 84: 204-210. DOI:10.1016/j.ibiod.2012.03.001 |
| [25] |
Han QX, Wang QZ, Zhang M. Study on biodegradability of modified PE film. China Plast Ind, 2009, 37(10): 48-51 (in Chinese). 韩秋霞, 王庆昭, 张萌. 改性PE膜的生物可降解性研究. 塑料工业, 2009, 37(10): 48-51. DOI:10.3321/j.issn:1005-5770.2009.10.014 |
| [26] |
Lu CX, Liu L, Li JH, et al. Isolation and characterization of a microorganism degrading starch/polyethylene blends. Chin J Appl Environ Biol, 2013, 19(4): 683-687 (in Chinese). 陆辰霞, 刘龙, 李江华, 等. 淀粉填充聚乙烯类塑料降解微生物的筛选和降解特性. 应用与环境生物学报, 2013, 19(4): 683-687. |
| [27] |
Sudhakar M, Doble M, Murthy PS, et al. Marine microbe-mediated biodegradation of low- and high-density polyethylenes. Int Biodeter Biodegr, 2008, 61(3): 203-213. DOI:10.1016/j.ibiod.2007.07.011 |
| [28] |
Harshvardhan K, Jha B. Biodegradation of low-density polyethylene by marine bacteria from pelagic waters, Arabian Sea, India. Mar Pollut Bull, 2013, 77(1/2): 100-106. |
| [29] |
Balasubramanian V, Natarajan K, Hemambika B, et al. High-density polyethylene (HDPE)-degrading potential bacteria from marine ecosystem of Gulf of Mannar, India. Lett Appl Microbiol, 2010, 51(2): 205-211. |
| [30] |
Syranidou E, Karkanorachaki K, Amorotti F, et al. Biodegradation of mixture of plastic films by tailored marine consortia. J Hazard Mater, 2019, 375: 33-42. DOI:10.1016/j.jhazmat.2019.04.078 |
| [31] |
Raghavan D, Torma AE. DSC and FTIR characterization of biodegradation of polyethylene. Polym Eng Sci, 2010, 32(6): 438-442. |
| [32] |
Miao SJ. Study dn bidldgical characteristics of Zophobas morio and its function on plastic degradation[D]. Yangling: Northwest A & F University, 2010. 苗少娟.大麦虫Zophobas morio的生物学特性及其对塑料降解作用的研究[D].杨凌: 西北农林科技大学, 2010. |
| [33] |
Miao SJ, Zhang YL. Feeding and degradation effect on plastic of Zophobas morio. J Environ Entomol, 2010, 32(4): 435-444 (in Chinese). 苗少娟, 张雅林. 大麦虫Zophobas morio对塑料的取食和降解作用研究. 环境昆虫学报, 2010, 32(4): 435-444. DOI:10.3969/j.issn.1674-0858.2010.04.001 |
| [34] |
Yang J, Yang Y, Wu WM, et al. Evidence of polyethylene biodegradation by bacterial strains from the guts of plastic-eating waxworms. Environ Sci Technol, 2014, 48(23): 13776-13784. DOI:10.1021/es504038a |
| [35] |
Zhang K, Hu RQ, Cai MM, et al. Degradation of plastic film containing polyethylene (PE) by yellow meal worms. Chem Bioeng, 2017, 34(4): 47-49 (in Chinese). 张可, 胡芮绮, 蔡珉敏, 等. 黄粉虫取食和消化降解PE塑料薄膜的研究. 化学与生物工程, 2017, 34(4): 47-49. DOI:10.3969/j.issn.1672-5425.2017.04.011 |
| [36] |
Luo BX. Screening and identification of strains degrading polyethylene (PE) and esearches on degradation characteristics of LBX-2 strian[D]. Chengdu: Sichuan Normal University, 2013. 罗贝旭.聚乙烯降解菌的筛选、鉴定和降解特性的研究[D].成都: 四川师范大学, 2013. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2301349 |
| [37] |
Cacciari I, Quatrini P, Zirletta G, et al. Isotactic polypropylene biodegradation by a microbial community: physicochemical characterization of metabolites produced. Appl Environ Microbiol, 1993, 59(11): 3695-3700. |
| [38] |
Arkatkar A, Arutchelvi J, Bhaduri S, et al. Degradation of unpretreated and thermally pretreated polypropylene by soil consortia. Int Biodeter Biodegr, 2009, 63(1): 106-111. DOI:10.1016/j.ibiod.2008.06.005 |
| [39] |
Arkatkar A, Juwarkar AA, Bhaduri S, et al. Growth of Pseudomonas and Bacillus biofilms on pretreated polypropylene surface. Int Biodeter Biodegr, 2010, 64(6): 530-536. DOI:10.1016/j.ibiod.2010.06.002 |
| [40] |
Jeyakumar D, Chirsteen J, Doble M. Synergistic effects of pretreatment and blending on fungi mediated biodegradation of polypropylenes. Bioresour Technol, 2013, 148: 78-85. DOI:10.1016/j.biortech.2013.08.074 |
| [41] |
Cregut M, Bedas M, Durand MJ, et al. New insights into polyurethane biodegradation and realistic prospects for the development of a sustainable waste recycling process. Biotechnol Adv, 2013, 31(8): 1634-1647. DOI:10.1016/j.biotechadv.2013.08.011 |
| [42] |
Wei R, Zimmermann W. Microbial enzymes for the recycling of recalcitrant petroleum-based plastics: how far are we?. Microb Biotechnol, 2017, 10(6): 1308-1322. DOI:10.1111/1751-7915.12710 |
| [43] |
Howard GT, Blake RC. Growth of Pseudomonas fluorescens on a polyester-polyurethane and the purification and characterization of a polyurethanase-protease enzyme. Int Biodeter Biodegr, 1998, 42(4): 213-220. DOI:10.1016/S0964-8305(98)00051-1 |
| [44] |
Nomura N, Shigeno-Akutsu Y, Nakajima-Kambe T, et al. Cloning and sequence analysis of a polyurethane esterase of Comamonas acidovorans TB-35. J Fermentat Bioeng, 1998, 86(4): 339-345. DOI:10.1016/S0922-338X(99)89001-1 |
| [45] |
Akutsu Y, Nakajima-Kambe T, Nomura N, et al. Purification and properties of a polyester polyurethane-degrading enzyme from Comamonas acidovorans TB-35. Appl Environ Microbiol, 1998, 64(1): 62-67. |
| [46] |
Stern RV, Howard GT. The polyester polyurethanase gene (pueA) from Pseudomonas chlororaphis encodes a lipase. FEMS Microbiol Lett, 2000, 185(2): 163-168. DOI:10.1111/j.1574-6968.2000.tb09056.x |
| [47] |
Howard GT, Crother B, Vicknair J. Cloning, nucleotide sequencing and characterization of a polyurethanase gene (pueB) from Pseudomonas chlororaphis. Int Biodeter Biodegr, 2001, 47(3): 141-149. DOI:10.1016/S0964-8305(01)00042-7 |
| [48] |
Ruiz C, Howard GT. Nucleotide sequencing of a polyurethanase gene (pulA) from Pseudomonas fluorescens. Int Biodeter Biodegr, 1999, 44(2): 127-131. |
| [49] |
Crabbe JR, Campbell JR, Thompson L, et al. Biodegradation of a colloidal ester-based polyurethane by soil fungi. Int Biodeter Biodegr, 1994, 33(2): 103-113. DOI:10.1016/0964-8305(94)90030-2 |
| [50] |
Russell JR, Huang J, Anand P, et al. Biodegradation of polyester polyurethane by endophytic fungi. Appl Environ Microbiol, 2011, 77(17): 6076-6084. DOI:10.1128/AEM.00521-11 |
| [51] |
Filip Z. Decomposition of polyurethane in a garbage landfill leakage water and by soil microorganisms. Eur J Appli Microbiol Biotechnol, 1978, 5(3): 225-231. DOI:10.1007/BF00579343 |
| [52] |
Urgun-Demirtas M, Singh D, Pagilla K. Laboratory investigation of biodegradability of a polyurethane foam under anaerobic conditions. Polyme Degrad Stabil, 2007, 92(8): 1599-1610. DOI:10.1016/j.polymdegradstab.2007.04.013 |
| [53] |
Webb HK, Arnott J, Crawford R, et al. Plastic degradation and its environmental implications with special reference to poly (ethylene terephthalate). Polymers, 2013, 5(1): 1-18. |
| [54] |
Marten E, Müller RJ, Deckwer WD. Studies on the enzymatic hydrolysis of polyesters I. Low molecular mass model esters and aliphatic polyesters. Polym Degrad Stabil, 2003, 80(3): 485-501. DOI:10.1016/S0141-3910(03)00032-6 |
| [55] |
Marten E, Müller RJ, Deckwer WD. Studies on the enzymatic hydrolysis of polyesters. Ⅱ. Aliphaticaromatic copolyesters. Polym Degrad Stabil, 2005, 88(3): 371-381. DOI:10.1016/j.polymdegradstab.2004.12.001 |
| [56] |
Liu RYF, Hu YS, Schiraldi D, et al. Crystallinity and oxygen transport properties of PET bottle walls. J Appl Polym Sci, 2004, 94(2): 671-677. DOI:10.1002/app.20905 |
| [57] |
Lee JH, Lim KS, Hahm WG, et al. Properties of recycled and virgin poly (ethylene terephthalate) blend fibers. J Appl Polym Sci, 2013, 128(2): 1250-1256. DOI:10.1002/app.38502 |
| [58] |
Alves N, Mano JF, Balaguer E, et al. Glass transition and structural relaxation in semi-crystalline poly (ethylene terephthalate): a DSC study. Polymer, 2002, 43(15): 4111-4122. DOI:10.1016/S0032-3861(02)00236-7 |
| [59] |
Parikh M, Gross RA, McCarthy SP. The effect of crystalline morphology on enzymatic degradation kinetics. Polym Mater Sci Eng, 1992, 66: 408-409. |
| [60] |
Ronkvist ÅM, Xie WC, Lu WH, et al. Cutinasecatalyzed hydrolysis of poly (ethylene terephthalate). Macromolecules, 2009, 42(14): 5128-5138. DOI:10.1021/ma9005318 |
| [61] |
Sulaiman S, You DJ, Kanaya E, et al. Crystal structure and thermodynamic and kinetic stability of metagenome-derived LC-cutinase. Biochemistry, 2014, 53(11): 1858-1869. DOI:10.1021/bi401561p |
| [62] |
Then J, Wei R, Oeser T, et al. A disulfide bridge in the calcium binding site of a polyester hydrolase increases its thermal stability and activity against polyethylene terephthalate. FEBS Open Bio, 2016, 6(5): 425-432. DOI:10.1002/2211-5463.12053 |
| [63] |
Wei R, Oeser T, Then J, et al. Functional characterization and structural modeling of synthetic polyester-degrading hydrolases from Thermomonospora curvata. AMB Expr, 2014, 4: 44. DOI:10.1186/s13568-014-0044-9 |
| [64] |
Eberl A, Heumann S, Brückner T, et al. Enzymatic surface hydrolysis of poly (ethylene terephthalate) and bis (benzoyloxyethyl) terephthalate by lipase and cutinase in the presence of surface active molecules. J Biotechnol, 2009, 143(3): 207-212. DOI:10.1016/j.jbiotec.2009.07.008 |
| [65] |
Liebminger S, Eberl A, Sousa F, et al. Hydrolysis of PET and bis-(benzoyloxyethyl) terephthalate with a new polyesterase from Penicillium citrinum. Biocatal Biotransformat, 2007, 25(24): 171-177. |
| [66] |
Vertommen MAME, Nierstrasz VA, van der Veer M, et al. Enzymatic surface modification of poly (ethylene terephthalate). J Biotechnol, 2005, 120(4): 376-386. DOI:10.1016/j.jbiotec.2005.06.015 |
| [67] |
Alisch-Mark M, Herrmann A, Zimmermann W. Increase of the hydrophilicity of polyethylene terephthalate fibres by hydrolases from Thermomonospora fusca and Fusarium solani f. sp. pisi. Biotechnol Lett, 2006, 28(10): 681-685. DOI:10.1007/s10529-006-9041-7 |
| [68] |
Araújoa R, Silva C, O'Neill A, et al. Tailoring cutinase activity towards polyethylene terephthalate and polyamide 6, 6 fibers. J Biotechnol, 2007, 128(4): 849-857. DOI:10.1016/j.jbiotec.2006.12.028 |
| [69] |
Chen S, Su LQ, Chen J, et al. Cutinase: characteristics, preparation, and application. Biotechnol Adv, 2013, 31(8): 1754-1767. DOI:10.1016/j.biotechadv.2013.09.005 |
| [70] |
Herrero AE, Ribitsch D, Steinkellner G, et al. Enzymatic surface hydrolysis of PET: Effect of structural diversity on kinetic properties of cutinases from Thermobifida. Macromolecules, 2011, 44(12): 4632-4640. DOI:10.1021/ma200949p |
| [71] |
Müller RJ, Schrader H, Profe J, et al. Enzymatic degradation of poly (ethylene terephthalate): rapid hydrolyse using a hydrolase from T. fusca. Macromol Rapid Commun, 2005, 26(17): 1400-1405. DOI:10.1002/marc.200500410 |
| [72] |
Nimchua T, Punnapayak H, Zimmermann W. Comparison of the hydrolysis of polyethylene terephthalate fibers by a hydrolase from Fusarium oxysporum LCH I and Fusarium solani f. sp. pisi. Biotechnol J, 2007, 2(3): 361-364. DOI:10.1002/biot.200600095 |
| [73] |
Wei R, Oeser T, Zimmermann W. Synthetic polyester-hydrolyzing enzymes from thermophilic actinomycetes. Adv Appl Microbiol, 2014, 89: 267-305. DOI:10.1016/B978-0-12-800259-9.00007-X |
| [74] |
Ribitsch D, Heumann S, Trotscha E, et al. Hydrolysis of polyethyleneterephthalate by p-nitrobenzylesterase from Bacillus subtilis. Biotechnol Progr, 2011, 27(4): 951-960. DOI:10.1002/btpr.610 |
| [75] |
Guebitz GM, Cavaco-Paulo A. Enzymes go big: surface hydrolysis and functionalisation of synthetic polymers. Trends Biotechnol, 2008, 26(1): 32-38. DOI:10.1016/j.tibtech.2007.10.003 |
| [76] |
Wei R, Oeser T, Schmidt J, et al. Engineered bacterial polyester hydrolases efficiently degrade polyethylene terephthalate due to relieved product inhibition. Biotechnol Bioeng, 2016, 113(8): 1658-1665. DOI:10.1002/bit.25941 |
| [77] |
Han X, Liu WD, Huang JW, et al. Structural insight into catalytic mechanism of PET hydrolase. Nat Commun, 2017, 8: 2106. DOI:10.1038/s41467-017-02255-z |
| [78] |
Joo S, Cho IJ, Seo H, et al. Structural insight into molecular mechanism of poly (ethylene terephthalate) degradation. Nat Commun, 2018, 9: 382. DOI:10.1038/s41467-018-02881-1 |
| [79] |
Austin HP, Allen MD, Donohoe BS, et al. Characterization and engineering of a plasticdegrading aromatic polyesterase. Proc Natl Acad Sci USA, 2018, 115(19): E4350-E4357. DOI:10.1073/pnas.1718804115 |
| [80] |
Then J, Wei R, Oeser T, et al. Ca2+ and Mg2+ binding site engineering increases the degradation of polyethylene terephthalate films by polyester hydrolases from Thermobifida fusca. Biotechnol J, 2015, 10(4): 592-598. DOI:10.1002/biot.201400620 |
| [81] |
Atthoff B, Hilborn J. Protein adsorption onto polyester surfaces: is there a need for surface activation?. J Biomed Mater Res B: Appl Biomater, 2007, 80(1): 121-130. |
| [82] |
Knoll M, Hamm TM, Wagner F, et al. The PHA depolymerase engineering database: a systematic analysis tool for the diverse family of polyhydroxyalkanoate (PHA) depolymerases. BMC Bioinformat, 2009, 10: 89. DOI:10.1186/1471-2105-10-89 |
| [83] |
Ribitsch D, Acero EH, Przylucka A, et al. Enhanced cutinase-catalyzed hydrolysis of polyethylene terephthalate by covalent fusion to hydrophobins. Appl Environ Microbiol, 2015, 81(11): 3586-3592. DOI:10.1128/AEM.04111-14 |
| [84] |
Furukawa M, Kawakami N, Oda K, et al. Acceleration of enzymatic degradation of poly (ethylene terephthalate) by surface coating with anionic surfactants. ChemSusChem, 2018, 11(23): 4018-4025. DOI:10.1002/cssc.201802096 |
| [85] |
Otake Y, Kobayashi T, Asabe H, et al. Biodegradation of low-density polyethylene, polystyrene, polyvinyl chloride, and urea formaldehyde resin buried under soil for over 32 years. J Appl Polym Sci, 1995, 56(13): 1789-1796. DOI:10.1002/app.1995.070561309 |
| [86] |
Ali Muhammad I, Ahmed S, Robson G, et al. Isolation and molecular characterization of polyvinyl chloride (PVC) plastic degrading fungal isolates. J Bas Microbiol, 2014, 54(1): 18-27. DOI:10.1002/jobm.201200496 |
| [87] |
Giacomucci L, Raddadi N, Soccio M, et al. Polyvinyl chloride biodegradation by Pseudomonas citronellolis and Bacillus flexus. New Biotechnol, 2019, 52: 35-41. DOI:10.1016/j.nbt.2019.04.005 |
 2019, Vol. 35
2019, Vol. 35