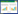中国科学院微生物研究所、中国微生物学会主办
文章信息
- 梁宗林, 秦亚玲, 王沛, 王保军, 刘征华, 尹华群, 刘双江, 姜成英
- Liang Zonglin, Qin Yaling, Wang Pei, Wang Baojun, Liu Zhenghua, Yin Huaqun, Liu Shuang-Jiang, Jiang Cheng-Ying
- 云南省蒙自酸性矿山排水微生物群落结构和功能
- Microbial community structure and function in acid mine drainage from Mengzi, Yunnan Province
- 生物工程学报, 2019, 35(11): 2035-2049
- Chinese Journal of Biotechnology, 2019, 35(11): 2035-2049
- 10.13345/j.cjb.190506
-
文章历史
- Received: November 12, 2019
- Accepted: November 15, 2019
2. 中国科学院大学, 北京 100049;
3. 中国科学院环境微生物技术联合实验室, 北京 100101;
4. 中南大学 资源加工与生物工程学院, 湖南 长沙 410083;
5. 中南大学 教育部生物冶金重点实验室, 湖南 长沙 410083
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. RCEES-IMCAS-UCAS Joint Laboratory for Environmental Microbial Technology, Chinese Academy of Sciences, Beijing 100101, China;
4. School of Minerals Processing and Bioengineering, Central South University, Changsha 410083, Hunan, China;
5. Key Laboratory of Biometallurgy of Ministry of Education, Central South University, Changsha 410083, Hunan, China
酸性矿山排水(Acid mine drainage, AMD)是矿区内常见的一种极端环境,由黄铁矿(FeS2)等硫化矿物在接触空气和水的条件下自发氧化及在微生物的参与下加速氧化溶解形成[1],通常呈红棕色或褐色,也称为酸性矿山废水,是矿山废水中污染范围最广且危害最为严重的一类[2]。其典型特征是随着氧化反应的进行,pH逐渐降低,通常为2–4,甚至小于2;具有高浓度的SO42−,以及铁、铝、铜、铅、砷等金属和类金属元素。由于废矿石和尾矿的大量积累,以及旧矿井的废弃,酸性矿山排水的产生已成为一个全球性的环境问题[3]。
黄铁矿是地球中含量最为丰富的硫化矿物[4],它的氧化性溶解是酸性矿山排水的主要成因[5],习惯性概括为总反应式:4FeS2+14H2O+15O2→ 4Fe(OH)3+8SO42−+16H+。值得注意的是,黄铁矿是酸不溶性矿物,其氧化性溶解包含一系列反应过程[1],Fe3+起主要的氧化作用。黄铁矿与水和氧气接触后发生氧化,产生Fe2+,并进一步被氧化为Fe3+;在pH低于4时,Fe2+自发氧化速度慢,它的氧化主要由嗜酸性铁氧化菌完成[4],且其速度超过自发氧化106倍,使Fe3+快速再生,这是促进硫化矿物氧化的关键步骤[6]。另外,硫化矿物氧化还生成还原型无机硫化合物和S0,为硫氧化菌的生长提供能源物质[7]。研究表明,酸性矿山排水自然条件严苛且地球化学因素相对简单,但具有丰富的嗜酸性铁、硫氧化及异养的细菌和古菌[8-9]。因此酸性矿山排水中的微生物及其铁、硫元素代谢一直备受关注[10-11]。近些年来,不依赖于培养的微生物群落分析方法如16S rRNA基因克隆文库、扩增子测序、宏基因组测序以及宏转录组测序技术等已广泛用于酸性矿山排水的研究[12-14],使人们对其中的微生物多样性、群落结构和功能以及微生物与环境的相互作用有了更全面的认识[15-16]。
酸性矿山排水生态系统包含酸矿水、沉积泥和生物膜等不同环境[13, 17],而目前关于其不同环境中微生物群落组成、分布及功能的研究依然较少[17]。本研究采用高通量测序技术分析了云南省蒙自某矿区酸矿水坑不同类型样本和周边溪水的原核微生物多样性及群落结构差异,探究了影响群落结构的主要因素,进而分析了菌群的分布和适应性及重要功能,旨在更全面地了解酸性矿山排水的形成和发展规律及其中的铁、硫等元素生物地球化学循环[18],发掘极端环境的微生物资源,同时为酸性矿山排水的治理和修复[4]以及微生物浸矿[19]提供科学依据和理论指导。
1 材料与方法 1.1 采样地点描述和样本采集中国云南省蒙自市矿产资源丰富,其中白牛厂银多金属矿床已探明银、锌储量达超大型规模,铅、锡储量达大型规模[20]。本研究于2017年9月22日采集该矿区内某地点(东经103°46′47″,北纬23°28′53″,海拔约1 847 m)自然形成的酸矿水坑中酸矿水(Drainage)、沉积泥(Sediment)和生物膜(Biofilm)等不同环境的样本,并根据pH高低将各酸矿水坑分为B、C、D区。B区采集3个酸矿水样本(BD)和5个沉积泥样本(BS),C、D区各采集3个酸矿水样本(CD和DD)和3个沉积泥样本(CS和DS),E区采集3个生物膜样本(EF)。酸矿水坑周边一处酸性水流(Rill)作为A区,采集3个周边溪水样本(AR)。
1.2 理化参数测定采用便携式水质多参数测量仪(Star A329, Orion, USA)测定采样点的温度、pH和电导率等参数。基于邻菲啰啉分光光度法(HJ/T 345-2007)和比浊法[21],采用便携式比色计(DR/890, HACH, USA)测定酸矿水样本的Fe2+、Fe3+和SO42−浓度。取适量样本经微波消解仪(TOPwave, Analytik Jena, Germany)消解及电热板加热赶酸后稀释到适宜浓度,采用ICP-OES (Optima 5300 DV, PerkinElmer, USA)测定元素含量[22]。
1.3 样本基因组提取采用DNA提取试剂盒(PowerMax Soil DNA Isolation Kit, Qiagen)提取样本DNA。采用溶剂过滤器和真空泵将酸矿水抽滤通过微孔滤膜(PES, Φ47–50 mm 0.22 μm, PALL)以收集样本中的微生物,用无菌手术剪和镊子将滤膜剪碎[23]后进行DNA提取。沉积泥和生物膜样本直接按照试剂盒操作说明进行提取。DNA溶解于Tris盐酸缓冲液(pH 8.0)中,−20 ℃冻存,由北京博奥汇玖生物科技有限公司完成16S rRNA基因扩增子测序。
1.4 16S rRNA基因扩增子测序选择16S rRNA基因的V4–V5区进行扩增子测序。取DNA样品与已融合样本标签(Barcodes)的引物515F (5′-GTGYCAGCMGCCGCGGTAA-3′)和926R (5′-CCGYCAATTYMTTTRAGTTT-3′)于预混液(HiFi HotStart ReadyMix, KAPA)中配制PCR反应体系。PCR反应条件为:95 ℃预变性3 min;98 ℃变性20 s,55 ℃退火15 s,72 ℃延伸15 s,共25个循环;72 ℃终延伸1 min。PCR产物经凝胶电泳和回收纯化后采用Qubit测定DNA浓度并连接测序接头完成文库构建。采用Agilent 2100生物分析仪检测文库片段长度分布和浓度,选择Illumina MiSeq PE250平台完成测序。
1.5 高通量测序数据分析序列数据不含测序接头,根据样本标签进行分样得到各样本上、下游单端序列。采用USEARCH v11[24]合并单端序列,并用Cutadapt v2.3[25]去除引物;滤除低质量序列和Singletons,并对高质量序列进行去冗余;按照97%相似性聚类(Cluster)形成可操作分类单元(Operational taxonomic units,OTUs)[26]以及通过去噪(Denoise)保留数目大于6的序列获得扩增子序列变体(Amplicon sequence variants,ASVs)[27],去除嵌合体,OTUs和ASVs统称为特征(Features);最终获得各特征的代表序列并生成特征表。在QIIME2[28]中基于引物对515F/926R和Silva 132 16S rRNA基因数据库训练RDP物种分类器[29],并按照0.7置信度阈值进行物种注释;根据注释结果去除线粒体和叶绿体序列并过滤特征表;基于样本最少序列数对特征表抽平后计算各样本α多样性指数和距离矩阵。采用R语言及“vegan”、“ggplot2”、“ComplexHeatmap”等R程序包进行统计分析及数据可视化。
2 结果与分析 2.1 样本理化特征周边溪水与酸矿水坑的物理化学特征差异明显。各采样区温度均约为25 ℃,A区pH为5.07±0.15,在各区中最高;B、C、D区pH逐渐降低且同区酸矿水pH略高于沉积泥(表 1);E区酸矿水坑采集到少量生物膜样本,pH为1.52±0.08。
| Physicochemical parameters | BS (n=5) | CS (n=3) | DS (n=3) | BD (n=3) | CD (n=3) | DD (n=3) |
| pH | 2.37±0.05 | 2.18±0.11 | 1.89±0.07 | 2.54±0.05 | 2.25±0.05 | 1.94±0.05 |
| Cond. (mS/cm) | NA | NA | NA | 6.06±0 | 17.2±0.05 | 28.55±0.04 |
| TDS (g/L) | NA | NA | NA | 2.97±0 | 8.43±0.02 | 13.98±0.02 |
| Fe2+ (mg/L) | NA | NA | NA | 15±1 | 6 393±111 | 15 630±42 |
| Fe3+ (mg/L) | NA | NA | NA | 3 159±3 | 12 485±17 | 46 475±46 |
| SO42− (mg/L) | NA | NA | NA | 6 933±306 | 33 667±577 | 134 000±2 000 |
| Al (mg/(kg or L)) | 57 264±2 373 | 54 787±340 | 55 093±1 148 | 42±2 | 444±21 | 1 547±57 |
| As (mg/(kg or L)) | 2 093.3±214.4 | 1 836.2±76.7 | 1 362.3±23.8 | 19.1±0.6 | 151.8±2.1 | 641.7±8.8 |
| B (mg/(kg or L)) | 351.9±53.8 | 389±53.9 | 380.1±32.8 | 8.9±1.4 | 50.9±8.6 | 182.3±24.3 |
| Ba (mg/(kg or L)) | 147.99±5.36 | 162.41±9.42 | 150.26±2.03 | 0.22±0.08 | 1.34±0.51 | 4.8±1.86 |
| Ca (mg/(kg or L)) | 8 301±1 283 | 4 129±903 | 3 677±294 | 406±13.8 | 464.2±2.2 | 658.3±20.6 |
| Cd (mg/(kg or L)) | 24.78±12.8 | 20.3±7.9 | 15.88±6.17 | 0.28±0.09 | 1.85±0.73 | 7.72±3.12 |
| Co (mg/(kg or L)) | 7.96±0.53 | 10.49±1.31 | 13.51±0.57 | 0.4±0.02 | 2.05±0.08 | 8.13±0.28 |
| Cr (mg/(kg or L)) | 152.25±12.38 | 142.45±23.26 | 145.64±13.63 | 2.16±0.52 | 12.47±2.75 | 52.27±12.31 |
| Cu (mg/(kg or L)) | 899.1±683.9 | 1 108.3±408.5 | 1 878.8±45.6 | 62.3±2.8 | 363.1±8 | 1 564.3±30.9 |
| Fe (mg/(kg or L)) | 101 355±3 067 | 120 140±1 394 | 115 157±6 740 | 3 107±10 | 17 910±266 | 65 250±2 198 |
| K (mg/(kg or L)) | 13 597±1 286 | 15 337±1 504 | 14 751±1 065 | 0.32±0.56 | 0.81±1.41 | 15.52±10.87 |
| Li (mg/(kg or L)) | 24.27±0.54 | 30.5±0.82 | 30.78±0.61 | 0.02±0.02 | 0.27±0.1 | 0.5±0.87 |
| Mg (mg/(kg or L)) | 4 709.4±218.8 | 6 347.8±738.4 | 6 513.1±578.1 | 254.8±2.5 | 848.3±17.5 | 2 988±87.8 |
| Mn (mg/(kg or L)) | 214.2±64.4 | 527.2±162 | 631.8±42.5 | 30.8±1.3 | 133.5±3 | 502.7±9.4 |
| Mo (mg/(kg or L)) | 2.59±1.12 | 0±0 | 2.6±2.25 | 0±0 | 0±0 | 0±0 |
| Na (mg/(kg or L)) | 445.2±26.3 | 475.7±41.2 | 446.6±18.8 | 2±0.6 | 3.4±1.2 | 8.7±15 |
| Ni (mg/(kg or L)) | 25.63±0.44 | 32.05±0.23 | 37.05±1.09 | 0.55±0 | 3.04±0.09 | 12.32±0.33 |
| P (mg/(kg or L)) | 3 456±216.6 | 3 215.3±125.3 | 3 482.6±185.6 | 76.8±2.1 | 609.2±19.3 | 2 236.5±76.5 |
| Pb (mg/(kg or L)) | 192.8±36.6 | 1 633.6±565 | 818.6±123.5 | 0.2±0.1 | 1.6±0.6 | 5.3±2.4 |
| Se (mg/(kg or L)) | 0±0 | 0±0 | 0±0 | 0±0 | 0±0 | 0±0 |
| Si (mg/(kg or L)) | 2 260±1 051 | 496±313 | 649±435 | 12.7±2 | 30.4±15.1 | 56±2.5 |
| S (mg/(kg or L)) | 20 690±2 024 | 34 682±3 276 | 43 438±3 151 | 2 430±47 | 11 041±332 | 37 882±1 644 |
| Sr (mg/(kg or L)) | 46.72±4.44 | 42.95±1.95 | 44.82±0.92 | 0.63±0.05 | 1.32±0.1 | 2.85±0.09 |
| V (mg/(kg or L)) | 145.54±10.8 | 137.13±17.02 | 131.05±8.45 | 0.62±0.45 | 6.74±1.13 | 45.73±13.76 |
| Zn (mg/(kg or L)) | 117.3±20.1 | 445.8±62.3 | 420.4±12.2 | 17.9±0.6 | 68.2±2.4 | 238.4±6 |
| Cond.: conductivity; TDS: total dissolved solids; NA: not available. | ||||||
酸矿水坑中含量最丰富的元素是Fe,沉积泥中还含有大量的Al、S、K、Mg、Ca、P、As、Cu、Pb等元素。随着pH的降低,酸矿水中Fe2+、Fe3+和SO42−的浓度,以及各元素(除Mo和Se)的含量明显升高(表 1)。D区酸矿水中总Fe离子浓度高达(65 250±2 198) mg/L,同时含有高浓度的SO42− ((134 000±2 000) mg/L)和有毒的类金属元素As ((641.7±8.8) mg/L)。如不及时加以控制,持续产生的酸性矿山排水将严重污染周围的水体与土壤。
2.2 酸性矿山排水环境微生物群落多样性DD组样本由于条件极端未提取到DNA,其他7组23个样本经测序共获得1 300 684条有效序列,通过聚类和去噪及去除线粒体和叶绿体等特征后共获得968个OTUs和801个ASVs (表 2)。比较发现,B、C、D区各组ASVs数明显高于OTUs数,表明酸矿水和沉积泥中具有较多序列相似性较高的16S rRNA基因,可能存在较多系统发生关系较近的物种,最近已研究采用去噪形成ASVs的方法分析酸性矿山排水中的微生物群落[30],因此本研究选择去噪的结果用于后续分析。
| Sample (n=23) | OTUs (t=968) | ASVs (t=801) | Counts | Alpha diversity metrics | ||||||
| ACE | Chao1 | Observed | Shannon | Evenness | PD | Coverage | ||||
| AR1 | 574 | 563 | 52 674 | 571.5 | 573.4 | 562 | 6.23 | 0.05 | 23.25 | 0.999 4 |
| AR2 | 569 | 561 | 55 473 | 573.9 | 589.3 | 557 | 6.18 | 0.05 | 21.58 | 0.999 0 |
| AR3 | 570 | 561 | 55 316 | 561.3 | 570.7 | 553 | 6.35 | 0.06 | 21.46 | 0.999 4 |
| BD1 | 74 | 131 | 47 596 | 155.6 | 161.0 | 131 | 4.01 | 0.06 | 7.99 | 0.999 5 |
| BD2 | 96 | 142 | 48 219 | 155.4 | 162.9 | 141 | 4.58 | 0.10 | 9.38 | 0.999 6 |
| BD3 | 92 | 146 | 49 285 | 161.3 | 169.3 | 146 | 4.36 | 0.08 | 9.09 | 0.999 6 |
| BS1 | 96 | 133 | 53 999 | 145.6 | 155.4 | 131 | 4.72 | 0.12 | 8.32 | 0.999 6 |
| BS2 | 89 | 132 | 53 510 | 166.3 | 155.0 | 130 | 4.67 | 0.11 | 7.87 | 0.999 5 |
| BS3 | 76 | 114 | 55 378 | 122.4 | 119.1 | 110 | 4.38 | 0.10 | 6.56 | 0.999 7 |
| BS4 | 68 | 112 | 54 756 | 119.0 | 114.0 | 108 | 4.35 | 0.10 | 6.09 | 0.999 7 |
| BS5 | 71 | 115 | 54 751 | 122.9 | 118.9 | 115 | 4.64 | 0.13 | 6.74 | 0.999 8 |
| CD1 | 60 | 99 | 55 213 | 107.3 | 105.7 | 97 | 3.03 | 0.04 | 7.18 | 0.999 7 |
| CD2 | 53 | 86 | 52 619 | 98.2 | 94.1 | 85 | 2.60 | 0.04 | 6.18 | 0.999 7 |
| CD3 | 50 | 83 | 55 706 | 98.4 | 98.1 | 81 | 2.68 | 0.05 | 5.40 | 0.999 7 |
| CS1 | 50 | 91 | 52 579 | 105.2 | 119.6 | 89 | 2.49 | 0.03 | 5.02 | 0.999 6 |
| CS2 | 46 | 83 | 54 247 | 93.4 | 100.2 | 82 | 2.27 | 0.04 | 5.20 | 0.999 7 |
| CS3 | 43 | 87 | 52 412 | 97.5 | 94.7 | 86 | 2.13 | 0.03 | 4.53 | 0.999 7 |
| DS1 | 18 | 34 | 55 202 | 39.0 | 37.5 | 34 | 0.85 | 0.04 | 2.63 | 0.999 9 |
| DS2 | 26 | 45 | 56 486 | 58.5 | 58.0 | 45 | 0.82 | 0.03 | 3.42 | 0.999 7 |
| DS3 | 32 | 52 | 56 898 | 53.3 | 63.0 | 49 | 1.27 | 0.03 | 3.87 | 0.999 8 |
| EF1 | 183 | 143 | 62 499 | 175.9 | 167.8 | 134 | 1.78 | 0.02 | 12.10 | 0.999 1 |
| EF2 | 209 | 167 | 60 881 | 207.6 | 213.3 | 158 | 1.90 | 0.02 | 13.97 | 0.998 9 |
| EF3 | 256 | 152 | 61 462 | 156.3 | 174.5 | 149 | 2.29 | 0.02 | 13.14 | 0.999 6 |
各样本的Alpha多样性指数见表 2。Observed指实际测到的特征数;Evenness为辛普森(Simpson)均匀度指数;香农(Shannon)指数同时衡量丰富度和均匀度,反映群落多样性情况;PD表示Faith[31]提出的谱系多样性(Phylogenetic diversity)。各样本覆盖度(Coverage)均高于99%,表明测序深度足够。AR群落丰富度(ACE和Chao1)最高,但均匀度较低;B、C、D区多样性逐渐降低;EF丰富度较高,但均匀度最低。总体来看,pH更低的采样区表现为群落多样性更低,而生物膜具有一定的特殊性。
2.3 酸性矿山排水环境微生物群落组成7组样本共注释到26个门、45个纲和96个目,通过堆叠柱形图展示平均相对丰度前20的微生物类群。在门水平(图 1A)上,AR中变形菌门(Proteobacteria) (66.58%)和拟杆菌门(Bacteroidetes) (31.41%)占主要优势,属于丰富类群(平均相对丰度≥1%);其次为芽单胞菌门(Gemmatimonadetes) (0.92%)和蓝细菌门(Cyanobacteria) (0.38%),但这两个门属于低丰度稀有类群(平均相对丰度 < 1%)。BD中变形菌门(63.98%)的丰度最高;广古菌门(Euryarchaeota) (17.28%)、硝化螺菌门(Nitrospirae) (11.82%)、酸杆菌门(Acidobacteria) (4.00%)和未分类的古菌(Unclassified Archaea) (1.34%)成为丰富类群;厚壁菌门(Firmicutes) (0.61%)、浮霉状菌门(Planctomycetes) (0.50%)、放线菌门(Actinobacteria) (0.35%)、Dependentiae (0.06%)和Diapherotrites (0.03%)的丰度增高。BS中广古菌门(50.12%)的丰度明显更高,在环境中占主导地位(平均相对丰度≥50%),未分类古菌(14.49%)、硝化螺菌门(12.52%)、放线菌门(3.04%)、Diapherotrites (0.74%)和绿弯菌门(Chloroflexi) (0.48%)相比于其他组中丰度最高。与B区相比,CD中广古菌门(65.51%)和Diapherotrites (0.45%)的丰度更高;随着酸矿水酸度和离子浓度的升高,CS和DS中广古菌门的丰度逐渐升高,分别为55.76%和86.40%。生物膜EF中,广古菌门的丰度高达96.40%,其他门为稀有类群。
|
| 图 1 酸性矿山排水环境微生物类群在不同分类水平上的相对丰度 Fig. 1 Relative abundance of microbial taxa in AMD environment at Phylum (A), Class (B) and Order (C) levels |
| |
在纲和目水平(图 1B–C)上,酸矿水坑样本中热原体纲(Thermoplasmata)热原体目(Thermoplasmatales)丰度最高且分布最广,其次为γ变形菌纲(Gammaproteobacteria)酸硫杆菌目(Acidithiobacillales)和未定位的类群;而γ变形菌纲中的β变形菌目(Betaproteobacteriales)和未培养的类群,以及α变形菌纲(Alphaproteobacteria)和拟杆菌纲(Bacteroidia)则主要存在于周边溪水AR中。硝化螺菌门中只检测到硝化螺菌纲(Nitrospira)的硝化螺菌目(Nitrospirales),主要存在于BD(11.82%)和BS(12.52%)中。α变形菌纲醋杆菌目(Acetobacterales)和酸杆菌纲(Acidobacteriia)酸杆菌目(Acidobacteriales)主要分布于BD (17.72%和4.00%)中。梭菌纲(Clostridia)梭菌目(Clostridiales)在酸矿水坑各组中保持较低丰度(0.29%–1.95%)。酸微菌纲(Acidimicrobiia)中未培养的类群和酸微菌目(Acidimicrobiales)主要分布于BS (1.82%和1.10%)中。δ变形菌纲(Deltaproteobacteria)的Sva0485[32]和微古菌纲(Micrarchaeia)的ARMAN-2主要位于BS (4.30%和0.72%)和CD (2.26%和0.45%)中。
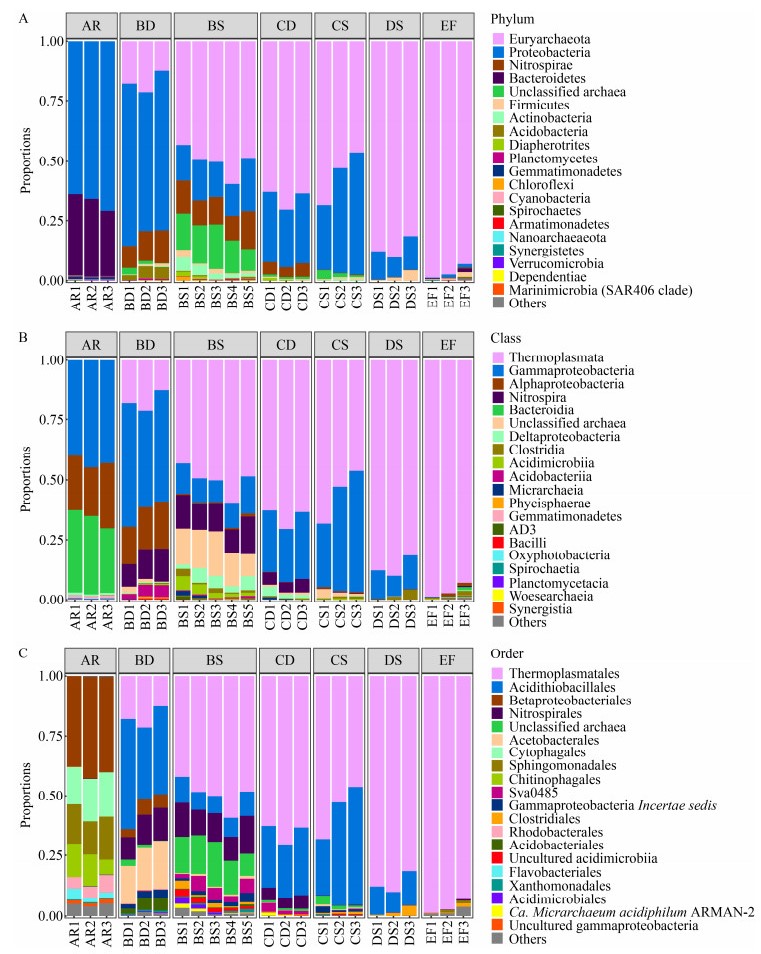
2.4 酸性矿山排水环境微生物群落结构差异基于Bray-Curtis相异和Jaccard相似距离矩阵进行主坐标分析(Principal coordinates analysis, PCoA),结果表明,考虑群落组成丰度(图 2A)时,各组群落结构间存在明显差异;而不考虑丰度(图 2B)时,相同采样区的酸矿水和沉积泥间差异明显减小。进一步基于Bray-Curtis距离作PERMANOVA检验,结果同样表明,各组群落结构存在极显著差异(R2=0.973, P=0.001),酸矿水和沉积泥间均具有明显或显著差异(表 3)。以上结果说明,各采样区和样本类型的群落结构存在明显差异,同区酸矿水和沉积泥的群落结构差异主要受丰度的影响。层次聚类(图 3)直观地展示各样本群落结构的相似程度,可见周边溪水和生物膜明显区别于其他组;同区酸矿水和沉积泥较为相似;C区与D区样本更相似。
|
| 图 2 基于Bray-Curtis相异和Jaccard相似距离矩阵的主坐标分析(PCoA) Fig. 2 Principal coordinates analysis (PCoA) based on Bray-Curtis dissimilarity (A) and Jaccard similarity (B) distance matrices |
| |

|
| 图 3 基于非加权组平均法(UPGMA)和Bray-Curtis距离矩阵对各样本群落结构相似性的层次聚类分析 Fig. 3 Hierarchical clustering based on UPGMA and Bray-Curtis distance matrix |
| |
| Groups | Bray-Curtis | |
| Variation (R2) | Pr (> F) | |
| BD/BS | 0.882 | 0.016 |
| BD/CD | 0.934 | 0.100 |
| BD/CS | 0.896 | 0.100 |
| BD/DS | 0.967 | 0.100 |
| BS/CD | 0.925 | 0.019 |
| BS/CS | 0.902 | 0.023 |
| BS/DS | 0.945 | 0.021 |
| CD/CS | 0.639 | 0.100 |
| CD/DS | 0.957 | 0.100 |
| CS/DS | 0.852 | 0.100 |
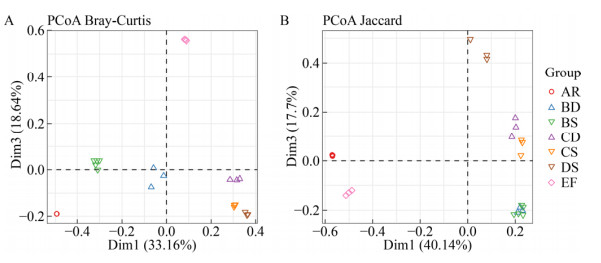
对各样本pH和Shannon指数进行线性回归分析(图 4),同时计算皮尔逊(Pearson)相关系数。结果显示,pH和Shannon指数呈极显著正相关(R=0.804, P≤0.001),表明群落多样性随pH降低而降低。
|
| 图 4 各样本pH和Shannon指数的线性回归分析及Pearson相关系数 Fig. 4 Linear regression and Pearson correlation coefficient between pH value and Shannon index of all samples. Level of confidence interval: 0.95; *** indicates P≤0.001 |
| |
进一步以纲水平(γ变形菌纲以目水平)群落组成及丰度进行冗余分析(Redundancy analysis, RDA),红色划线表示平均相对丰度(> 0.05%)前20的微生物类群(图 5)。RDA1和RDA2轴的解释率分别为54.55% (P≤0.05)和19.97% (P≤0.05),表明群落结构主要趋势已被pH (P≤0.05)和样本类型(P≤0.05)解释,pH及周边溪水和生物膜对群落结构具有重大影响。包括α变形菌纲、拟杆菌纲、热原体纲、梭菌纲、β变形菌目和γ变形菌纲未培养的类群在内的6个微生物类群对第一轴有重要贡献,热原体纲只与梭菌纲呈正相关;其余14个类群则对第二轴有主要的贡献,之间多呈正相关。
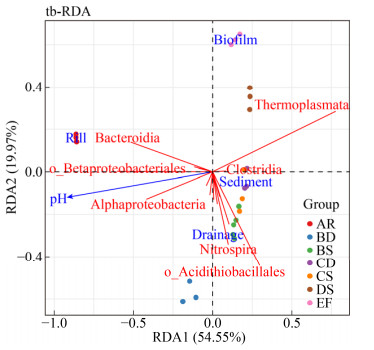
|
| 图 5 纲水平微生物类群的冗余分析(RDA) Fig. 5 Redundancy analysis (RDA) based on the Hellinger-transformed microbial taxa relative abundance at class level constrained by variables pH and sample types, scale=FALSE, scaling=2 |
| |
在属水平(图 6)上,AR中的菌群与其他组具有明显差异,特有的高丰度类群主要归为α变形菌纲、β变形菌目和拟杆菌纲,且明显聚为一簇,其他低丰度的嗜酸性微生物极有可能迁移自酸性矿山排水。

|
| 图 6 属水平上微生物类群的相对丰度及与酸度的Pearson相关性组合热图 Fig. 6 Heatmaps based on relative abundance of the top 49 microbial taxa (mean relative abundance > 0.1%) at genus level and their Pearson correlation with acidity. Row clustering distance: Euclidean. Hierarchical clustering method: Complete. P-values were adjusted using the "BH" method. "." indicate P≤0.1, * indicate P≤0.05, ** indicate P≤0.01 |
| |
整体来看,酸矿水坑中的菌群组成极为相似,亚铁原体属Ferroplasma、酸硫杆菌属Acidithiobacillus、钩端螺旋菌属Leptospirillum等丰度高且分布广的类群明显聚为一簇。亚铁原体属(6.60%–86.34%)的丰度与酸度呈显著正相关,在条件极端的DS中达到最高,此时硫化芽胞杆菌属Sulfobacillus的丰度也最高(1.58%)。酸硫杆菌属在酸矿水和沉积泥(pH 2.0–2.5)中的丰度达8.75%–38.29%。钩端螺旋菌属主要分布于BD和BS (11.82%和12.52%)中。此外,BD中分布有最高丰度的酸杆菌属Acidibacter (2.44%)、醋杆菌科Acetobacteraceae未培养的类群(5.90%)、嗜酸菌属Acidiphilium (11.12%)、铁卵形菌属Ferrovum (5.26%)和Acidicapsa (2.16%)等,而BS中主要为热原体科(Thermoplasmataceae)的未培养类群(15.81%)、Cuniculiplasma (21.64%)、Metallibacterium (0.63%)、A原体(A-plasma) (3.17%)和E原体(E-plasma) (1.26%)[33]以及铁线菌属Ferrithrix (1.04%)等,这些微生物类群多与酸度呈显著或极显著负相关。EF中主要为亚铁原体属和酸原体属Acidiplasma (79.26%和16.97%),几乎不存在(平均相对丰度 < 0.01%)或未测到酸硫杆菌属和Cuniculiplasma。
3 讨论 3.1 酸性矿山排水环境菌群的分布及适应性结合群落结构及菌群与环境因子相关性分析发现,本研究蒙自酸矿水坑不同环境与周边溪水群落组成差异较大,起主要作用的环境因子为pH。周边溪水pH最高,群落丰富度也最高,主要有变形菌门β变形菌目和α变形菌纲以及拟杆菌门拟杆菌纲,其特有的高丰度类群在酸矿水坑中难以存活,只有少量存在于生物膜中(图 6),生物膜中可能形成了特殊的微环境。拟杆菌纲几乎只分布于周边溪水中,与pH呈明显正相关(图 5)。通常拟杆菌门在酸性矿山排水中不常见,在环境酸度较低或测序深度足够大的情况下偶有报道[22, 34],其仅有的耐酸类群最适pH大于5.0[35]。可认为拟杆菌门不适应酸性矿山排水环境,但与拟杆菌纲呈明显正相关的β变形菌目和α变形菌纲中具有适应性较好的铁卵形菌属和醋杆菌目,这些微生物主要分布于B区酸矿水中。
广古菌门、变形菌门(α、γ和δ变形菌纲)、硝化螺菌门、酸杆菌门、放线菌门和厚壁菌门等广泛分布于蒙自酸矿水坑中,至少在一组中为丰富类群,这些微生物常见于酸性矿山排水环境且表现出很强的适应性[22],此外,Diapherotrites、浮霉状菌门、绿弯菌门、Dependentiae (TM6)[36]和蓝细菌门也存在于酸矿水坑不同环境中,最近已有研究报道这些类群分布于不同的酸性矿山排水中[30, 37]。本研究发现热原体纲热原体科未培养的类群(ASV_8, 16, 19, 20)与A、E原体和Cuniculiplasma在沉积泥中丰度均较高,其分布特征与钩端螺旋菌属最为近似。酸杆菌门的Acidicapsa主要存在于酸矿水中,而有研究发现该门的类群在沉积泥中丰度也较高[37]。值得关注的是,本研究酸矿水坑中具有高丰度的未分类的古菌(ASV_7, 18, 25),其与Rio Tinto河酸性矿山排水中未培养的古菌[38]聚为一个OTU,它们在环境中的作用和适应性有待进一步研究。ARMAN-2是该环境中主要的Diapherotrites类群,在沉积泥和酸度更低的酸矿水中均有分布,而在生物膜中几乎不存在;其他未培养的类群Sva0485和浮霉状菌门的CPla-3在酸矿水和沉积泥中均有分布,而绿弯菌门的AD3主要分布于沉积泥中,这些类群在酸性矿山排水中较少有报道[13, 39]。
研究表明,pH对酸性矿山排水环境微生物群落结构具有重大影响。随着硫化矿物的氧化性溶解,酸性矿山排水的pH不断降低,进而溶解更多的金属和类金属元素,离子浓度不断升高,群落结构及菌群分布可能也发生着适应性的变化。本研究发现同区酸矿水和沉积泥的群落结构差异主要表现在群落组成丰度上,随着pH的降低,酸矿水坑微生物群落多样性明显降低,表现为群落组成和结构均趋于简单,主要原因可能为热原体纲对该环境的适应性极强,其丰度与pH呈明显负相关,在极端条件下对群落结构起主导作用,在属水平上表现为亚铁原体属在低pH采样区中丰度明显升高。
3.2 酸性矿山排水环境菌群的功能分析铁卵形菌属主要分布于B区酸矿水(pH 2.54±0.05)中,在酸度和离子浓度更高的分组中均未测到。该属成员以Fe2+和黄铁矿自养生长,对有机物敏感[40],广泛分布于酸性矿山排水环境,中度嗜酸,在pH 2.0–4.0的条件下[22, 41]和一些特殊生境[42]中可占主要优势。推测铁卵形菌属主要在酸性矿山排水形成初期进行铁氧化,促进矿石溶解,而随着环境酸度和离子浓度的升高,其生态地位逐渐被其他铁氧化菌取代。
酸硫杆菌属在酸矿水和沉积泥中均有较高的丰度(8.75%–38.29%),可利用还原型无机硫化合物和S0作为能源自养生长,部分种(At. ferrooxidans等)还可利用Fe2+和黄铁矿等硫化矿物,以及还原Fe3+[43]。该属是酸性矿山排水中常见的嗜酸性铁、硫氧化细菌,最适pH小于3。本研究从酸矿水样本中分离得到了该属多株菌,包含潜在新种,可应用于生物浸矿中,促进硫化矿物的氧化。钩端螺旋菌属(0.09%–12.52%)的丰度仅次于酸硫杆菌属,Leptospirillum spp.是严格好氧的嗜酸性铁氧化细菌,以Fe2+作为唯一能源严格自养生长,对有机物极其敏感。相比于酸硫杆菌属,该属适应更低的pH,在靠近矿石溶解的区域通过再生Fe3+对硫化矿物的氧化可能发挥着主要的促进作用[44]。
亚铁原体属(6.60%–86.34%)在酸矿水坑中分布最广,该属在极端低pH和高浓度金属离子的环境中广泛分布且可占主要优势[12, 45],在生物浸矿体系和酸性矿山排水中相继得到分离培养,极端嗜酸,好氧且兼性厌氧,能氧化Fe2+及还原Fe3+,同化CO2和有机碳源[46-47]。本课题组的一些研究表明,该属铁氧化能力极强。这类不具细胞壁的古菌在酸性矿山排水的形成和发展中发挥着极其重要的作用[48]。
除铁、硫氧化菌,酸矿水坑中还广泛分布着酸杆菌属、嗜酸菌属、Metallibacterium、铁线菌属、Acidicapsa和硫化芽胞杆菌属等嗜酸、中度嗜酸或耐酸的异养菌,通过利用环境中的有机物,驱动C元素循环,促进铁、硫氧化菌的生长。此外,Acidiphilium acidophilum[49]、Sulfobacillus spp.和Ferrithrix thermotolerans[50]可利用还原型无机硫化合物和S0以及Fe2+或硫化矿物兼性化能无机营养或混合营养生长,促进矿石的氧化性溶解;Acidibacter spp.[51]、Acidiphilium cryptum[52]、Metallibacterium scheffleri[53]、Ft. thermotolerans、酸性矿山排水来源的Acidicapsa spp.[54]和Sulfobacillus spp.中多个成员[55-56]均可在微好氧或厌氧的条件下还原Fe3+,直接参与Fe循环,产生Fe2+供铁氧化菌利用及催化三价铁矿的还原性溶解。结合成熟酸性矿山排水中的一些研究[57]及本研究微生物类群相关性分析的结果推测,Sulfobacillus spp.在极端条件下可能与亚铁原体属协同作用以溶解矿石。
4 结论云南省蒙自某矿区酸性矿山排水环境酸矿水坑和周边溪水的群落结构存在明显差异,pH是对其影响最大的环境因素。酸矿水坑群落多样性较低,但具有丰富的嗜酸性铁、硫氧化菌,以及代谢能力多样的嗜酸性异养菌,是铁、硫元素代谢和生物浸矿等研究和应用的极端菌种资源库。在严苛的环境条件下,酸矿水坑中还蕴含着较多未分类和未培养的微生物类群,它们的生理代谢和环境功能可通过高通量测序数据进行分析,但仍依赖于菌株分离培养来进一步验证;环境中更为复杂的微生物之间的相互作用及其对群落结构的影响也有待进一步研究。另外,嗜酸菌的迁移及其对环境的影响也需引起足够的关注。
| [1] |
Johnson DB. Chemical and microbiological characteristics of mineral spoils and drainage waters at abandoned coal and metal mines. Water Air Soil Pollut, 2003, 3(1): 47-66. |
| [2] |
Akcil A, Koldas S. Acid mine drainage (AMD): causes, treatment and case studies. J Clean Prod, 2006, 14(12/13): 1139-1145. |
| [3] |
Kefeni KK, Msagati TAM, Mamba BB. Acid mine drainage: prevention, treatment options, and resource recovery: a review. J Clean Prod, 2017, 151: 475-493. DOI:10.1016/j.jclepro.2017.03.082 |
| [4] |
Johnson DB, Hallberg KB. Acid mine drainage remediation options: a review. Sci Total Environ, 2005, 338(1/2): 3-14. |
| [5] |
Evangelou VP, Zhang YL. A review: pyrite oxidation mechanisms and acid mine drainage prevention. Crit Rev Environ Sci Technol, 1995, 25(2): 141-199. DOI:10.1080/10643389509388477 |
| [6] |
Singer PC, Stumm W. Acidic mine drainage: the rate-determining step. Science, 1970, 167(3921): 1121-1123. DOI:10.1126/science.167.3921.1121 |
| [7] |
Schippers A, Sand W. Bacterial leaching of metal sulfides proceeds by two indirect mechanisms via thiosulfate or via polysulfides and sulfur. Appl Environ Microbiol, 1999, 65(1): 319-321. |
| [8] |
Baker BJ, Banfield JF. Microbial communities in acid mine drainage. FEMS Microbiol Ecol, 2003, 44(2): 139-152. DOI:10.1016/S0168-6496(03)00028-X |
| [9] |
Johnson DB, Hallberg KB. The microbiology of acidic mine waters. Res Microbiol, 2003, 154(7): 466-473. DOI:10.1016/S0923-2508(03)00114-1 |
| [10] |
Bonnefoy V, Holmes DS. Genomic insights into microbial iron oxidation and iron uptake strategies in extremely acidic environments. Environ Microbiol, 2012, 14(7): 1597-1611. DOI:10.1111/j.1462-2920.2011.02626.x |
| [11] |
Dopson M, Johnson DB. Biodiversity, metabolism and applications of acidophilic sulfur-metabolizing microorganisms. Environ Microbiol, 2012, 14(10): 2620-2631. DOI:10.1111/j.1462-2920.2012.02749.x |
| [12] |
Bond PL, Druschel GK, Banfield JF. Comparison of acid mine drainage microbial communities in physically and geochemically distinct ecosystems. Appl Environ Microbiol, 2000, 66(11): 4962-4971. DOI:10.1128/AEM.66.11.4962-4971.2000 |
| [13] |
Méndez-García C, Mesa V, Sprenger RR, et al. Microbial stratification in low pH oxic and suboxic macroscopic growths along an acid mine drainage. ISME J, 2014, 8(6): 1259-1274. DOI:10.1038/ismej.2013.242 |
| [14] |
Chen LX, Hu M, Huang LN, et al. Comparative metagenomic and metatranscriptomic analyses of microbial communities in acid mine drainage. ISME J, 2015, 9(7): 1579-1592. DOI:10.1038/ismej.2014.245 |
| [15] |
Méndez-García C, Peláez AI, Mesa V, et al. Microbial diversity and metabolic networks in acid mine drainage habitats. Front Microbiol, 2015, 6: 475. |
| [16] |
Kuang JL, Huang LN, He ZL, et al. Predicting taxonomic and functional structure of microbial communities in acid mine drainage. ISME J, 2016, 10(6): 1527-1539. DOI:10.1038/ismej.2015.201 |
| [17] |
Chen LX, Huang LN, Méndez-García C, et al. Microbial communities, processes and functions in acid mine drainage ecosystems. Curr Opin Biotechnol, 2016, 38: 150-158. DOI:10.1016/j.copbio.2016.01.013 |
| [18] |
Johnson DB, Hallberg KB. Carbon, iron and sulfur metabolism in acidophilic micro-organisms. Adv Microb Physiol, 2008, 54: 201-255. DOI:10.1016/S0065-2911(08)00003-9 |
| [19] |
Rawlings DE, Johnson DB. The microbiology of biomining: development and optimization of mineral-oxidizing microbial consortia. Microbiology, 2007, 153(2): 315-324. DOI:10.1099/mic.0.2006/001206-0 |
| [20] |
Li KW, Zhang Q, Wang DP, et al. Isotope geochemistry of Bainiuchang silver polymetallic deposits in Mengzi, Yunnan. Mineral Deposits, 2010, 29(S1): 462-463 (in Chinese). 李开文, 张乾, 王大鹏, 等. 云南蒙自白牛厂银多金属矿床同位素地球化学研究. 矿床地质, 2010, 29(S1): 462-463. |
| [21] |
Chesnin L, Yien CH. Turbidimetric determination of available sulfates. Soil Sci Soc Am Proc, 1950, 15: 149-151. |
| [22] |
Kuang JL, Huang LN, Chen LX, et al. Contemporary environmental variation determines microbial diversity patterns in acid mine drainage. ISME J, 2013, 7(5): 1038-1050. DOI:10.1038/ismej.2012.139 |
| [23] |
Fuhrman JA, Comeau DE, Hagstr m , et al. Extraction from natural planktonic microorganisms of DNA suitable for molecular biological studies. Appl Environ Microbiol, 1988, 54(6): 1426-1429. |
| [24] |
Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [25] |
Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet J, 2011, 17(1): 3. DOI:10.14806/ej.17.1.202 |
| [26] |
Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nat Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [27] |
Edgar RC. UNOISE2: improved error-correction for Illumina 16S and ITS amplicon sequencing. BioRxiv, 2016, 081257. |
| [28] |
Bolyen E, Rideout JR, Dillon MR, et al. Reproducible, interactive, scalable and extensible microbiome data science using QⅡME 2. Nat Biotechnol, 2019, 37(8): 852-857. DOI:10.1038/s41587-019-0209-9 |
| [29] |
Wang Q, Garrity GM, Tiedje JM, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Appl Environ Microbiol, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [30] |
Korzhenkov AA, Toshchakov SV, Bargiela R, et al. Archaea dominate the microbial community in an ecosystem with low-to-moderate temperature and extreme acidity. Microbiome, 2019, 7: 11. DOI:10.1186/s40168-019-0623-8 |
| [31] |
Faith DP. Conservation evaluation and phylogenetic diversity. Biol Conservat, 1992, 61(1): 1-10. DOI:10.1016/0006-3207(92)91201-3 |
| [32] |
Tan S, Liu J, Fang Y, et al. Insights into ecological role of a new deltaproteobacterial order Candidatus Acidulodesulfobacterales by metagenomics and metatranscriptomics. ISME J, 2019, 13(8): 2044-2057. DOI:10.1038/s41396-019-0415-y |
| [33] |
Yelton AP, Comolli LR, Justice NB, et al. Comparative genomics in acid mine drainage biofilm communities reveals metabolic and structural differentiation of co-occurring archaea. BMC Genomics, 2013, 14: 485. DOI:10.1186/1471-2164-14-485 |
| [34] |
Cao ZM, Banda JF, Pei LX, et al. Microbial community structure characteristics in different mine drainage lakes of an iron mine in Anhui Province. Acta Microbiol Sin, 2019, 59(6): 1076-1088 (in Chinese). 曹子敏, Banda JF, 裴理鑫, 等. 安徽某铁矿不同矿山废水库中微生物群落结构特征. 微生物学报, 2019, 59(6): 1076-1088. |
| [35] |
Sánchez-Andrea I, Sanz JL, Stams AJM. Microbacter margulisiae gen. nov., sp. nov., a propionigenic bacterium isolated from sediments of an acid rock drainage pond. Int J Syst Evol Microbiol, 2014, 64(12): 3936-3942. |
| [36] |
Yeoh YK, Sekiguchi Y, Parks DH, et al. Comparative genomics of candidate phylum TM6 suggests that parasitism is widespread and ancestral in this lineage. Mol Biol Evol, 2016, 33(4): 915-927. DOI:10.1093/molbev/msv281 |
| [37] |
Mesa V, Gallego JLR, González-Gil R, et al. Bacterial, archaeal, and eukaryotic diversity across distinct microhabitats in an acid mine drainage. Front Microbiol, 2017, 8: 1756. DOI:10.3389/fmicb.2017.01756 |
| [38] |
Amaral-Zettler LA, Zettler ER, Theroux SM, et al. Microbial community structure across the tree of life in the extreme Río Tinto. ISME J, 2011, 5(1): 42-50. DOI:10.1038/ismej.2010.101 |
| [39] |
Gavrilov SN, Korzhenkov AA, Kublanov IV, et al. Microbial communities of polymetallic deposits' acidic ecosystems of continental climatic zone with high temperature contrasts. Front Microbiol, 2019, 10: 1573. DOI:10.3389/fmicb.2019.01573 |
| [40] |
Johnson DB, Hallberg KB, Hedrich S. Uncovering a microbial enigma: isolation and characterization of the streamer-generating, iron-oxidizing, acidophilic bacterium "Ferrovum myxofaciens". Appl Environ Microbiol, 2014, 80(2): 672-680. DOI:10.1128/AEM.03230-13 |
| [41] |
Hua ZS, Han YJ, Chen LX, et al. Ecological roles of dominant and rare prokaryotes in acid mine drainage revealed by metagenomics and metatranscriptomics. ISME J, 2015, 9(6): 1280-1294. DOI:10.1038/ismej.2014.212 |
| [42] |
Hallberg KB, Coupland K, Kimura S, et al. Macroscopic streamer growths in acidic, metal-rich mine waters in north wales consist of novel and remarkably simple bacterial communities. Appl Environ Microbiol, 2006, 72(3): 2022-2030. DOI:10.1128/AEM.72.3.2022-2030.2006 |
| [43] |
Falagán C, Moya-Beltrán A, Castro M, et al. Acidithiobacillus sulfuriphilus sp. nov.: an extremely acidophilic sulfur-oxidizing chemolithotroph isolated from a neutral pH environment. Int J Syst Evol Microbiol, 2019, 69(9): 2907-2913. DOI:10.1099/ijsem.0.003576 |
| [44] |
Schrenk MO, Edwards KJ, Goodman RM, et al. Distribution of Thiobacillus ferrooxidans and Leptospirillum ferrooxidans: implications for generation of acid mine drainage. Science, 1998, 279(5356): 1519-1522. DOI:10.1126/science.279.5356.1519 |
| [45] |
Golyshina OV, Timmis KN. Ferroplasma and relatives, recently discovered cell wall-lacking archaea making a living in extremely acid, heavy metal-rich environments. Environ Microbiol, 2005, 7(9): 1277-1288. DOI:10.1111/j.1462-2920.2005.00861.x |
| [46] |
Golyshina OV, Pivovarova TA, Karavaiko GI, et al. Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea. nt J Syst Evol Microbiol, 2000, 50(3): 997-1006. DOI:10.1099/00207713-50-3-997 |
| [47] |
Dopson M, Baker-Austin C, Hind A, et al. Characterization of Ferroplasma isolates and Ferroplasma acidarmanus sp. nov., extreme acidophiles from acid mine drainage and industrial bioleaching environments. Appl Environ Microbiol, 2004, 70(4): 2079-2088. DOI:10.1128/AEM.70.4.2079-2088.2004 |
| [48] |
Edwards KJ, Bond PL, Gihring TM, et al. An archaeal iron-oxidizing extreme acidophile important in acid mine drainage. Science, 2000, 287(5459): 1796-1799. DOI:10.1126/science.287.5459.1796 |
| [49] |
Hiraishi A, Nagashima KV, Matsuura K, et al. Phylogeny and photosynthetic features of Thiobacillus acidophilus and related acidophilic bacteria: its transfer to the genus Acidiphilium as Acidiphilium acidophilum comb. nov. Int J Syst Bacteriol, 1998, 48(4): 1389-1398. DOI:10.1099/00207713-48-4-1389 |
| [50] |
Johnson DB, Bacelar-Nicolau P, Okibe N, et al. Ferrimicrobium acidiphilum gen. nov., sp. nov. and Ferrithrix thermotolerans gen. nov., sp. nov.: heterotrophic, iron-oxidizing, extremely acidophilic actinobacteria. Int J Syst Evol Microbiol, 2009, 59(5): 1082-1089. DOI:10.1099/ijs.0.65409-0 |
| [51] |
Falagán C, Johnson DB. Acidibacter ferrireducens gen. nov., sp. nov.: an acidophilic ferric iron-reducing gammaproteobacterium. Extremophiles, 2014, 18(6): 1067-1073.
|
| [52] |
Küsel K, Dorsch T, Acker G, et al. Microbial reduction of Fe(Ⅲ) in acidic sediments: Isolation of Acidiphilium cryptum JF-5 capable of coupling the reduction of Fe(Ⅲ) to the oxidation of glucose. Appl Environ Microbiol, 1999, 65(8): 3633-3640. |
| [53] |
Ziegler S, Waidner B, Itoh T, et al. Metallibacterium scheffleri gen. nov., sp. nov., an alkalinizing gammaproteobacterium isolated from an acidic biofilm. Int J Syst Evol Microbiol, 2013, 63(4): 1499-1504. |
| [54] |
Falagan C, Foesel B, Johnson B. Acidicapsa ferrireducens sp. nov., Acidicapsa acidiphila sp. nov., and Granulicella acidiphila sp. nov.: novel acidobacteria isolated from metal-rich acidic waters. Extremophiles, 2017, 21(3): 459-469. DOI:10.1007/s00792-017-0916-4 |
| [55] |
Bridge TAM, Johnson DB. Reduction of soluble iron and reductive dissolution of ferric iron-containing minerals by moderately thermophilic iron-oxidizing bacteria. Appl Environ Microbiol, 1998, 64(6): 2181-2186. |
| [56] |
Johnson DB, Joulian C, d'Hugues P, et al. Sulfobacillus benefaciens sp. nov., an acidophilic facultative anaerobic Firmicute isolated from mineral bioleaching operations. Extremophiles, 2008, 12(6): 789-798. DOI:10.1007/s00792-008-0184-4 |
| [57] |
Druschel GK, Baker BJ, Gihring TM, et al. Acid mine drainage biogeochemistry at Iron Mountain, California. Geochem Trans, 2004, 5: 13. DOI:10.1186/1467-4866-5-13 |
 2019, Vol. 35
2019, Vol. 35