中国科学院微生物研究所、中国微生物学会主办
文章信息
- 蔺玉萍, 王钦宏
- Lin Yuping, Wang Qinhong
- 工业微生物遗传和环境扰动的调控和适应进化
- Regulation and adaptive evolution of industrial microorganisms towards genetic and environmental disturbances
- 生物工程学报, 2019, 35(10): 1925-1941
- Chinese Journal of Biotechnology, 2019, 35(10): 1925-1941
- 10.13345/j.cjb.190247
-
文章历史
- Received: June 12, 2019
- Accepted: August 30, 2019
- Published: September 17, 2019
在自然环境条件下,地球的生命经历了上亿年的进化,拥有了精细而复杂的遗传调控系统,以支撑其适者生存和繁殖的性状。然后在人类上千年的驯化过程中,一些天然动植物和微生物被自觉与不自觉地改造,演化出积累特定产物以满足人类需要的性状,彼时人类对性状与遗传之间的关系还知之甚少,这个过程主要借助于自然环境的筛选压力[1]。生物经过长期进化,获得特有的生物鲁棒性,从而具备在特定环境下的生存能力和竞争优势。生物鲁棒性是指生物系统在受到环境变化、随机事件(或细胞内噪声)和遗传变异等不确定干扰时保持其表型稳定性的一种特性,是所有生物系统所进化出来的固有特性[2-3]。伴随百年工业发展史和生产规模的不断扩大,工业微生物需要暴露在更为复杂更为严苛的工业环境条件下完成高产高效的生产任务。工业微生物的鲁棒性状体现在能够应对复杂多样的恶劣工业环境,并且具有与正常环境下可比拟的生产性能[4-5]。现代生物产业不仅要求工业微生物具有高产高效的鲁棒性状,还能够兼容扩展底物谱和产物谱,以满足人类社会可持续发展的需求[2]。解析工业微生物鲁棒性状和适配性的遗传调控规律,是构建高性能工业微生物以满足现代生物制造需求的基础。现代生命科学的发展正从认知生命迈向创造生命,不仅为解析工业环境条件下的生物遗传和适应机制提供了强有力工具,而且为人工创制工业微生物的新性状提供依据。
虽然针对生物复杂性状的遗传调控机制已经开展了大量的研究,但是由于细胞代谢、基因调控和信号转导等网络及其之间相互关系的复杂性,加之工业环境条件下细胞暴露在各种胁迫压力下,对于工业环境下细胞的响应和适应机制,我们的理解尚有限。常规的育种手段耗时、耗成本、耗劳动力,想要克服这些问题,需要从系统全局的角度出发,整合最先进的系统生物学、合成生物学和进化工程工具并不断迭代来克服[7-8],不断加深对工业微生物遗传调控机制的理解,从而开发遗传稳定的、鲁棒的、适配的工业微生物。本文从基础研究和实际应用两个角度总结了工业微生物遗传调控和胁迫响应机制以及系统全局改造的研究进展,进而探讨未来工业微生物学在性状的遗传解析和改造方面所面临的挑战和发展方向。
1 工业微生物鲁棒性状的遗传调控与胁迫响应机制伴随系统生物学和合成生物学的发展,我们所能驾驭的工业微生物已经开始突破原有的局限,不仅仅局限于传统发酵所使用的天然生产者和常规代谢工程的底盘细胞[9-11],比如大肠杆菌[12]、酿酒酵母[13]、放线菌[14]、枯草芽孢杆菌[15]等,还包括近年来广受重视的非常规工业微生物,比如解脂耶氏酵母[16]、假单胞菌[17]、需钠弧菌[18]等,从而大大扩展了工业微生物技术的底物谱和产品谱。常规底盘细胞具有相对成熟的工业应用和长期的工程改造研究。与之相比,非常规工业微生物的应用潜力近年来才逐渐被鉴定、认识和开发。以研发用于生产生物燃料的工业微生物为例,通过利用和改造微生物的脂肪酸生物合成来生产高级生物燃料已经有大量的研究。这些研究主要是以大肠杆菌和酿酒酵母为宿主菌,通过引入外源或强化内源脂肪酸合成途径、抑制竞争途径以及体内群体质量控制系统(PopQC)持续筛选高性能菌株等手段,提高工程菌株积累各种高级生物燃料的生产性能[19-22]。然而,目前这些菌株的生产性能尚不能完全满足产业化的要求。近几年来,研究人员把目光投向产油微生物的开发,其中最具代表性的是解脂耶氏酵母Yarrowia lipolytica[23]和不透明红球菌Rhodococcus opacus[24]。解脂耶氏酵母被认为是非致病性的安全菌种。围绕解脂耶氏酵母的改造,其合成生物学工具已经日益成熟,包括DNA组装技术、构建表达盒的DNA元件、基因组编辑技术以及基因组尺度代谢模型的构建等,使得该菌也可以作为生产医药化合物和食品添加剂的良好底盘细胞[25]。最近,韩国KAIST研究所的研究人员采用系统代谢工程的策略,将不透明红球菌改造为高级生物燃料的高产菌,从而为化学品和燃料的可持续生产提供了新的可能性[24]。此外,非常规微生物还可以作为新型功能元件的来源。木糖发酵真菌的比较基因组学研究挖掘了一些与木糖代谢有关的基因,能够显著提高酿酒酵母工程菌的木糖利用,从而促进生物质转化生产生物燃料过程中菌株的研发[26]。高度多样化的子囊菌酵母能够将多种底物转化成乙醇、脂类、维生素等具有工业应用潜力的产物,并且能够在极端的温度、高盐和pH条件下生长。对这些酵母的比较基因组学研究揭示了与有用的代谢性状所关联的基因和遗传基础,不仅有助于相关生物合成途径的工程改造,而且为筛选有应用潜力的工业细胞底盘提供依据[27]。因此,非常规微生物相关系统生物学和合成生物学的研发,对于拓展工业微生物的应用以及揭示其鲁棒性状的遗传基础都将是不可或缺的。
总的来讲,对于天然生产者而言,比如用于食品和饮料发酵的微生物,通常在特定的人为环境下不断驯化,获得了与之相适应的鲁棒性能,包括高效利用特定底物、应对特定工业压力以及积累理想的化合物。而且不同工业环境来源的同一物种的不同谱系,呈现基因型和表型多样性。基于大规模的基因组测序,逐渐揭示这些多样性产生的遗传调控方式包括:种间杂交、基因水平转移、拷贝数变异、基因组衰退、染色体重排等[1]。以被人类长期驯养的、用来生产啤酒和葡萄酒的酵母为例,用于生产拉格(Lager)啤酒的工业菌株巴氏酵母Saccharomyces pastorianus,是酿酒酵母Saccharomyces cerevisiae和贝酵母Saccharomyces bayanus的杂交种,融合了前者强劲的发酵能力和后者的耐寒性状,从而能够进行低温发酵[28]。在许多用于生产葡萄酒的酿酒酵母菌株的基因组中,有一段约158 kb的区域是通过基因水平转移来自于另外一种酵母菌小捕有胞圆酵母Torulaspora microellipsoides,该区域含有编码寡聚肽转运蛋白的FOT基因,能使菌株利用寡聚肽为氮源,从而可以在氮源受限的葡萄酒发酵环境中依然具有发酵活力[29]。此外,葡萄酒酵母耐受亚硫酸盐性能与驯养过程中染色体间发生重排有关,SSU1和ECM34基因上游5′区域非常短的序列之间发生微同源末端重组介导的不等交换,导致8号和16号染色体之间发生易位,从而使SSU1的表达水平提高。SSU1基因编码亚硫酸盐泵,进而赋予菌株更高的亚硫酸盐抗性[30]。葡萄酒酵母的铜胁迫抗性和啤酒酵母高效发酵麦芽糖和麦芽三糖的性能则分别是由于其基因组中存在高拷贝的CUP1基因(编码铜结合蛋白)[31]和MAL基因(编码麦芽糖转运蛋白和水解酶)[32]。用于乳制品生产的乳酸乳球菌Lactococcus lactis,其在牛奶环境的驯化过程同样也伴随着基因组衰退以及通过基因水平转移获得特异利用蛋白质和乳糖的基因[33]。对于用于工程改造的底盘细胞而言,所引入的外源代谢途径往往需要遵循宿主本身的遗传调控规律,包括遗传操作系统、启动子和终止子、编码区的密码子偏好性、转录翻译及其调控网络,或者通过引入合成的调控系统,最终与细胞内源的代谢相平衡[5, 34-36]。
与自然界环境相比,工业微生物所面临的胁迫压力更复杂更严苛,主要总结为以下几个方面(图 1)[37-38]:1)外源代谢途径的引入,可能会造成合成外源蛋白的翻译负担、非折叠蛋白反应和内质网应激、蛋白质稳态失衡、蛋白分泌系统压力、代谢通量负担、破坏氧化还原平衡等;2)与底物相关的胁迫,包括高浓度底物造成的高渗压力、抑制物等[39];3)中间代谢产物和/或终产物积累带来的压力和毒性;4)环境压力:异常温度、异常pH、有机无机酸、抑制化合物、营养缺乏、静压、氧化胁迫、其他生物竞争等。一方面,作为系统生物学的一项技术,组学分析被广泛应用于从系统和网络的层面解析细胞应对不同水平胁迫压力的响应机制;另一方面,反向代谢工程获得的突变菌株,经过组学分析,揭示突变表型背后的遗传基础和调控网络,鉴定和挖掘与提高胁迫抗性和生产性能相关的候选目标。相关的胁迫响应机制包括(图 1)[40-41]:1)细胞壁和细胞膜的屏障作用:细胞壁重构、细胞膜流动性降低、海藻糖聚集等;2)转运系统清理有毒物质;3)信号转导系统的激活和级联调控作用,例如MAPK、TOR等信号转导途径,进而调控转录和翻译;4) DNA遗传水平发生损伤修复突变;5)特定的转录因子被激活或失活,进而影响更多功能基因的表达;6)非折叠蛋白应激,比如热激蛋白分子伴侣促进蛋白折叠;7)线粒体胁迫响应;8)代谢水平的调控,比如平衡氧化还原稳态。
另外,这些胁迫在实际工业生产过程中,可能是组合或者顺序发生,因此细胞应对多重胁迫响应的机制可能涉及上述细胞不同层面的重塑和调控[42]。为了从多个层面解析工业酿酒酵母菌株进行高温浓醪乙醇发酵时的胁迫响应机制,本实验室陆续开展了一些研究。通过对高温发酵条件下的酵母细胞进行蛋白质组分析,发现工业酵母的长时高温胁迫响应不同于实验室菌株的热激响应,在高温条件下,工业酵母的磷酸戊糖途径蛋白表达量降低,而糖酵解途径蛋白表达量提高,这意味着工业酵母进行高温发酵时发生了代谢调控,同时鉴定了一些特异响应长时高温胁迫的蛋白和转录因子[43-44]。通过对耐热特异响应转录因子的敲除菌株进行转录组测序,解析了与耐热相关的基因转录水平上的层级调控网络(未发表)。另外,基于孢子池全基因组测序和数量性状基因座定位策略高效挖掘了与工业酵母高温发酵性能相关的遗传变异,揭示了海藻糖积累和膜流动性降低有利于工业酵母的高温发酵性能的分子基础[45]。这些结果为改造和选育适用于高温浓醪发酵的工业酵母菌种提供了理论依据。
2 工业微生物基因组全局扰动与多位点快速进化目前,绝大多数重要的工业微生物是通过长期的人工驯化和诱变筛选获得。基于物理和化学诱变的育种技术,尽管在一定程度上有效,但存在改造周期长、非定向、遗传机制不清晰和不能突破代谢网络刚性的特点,在创造具有卓越生产性能的工业微生物方面,正面临着许多无法逾越的障碍。伴随分子生物学的发展,针对单基因的基因工程或针对单一代谢途径的代谢工程的学术研究已经很多,但对生产性能的提升效果有限,较难在工业生产上推广使用。由于工业微生物的高产性状通常对应着复杂的基因型,传统育种手段对涉及到十几个、甚至是数十个基因的表达优化无能为力。随着基因组学、系统生物学、合成生物学的发展和高通量分析技术的进步,以全局转录机器工程(Global transcription machinery engineering,gTME)、多位点自动基因组工程(Multiplex automated genome engineering, MAGE)、可跟踪多轮重组工程(Trackable multiplex recombineering, TRMR)和核酸酶介导的基因组编辑为代表的基因组工程技术日益成熟,可以在单一宿主细胞中实现多个不同性状的同时改造,大幅度缩短了改造时间。基因组尺度的理性设计、途径重建、网络重构、全局扰动、多位点快速进化和基因组删减技术可以对工业微生物基因组进行多位点、高通量、高效率修饰。从而赋予工业微生物粗原料利用、新产品合成等新型生产功能,优化工业微生物的发酵生产能力,极大加快工业微生物的改造进程,显著提高改进效果。这些技术已经在氨基酸、大宗化学品、天然化合物药物、蛋白质/工业酶等生产领域取得了一系列标志性成果,为系统改造工业微生物的生产性状提供了全新手段。
2.1 转录因子全局扰动针对细胞非特定生化途径所关联的复杂性状,比如高产和胁迫抗性,其改造提升往往需要对细胞内在的代谢进行彻底的重编程,这就需要同时改变很多基因的表达水平,难以通过连续的多基因修饰来实现。全局转录机器工程(gTME)是通过改造和进化全局转录因子、转录机器等关键蛋白,构建高度多样、复杂的转录调控突变文库,在转录水平上产生新型的丰富多样性,实现对基因表达网络和细胞代谢重编程,并以定向进化方式加以迭代,从而使细胞获得所期望的表型[46]。gTME是一种从转录全局角度工程改造复杂性状的普适性方法。自2006年开发以来[47],该方法已成功地应用于原核生物和真核生物系统,比如酿酒酵母、马克斯克鲁维酵母、大肠杆菌、植物乳杆菌和运动发酵单胞菌等,从而改善细胞的环境胁迫耐受性、代谢产物产量和底物利用效率(表 1)。
| Organism | Transcription factor | Enhanced phenotype | Representative effect | Reference |
| Saccharomyces cerevisiae | Spt15, Taf25 | Ethanol tolerance | 69% increase in volumetric ethanol productivity due to Spt15F177S, Y195H, K218R | [47-48] |
| Spt15 | Xylose fermentation | Mutant strain spt15-25 converted 93.5% of 50 g/L xylose to 15.3 g/L ethanol | [49] | |
| Spt15 | Corn cob acid hydrolysate tolerance | Mutant strain spt15-29 can ferment corn cob hydrolysate containing 68.41 g/L xylose and 7.67 g/L glucose to 11.9 g/L ethanol | [50] | |
| Rpb7 | Ethanol production and tolerance | Improved resistance towards 8 and 10% ethanol, and 40% increase in ethanol titre (~122 g/L) due to Rpb7Y25N, A76T | [51] | |
| Kluyveromyces marxianus | Spt15 | Ethanol production and tolerance | 22.05% increase in ethanol titre (57.29 g/L) due to Spt15K31E when fermenting 140 g/L glucose at 45 C for 48 h | [52] |
| Escherichia coli | Sigma factors | Ethanol tolerance | Improved tolerance to up to 70 g/L ethanol | [53] |
| cAMP receptor protein (CRP) | Lycopene production | Integration of the mutant crpD8V gene resulted in an ~25% increase in lycopene yield of 128 mg/L in a 10-L bench-top fermentor | [54] | |
| cAMP receptor protein (CRP) | Isobutanol tolerance | Mutant CRPS179P, H199R resulted in much better growth (0.18 h-1) than the control (0.05 h-1) in 1.2% (V/V) isobutanol (9.6 g/L) | [55] | |
| Catabolite repressor/activator (Cra) | Succinate production | The strain with Cra mutations at R57K, A58G, G59Q, R60Q, S75H, T76Y, D148I, R149I showed the best succinate production of 79.8 g/L, 22.8% greater than control | [56-57] | |
| Sigma factor σ70 | Solvent tolerance | Improved tolerance to up to 69% cyclohexane due to σ70 mutations at D39E, A72V, T94M and a stop codon mutation at residue 123 | [58] | |
| Histone-like nucleoid structuring factor (H-NS) | Acid tolerance | Five H-NS mutants containing multiple mutations in the promoter and coding regions resulted in enhanced cell growth by 26%–53%, and survival rates by 10- to 100-fold at pH 2.5 | [59] | |
| Lactobacillus plantarum | Sigma factor | Lactic and inorganic acid tolerance | One σ-factor mutation at Q345K resulted in a 3.5-fold faster growth and up to a 5-fold higher OD in the lactic acid condition. The other σ-factor mutation at at Q345K or at T44A, R74K, D114A, and S119A resulted in a 86.4% higher OD and a 25% higher growth rate in the HCl condition | [60] |
| Zymomonas mobilis | Sigma factor σ70 | Ethanol tolerance | Four mutants (P195T; D203V, R324H, A592V, L606S; G97S, D206E, M369L, E370D, E573G;Q57L, G426C, I448N) resulted in elevated ethanol tolerance | [61] |
在自然界没有人工参与的情况下,生物体受自然诱变剂的作用或在DNA复制、转录、修复时以一定频率(约10–9–10–6)自然发生突变,产生遗传和表型多样性,推进生物进化。但这个过程非常缓慢,通过人工模拟自然进化过程,改造和突变基因组的进化动力,可以建立加快进化速率的基因组全局扰动新技术[62]。该技术会让上百种或上千种变异同时进行,从而制造出十亿种不同的DNA序列。利用细胞自身的进化机制,经过无数次的变异,使得细胞在短期内被赋予所期望的特性,使其生产特定产物具有更为低廉的成本和更短的研发周期。
多位点自动基因组工程(MAGE)是近年来发展的针对基因组中多条相关代谢途径进行快速改造的重要方法[63-64]。MAGE基于λ噬菌体Red重组酶的同源重组系统,针对与某条特定的代谢途径或局部的代谢网络进行理性设计,人工合成的多个兼并寡核苷酸靶向基因组上的特定序列,通过同源重组的方式靶向序列,将许多已知具有最为优良功能的基因整合在一起,从而获得具有最优工业性能的工业微生物。例如,现已知至少有20种基因影响番茄红素的生产,一次改写一个或一部分基因的常规基因工程技术显然不能满足需求。为了解决这一问题,哈佛大学的研究人员利用MAGE改造大肠杆菌生产番茄红素[63]。此外,为了进一步提高MAGE进行基因组进化的效率,通过引入co-marks建立改进后的共选择Co-selection MAGE,可以修饰大肠杆菌基因组80个位点。通过破坏复制体中引发酶和解旋酶之间的相互作用,提高ssDNA在复制叉处的丰度,进一步提高了每轮操作的修饰效率[65-66]。MAGE技术应用于酿酒酵母中,建立的eMAGE技术独立于Rad51介导的同源重组,避免了DNA双链断裂造成的意外突变,一次可以同时转化具有60个靶向突变的12个寡核苷酸,迭代转化寡核苷酸混合库,能够快速产生大于105个的基因组组合突变。该技术在异源胡萝卜素生物合成途径基因的启动子、基因和终止子引入精确突变,产生遗传多样性,从而调控类胡萝卜素的产物水平[67]。为了同时鉴定MAGE基因组进化过程中与性状相关联的遗传基础,进而发展了可跟踪多轮重组工程(TRMR)技术,通过合成带有条形码的突变序列,转化宿主细胞,然后利用分子条码和微阵列技术追踪相关突变,量化突变率,并对遗传性状进行定位。该技术能在一天之内修饰大肠杆菌95%的基因,并在一周内完成影响细胞不同培养基(丰富、基础和纤维素水解液)和抑制物(β-糖苷、D-岩藻糖、缬氨酸和丙酮醛)条件下生长的近4 000个基因的遗传定位[68]。
基因组复制工程辅助的连续进化(Genome replication engineering assisted continuous evolution, GREACE技术)[69]是将保真性下降的DNA聚合酶元件引入到大肠杆菌中,诱发细胞进入高突变态,从而在复制过程中不断产生基因组突变,在环境压力下,积累有益突变的子代细胞被筛选出来。不断提高环境压力就可以使大肠杆菌不断地适应新的压力,达到连续进化的目的。利用该技术可以提高大肠杆菌的耐热性和甲醇利用能力[69-70]。
基于合成染色体和Cre-loxP位点特异性重组机制,最近开发的基因组重排系统(Synthetic chromosome rearrangement and modification by loxP-mediated evolution,SCRaMbLE)可以产生全基因组尺度的、大片段的DNA缺失、重复、易位、倒位和复杂的基因组重排事件,实现酿酒酵母基因组的快速进化[71-72]。利用该技术可以提高底盘细胞与外源路径间的适配性,实现代谢通路的优化,提高了酿酒酵母细胞工厂类胡萝卜素、紫罗兰素、紫色杆菌素、青霉素的生产能力[73-75]以及耐受环境胁迫的能力[76-77]。另外,通过对基因组序列结构变化的解析可以鉴定与表型相关的基因型。
2.3 基因组删减优化模式生物基因组测序和分析揭示了大量的生存非必需的序列,以及大量未知功能的基因。为了理解基因组的功能,构建最小基因组的细胞工厂,在大肠杆菌、枯草芽孢杆菌、酿酒酵母、裂殖酵母等模式微生物和有工业应用价值的微生物(如谷氨酸棒杆菌)中已经开展了基因组删减优化的工作[78-81]。所采用的DNA敲除技术从早期的转座子随机整合/缺失、转座子协同Cre/loxP切除,到最近发展的Red介导/双链断裂促进的重组、无疤痕缺失等。如Cre/loxP系统可以用于染色体间的基因重排,为研究多种最小基因组的可能性提供了一个很好的技术手段[82]。新的技术能够做到高效、精确和连续的DNA片段的敲除,目前最多的已敲除了约30%的基因组序列。随着基因组计算机辅助设计软件的开发,大片段DNA拼接合成技术的发展,可以在计算机上进行基因组的删减优化,包括删除转座子、去除冗余DNA片段以及重新安置易造成基因组不稳定的tRNA基因等方式,之后采取大片段DNA从头合成并转入酵母细胞逐步置换的方法,创建数字化基因组删减与逐步置换合成相结合的基因组改造创新技术体系[83]。Ikeda研究小组将一株原来不产链霉素、头霉素和普拉地内酯的链霉菌,分别改造为高产这3种抗生素的工业菌株,并使其基因组缩小至原野生菌株的83.12%–81.46%,不仅极大地提高了菌株的生长速度,而且困扰这些抗生素多年的无效、有毒副产物均未检测到[84]。
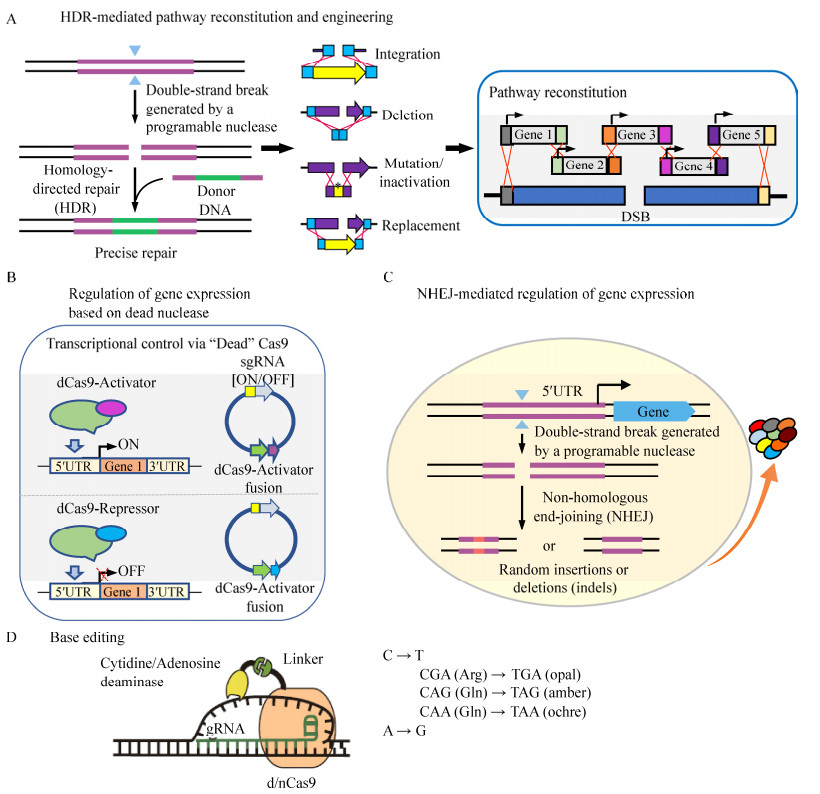
2.4 基因组多位点编辑进化近年来,基于核酸酶的基因组编辑技术,特别是CRISPR技术,被广泛应用于基础研究、生物技术、基因治疗等领域[85]。基因组多位点编辑进化,包括在全基因组尺度上搜索有利于目标产物合成的基因表达或基因敲除,并通过循环筛选得到优化的基因表达和敲除组合;快速实现在基因组上多个位点同时进行突变、缺失或插入短序列等修饰。根据是否引入DNA损伤以及细胞内源组分的参与情况,核酸酶介导的基因组编辑技术在生物技术领域中的应用归纳为以下4个方面(图 2)。
|
| 图 2 核酸酶介导的基因组编辑技术在生物技术领域的应用(参考文献[85]修改) Fig. 2 Applications of nuclease-mediated genome engineering in biotechnology (modified from [85]). (A) HDR-mediated pathway reconstitution and engineering. (B) Regulation of gene expression based on dead nuclease. (C) NHEJ-mediated regulation of gene expression. (D) Base editing. |
| |
1) 同源重组介导的途径构建与改造(图 2A):编辑系统的核酸酶切割靶向DNA位点,产生DNA双链断裂(DSB),在有与靶向位点同源臂的外源DNA片段供体存在的情况下,DSB经由细胞内源同源重组系统(HDR)进行修复,实现同源片段的整合、敲除、突变、失活或者替换,进而实现生物合成途径的构建与改造[86]。
2) 基于失活核酸酶的基因表达调控(图 2B):利用失活的核酸酶,融合细胞内源激活子或抑制子,对基因表达进行调控,从而实现外源生物合成途径以及与之适配的底盘细胞的优化。最近,哈佛大学Church实验室通过CRISPRi确定了需钠弧菌快速生长所需的最小基因集[87]。
3) 非同源末端重组介导的基因表达调控(图 2C):在没有外源同源重组DNA片段存在的情况下,核酸酶切割产生的DSB主要经由细胞内源非同源末端重组(NHEJ)系统进行修复,引入插入或缺失随机突变,当靶向基因启动子区时,能够引起编辑基因的差异表达,形成多性状突变库,从而为工业微生物突变育种提供了新策略。本实验室基于TALENs特异性识别修饰真核启动子区保守序列TATA框与GC框之间的关键区域,引起不同基因的差异表达,形成多性状菌体库,在酿酒酵母中建立了TALENs介导的多位点基因组编辑技术,并辅助酵母胁迫抗性进化育种,提高了工业菌株耐受高温和高渗的性能[88-89]。
4) 碱基编辑(图 2D):将CRISPR/Cas9 (或Cpf1)系统中的核酸酶失活或突变为切口酶,并融合胞嘧啶或腺苷酸脱氨酶,利用CRISPR系统的定位功能与脱氨酶的碱基编辑功能,可在染色体靶位点实现C到T或A到G的编辑,实现碱基突变,当碱基位于基因编码区时则有可能造成氨基酸突变,当靶向基因编码区中编码精氨酸的CGA、编码谷氨酰胺的CAG和CAA中的C突变为T时,分别会产生TGA、TAG和TAA终止密码子,从而造成基因失活。最近,在重要工业平台微生物谷氨酸棒杆菌中开发的多元自动化基因组编辑方法MACBETH (Multiplex automated corynebacterium glutamicum base editing method)[90],结合CRISPR/Cas9系统的定位功能与胞嘧啶脱氨酶(AID)的碱基编辑功能,可在染色体靶位点实现C到T的编辑,效率高达90%。MACBETH可同时在多个基因中生成提前的终止密码子,以失活靶基因。同时借助自动化平台,可实现从质粒构建、基因组编辑、获取正确突变株和表型验证的全流程自动化操作,编辑能力可达到每月数千突变株,为将谷氨酸棒杆菌改造为通用的微生物底盘提供强大的技术支持。
2.5 基因组适应性进化对于遗传基础复杂难以确定的性状改造,基因组适应性进化也是一种行之有效的方法,同时通过蛋白、转录和基因组学技术解析在适应性进化过程中发生的响应与调控机制,再通过反向代谢工程等手段,进一步稳定或增强这种特性以达到获得优良性状的目的。借助于基因组适应性进化,一些大肠杆菌、酿酒酵母、谷氨酸棒杆菌、肠膜明串珠菌等细胞工厂的工业生产效率和胁迫耐受性都得到了很大的提升[91-92]。系列传代和连续培养是适应性进化的两种经典设计,针对特定的工业性状育种目标,施加单一的或多重的或变化的环境筛选压力,使得突变在全基因组尺度上广泛积累,然后通过特定的生理指标检测筛选目标突变体[93]。值得一提的是,相关自动化设备以及高通量的突变体分离和筛选方法已被大量开发和应用,从而节省适应性进化实验的人力成本并提高筛选效率[91, 93]。本实验室设计了一款自动化程序控制、培养体积小且可并行运行多种筛选培养条件的微生物适应性进化培养设备[94]。该设备包括中央控制系统与若干执行单元(培养条件控制单元、培养瓶单元、原料储罐及培养物收集罐单元),具有程序编辑与控制模块以及若干个蠕动泵。培养条件控制单元由一个培养温度控制的循环水槽和一组磁力搅拌器组成;培养瓶单元包括带刻度的瓶体、瓶盖及与之相连的管线及监测部件,培养瓶单元放置于循环水槽中,磁力搅拌器转子置于所述培养瓶,培养瓶通过其管路与蠕动泵连接;原料储罐与培养物收集罐单元具有若干个培养基原料储罐及培养物排出液收集罐,培养瓶通过其管路与培养基原料储罐及培养物排出液收集罐相连接。该设备能够用来进行细菌、酵母、真菌等微生物及可悬浮培养细胞的系列传代培养工作,适用于生物、医药、农业等科研或工厂实验室中,并用来替代目前常用的传统的适应性进化培养实验工作,节省人力成本并实现实验过程的标准化高效运行。利用该设备,我们通过耐高温驯化和/或模拟工业发酵条件对工业酿酒酵母菌株进行适应性进化,获得一系列适应性进化菌株,同时利用基因组重测序揭示了进化菌株中积累的非同义突变和大片段缺失[95]。相较于普通产乙醇工业酵母发酵生产乙醇,进化菌株的乙醇产量均明显提高,能够耐受高温、高浓醪和抑制物等多重胁迫,解决高温浓醪发酵淀粉生产酒精中酵母细胞多重胁迫耐性不足的缺陷。在高温下发酵生产乙醇,可大幅度降低工业用水的用量,从而减少能量消耗,降低乙醇生产成本,而且进化菌株能够利用工业用水进行大规模发酵生产乙醇,进一步降低了乙醇的生产成本,在大规模工业生产中具有很大的潜力。
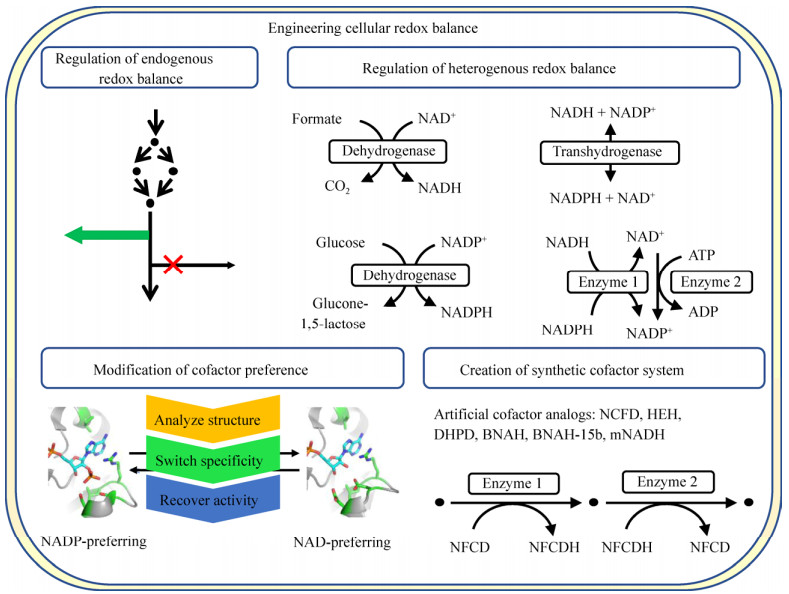
3 工业微生物细胞水平氧还平衡的全局扰动辅因子对NADH/NAD+和NADPH/NADP+在所有生物有机体中起着重要的电子供体或受体的作用,驱动大量的分解和合成代谢反应,参与细胞的一系列生理功能,包括调节能量代谢、细胞内氧化还原状态、控制碳流量、提高线粒体的功能和活性、调节细胞生命周期和细胞毒力等。工业微生物工程改造所改变或引入的代谢途径,特别是涉及消耗、生成或转化辅因子的代谢途径,往往会引起氧化还原状态的波动,从而严重阻碍细胞代谢,导致细胞生长和生物合成能力的下降。因此,除了途径改造外,氧化还原平衡也是工程改造细胞利用特定底物生产目标产物的重要考虑因素。通过辅因子工程,维持胞内氧化还原平衡,增加目标产物生物合成途径的代谢通量,提高生产效率,是较早发展起来的系统改造手段之一,广泛应用于提高酶、药物、生物燃料、大宗化学品等细胞工厂的生产性能。一些综述文献总结了辅因子工程的相关策略和应用[96-101],4种基本策略包括(图 3):
1) 调节内源辅因子系统,改善自平衡,比如辅因子竞争途径的消除或阻断,辅因子生成途径的加强,以及微调氧化还原相关转录调控因子等。例如,NADPH是一些高附加值化学品生物合成途径所需的重要辅因子。细胞能否有足够的NADPH是构建这类细胞工厂的限制因素之一。大肠杆菌转录调控因子HdfR,其功能缺失能够提高细胞内NADPH的含量,从而提高3-羟基丙酸的产量[102]。
2) 在宿主中增补异源的辅因子再生系统,比如增加NAD与NADP之间转化的酶或途径,增强NAD合成等,反之亦然。Ng等基于运动发酵单胞菌的Entner-Doudoroff (ED)途径,对其进行理性设计,并引入到大肠杆菌中,将NADPH产生效率提高了25倍[103]。
3) 替换具有特定辅因子特异性的酶或改造酶的辅因子特异性。最近,Arnold实验室开发了一个将酶对NADP依赖性逆转为NAD依赖性的在线工具(Cofactor specificity reversal-structural analysis and library design,CSR-SALAD),基于蛋白结构分析和半理性设计,成功将4种结构不同的NADP依赖酶(乙醛酸还原酶、肉桂醇脱氢酶、木糖还原酶和含铁醇脱氢酶)的辅因子特异性逆转为NAD依赖[104]。
4) 创建合成的辅因子系统,比如利用人工的辅因子类似物。基于氧化还原反应的生物转化对于工业生物制造具有潜在的吸引力,但需要依赖于不稳定且昂贵的辅因子。Knaus等鉴定了一些人工合成的辅因子类似物,其催化活性甚至比NAD(P)H还好[105]。另外,这些策略的组合也具有可行性和有效性。
4 展望开发和改进工业微生物的鲁棒性和适配性,比如高产和胁迫抗性,是发展生物产业的基础。然而这些性状是由多层次的复杂调控网络控制,仅仅基于对模式生物和非工业环境所开展的研究尚不能全面解析相关的遗传调控机理。未来依然需要借助于系统生物学的分析手段,加大对工业微生物的基因组学和功能基因组学分析,揭示重要工业微生物遗传操作的限制修饰系统,分析工业环境下高产性状的分子机制,挖掘高产性状相关的特殊遗传标记与基因,构建基因组规模的代谢网络以及调控网络模型,最终为实现可预测、可控制的工业微生物提出更为全面可靠的理性设计改造方案。同时,由于传统单一基因、单一途径、单一策略的代谢工程改造对于复杂性状提升的空间有限,需要借助合成生物学的工程改造手段,对工业微生物开展全基因组水平的、系统的、全局的工程改造扰动,同时结合自动化和高通量分析手段,多次迭代,从而缩短育种周期,获得工业生产所需的优良工业微生物。另外,人工创制生物在工业应用时涉及到转基因的问题,如何加强人工创制生物的安全性管控和策略以及提高民众对转基因生物的认知也至关重要。
| [1] |
Steensels J, Gallone B, Voordeckers K, et al. Domestication of industrial microbes. Curr Biol, 2019, 29(10): R381-R393. DOI:10.1016/j.cub.2019.04.025 |
| [2] |
Kitano H. Biological robustness. Nat Rev Genet, 2004, 5(11): 826-837. DOI:10.1038/nrg1471 |
| [3] |
Stelling J, Sauer U, Szallasi Z, et al. Robustness of cellular functions. Cell, 2004, 118(6): 675-685. DOI:10.1016/j.cell.2004.09.008 |
| [4] |
Zhu LJ, Zhu Y, Zhang YP, et al. Engineering the robustness of industrial microbes through synthetic biology. Trends Microbiol, 2012, 20(2): 94-101. DOI:10.1016/j.tim.2011.12.003 |
| [5] |
Gong ZW, Nielsen J, Zhou YJ. Engineering robustness of microbial cell factories. Biotechnol J, 2017, 12(10): 1700014. DOI:10.1002/biot.201700014 |
| [6] |
Adrio JL, Demain AL. Recombinant organisms for production of industrial products. Bioeng Bugs, 2010, 1(2): 116-131. DOI:10.4161/bbug.1.2.10484 |
| [7] |
Lee SY, Kim HU. Systems strategies for developing industrial microbial strains. Nat Biotechnol, 2015, 33(10): 1061-1072. DOI:10.1038/nbt.3365 |
| [8] |
Gustavsson M, Lee SY. Prospects of microbial cell factories developed through systems metabolic engineering. Microb Biotechnol, 2016, 9(5): 610-617. DOI:10.1111/1751-7915.12385 |
| [9] |
Friedman DC, Ellington AD. Industrialization of biology. ACS Synth Biol, 2015, 4(10): 1053-1055. DOI:10.1021/acssynbio.5b00190 |
| [10] |
Calero P, Nikel PI. Chasing bacterial chassis for metabolic engineering: a perspective review from classical to non-traditional microorganisms. Microb Biotechnol, 2019, 12(1): 98-124. |
| [11] |
Steensels J, Snoek T, Meersman E, et al. Improving industrial yeast strains: exploiting natural and artificial diversity. FEMS Microbiol Rev, 2014, 38(5): 947-995. DOI:10.1111/1574-6976.12073 |
| [12] |
Castiñeiras TS, Williams SG, Hitchcock AG, et al. E. coli strain engineering for the production of advanced biopharmaceutical products. FEMS Microbiol Lett, 2018, 365(15): fny162. DOI:10.1093/femsle/fny162 |
| [13] |
Nielsen J, Larsson C, van Maris A, et al. Metabolic engineering of yeast for production of fuels and chemicals. Curr Opin Biotechnol, 2013, 24(3): 398-404. DOI:10.1016/j.copbio.2013.03.023 |
| [14] |
Palazzotto E, Tong YJ, Lee SY, et al. Synthetic biology and metabolic engineering of actinomycetes for natural product discovery. Biotechnol Adv, 2019, 37(6): 107366. DOI:10.1016/j.biotechadv.2019.03.005 |
| [15] |
Liu YF, Liu L, Li JH, et al. Synthetic biology toolbox and chassis development in Bacillus subtilis. Trends Biotechnol, 2019, 37(5): 548-562. DOI:10.1016/j.tibtech.2018.10.005 |
| [16] |
Zhu Q, Jackson EN. Metabolic engineering of Yarrowia lipolytica for industrial applications. Curr Opin Biotechnol, 2015, 36: 65-72. DOI:10.1016/j.copbio.2015.08.010 |
| [17] |
Nikel PI, Martínez-García E, de Lorenzo V. Biotechnological domestication of pseudomonads using synthetic biology. Nat Rev Microbiol, 2014, 12(5): 368-379. DOI:10.1038/nrmicro3253 |
| [18] |
Weinstock MT, Hesek ED, Wilson CM, et al. Vibrio natriegens as a fast-growing host for molecular biology. Nat Methods, 2016, 13(10): 849-851. DOI:10.1038/nmeth.3970 |
| [19] |
Peralta-Yahya PP, Keasling JD. Advanced biofuel production in microbes. Biotechnol J, 2010, 5(2): 147-62. DOI:10.1002/biot.200900220 |
| [20] |
Meadows CW, Kang A, Lee TS. Metabolic engineering for advanced biofuels production and recent advances toward commercialization. Biotechnol J, 2018, 13(1). DOI:10.1002/biot.201600433 |
| [21] |
Zhou YJ, Buijs NA, Zhu ZW, et al. Production of fatty acid-derived oleochemicals and biofuels by synthetic yeast cell factories. Nat Commun, 2016, 7: 11709. DOI:10.1038/ncomms11709 |
| [22] |
Xiao Y, Bowen CH, Liu D, et al. Exploiting nongenetic cell-to-cell variation for enhanced biosynthesis. Nat Chem Biol, 2016, 12(5): 339-344. DOI:10.1038/nchembio.2046 |
| [23] |
Ekas H, Deaner M, Alper HS. Recent advancements in fungal-derived fuel and chemical production and commercialization. Curr Opin Biotechnol, 2019, 57: 1-9. |
| [24] |
Kim HM, Chae TU, Choi SY, et al. Engineering of an oleaginous bacterium for the production of fatty acids and fuels. Nat Chem Biol, 2019, 15(7): 721-729. DOI:10.1038/s41589-019-0295-5 |
| [25] |
Larroude M, Rossignol T, Nicaud JM, et al. Synthetic biology tools for engineering Yarrowia lipolytica. Biotechnol Adv, 2018, 36(8): 2150-2164. DOI:10.1016/j.biotechadv.2018.10.004 |
| [26] |
Wohlbach DJ, Kuo A, Sato TK, et al. Comparative genomics of xylose-fermenting fungi for enhanced biofuel production. Proc Natl Acad Sci USA, 2011, 108(32): 13212-13217. DOI:10.1073/pnas.1103039108 |
| [27] |
Riley R, Haridas S, Wolfe KH, et al. Comparative genomics of biotechnologically important yeasts. Proc Natl Acad Sci USA, 2016, 113(35): 9882-9887. DOI:10.1073/pnas.1603941113 |
| [28] |
Dunn B, Sherlock G. Reconstruction of the genome origins and evolution of the hybrid lager yeast Saccharomyces pastorianus. Genome Res, 2008, 18(10): 1610-1623. DOI:10.1101/gr.076075.108 |
| [29] |
Marsit S, Mena A, Bigey F, et al. Evolutionary advantage conferred by an eukaryote-to-eukaryote gene transfer event in wine yeasts. Mol Biol Evol, 2015, 32(7): 1695-1707. DOI:10.1093/molbev/msv057 |
| [30] |
Pérez-Ortín JE, Querol A, Puig S, et al. Molecular characterization of a chromosomal rearrangement involved in the adaptive evolution of yeast strains. Genome Res, 2002, 12(10): 1533-1539. DOI:10.1101/gr.436602 |
| [31] |
Strope PK, Skelly DA, Kozmin SG, et al. The 100-genomes strains, an S. cerevisiae resource that illuminates its natural phenotypic and genotypic variation and emergence as an opportunistic pathogen. Genome Res, 2015, 25(5): 762-774. DOI:10.1101/gr.185538.114 |
| [32] |
Gallone B, Steensels J, Prahl T, et al. Domestication and divergence of Saccharomyces cerevisiae beer yeasts. Cell, 2016, 166(6): 1397-1410. DOI:10.1016/j.cell.2016.08.020 |
| [33] |
Cavanagh D, Fitzgerald GF, McAuliffe O. From field to fermentation: the origins of Lactococcus lactis and its domestication to the dairy environment. Food Microbiol, 2015, 47: 45-61. DOI:10.1016/j.fm.2014.11.001 |
| [34] |
Keasling JD. Manufacturing molecules through metabolic engineering. Science, 2010, 330(6009): 1355-1358. DOI:10.1126/science.1193990 |
| [35] |
Woolston BM, Edgar S, Stephanopoulos G. Metabolic engineering: past and future. Annu Rev Chem Biomol Eng, 2013, 4: 259-288. DOI:10.1146/annurev-chembioeng-061312-103312 |
| [36] |
Nielsen J, Keasling JD. Engineering cellular metabolism. Cell, 2016, 164(6): 1185-1197. DOI:10.1016/j.cell.2016.02.004 |
| [37] |
Guan NZ, Li JH, Shin HD, et al. Microbial response to environmental stresses: from fundamental mechanisms to practical applications. Appl Microbiol Biotechnol, 2017, 101(10): 3991-4008. DOI:10.1007/s00253-017-8264-y |
| [38] |
Takagi H, Kitagaki H. Stress Biology of Yeasts and Fungi: Applications for Industrial Brewing and Fermentation. New York: Springer, 2015, 1-218. |
| [39] |
Wendisch VF, Brito LF, Gil Lopez M, et al. The flexible feedstock concept in industrial biotechnology: Metabolic engineering of Escherichia coli, Corynebacterium glutamicum, Pseudomonas, Bacillus and yeast strains for access to alternative carbon sources. J Biotechnol, 2016, 234: 139-157. DOI:10.1016/j.jbiotec.2016.07.022 |
| [40] |
Fu Y, Chen L, Zhang W. Regulatory mechanisms related to biofuel tolerance in producing microbes. J Appl Microbiol, 2016, 121(2): 320-332. DOI:10.1111/jam.13162 |
| [41] |
Teixeira MC, Mira NP, Sá-Correia I. A genome-wide perspective on the response and tolerance to food-relevant stresses in Saccharomyces cerevisiae. Curr Opin Biotechnol, 2011, 22(2): 150-156. DOI:10.1016/j.copbio.2010.10.011 |
| [42] |
Kitichantaropas Y, Boonchird C, Sugiyama M, et al. Cellular mechanisms contributing to multiple stress tolerance in Saccharomyces cerevisiae strains with potential use in high-temperature ethanol fermentation. AMB Express, 2016, 6: 107. DOI:10.1186/s13568-016-0285-x |
| [43] |
Shui WQ, Xiong Y, Xiao WD, et al. Understanding the mechanism of thermotolerance distinct from heat shock response through proteomic analysis of industrial strains of Saccharomyces cerevisiae. Mol Cell Proteom, 2015, 14(7): 1885-1897. DOI:10.1074/mcp.M114.045781 |
| [44] |
Xiao WD, Duan XX, Lin YP, et al. Distinct proteome remodeling of industrial Saccharomyces cerevisiae in response to prolonged thermal stress or transient heat shock. J Proteome Res, 2018, 17(5): 1812-1825. DOI:10.1021/acs.jproteome.7b00842 |
| [45] |
Wang Z, Qi Q, Lin YP, et al. QTL analysis reveals genomic variants linked to high-temperature fermentation performance in the industrial yeast. Biotechnol Biofuels, 2019, 12: 59. DOI:10.1186/s13068-019-1398-7 |
| [46] |
Lanza AM, Alper HS. Using transcription machinery engineering to elicit complex cellular phenotypes//Weber W, Fussenegger M, Eds. Synthetic Gene Networks. New York: Humana Press, 2012, 813: 229-248.
|
| [47] |
Alper H, Moxley J, Nevoigt E, et al. Engineering yeast transcription machinery for improved ethanol tolerance and production. Science, 2006, 314(5805): 1565-1568. DOI:10.1126/science.1131969 |
| [48] |
El-Rotail AAMM, Zhang L, Li YR, et al. A novel constructed SPT15 mutagenesis library of Saccharomyces cerevisiae by using gTME technique for enhanced ethanol production. AMB Express, 2017, 7: 111. DOI:10.1186/s13568-017-0400-7 |
| [49] |
Liu HM, Yan M, Lai CG, et al. gTME for improved xylose fermentation of Saccharomyces cerevisiae. Appl Biochem Biotechnol, 2010, 160(2): 574-582. DOI:10.1007/s12010-008-8431-9 |
| [50] |
Liu HM, Liu K, Yan M, et al. gTME for improved adaptation of Saccharomyces cerevisiae to corn cob acid hydrolysate. Appl Biochem Biotechnol, 2011, 164(7): 1150-1159. DOI:10.1007/s12010-011-9201-7 |
| [51] |
Qiu ZL, Jiang RR. Improving Saccharomyces cerevisiae ethanol production and tolerance via RNA polymerase Ⅱ subunit Rpb7. Biotechnol Biofuels, 2017, 10: 125. DOI:10.1186/s13068-017-0806-0 |
| [52] |
Li PS, Fu XF, Li SZ, et al. Engineering TATA-binding protein Spt15 to improve ethanol tolerance and production in Kluyveromyces marxianus. Biotechnol Biofuels, 2018, 11: 207. DOI:10.1186/s13068-018-1206-9 |
| [53] |
Alper H, Stephanopoulos G. Global transcription machinery engineering: a new approach for improving cellular phenotype. Metab Eng, 2007, 9(3): 258-267. DOI:10.1016/j.ymben.2006.12.002 |
| [54] |
Huang L, Pu Y, Yang XL, et al. Engineering of global regulator cAMP receptor protein (CRP) in Escherichia coli for improved lycopene production. J Biotechnol, 2015, 199: 55-61. DOI:10.1016/j.jbiotec.2015.02.006 |
| [55] |
Chong HQ, Geng HF, Zhang HF, et al. Enhancing E. coli isobutanol tolerance through engineering its global transcription factor cAMP receptor protein (CRP). Biotechnol Bioeng, 2014, 111(4): 700-708. DOI:10.1002/bit.25134 |
| [56] |
Wei LN, Zhu LW, Tang YJ. Succinate production positively correlates with the affinity of the global transcription factor Cra for its effector FBP in Escherichia coli. Biotechnol Biofuels, 2016, 9: 264. DOI:10.1186/s13068-016-0679-7 |
| [57] |
Zhu LW, Xia ST, Wei LN, et al. Enhancing succinic acid biosynthesis in Escherichia coli by engineering its global transcription factor, catabolite repressor/activator (Cra). Sci Rep, 2016, 6: 36526. DOI:10.1038/srep36526 |
| [58] |
Zhang F, Qian XH, Si HM, et al. Significantly improved solvent tolerance of Escherichia coli by global transcription machinery engineering. Microb Cell Fact, 2015, 14: 175. DOI:10.1186/s12934-015-0368-4 |
| [59] |
Gao XX, Yang XF, Li JH, et al. Engineered global regulator H-NS improves the acid tolerance of E. coli. Microb Cell Fact, 2017, 17: 118. DOI:10.1186/s12934-018-0966-z |
| [60] |
Klein-Marcuschamer D, Stephanopoulos G. Assessing the potential of mutational strategies to elicit new phenotypes in industrial strains. Proc Natl Acad Sci USA, 2008, 105(7): 2319-2324. DOI:10.1073/pnas.0712177105 |
| [61] |
Tan FR, Wu B, Dai LC, et al. Using global transcription machinery engineering (gTME) to improve ethanol tolerance of Zymomonas mobilis. Microb Cell Fact, 2016, 15: 4. DOI:10.1186/s12934-015-0398-y |
| [62] |
Yang J, Kim B, Kim GY, et al. Synthetic biology for evolutionary engineering: from perturbation of genotype to acquisition of desired phenotype. Biotechnol Biofuels, 2019, 12: 113. DOI:10.1186/s13068-019-1460-5 |
| [63] |
Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894-898. DOI:10.1038/nature08187 |
| [64] |
Singh V, Braddick D. Recent advances and versatility of MAGE towards industrial applications. Syst Synth Biol, 2015, 9(S1): 1-9. DOI:10.1007/s11693-015-9184-8 |
| [65] |
Carr PA, Wang HH, Sterling B, et al. Enhanced multiplex genome engineering through co-operative oligonucleotide co-selection. Nucleic Acids Res, 2012, 40(17): e132. DOI:10.1093/nar/gks455 |
| [66] |
Lajoie MJ, Gregg CJ, Mosberg JA, et al. Manipulating replisome dynamics to enhance lambda Red-mediated multiplex genome engineering. Nucleic Acids Res, 2012, 40(22): e170. DOI:10.1093/nar/gks751 |
| [67] |
Barbieri EM, Muir P, Akhuetie-Oni BO, et al. Precise editing at DNA replication forks enables multiplex genome engineering in eukaryotes. Cell, 2017, 171(6): 1453-1467. DOI:10.1016/j.cell.2017.10.034 |
| [68] |
Warner JR, Reeder PJ, Karimpour-Fard A, et al. Rapid profiling of a microbial genome using mixtures of barcoded oligonucleotides. Nat Biotechnol, 2010, 28(8): 856-862. DOI:10.1038/nbt.1653 |
| [69] |
Luan GD, Cai Z, Li Y, et al. Genome replication engineering assisted continuous evolution (GREACE) to improve microbial tolerance for biofuels production. Biotechnol Biofuels, 2013, 6: 137. DOI:10.1186/1754-6834-6-137 |
| [70] |
Wang XL, Wang Y, Liu J, et al. Enhanced methanol utilization in genetically engineered Escherichia coli by directed evolution. Biotech Bull, 2017, 33(9): 101-109 (in Chinese). 王晓璐, 王钰, 刘娇, 等. 利用定向进化提高基因工程大肠杆菌的甲醇利用能力. 生物技术通报, 2017, 33(9): 101-109. |
| [71] |
Chai MZ, Jia B, Li BZ, et al. Advances in synthetic genome and genome rearrangement. Chin Bull Life Sci, 2019, 31(4): 364-371 (in Chinese). 柴梦哲, 贾斌, 李炳志, 等. 人工基因组合成与重排研究进展. 生命科学, 2019, 31(4): 364-371. |
| [72] |
Huang YQ, Liu D, Jia B, et al. SCRaMbLE of synthetic yeast chromosomes and applications. Biotech Bus, 2019(1): 62-66 (in Chinese). 黄耀庆, 刘夺, 贾斌, 等. 合成型酿酒酵母染色体重排技术及应用. 生物产业技术, 2019(1): 62-66. DOI:10.3969/j.issn.1674-0319.2019.01.008 |
| [73] |
Liu W, Luo ZQ, Wang Y, et al. Rapid pathway prototyping and engineering using in vitro and in vivo synthetic genome SCRaMbLE-in methods. Nat Commun, 2018, 9: 1936. DOI:10.1038/s41467-018-04254-0 |
| [74] |
Jia B, Wu Y, Li BZ, et al. Precise control of SCRaMbLE in synthetic haploid and diploid yeast. Nat Commun, 2018, 9: 1933. DOI:10.1038/s41467-018-03084-4 |
| [75] |
Blount BA, Gowers GF, Ho JCH, et al. Rapid host strain improvement by in vivo rearrangement of a synthetic yeast chromosome. Nat Commun, 2018, 9: 1932. DOI:10.1038/s41467-018-03143-w |
| [76] |
Shen MJ, Wu Y, Yang K, et al. Heterozygous diploid and interspecies SCRaMbLEing. Nat Commun, 2018, 9: 1934. DOI:10.1038/s41467-018-04157-0 |
| [77] |
Luo ZQ, Wang LH, Wang Y, et al. Identifying and characterizing SCRaMbLEd synthetic yeast using ReSCuES. Nat Commun, 2018, 9: 1930. DOI:10.1038/s41467-017-00806-y |
| [78] |
Choi JW, Yim SS, Kim MJ, et al. Enhanced production of recombinant proteins with Corynebacterium glutamicum by deletion of insertion sequences (IS elements). Microb Cell Fact, 2015, 14: 207. DOI:10.1186/s12934-015-0401-7 |
| [79] |
Annaluru N, Ramalingam S, Chandrasegaran S. Rewriting the blueprint of life by synthetic genomics and genome engineering. Genome Biol, 2015, 16: 125. DOI:10.1186/s13059-015-0689-y |
| [80] |
Juhas M, Reuß DR, Zhu BY, et al. Bacillus subtilis and Escherichia coli essential genes and minimal cell factories after one decade of genome engineering. Microbiology, 2014, 160(11): 2341-2351. |
| [81] |
Sasaki M, Kumagai H, Takegawa K, et al. Characterization of genome-reduced fission yeast strains. Nucleic Acids Res, 2013, 41(10): 5382-5399. DOI:10.1093/nar/gkt233 |
| [82] |
Yu BJ, Kang KH, Lee JH, et al. Rapid and efficient construction of markerless deletions in the Escherichia coli genome. Nucleic Acids Res, 2008, 36(14): e84. DOI:10.1093/nar/gkn359 |
| [83] |
Wang L, Maranas CD. MinGenome: an In silico top-down approach for the synthesis of minimized genomes. ACS Synth Biol, 2018, 7(2): 462-473. DOI:10.1021/acssynbio.7b00296 |
| [84] |
Komatsu M, Uchiyama T, Ōmura S, et al. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism. Proc Natl Acad Sci USA, 2010, 107(6): 2646-2651. DOI:10.1073/pnas.0914833107 |
| [85] |
Komor AC, Badran AH, Liu DR. CRISPR-based technologies for the manipulation of eukaryotic genomes. Cell, 2017, 168(1/2): 20-36. |
| [86] |
Lian JZ, Bao ZH, Hu SM, et al. Engineered CRISPR/Cas9 system for multiplex genome engineering of polyploid industrial yeast strains. Biotechnol Bioeng, 2018, 115(6): 1630-1635. DOI:10.1002/bit.26569 |
| [87] |
Lee HH, Ostrov N, Wong BG, et al. Functional genomics of the rapidly replicating bacterium Vibrio natriegens by CRISPRi. Nat Microbiol, 2019, 4(7): 1105-1113. DOI:10.1038/s41564-019-0423-8 |
| [88] |
Zhang GQ, Lin YP, Qi XN, et al. TALENs-assisted multiplex editing for accelerated genome evolution to improve yeast phenotypes. ACS Synth Biol, 2015, 4(10): 1101-1111. DOI:10.1021/acssynbio.5b00074 |
| [89] |
Gan YM, Lin YP, Guo YF, et al. Metabolic and genomic characterisation of stress-tolerant industrial Saccharomyces cerevisiae strains from TALENs-assisted multiplex editing. FEMS Yeast Res, 2018, 18(5): foy045. DOI:10.1093/femsyr/foy045 |
| [90] |
Wang Y, Liu Y, Liu J, et al. MACBETH: Multiplex automated Corynebacterium glutamicum base editing method. Metab Eng, 2018, 47: 200-210. DOI:10.1016/j.ymben.2018.02.016 |
| [91] |
Zhu ZM, Zhang J, Ji XM, et al. Evolutionary engineering of industrial microorganisms-strategies and applications. Appl Microbiol Biotechnol, 2018, 102(11): 4615-4627. DOI:10.1007/s00253-018-8937-1 |
| [92] |
Dragosits M, Mattanovich D. Adaptive laboratory evolution-principles and applications for biotechnology. Microb Cell Fact, 2013, 12: 64. DOI:10.1186/1475-2859-12-64 |
| [93] |
van den Bergh B, Swings T, Fauvart M, et al. Experimental design, population dynamics, and diversity in microbial experimental evolution. Microbiol Mol Biol Rev, 2018, 82(3): e00008-18. DOI:10.1128/MMBR.00008-18 |
| [94] |
Wang QH, Wang BW, Ma YH. A programmed control equipment for adaptation, screening and cultivation: CN, 203373351U. 2014-01-01 (in Chinese). 王钦宏, 汪保卫, 马延和.一种程序化控制的驯化筛选培养设备: CN, 203373351U. 2014-01-01. |
| [95] |
Wang QH, Lin YP, Qi XN, et al. Saccharomyces cerevisiae and its breeding methods and applications in ethanol production by industrial fermentation: CN, 107603898A. 2018-01-19 (in Chinese). 王钦宏, 蔺玉萍, 齐显尼, 等.酿酒酵母及其育种方式及工业化发酵生产乙醇的应用: CN, 107603898A. 2018-01-19. |
| [96] |
Liu JH, Li HL, Zhao GR, et al. Redox cofactor engineering in industrial microorganisms: strategies, recent applications and future directions. J Ind Microbiol Biotechnol, 2018, 45(5): 313-327. DOI:10.1007/s10295-018-2031-7 |
| [97] |
Wang M, Chen BQ, Fang YM, et al. Cofactor engineering for more efficient production of chemicals and biofuels. Biotechnol Adv, 2017, 35(8): 1032-1039. DOI:10.1016/j.biotechadv.2017.09.008 |
| [98] |
Chen XL, Li SB, Liu LM. Engineering redox balance through cofactor systems. Trends Biotechnol, 2014, 32(6): 337-343. DOI:10.1016/j.tibtech.2014.04.003 |
| [99] |
Zhao CH, Zhao QW, Li Y, et al. Engineering redox homeostasis to develop efficient alcohol-producing microbial cell factories. Microb Cell Fact, 2017, 16(1): 115. DOI:10.1186/s12934-017-0728-3 |
| [100] |
Akhtar MK, Jones PR. Cofactor engineering for enhancing the flux of metabolic pathways. Front Bioeng Biotechnol, 2014, 2: 30. DOI:10.3389/fbioe.2014.00030 |
| [101] |
Wang YP, San KY, Bennett GN. Cofactor engineering for advancing chemical biotechnology. Curr Opin Biotechnol, 2013, 24(6): 994-999. DOI:10.1016/j.copbio.2013.03.022 |
| [102] |
Reynolds TS, Courtney CM, Erickson KE, et al. ROS mediated selection for increased NADPH availability in Escherichia coli. Biotechnol Bioeng, 2017, 114(11): 2685-2689. DOI:10.1002/bit.26385 |
| [103] |
Ng CY, Farasat I, Maranas CD, et al. Rational design of a synthetic Entner-Doudoroff pathway for improved and controllable NADPH regeneration. Metab Eng, 2015, 29: 86-96. DOI:10.1016/j.ymben.2015.03.001 |
| [104] |
Cahn JK, Werlang CA, Baumschlager A, et al. A general tool for engineering the NAD/NADP cofactor preference of oxidoreductases. ACS Synth Biol, 2017, 6(2): 326-333. DOI:10.1021/acssynbio.6b00188 |
| [105] |
Knaus T, Paul CE, Levy CW, et al. Better than nature: nicotinamide biomimetics that outperform natural coenzymes. J Am Chem Soc, 2016, 138(3): 1033-1039. DOI:10.1021/jacs.5b12252 |
 2019, Vol. 35
2019, Vol. 35