中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘大文, 程海荣, 邓子新
- Liu Dawen, Cheng Hairong, Deng Zixin
- 解脂耶氏酵母表面展示β-淀粉酶与α-葡萄糖转苷酶及一步法由淀粉合成低聚异麦芽糖
- One step production of isomalto-oligosaccharides by engineered Yarrowia lipolytica yeast co-displayed β-amylase and α-transglucosidase
- 生物工程学报, 2019, 35(1): 121-132
- Chinese Journal of Biotechnology, 2019, 35(1): 121-132
- 10.13345/j.cjb.180077
-
文章历史
- Received: March 9, 2018
- Accepted: May 16, 2018
低聚异麦芽糖(Isomalto-oligosaccharides,简称IMO)是一类含有一个或多个α-1, 6糖苷键的多聚葡萄糖(Glucose oligomers),除了含有α-1, 6糖苷键外,还可以同时含有α-1, 4糖苷键。IMO通常包括异麦芽糖、潘糖、异潘糖、异麦芽三糖、异麦芽四糖、异麦芽五糖以及更长糖链的低聚糖[1]。另外,国际上通常把含有α-1, 2糖苷键或α-1, 3糖苷键的多聚葡萄糖也划分为低聚异麦芽糖的范畴[2]。IMO的聚合度通常为2–10,但目前由酶转化的IMO聚合度通常为2–4,即异麦芽糖、潘糖、异潘糖、异麦芽三糖以及异麦芽四糖。IMO在食品、医药以及化妆品领域都有应用,在食品领域应用最广泛。作为一种优良的益生元,IMO能显著提高机体消化道内益生菌的群体数量,维持稳定健康的微生态,促进消化道对矿物质的吸收,调节胆固醇与甘油三酯的水平,还能提高机体的免疫功能[3-6]。基于IMO的生理功能,IMO已经在乳制品、酵素、功能饮品、黄酒等食品中得到非常广泛的应用。目前世界很多国家都已批准IMO在食品中的应用,因此市场占有量逐年上升[7]。
典型的IMO的合成方法是采用多酶协同法,出发原料为玉米淀粉。首先,玉米淀粉经过乳化加入中温或者高温α-淀粉酶液化为麦芽糊精(Maltodextrin),再加入真菌β-淀粉酶(β-amylase)与普鲁兰酶(Pullulanase)进行糖化,糖化液含有50%以上含量的麦芽糖,最后加入曲霉来源的α-葡萄糖转苷酶(α-transglucosidase)在50 ℃下进行转糖基反应,生成含有α-1, 6糖苷键的低聚异麦芽糖[8],这种典型的三步法合成IMO步骤多,使用酶多且过程长。后来又开发出两步酶转化法[9]:首先玉米淀粉经过乳化加入中温或者高温α-淀粉酶液化为麦芽糊精(Maltodextrin),然后加入具有水解与转苷酶活性的麦芽糖淀粉酶(Maltogenic α-amylase),合成IMO。我国学者Lin等[10]利用水稻淀粉为底物,利用新普鲁兰酶(Neopullulanase)与糖化淀粉酶(Saccharifying α-amylase)组合,加入到水稻淀粉乳中,57 ℃反应72 h能得到纯度为59.2%的IMO。该方法不用单独在高温下液化淀粉,直接在淀粉乳中加入上述两种酶,较两步法更为简便。
虽然上述合成IMO的方法能够以淀粉为原料合成IMO,但均首先需要通过发酵的方法制备酶,制备酶的过程较为费时而且成本较高,转化时间也较长。因此,开发一种无需制备纯酶、转化时间又短的合成方法对于进一步降低成本具有重要的意义。本研究采用食品安全的解脂耶氏酵母Yarrowia lipolytica为出发菌株,通过将β-淀粉酶与α-葡萄糖转苷酶融合在一起,利用酵母展示系统将融合酶固定在细胞表面,培养酵母细胞,转化液化的淀粉一步法合成IMO。
1 材料与方法 1.1 菌株与培养基大肠杆菌Top 10作为质粒扩增宿主,BL21 (DE3)作为质粒表达宿主,均为本实验室保存。LB培养基(10 g/L蛋白胨,5 g/L酵母提取物,10 g/L NaCl) 37 ℃培养,LB中添加100 μg/mL氨苄青霉素用作筛选。
解脂耶氏酵母Yarrowia lipolytica CGMCC7326作为酶表面展示宿主,为本实验室保存。YPD培养基(10 g/L蛋白胨,5 g/L酵母提取物,20 g/L葡萄糖) 30 ℃培养,YPD中添加200 μg/mL潮霉素B作为筛选使用。
1.2 菌株的构建 1.2.1 构建包含β-淀粉酶基因(β-amylase)的酵母展示菌株耐热β-淀粉酶基因来自热硫嗜热厌氧杆菌Thermoanaerobacterium thermosulfurigenes (GenBank Accession No. M22471),基因大小为1 668 bp[11]。该基因按照酵母Y. lipolytica密码子进行优化并委托安徽通用生物技术有限公司进行全基因合成,合成后亚克隆到用BamHⅠ与KpnⅠ双酶切的载体pINA1313[12]中,构成新质粒pINA1313-β-amylase。Y. lipolytica的锚定蛋白Pir1基因(GenBank Accession No. AF336989,861 bp)采用一对引物Ppir1-F与Ppir1-R进行PCR扩增并亚克隆到PmlⅠ与BamHⅠ双酶切的质粒pINA1313- β-amylase中,构成新质粒pINA1313-Pir1-β-amylase。再采用一对引物Php4d-F与Phph-R,以质粒pUB4-CRE为模板[13],将抗性筛选标记基因片段扩增(大小1.5 kb)后连接到用SalⅠ与ClaⅠ双酶切的载体pINA1313-Pir1-β-amylase中,最后构建成含β-淀粉酶表面展示表达盒与抗性筛选标记的Y. lipolytica酵母整合表达载体pSWV-β-amylase。
1.2.2 构建包含α-葡萄糖苷酶基因(α- transglucosidase,GTase)的酵母展示菌株耐热α-葡萄糖转苷酶基因来自黑曲酶Aspergillus niger (GenBank Accession No. D45356,去除信号肽57个碱基后为2 958 bp)[14]。该基因按照酵母Y. lipolytica密码子进行优化并委托安徽通用生物技术有限公司进行全基因合成,并亚克隆到用BamHⅠ与KpnⅠ双酶切的载体pSWV-β-amylase中,构成新质粒pSWV-GTase。
1.2.3 β-amylase与GTase融合基因质粒构建β-amylase与GTase基因融合参考Wang等[15]引入linker (5'-TTAGGATCTAGATCTGCCGAATT A-3'),融合方式一种是5'-β-amylase-linker-GTase-3' (简称为βa-GT),另一种是5'-GTase-linker-β-amylase-3' (简称为GT-βa),融合所用的引物序列见表 1。先以质粒pSWV-β-amylase为模板,Pβ1-F/Pβ1-R一对引物扩出β-amylase基因;以pSWV-GTase为模板,以PG1-F/PG1-R一对引物扩出GTase基因;以上述两轮PCR产物作为混合模板(含有GTase基因与β-amylase基因),以Pβ1-F与PG1-R一对引物扩出融合基因βa-GT,采用重组酶克隆法克隆到BamHⅠ与XhoⅠ双切的载体pSWV-β-amylase中,构成重组质粒pSWV-βa-GT。
| Primers name | Primer sequence (5′–3′) | Enzyme sites |
| Ppir1-F | ACACACGTGATGCTCTTCAAGTCCGCTGCCGTCTCTC | PmlⅠ |
| Ppir1-R | ACAGGATCCTTAACAGTCCTCGAGGTTGACGATGGC | BamHⅠ |
| Php4d-F | ACAATCGATCTGAGGTGTCTACCAAGTGCCGTGC | ClaⅠ |
| Phph-R | ACAGTCGACCTATTCCTTTGCCCTCGGACGAGTGCTGG | SalⅠ |
| Pβ1-F | ACGACAAGGTCGACGGATCCATGATTGGAGCTTTTAAGCGACTG | BamHⅠ |
| Pβ1-R | TAATTCGGCAGATCTAGATCCTAAGTTCTGCCAGGTAATGGTGACAG | Linker |
| PG1-F | TTAGGATCTAGATCTGCCGAATTAATGGTCAAGCTGACGCATCTCC | Linker |
| PG1-R | TATCTGTTAATTGCCTCGAGCTACCACTCCAGCACCCAGTTCTC | XhoⅠ |
| PG2-F | CGACAAGGTCGACGGATCCATGGTCAAGCTGACGCATCTCC | BamHⅠ |
| PG2-R | TAATTCGGCAGATCTAGATCCTAACCACTCCAGCACCCAGTTCTCGGC | Linker |
| Pβ2-F | TTAGGATCTAGATCTGCCGAATTAATGATTGGAGCTTTTAAGCGACTG | Linker |
| Pβ2-R | TATCTGTTAATTGCCTCGAGTTAGTTCTGCCAGGTAATGGTGACAG | XhoⅠ |
| PβG-F | GTGCCGCGCGGCAGCCATATGATTGGAGCTTTTAAGCGACTG | NcoⅠ |
| PβG-R | GTGGTGGTGGTGGTGGTGCTCGAGCTACCACTCCAGCACCCAGTTCTC | XhoⅠ |
| PGβ-F | GTGCCGCGCGGCAGCCATATG-GTCAAGCTGACGCATCTCC | NcoⅠ |
| PGβ-R | GTGGTGGTGGTGGTGGTGCTCGAGTTAGTTCTGCCAGGTAATGGTGACAG | XhoⅠ |
| Note: Underlines indicate restriction enzyme recognition sites. | ||
以pSWV-GTase为模板,PG2-F/PG2-R一对引物扩增出GTase基因;以pSWV-β-amylase为模板,Pβ2-F/Pβ2-R为引物扩出β-amylase基因。以上两轮PCR产物作为混合模板,以PG2-F与Pβ2-R作为引物扩出融合基因GT-βa,采用重组酶克隆法克隆到BamHⅠ与XhoⅠ双切的载体pSWV-β- amylase中,构建重组质粒pSWV-GT-βa。构建示意图如图 1所示。
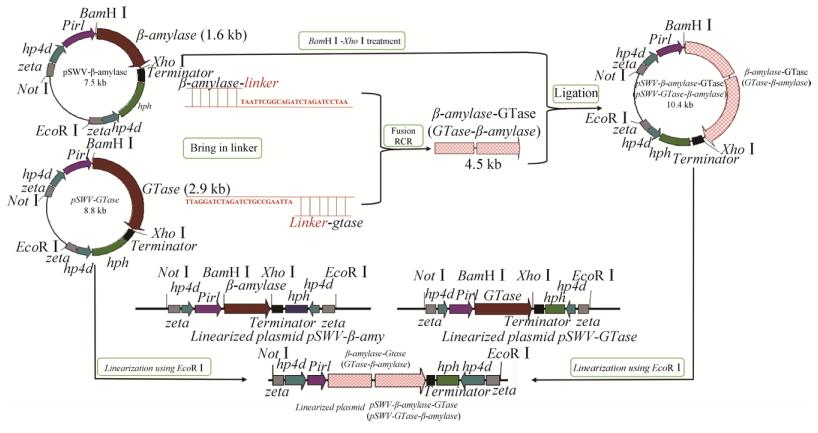
|
| 图 1 β-amylase-GTase与GTase-β-amylase基因合成、质粒构建及线性化流程图 Fig. 1 Scheme of the synthesis of β-amylase-GTase and GTase-β-amylase plasmid and the linearized functional elements. |
| |
以融合基因片段βa-GT和GT-βa为模板设计引物见表 1,引入酶切位点NcoⅠ和XhoⅠ。以βa-GT为模板,PβG-F和PβG-R作为引物PCR获得片段,重组连接入NcoⅠ和XhoⅠ双酶切载体pET32b得到重组质粒pET32b-βa-GT。以GT-βa为模板,PGβ-F和PGβ-R作为引物PCR获得片段,重组连接入双酶切载体pET32b得到重组质粒pET32b-GT-βa。以pSWV-β-amylase质粒为模板,PβG-F和PGβ-R为引物PCR获得单个基因片段,连入载体得到重组质粒pET32b-β-amylase。以pSWV-GTase质粒为模板,PGβ-F和PβG-R为引物PCR获得单个基因片段,连入载体得到重组质粒pET32b-GTase。
1.3 菌株的纯化与表达 1.3.1 酵母菌株的纯化与鉴定将上述质粒pSWV-β-amylase、pSWV-GTase、pSWV-βa-GT和pSWV-GT-βa使用EcoRⅠ单酶切获得线性化片段,参考Chen等[16]报道的“醋酸锂一步转化法”转化解脂耶氏酵母,将Y. lipolytica CGMCC7326在30 ℃平板活化生长一夜,取活化的菌株适量加入提前配置好的200 μL转化缓冲液中(400 g/L PEG 4 000,150 mmol/L pH 6.0醋酸锂,150 mmol/L二硫苏糖醇,0.2 mg/mL单链DNA),加入线性化片段3−5 μg,同时用ddH2O作为空白对照,39 ℃孵育90 min,将反应混合物涂布在潮霉素抗性YPD平板进行筛选。30 ℃稳定生长3−5 d,可获得单菌落200−300个,取新的抗性平板纯化3−4次后,获得稳定遗传转化子约50个。挑取转化子在50 mL YPD液体培养基,30 ℃、200 r/min培养1 d,按照Cheng等[17]报道的玻璃珠提取基因组的方法提取酵母转化子基因组,将pSWV-β-amylase以Pβ1-F和Pβ1-R为引物、pSWV-GTase以PG1-F和PG1-R为引物、pSWV-βa- GT以Pβ1和PG1-R为引物、pSWV-GT-βa以PG2-F和Pβ2-R为引物分别进行PCR筛选阳性转化子,将以上各质粒转化酵母获得的转化子命名为Yβa、YGT、Yβa-GT和YGT-βa。
1.3.2 酵母菌株活性的表达为测定不同转化子活力,Yβa、Yβa-GT、YGT-βa各挑选数个转化子接种于50 mL YPD液体培养基中,30 ℃培养2 d,待OD600达到15−20时,将菌液全部倒入50 mL离心管内,10 000 r/min离心7 min,除去上清,得沉淀细胞,使用无菌水洗涤细胞2次,尽量除去水分后得纯净细胞,湿重约250 mg。配制3%可溶性淀粉溶液作为底物(即3 g淀粉溶解于ddH2O中并定容至100 mL,淀粉需加热溶解成透明状),取15 mL加入上述含纯净细胞的离心管,放在50 ℃、200 r/min反应,定时取1 mL反应液入EP管内–20 ℃暂存。
同样将YGT、Yβa-GT、YGT-βa各转化子得到的纯净细胞加入配制的底物40%麦芽糖溶液中(即40 g麦芽糖溶于100 mL ddH2O),50 ℃、200 r/min反应,定时取1 mL反应液入EP管内–20 ℃暂存以备后续监测产物。根据产物生成量计算出转化子的产率,选择出最优转化子。
因β-淀粉酶在50–80 ℃条件下可达到60%以上活性,α-葡萄糖苷酶在40–55 ℃条件下可达到90%以上活性,选择两者适宜温度50 ℃作为实验温度,为两者融合创造最适宜温度。实验中以不含任何质粒的Y. lipolytica酵母细胞作为空白对照,证明酶是否表达。
1.3.3 大肠杆菌菌株活性的表达大肠杆菌BL21 (DE3)作为质粒表达宿主,利用载体所含抗性基因,将含质粒pET32b-β- amylase、pET32b-GTase、pET32b-βa-GT和pET32b-GT-βa的菌液分别涂布在氨苄抗性LB平板获得大肠杆菌转化子,分别命名为Eβa、EGT、Eβa-GT和EGT-βa。
随机挑取转化子在5 mL LB抗性液体培养基中,37 ℃、200 r/min培养约3 h,再将上述5 mL菌液接种于含50 mL抗性液体培养基中,37 ℃、200 r/min培养约6 h,直到大肠杆菌OD600达到约1.0时,加入1 mmol/L IPTG作为诱导剂,转移至16 ℃、200 r/min培养过夜,实验选取5个转化子作为平行对照,含pET32b质粒的菌株作为阴性对照,不含质粒的菌株作为空白对照。
为测定大肠杆菌融合基因的表达,对Eβa-GT、EGT-βa所表达酶进行SDS-PAGE分析,具体操作如下:将诱导后的菌株细胞等细胞量各取两个平行样品,10 000 r/min离心2 min,弃上清,用pH 8.0 Tris-HCl缓冲液洗涤细胞一次后加入20 μL悬浮细胞,加入上样缓冲液(50 mmol/L pH 6.8 Tris-HCl,100 mmol/L二硫苏糖醇,2% SDS,0.1%溴酚蓝,10%甘油)在沸水中煮10 min,离心所得上清即为上样样品。电泳使用金斯瑞ExpressPlus™蛋白预制胶,12%聚丙烯酰胺凝胶,一切操作流程按照说明书具体要求操作。
将诱导后的菌液倒入50 mL离心管内,6 000 r/min离心5 min,去除上清培养基成分,加入无菌水洗涤细胞2次,获得纯净大肠杆菌细胞约100 mg,在Eβa、Eβa-GT、EGT-βa细胞中加入5 mL 3%可溶性淀粉溶液,EGT、Eβa-GT、EGT-βa细胞中加入5 mL 40%麦芽糖溶液,分装于1.5 mL EP管内,每管约800 μL,50 ℃、200 r/min反应,定时取样品–20 ℃暂存。
1.3.4 一步法由淀粉合成低聚异麦芽糖(IMO)取6个500 mL烧杯,分别加入150 mL ddH2O和40 g玉米淀粉,95 ℃预热5 min,加入0.2 g耐高温α-淀粉酶(Sigma-A3306,> 10 000 U/mL) 95 ℃继续反应,用玻璃棒搅拌使反应充分,在2、4、6、10、15、20 min时分别从中取出一只烧杯,加入HCl使酶失去活性,NaOH调pH至7.0,降至室温,用ddH2O定容至200 mL。取1 mL样品4 ℃暂存待测,确定最适的玉米淀粉液化时间。将玉米淀粉按最适时间液化,获得高浓度液化液,加入到依照上述实验方法处理得到的Yβa-GT、YGT-βa纯净酵母细胞中,50 ℃、200 r/min反应,定时取1 mL反应液入EP管内4 ℃暂存。
1.4 产物的分析方法及酶活检测将上述–20 ℃暂存样品室温溶解,10 000 r/min离心10 min,取上清入新EP管内离心2次,获得纯净样品,使用HPLC检测产物成分。色谱柱1:Shodex KS802 (8 mm×300 mm),RI101型示差折光检测器,流动相ddH2O;流速1.0 mL/min,柱温70 ℃,进样量20 μL。色谱柱2:Hydrophobic polystyrene gel (Shodex Rspak DC-613),RI101型示差折光检测器,流动相75%乙腈,流速1.0 mL/min,柱温40 ℃,进样量20 μL。
β-淀粉酶的一个活力单位(U)指在50 ℃反应条件下每分钟产生1 μmol麦芽糖所需的酵母细胞或者大肠杆菌细胞干重(mg);α-葡萄糖转苷酶的一个活力单位(U)指在50 ℃反应条件下每分钟转移1 μmol麦芽糖所需的细胞干重(mg)。
2 结果与分析 2.1 融合基因质粒表达盒的构建与表达构建融合基因质粒表达盒需先扩增β-amylase与GTase基因片段,如图 2所示,左为1.6 kb大小的β-amylase片段与2.9 kb大小的GTase片段,右为融合PCR获得的融合片段βa-GT与GT-βa约4.5 kb,且融合质粒上下游测序结果与NCBI-GenBank进行匹配一致性为100%,说明序列正确。在β-amylase-GTase融合中,β-amylase的C端与GTase的N端融合,GTase-β-amylase的融合方式与之相反。Linker序列的加入目的是加快重组DNA的构建,且不删除任何氨基酸,插入的片段表达的氨基酸为LGSRSAEL[15]。
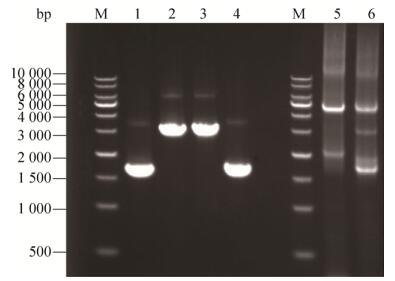
|
| 图 2 βa-GT和GT-βa基因扩增 Fig. 2 DNA gel electrophoresis of βa-GT and GT-βa gene. Lanes 1–2 : two parts PCR product of βa-GT, β-amylase and GTase; lanes 3–4: two parts PCR product of GT-βa, GTase and β-amylase; lanes 5–6: the whole PCR product of βa-GT and GT-βa. |
| |
融合质粒在E. coli胞内表达蛋白SDS-PAGE分析如图 3所示,Lanes 1–2为β-amylase-GTase蛋白,Lanes 3和4为GTase-β-amylase蛋白,去掉pET32b自身载体蛋白后大小约为170 kDa,与理论值相一致。
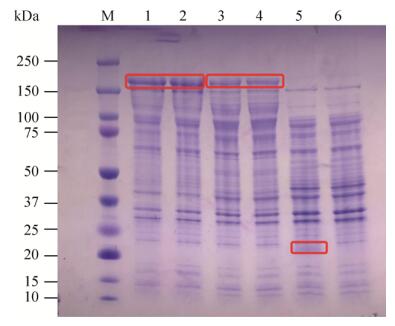
|
| 图 3 E. coli表达βa-GT和GT-βa蛋白SDS-PAGE分析 Fig. 3 SDS-PAGE of the fusion protein βa-GT and GT-βa. Lanes 1–2, 3–4, 5: the supernatant (soluble proteins) of E. coli containing pET32b-βa-GT, pET32b-GT-βa and pET32b; lane 6: the supernatant (soluble proteins) of E. coli. |
| |
经过潮霉素抗性平板筛选得到约300个转化子,选取50个较大的转化子进一步纯化,PCR验证获得阳性克隆30−40个不等。转化子进行底物转化实验,根据转化效率选出最优转化子。如图 4A所示,32个Yβa转化子中有10个转化子与空白对照相同,未表现出活性,其他由底物可溶性淀粉可产生约62%麦芽糖产率,选出最优转化子Yβa19,其酶活力为0.03 U/mg。通过对YGT (图 4B)共40个转化子添加底物40%麦芽糖溶液反应检测,除了5个转化子只表现出水解酶活性,即将麦芽糖水解产生葡萄糖外,其他均有IMO产生,产率在30%上下浮动,选出最优转化子YGT7,其酶活力约为0.023 U/mg。
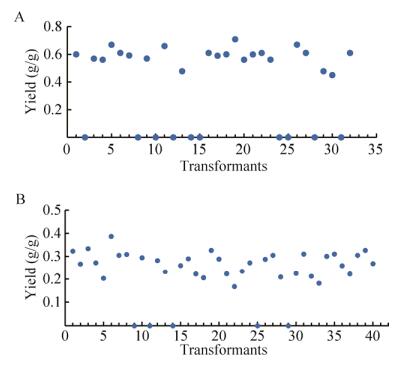
|
| 图 4 不同酵母转化子转化淀粉或麦芽糖合成产物的差异分析 Fig. 4 Comparative analysis of different Y. lipolytica transformants. (A) Yields were obtained when various Yβa transformants converting 3% soluble starch to maltose. (B) Yields were obtained when various YGT transformants converting 40% maltose to IMO. |
| |
含融合基因酵母展示菌株经多次纯化获得稳定遗传转化子,对于YGT-βa,经多次转化获得不同批次转化子约100个均进行了底物转化实验,未见反应,怀疑是GTase基因先表达影响了整体的活性。
对于Yβa-GT,选取30个转化子以3%可溶性淀粉溶液作为底物的产物组成见图 5A,不同的转化子产物表现各异,除去无反应的和只有麦芽糖一种产物的转化子,有10个转化子的产物中观测到了麦芽糖与葡萄糖,且葡萄糖占比约为麦芽糖的2倍;有11个在产物中只观测到了葡萄糖,约占43% (淀粉不能完全转化,有一部分底物残留)。由此可认为此21个转化子是两个基因均发挥了作用,首先β-淀粉酶将淀粉水解产生麦芽糖,而耐热α-葡萄糖苷酶对于低浓度的麦芽糖转苷酶活性较弱,主要发挥了水解酶的活性,将得到的麦芽糖进一步水解产生葡萄糖。挑选其中的转化子以麦芽糖作为底物实验得到的结果与单个YGT7菌株基本无异(图 5B),证明了Yβa-GT融合菌株的确是两个基因均可以表达,选出最优转化子Yβa-GT29。
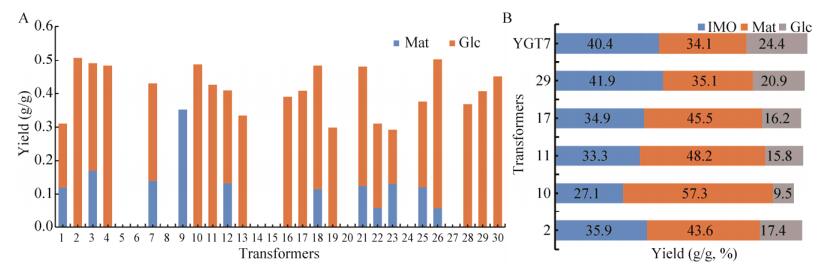
|
| 图 5 选取不同的酵母转化子转化淀粉合成产物的分析 Fig. 5 Comparative analysis of different Y. lipolytica transformants. (A) Yield of products by the Yβa-GT transformants with the 3% starch. (B) Yield of products by the Yβa-GT 2, 10, 11, 17, 29 transformants and YG7 with the 40% maltose. |
| |
以上转化子经PCR验证基因组中均含有相应目的基因表达盒,但是不同的转化子底物合成产物的性能存在较大的差异。图 4A中第19号含β-amylase基因的转化子(Yβa19),由可溶淀粉转化生成麦芽糖产率为71%,而有的转化子虽然含有目的基因却不能合成IMO。对于含GTase基因的转化子(图 4B)以及含融合基因βa-GT的转化子(图 5A)情况类似,不同转化子合成产物的性能差距很大。产生这种现象的原因可能是表达盒虽然整合到了Y. lipolytica基因组中,但其表达差异很大。我们选取了3个均含有β-amylase基因的转化子(Yβa2、Yβa13、Yβa19)提取总RNA,反转录进行β-amylase基因表达水平荧光定量分析,发现Yβa2转化子的β-amylase基因相对表达水平为0,即没有得到表达,Yβa19的相对表达水平为4.2,Yβa13的相对表达水平为1.7,与产物产率相一致。因此可以看出,虽外源基因均整合到基因组中,但由于其表达水平的差异而导致不同转化子的转化性能差异。产生这些差异的根本原因很可能是基因插入到基因组中的位置效应而导致的。染色体中有的位置基因活跃,而有的位置沉默,外源基因所处的位置对于其表达水平至关重要。Wu等[18]利用荧光蛋白作为标记研究了酿酒酵母6条染色体中的不同的位置对于荧光蛋白基因的表达水平的影响,发掘了一批活跃表达与沉默的位置,为外源基因在酿酒酵母中的表达奠定了基础。因此,为了获得外源基因的最高水平的表达,应该将外源基因插入到染色体的活跃区域。
2.3 E. coli工程菌合成IMO 2.3.1 含β-amylase与GTase单基因的大肠杆菌菌株Eβa和EGT合成麦芽糖与IMO的性能为比较酵母与大肠杆菌细胞的表达差异,将单个基因利用载体pET32b在BL21 (DE3)表达宿主内表达。值得注意的是,EGT菌株在添加麦芽糖作为底物后,完全没有产物IMO产生,原因可能是GTase基因为真菌米曲霉的基因,在大肠杆菌中不能正确折叠因而没有活性。含β-amylase基因的Eβa细胞转化淀粉的效率反而大幅度提高,如图 6,在加入底物的瞬间即能检测到麦芽糖产物的产生,20 min时已有60.5%麦芽糖产生,其酶活为0.398 U/mg,为酵母展示菌株的13.3倍,可用作由淀粉生产麦芽糖的工程菌株。
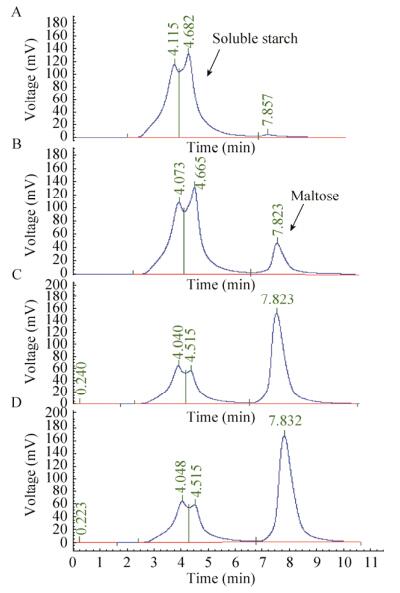
|
| 图 6 大肠杆菌Eβa转化可溶性淀粉时产物生成色谱图 Fig. 6 HPLC analysis of maltose products by E. coli Eβa cells with soluble starch. (A) 0 min. (B) 5 min. (C) 20 min. (D) 40 min. |
| |
含β-淀粉酶与α-葡糖糖转苷酶基因的酵母菌株Yβa-GT具有良好活性(图 5),能将液化的淀粉转化为IMO。而对于含β-淀粉酶与α-葡糖糖转苷酶基因的E. coli菌株,不同融合方式的EGT-βa、Eβa-GT菌株均能转化可溶性淀粉生成麦芽糖,表明EGT-βa、Eβa-GT菌株的β-amylase基因均能表达成具有活性的淀粉酶;但是均不能转化麦芽糖生成IMO,表明GTase基因虽然能得到融合表达(图 3),但是无活性,与单基因GTase在大肠杆菌内表达的结果相符。值得注意的是,β-淀粉酶基因与α-葡糖糖转苷酶基因以GTase-β-amylase顺序在酵母中融合表达时,这两个基因均未表现出活性,而在大肠杆菌中融合表达时,β-amylase有活性,而GTase没有活性。
2.3.3 不同的基因融合顺序对表达的酶活性的影响将基因进行融合共同表达成为多功能融合酶,在蛋白质工程中具有很大的潜力。融合酶由于其物理邻近效应[19],可以通过促进反应中间体转移到下一种酶的活性位点使得融合酶催化的反应速率大幅度提高,该技术已经在海藻糖以及长链低聚异麦芽糖的合成得到应用[15, 20-21]。也可以使用两种展示蛋白将两个酶进行共展示在细胞表面,但融合酶由于更加邻近,因而在反应中间体的转移过程中可更快速到达下一个酶,反应效率更高。
本研究将合成低聚异麦芽糖的两种关键酶β-淀粉酶基因与α-葡萄糖苷酶基因进行融合,在Y. lipolytica中进行表面展示表达以及在E. coli中进行胞内表达。有趣的是,融合基因在酵母表面展示表达时,基因的不同融合顺序对酶的活性具有很大的差异。当以β-amylase-GTase的先后顺序进行融合表达时,两种酶均有活性;而当以GTase-β-amylase的先后顺序在酵母表面进行融合表达时,均未发现有催化活性。然而,当融合基因在大肠杆菌进行胞内融合表达时,无论以哪种融合顺序(GTase-β-amylase或β-amylase-GTase),发现β-amylase均有催化活性,而GTase均无活性(表 2)。造成这种酶活性因融合顺序不同而不同的原因,很可能是融合顺序影响了GTase的正确折叠。无论是GTase-β-amylase或β-amylase- GTase融合方式,在大肠杆菌中GTase均不能正确折叠,即使能够表达也没有活性。而β-amylase能够正确折叠,从而在这两种不同的融合方式中均表现出活性。因此,在进行酶融合表达研究时,需要对不同的融合顺序研究,以确定最佳的融合方式。
| Genes | Expressed enzymes | Y. lipolytica | E. coli |
| β-amylase | β-amylase | + | + |
| GTase | GTase | + | – |
| β-amylase-GTase (fused) | β-amylase | + | + |
| GTase | + | – | |
| GTase-β-amylase (fused) | β-amylase | – | + |
| GTase | – | – | |
| (+):active; (–):inactive. | |||
将优选出的Yβa-GT29酵母菌株培养获得大量细胞,使用液化的玉米淀粉作为底物。根据玉米淀粉乳液化时间图谱分析选定15 min为最适液化时间,图 7中A、B、C、D为添加Yβa-GT29细胞在50 ℃反应时所得产物的HPLC分析图,淀粉被转化生成低聚葡萄糖。用色谱柱Shodex DC613、乙腈作为流动相将7 D产物进行分析,可清晰看出各低聚糖的含量(图 8A)。将异麦芽糖、潘塘、异麦芽三糖、异麦芽四糖及以上的寡糖归为IMO,可占总产物的75.3%,即200 g/L玉米淀粉最终可产150.6 g/L IMO。转化过程中IMO各组分含量随时间变化见图 8B,麦芽糖在7 h后开始减少,慢慢转化成其他低聚糖。
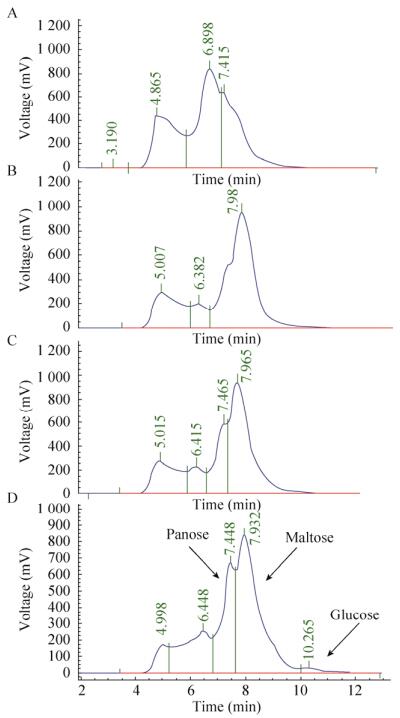
|
| 图 7 Yβa-GT29细胞转化玉米淀粉液化液生成产物的色谱图 Fig. 7 HPLC analysis of products obtained by Yβa-GT29 cells with the liquefied maize starch. (A) 0 h. (B) 5 h. (C) 10 h. (D) 20 h. |
| |
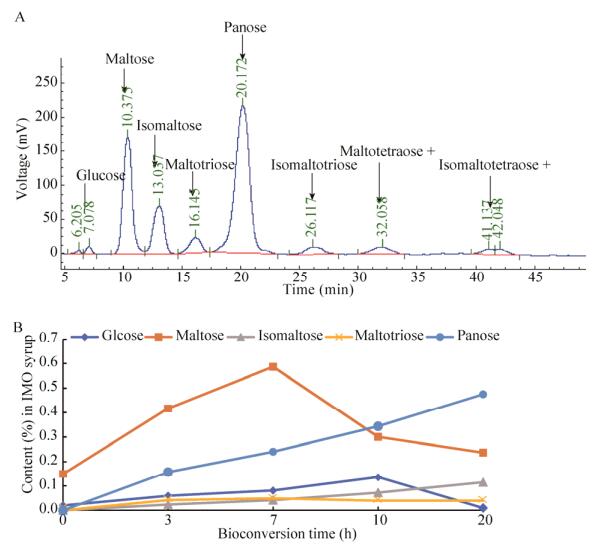
|
| 图 8 Yβa-GT29细胞转化玉米淀粉液化液的IMO各组分色谱图(A)及时间变化图(B) Fig. 8 HPLC analysis of carbohydrates in the IMO syrup obtained by Yβa-GT29 cells with the liquefied maize starch (A) and changes in the content during the bioconversion time (B). |
| |
如前言所说,低聚异麦芽糖目前通过淀粉以多酶有序进行转化合成,步骤较多,操作比较复杂,增加了生产成本。由于α-葡萄糖转苷酶是合成低聚异麦芽糖的关键酶,也是成本较高的酶之一,为了减少该酶的成本,有人研究采用化学戊二醛酶固定化的方式增加转苷酶的使用次数[22-23],但是当酶失效后,产生大量的固定化介质废弃物难以处理或增加废弃物处理成本。而采用本研究开发的双酶融合共表达并展示固定在酵母表面的方法,能够减少酶纯化的成本,无需化学固定酶的步骤,展示的酶能够多次反复利用。当酶失效后,酵母细胞仍然能够制成酵母干粉或者酵母浸膏,当成培养基使用,整个过程持续循环,绿色环保,基本无废弃物排放(Waste-Zero)。
解脂耶氏酵母Y. lipolytica是一种研究最广泛的酵母之一,被美国食品药品监督局认定为公认安全的食品微生物,已经在食品工业得到广泛的应用,如合成有机酸、赤藓糖醇、Ω-3多不饱和脂肪酸(如二十碳五烯酸EPA)、β-胡萝卜素等[24-28]。我们研究小组利用该酵母作为微生物细胞工厂,将果糖基转移酶、半乳糖基转移酶、海藻糖合成酶以及异麦芽酮糖合成酶表面展示在解脂耶氏酵母细胞表面,分别得到合成低聚果糖、低聚半乳糖、海藻糖以及异麦芽酮糖的工程菌株[29-32]。本研究将β-淀粉酶与α-葡萄糖转苷酶进行融合,同时表面展示在解脂耶氏酵母细胞上,对转化子进行筛选得到一株由液化淀粉为底物一步法合成低聚异麦芽糖的菌株Yβa-GT29,转化率达到75.3%,为工业合成IMO提供了一种新的方法。
| [1] | Goffin D, Wathelet B, Blecker C, et al. Comparison of the glucooligosaccharide profiles produced from maltose by two different transglucosidases from Aspergillus niger. Biotechnol Agronom Soci Environ, 2010, 14(4): 607–616. |
| [2] | Konishi Y, Shindo K. Production of nigerose, nigerosyl glucose, and nigerosyl maltose by Acremonium sp. S4G13. Biosci Biotechnol Biochem, 1997, 61(3): 439–442. DOI: 10.1271/bbb.61.439 |
| [3] | Florowska A, Krygier K, Florowski T, et al. Prebiotics as functional food ingredients preventing diet-related diseases. Food Funct, 2016, 7(5): 2147–2155. DOI: 10.1039/C5FO01459J |
| [4] | Yen CH, Tseng YH, Kuo YW, et al. Long-term supplementation of isomalto-oligosaccharides improved colonic microflora profile, bowel function, and blood cholesterol levels in constipated elderly people—A placebo-controlled, diet-controlled trial. Nutrition, 2011, 27(4): 445–450. DOI: 10.1016/j.nut.2010.05.012 |
| [5] | Swennen K, Courtin CM, Delcour JA. Non-digestible oligosaccharides with prebiotic properties. Crit Rev Food Sci Nutrit, 2006, 46(6): 459–471. DOI: 10.1080/10408390500215746 |
| [6] | Panesar PS, Kumari S, Panesar R. Biotechnological approaches for the production of prebiotics and their potential applications. Crit Rev Biotechnol, 2013, 33(4): 345–364. |
| [7] | Goffin D, Delzenne N, Blecker C, et al. Will isomalto-oligosaccharides, a well-established functional food in Asia, break through the European and American market? The status of knowledge on these prebiotics. Crit Revi Food Sci Nutrit, 2011, 51(5): 394–409. DOI: 10.1080/10408391003628955 |
| [8] | Kim YW, Choi JH, Kim JW, et al. Directed evolution of Thermus maltogenic amylase toward enhanced thermal resistance. Appl Environ Microbiol, 2003, 69(8): 4866–4874. DOI: 10.1128/AEM.69.8.4866-4874.2003 |
| [9] | Pan YC, Lee WC. Production of high-purity isomalto-oligosaccharides syrup by the enzymatic conversion of transglucosidase and fermentation of yeast cells. Biotechnol Bioeng, 2007, 89(7): 797–804. |
| [10] | Lin QL, Xiao HX, Zhao J, et al. Production of isomalto‐oligosaccharide syrup from rice starch using an one‐step conversion method. Int J Food Sci Technol, 2011, 46(6): 1194–1200. |
| [11] | Kitamoto N, Yamagata H, Kato T, et al. Cloning and sequencing of the gene encoding thermophilic β-amylase of Clostridium thermsulfurgenes. J Bacteriol, 1989, 170(12): 5848–5854. |
| [12] | Nicaud JM, Madzak C, Van Den Broek BP, et al. Protein expression and secretion in the yeast Yarrowia lipolytica. FEMS Yeast Res, 2002, 2(3): 371–379. |
| [13] | Fickers P, Le Dall MT, Gaillardin C, et al. New disruption cassettes for rapid gene disruption and marker rescue in the yeast Yarrowia lipolytica. J Microbiol Methods, 2003, 55(3): 727–737. |
| [14] | Nakamura A, Nishimura I, Yokoyama A, et al. Cloning and sequencing of an α-glucosidase gene from Aspergillus niger and its expression in A. nidulans. J Biotechnol, 1997, 53(1): 75–84. |
| [15] | Wang JH, Tsai MY, Lee GC, et al. Construction of a recombinant thermostable β-amylase-trehalose synthase bifunctional enzyme for facilitating the conversion of starch to trehalose. J Agric Food Chem, 2007, 55(4): 1256–1263. |
| [16] | Chen DC, Beckerich JM, Gaillardin C. One-step transformation of the dimorphic yeast Yarrowia lipolytica. Appl Microbiol Biotechnol, 1997, 48(2): 232–235. |
| [17] | Cheng HR, Jiang N. Extremely rapid extraction of DNA from bacteria and yeasts. Biotechnol Lett, 2006, 28(1): 55–59. |
| [18] | Wu XL, Li BZ, Zhang WZ, et al. Genome-wide landscape of position effects on heterogeneous gene expression in Saccharomyces cerevisiae. Biotechnol Bio, 2017, 10(1): 189. |
| [19] | Meek TD, Garvey EP, Santi DV. Purification and characterization of the bifunctional thymidylate synthetase-dihydrofolate reductase from methotrexate-resistant Leishmania tropica. Biochemistry, 1985, 24(3): 678–686. |
| [20] | de Pascale DP, Di Lernia I, Sasso M, et al. A novel thermophilic fusion enzyme for trehalose production. Extremophiles, 2002, 6(6): 463–468. DOI: 10.1007/s00792-002-0283-6 |
| [21] | Ryu HJ, Jin XJ, Lee JH, et al. Optimal expression and characterization of a fusion enzyme having dextransucrase and dextranase activities. Enzyme Microb Technol, 2010, 47(5): 212–215. |
| [22] | Zhang L, Jiang YJ, Jiang ZY, et al. Immobilized transglucosidase in biomimetic polymer-inorganic hybrid capsules for efficient conversion of maltose to isomaltooligosaccharides. Biochem Eng J, 2009, 46(2): 186–192. |
| [23] | Sheu DC, Huang CI, Duan KJ. Production of isomaltooligosaccharides by a-glucosidase immobilized in chitosan beads and by polyethyleneimine-glutaraldehyde treated mycelia of Aspergillus carbonarious. Biotechnol Technol, 1997, 11(5): 287–291. DOI: 10.1023/A:1018407009503 |
| [24] | Finogenova TV, Morgunov IG, Kamzolova SV, et al. Organic acid production by the yeast Yarrowia lipolytica: a review of prospects. Appl Biochem Microbiol, 2005, 41(5): 418–425. DOI: 10.1007/s10438-005-0076-7 |
| [25] | Xue ZX, Sharpe PL, Hong SP, et al. Production of omega-3 eicosapentaenoic acid by metabolic engineering of Yarrowia lipolytica. Nat Biotechnol, 2013, 31(8): 734–740. DOI: 10.1038/nbt.2622 |
| [26] |
Cheng HR, Lv JY, Wang B, et al. Yarrowia lipolytica strain and method thereof for synthesizing erythritol: CN, 103374534 A[P]. 2013 (in Chinese). 程海荣, 吕霁烊, 王犇, 等.解脂亚罗酵母菌株及其用于合成赤藓糖醇的方法: CN, 103374534A[P]. 2013. |
| [27] | Wang SQ, Wang HW, Lv JY, et al. Highly efficient erythritol recovery from waste erythritol mother liquor by a yeast-mediated biorefinery process. J Agric Food Chem, 2017, 65(50): 11020–11028. DOI: 10.1021/acs.jafc.7b04112 |
| [28] | Gao SL, Tong YY, Zhu L, et al. Iterative integration of multiple-copy pathway genes in Yarrowia lipolytica for heterologous β-carotene production. Metabol Eng, 2017, 41: 192–201. DOI: 10.1016/j.ymben.2017.04.004 |
| [29] | Zhang LB, An J, Li LJ, et al. Highly efficient fructooligosaccharides production by an erythritol-producing yeast Yarrowia lipolytica displaying fructosyltransferase. J Agric Food Chem, 2016, 64(19): 3828–3837. DOI: 10.1021/acs.jafc.6b00115 |
| [30] | An J, Zhang LB, Li LJ, et al. An alternative approach to synthesizing galactooligosaccharides by cell-surface display of β-galactosidase on Yarrowia lipolytica. J Agric Food Chem, 2016, 64(19): 3819–3827. DOI: 10.1021/acs.jafc.5b06138 |
| [31] | Li N, Wang HW, Li LJ, et al. Integrated approach to producing high-purity trehalose from maltose by the yeast Yarrowia lipolytica Displaying Trehalose Synthase (TreS) on the cell surface. J Agric Food Chem, 2016, 64(31): 6179–6187. DOI: 10.1021/acs.jafc.6b02175 |
| [32] | Li LJ, Wang HW, Cheng HR, et al. Isomaltulose production by yeast surface display of sucrose isomerase from Pantoea dispersa on Yarrowia lipolytica. J Funct Foods, 2017, 32: 208–217. DOI: 10.1016/j.jff.2017.02.036 |
 2019, Vol. 35
2019, Vol. 35




