中国科学院微生物研究所、中国微生物学会主办
文章信息
- 高洁, 李益民, 杜聪, 裴绪泽, 卢慧敏, 赵孝阳, 袁文杰
- Gao Jie, Li Yimin, Du Cong, Pei Xuze, Lu Huimin, Zhao Xiaoyang, Yuan Wenjie
- 褐藻胶裂解酶基因的克隆表达与酶学性质
- Cloning and expression of alginate lyase genes from Vibrio alginolyticus and characterization of the alginate lyase
- 生物工程学报, 2018, 34(7): 1178-1188
- Chinese Journal of Biotechnology, 2018, 34(7): 1178-1188
- 10.13345/j.cjb.180027
-
文章历史
- Received: January 16, 2018
- Accepted: April 2, 2018
大型海藻生长在潮间带或亚潮带,生存范围广,生长迅速,并且不与陆生植物竞争耕地、水源、肥料等资源,还可以固定大气和海水中的二氧化碳,能有效缓解温室效应和海水酸化的问题,具有良好的环境效益。海藻生物质的主要成分是褐藻多糖,占褐藻植物干重的10%−40%[1-2],广泛应用于食品、生物医学材料、化妆品和生物燃料等领域。
褐藻胶是一种含量最丰富的褐藻多糖[2-4],由L-古罗糖醛酸(α-L-guluronic acid,简称G)和D-甘露糖醛酸(β-D-mannuronic acid,简称M)两种单元通过1, 4-糖苷键连接而成。其组合方式有以下3种,即:均聚古罗糖醛酸片段(Poly- gulutonate,Poly-G)、均聚甘露糖醛酸片段(Poly- mannuronate,Poly-M)和甘露糖醛酸-古洛糖醛酸混合嵌合片段(Poly-MG)[5]。目前,褐藻胶降解的方法主要有化学降解法[6-7]、物理降解法[8-9]和酶解法[10-11],相比而言,酶促降解褐藻胶更有优势[12],其效率高、无毒副产物,更适于褐藻胶寡糖的制备。通过酶法降解获得的源自褐藻胶的各种寡糖具有促进植物生长、抗氧化、抗凝血、抗肿瘤、抗过敏、抗增殖和抗过敏活性等生理功能和生物活性[13-15]。低聚糖和寡糖因分子量小,容易被利用,其功能活性往往较多糖高,应用更为广泛[16-17]。
以微藻和大型褐藻为底物的第三代生物能源,更是目前引起国内外广泛关注的热点[18-19]。海藻是生产生物燃料的一种理想原料,具有很大的潜力。海藻的生物构造特征赋予它一种优于木质纤维素生物质的优势,易于高产,且避开了发酵前的耗能性预处理和水解过程。然而,目前尚未实现由海藻生产乙醇的全部潜能,主要是因为工业微生物不能代谢海藻多糖组分,必须先降解为单糖或寡糖后才能被微生物利用。
因此,褐藻胶裂解酶温和降解褐藻胶成为大型褐藻利用的关键点。文中对海藻酸裂解酶的酶学性质和机理方面进行研究,进而通过基因工程手段来提高褐藻胶裂解酶的产量,获得高活力低成本的褐藻胶裂解酶。
1 材料与方法 1.1 菌株和质粒菌株:解藻酸弧菌BH17 (Vibrio alginolyticus BH17),本实验室从腐烂海带中筛选鉴定并保藏;大肠杆菌Escherichia coli DH5α、E. coli BL21为本实验室保存。质粒载体pET29a (+)购自大连TaKaRa公司。
1.2 主要仪器和试剂仪器:Image Lab凝胶成像系统(美国BIO-RAD公司);Mini-PROTEAN Tetra蛋白电泳仪(美国BIO-RAD公司);蛋白纯化重力柱,购自TaKaRa公司。
试剂:PCR纯化试剂盒、质粒提取试剂盒,均购自OMEGA BIO-TEK公司;限制性内切酶、T4 DNA连接酶等购自TaKaRa公司;蛋白胶配制试剂盒购自BIO-RAD公司;海藻酸钠购自天津市大茂化学试剂厂,分析纯(AR);Poly-M、Poly-G购自青岛博智汇力生物科技有限公司。
1.3 褐藻胶裂解酶基因的克隆 1.3.1 褐藻胶裂解酶基因引物的合成根据文献报道,查找解藻酸弧菌上可能的褐藻胶裂解酶基因[20],如图 1所示,以此基因序列及载体的多克隆位点,采用Primer Premier 6.0软件设计上下游引物序列,引物序列见表 1。
| Primer name | Primer sequence (5′–3′) |
| PalgV1 F | CGCGGATCCTTTCAATTTTGATTTA |
| PalgV1 R | CGCCATATGAAACTCAATCTACTCGT |
| PalgV2 F | CGCGGATCCTTACCAAAAGATTGTT |
| PalgV2 R | GCGCATATGAAGCATATTTTCTTCA |
| PalgV3 F | CACCGCGGATCCGTGTTTATAAAATCAACTAA |
| PalgV3 R | CACCCGCTCGAGTTGATGAAGAGTGCTCAAAG |
| PalgV4 F | GGAGAAATACATATGAGCTACCAAACCC |
| PalgV4 R | AAGGATCCCCGATTTTTACATT |
| PalgV5 F | CTCTCCCCATATGCCACAAAAACTA |
| PalgV5 R | GGTGGATCCTTATTATTTCTCCTGC |
| Underline represent the restriction site. | |
将设计好的引物交由上海生工生物工程公司合成,根据引物OD值,用无菌水将其稀释至10 μmo1/L,于–20 ℃储存备用。将产褐藻胶裂解酶的菌株过夜培养,以此为模板,构建50 μL PCR反应体系,具体见表 2。所得PCR产物通过OMEGA BIO-TEK公司的DNA产物纯化试剂盒进行纯化,纯化产物备用,或克隆至pET29a质粒后测序并提取重组质粒备用。
| PCR reaction system | Volume (μL) |
| 5×PS buffer | 10.0 |
| dNTPs | 4.0 |
| Primer (F) | 1.5 |
| Primer (R) | 1.5 |
| ddH2O | 31.0 |
| Template | 1.5 |
| PrimeSTAR HS DNA polymerase | 0.5 |
| Total | 50 |
将纯化后的目的基因与载体按照连接体系(表 3)于16 ℃过夜连接,并用热击法转入扩增宿主E. coli DH5α中。检测阳性克隆,双酶切验证(37 ℃,1−3 h或过夜)正确后送生工生物工程(上海)股份有限公司检测序列。阳性克隆中的目的基因序列无突变时,提取重组质粒,并采用热击法转入表达宿主BL21。
| Connection system | Volume (μL) |
| 10×T4 buffer | 1 |
| T4 ligase | 1 |
| Vector | 2 |
| Target gene | 6 |
| Total | 10 |
| Double digestion system | Volume (μL) |
| 10×CutSmart buffer | 1.0 |
| ddH2O | 3.6 |
| Restriction enzyme 1 | 0.2 |
| Restriction enzyme 2 | 0.2 |
| Plasmid/Gene | 5.0 |
| Total | 10.0 |
将构建成功的5株携带重组质粒的宿主菌过夜活化后,1%接种量转接于含终浓度50 μg/mL卡那抗生素的LB液体培养基中。于37 ℃摇床中培养至OD620 0.6−0.7,加入终浓度60 mmol/L IPTG于18 ℃过夜培养。离心收集菌体,用0.05 mmol/L PBS缓冲溶液稀释菌体并在冰上超声破碎(功率300 W,工作3 s,间歇3 s,30 min),全菌体于4 ℃离心分离得上清和沉淀。
SDS-PAGE检测表达量:取待测样品30 μL,加入10 μL的4×上样缓冲液,混匀后煮沸10 min。300 V电压20−30 min后,取出蛋白胶通过凝胶检测系统进行检测。比较对应位置处蛋白条带鉴定重组酶是否表达或纯化。
1.4.2 重组酶的酶活测定目前,褐藻胶裂解酶的酶活力测定方法主要有硫代巴比妥酸法(TBA)[21]、紫外吸收法[22-23]、粘度法[24]和还原糖法(DNS)[25-26]。紫外吸收法是基于酶解产生的不饱和糖醛酸在230−240 nm处有较高的吸收峰,这种方法比较灵敏,是目前褐藻胶裂解酶酶活力测定的主要方法。
酶活力单位的定义为:235 nm下吸光值每分钟增加0.1所需的酶量作为一个酶活单位。底物配制:50 mmol/L磷酸钠缓冲液(pH 8.0),0.5%海藻酸钠,0.4 mol/L氯化钠。0.3 mL底物加0.l mL适当稀释的酶液于40 ℃水浴保温20 min后,加入0.6 mL HCl终止反应,在235 nm下测定其吸光值。
1.5 Gu柱亲和层析法纯化重组酶采用重力柱(购自大连TaKaRa公司)对重组酶进行纯化,缓冲液体系见表 5,具体操作步骤见说明书。
| Na2HPO4-NaH2PO4 buffer (mmol/L) |
NaCl (mmol/L) |
Imidazole (mmol/L) |
pH | |
| Equilibration buffer |
50 | 300 | / | 7.4 |
| Wash buffer |
50 | 300 | 10 | 7.4 |
| Elution buffer |
50 | 300 | 150 | 7.4 |
收集的流出液通过SDS-PAGE检测纯度:取待测样品30 μL,加入10 μL的4×上样缓冲液,混匀后煮沸10 min。300 V电压20−30 min后,取出蛋白胶通过凝胶检测系统进行检测。比较对应位置处蛋白条带检测纯化效果。
1.6 重组酶酶学性质的研究 1.6.1 重组酶rAlgV3的最适温度及其稳定性(1) 等体积的酶与底物混合,分别在4 ℃、20 ℃、30 ℃、35 ℃、40 ℃、45 ℃、50 ℃、60 ℃和70 ℃水浴中反应20 min测定酶活。
(2) 对于热稳定性,将重组酶酶液在4 ℃冰箱及20 ℃、30 ℃、35 ℃、40 ℃、45 ℃、50 ℃、60 ℃和70 ℃水浴1 h后,迅速冷却至4 ℃后测定酶活。
1.6.2 重组酶rAlgV3的最适pH及其稳定性(1) 分别用以下缓冲液调节反应体系的pH:乙酸-乙酸钠缓冲液(pH 4.5、5.0、5.5、6.0),Na2HPO4-NaH2PO4缓冲液(pH 6.0、6.5、7.0、7.5、8.0),Tris-HCl缓冲液(pH 8.0、8.5、9.0)和硼砂-氢氧化钠缓冲液(pH 9.0、9.5、10.0),测定褐藻胶裂解酶在不同pH条件下的酶活力。
(2) 将酶分别加入到上述几种缓冲液体系中,并于4 ℃保存24 h,测定酶活力。
1.6.3 不同离子对重组酶的影响在酶与底物的反应体系中分别加入终浓度为1 mmol/L的SDS、CuCl2、MnCl2、NaCl、KCl、(NH4)2SO4、MgCl2、FeSO4、FeCl3、EDTA溶液,未加任何离子反应体系的酶活定义为100%,测定其他反应体系中的酶活力。
1.6.4 不同NaCl浓度对酶活的影响在酶与底物的反应体系中,分别加入终浓度为0、100、300、500、700、900 mmol/L的NaCl溶液,测定酶活力。
1.6.5 重组酶底物偏好性分析将2 μL褐藻胶裂解酶液和携带空载体的裂解菌液分别加在含0.5%褐藻酸钠、Poly-G、Poly-M的平板上,40 ℃培养24 h后,加入CaCl2溶液观察酶对底物分解情况进行分析。
1.7 褐藻胶裂解酶降解产物ESI-MS分析为了确定降解产物的聚合度,利用ESI-MS进行质谱分析。所用仪器型号:LTQ Orbitrap XL,加热器温度:400 ℃,载气流量:10 L/min,辅助气体流量:10/3 L/min,毛细管温度:350 ℃,毛细管电压:39 V,扫描质量范围150–1 500 m/z。
2 结果与分析 2.1 褐藻胶裂解酶基因的克隆及表达载体的构建AlgV1–algV5基因PCR扩增产物和阳性克隆鉴定结果见图 2,条带清晰,与理论大小值相符(algV1–algV5碱基数分别为1 044、1 554、1 752、2 166和2 256 bp)。5个重组质粒经生工生物工程(上海)股份有限公司测序,其中插入基因的序列与已知基因组测序的序列相似度均大于98%,这表明algV1–algV5序列正确。

|
| 图 2 AlgV1–algV5的PCR结果 Figure 2 PCR identification of the gene algV1−algV5. M: DNA marker; lane 1: PCR fragment of algV1; lane 2: PCR fragment of algV2; lane 3: PCR fragment of algV3; lane 4: PCR fragment of algV4; lane 5: PCR fragment of algV5. |
| |
经诱导表达后,各重组菌超声破碎后超声上清液和超声沉淀中重组蛋白表达情况如图 3所示。由图可以看出rAlgV1–rAlgV5在相应位置均有表达,与经诱导携带空载体菌液相比,重组酶rAlgV2–rAlgV5均产生了可溶性蛋白。rAlgV1表达产物均在沉淀中,可能是由于重组酶产生了包涵体等不溶蛋白。

|
| 图 3 重组酶rAlgV1–rAlgV5的SDS-PAGE分析 Figure 3 SDS-PAGE analysis of recombinant protein rAlgV1–rAlgV5. (A) SDS-PAGE analysis of rAlgV1–rAlgV5 supernatant after ultrasonication. lane 1: rAlgV1; lane 2: rAlgV2; lane 3: rAlgV3; lane 4: rAlgV4; lane 5: rAlgV5; lane 6: control; M: protein marker. (B) SDS-PAGE analysis of rAlgV1–rAlgV5 sediment after ultrasonication. M: protein marker; lane 1: control; lane 2: rAlgV1; lane 3: rAlgV2; lane 4: rAlgV3; lane 5: rAlgV4; lane 6: rAlgV5. |
| |
通过紫外吸收法测定5株重组菌发酵上清和超声破碎后上清的酶活,结果见表 6。
| Strain/BL21/BL21 | Fermentation supernatant (U/L) |
Ultrasound supernatant (U/L) |
Total enzyme activity (U/L) |
| pET29a-AlgV1 | 179 | Not detected | 179 |
| pET29a-AlgV2 | 227 | 5 416 | 5 643 |
| pET29a-AlgV3 | 111 | 23 266 | 23 377 |
| pET29a-AlgV4 | Not detected | Not detected | Not detected |
| pET29a-AlgV5 | Not detected | 167 | 167 |
通过体外测定酶活验证algV1、algV2、algV3、algV5基因为海藻酸裂解酶编码基因;其中rAlgV1–rAlgV3这3个重组菌均有胞外褐藻胶酶活性,这与胥晴晴[20]研究结果一致,但是胞外酶活性相对较低,应用价值较小;由图 3可以看出rAlgV4和rAlgV5均进行了大量可溶性表达,但是rAlgV4胞内外均未检测到酶活,原因可能是其褐藻胶裂解酶的活性低,褐藻胶裂解酶活性的定量测活方法不灵敏导致未能测出海藻酸裂解酶的活性,也可能其本身就无海藻酸裂解酶活性,而与褐藻胶异构酶的活性相关;同样rAlgV5仅在胞内检测到少量酶活,可能原因与rAlgV4相同;重组蛋白rAlgV2表达量较高,酶活也较高,具有一定的应用潜力;重组酶rAlgV3的酶活性很高,尤其是胞内酶活,这比文献报道结果高了23倍,具有很高的应用价值[20]。接下来的实验选择rAlgV3为研究对象,通过优化诱导条件,提高rAlgV3的表达量,进而对该酶进行分离纯化与酶性质的表征。
2.2.2 rAlgV3诱导条件的优化为了提高rAlgV3的表达量,本研究对诱导温度、诱导OD、诱导剂IPTG浓度以及诱导时间4个方面进行优化,结果见图 4。由优化结果可知,最佳诱导条件为37 ℃培养菌体OD达到0.4,于25 ℃进行诱导6 h。诱导剂IPTG对酶活影响不大。优化后重组酶rAlgV3表达量提高,酶活达到1.68×105 U/L,比优化前提高了7.3倍。

|
| 图 4 重组酶rAlgV3诱导条件的优化 Figure 4 Optimization of induction conditions on the recombinant alginate lyase rAlgV3. (A) Effect of temperature on the recombinant alginate lyase rAlgV3. (B) Effect of OD on the recombinant alginate lyase rAlgV3. (C) Effect of IPTG on the recombinant alginate lyase rAlgV3. (D) Effect of induction time on the recombinant alginate lyase rAlgV3. |
| |
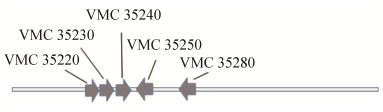
由于重组酶rAlgV3带有His-Tag,通过TaKaRa公司的重力柱进行纯化,收集的流出液进行SDS-PAGE (图 5),发现纯化的褐藻胶裂解酶有两条相近条带,通过Western blotting发现两条带均带有组氨酸标签,可能原因为质粒pET29a与目的基因均存在起始密码子,或者蛋白酶的部分降解导致。对收集到的纯酶进行活性测定为398 U/mg。
|
| 图 5 重组酶rAlgV3重力柱纯化SDS-PAGE分析 Figure 5 SDS-PAGE analysis of purified protein rAlgV3. M: protein marker; lane 1: control; lane 2: supernatant before purification; lane 3: fluid through the gravity column; lane 4–6: eluent after purification. |
| |
重组酶rAlgV3的最适温度和热稳定性:等体积的酶rAlgV3与底物混合后,分别在4 ℃、20 ℃、30 ℃、35 ℃、40 ℃、45 ℃、50 ℃、60 ℃和70 ℃水浴中反应20 min测定酶活,结果见图 6A。由结果可知,该酶40 ℃酶活最高,而在4−70 ℃均有酶活,可见该酶反应的温度范围较广,可在多种反应温度中应用。

|
| 图 6 重组酶rAlgV3的最适温度(A)和热稳定性(B) Figure 6 The optimal temperature (A) and thermal stability (B) of the recombinant rAlgV3. |
| |
对于热稳定性,将重组酶rAlgV3酶液在4 ℃冰箱及20 ℃、30 ℃、35 ℃、40 ℃、45 ℃、50 ℃、60 ℃和70 ℃水浴1 h后,迅速冷却至4 ℃后测定相对酶活,结果见图 6B。由结果可知,该重组酶rAlgV3在20 ℃以内酶活变化较小,具有很好的稳定性,30 ℃以上酶活下降,到60 ℃以上酶活性完全丧失。这说明重组酶rAlgV3不宜在高温环境中长期保存。
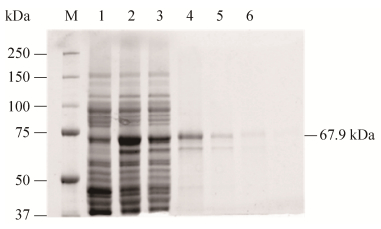
重组酶rAlgV3的最适pH和pH稳定性:分别用以下缓冲液调节反应体系的pH:乙酸-乙酸钠缓冲液(pH 4.5、5.0、5.5、6.0),Na2HPO4-NaH2PO4缓冲液(pH 6.0、6.5、7.0、7.5、8.0),Tris-HCl缓冲液(pH 8.0、8.5、9.0)和硼砂-氢氧化钠缓冲液(pH 9.0、9.5、10.0),测定褐藻胶裂解酶不同pH条件下的相对酶活,结果见图 7A。重组酶rAlgV3在pH 5.0以下几乎没有酶活,直至pH 8.0达到最大值,当pH大于8.0后酶活下降,当pH为10时,酶活性完全丧失。由图可知,重组酶rAlgV3适合反应的pH为6.5−9,具有广泛pH特性。
|
| 图 7 重组酶rAlgV3的最适pH值(A)和pH稳定性(B) Figure 7 Effects of pH on the recombinant rAlgV3. (A) The optimal pH of the recombinant rAlgV3. (B) The pH stability of the recombinant rAlgV3. |
| |
将酶分别加入到以上3种缓冲液体系中,并放于4 ℃冰箱中保存24 h,测定相对酶活,结果见图 7B,pH 4.5−9.5时相对酶活变化不大,而pH为10时,酶活几乎完全丧失,由其变化趋势可知,重组酶rAlg2的pH稳定范围为4.5−9.5,可见该酶具有耐酸耐碱的特性,在不同pH中稳定性很好,与目前文献报道的酶活pH稳定性相比具有明显优势,具有巨大的工业应用潜力。
不同离子对重组酶rAlgV3酶活的影响:在酶与底物的反应体系中分别加入终浓度为1 mmol/L的SDS、CuCl2、MnCl2、NaCl、KCl、(NH4)2SO4、MgCl2、FeSO4、FeCl3、EDTA溶液,未加任何离子反应体系的酶活定义为100%,测定反应体系中的相对酶活,结果见图 8。结果表明,SDS、Cu2+具有明显抑制酶活作用,而Fe2+、Fe3+有促进酶活的作用,其他离子影响不大。
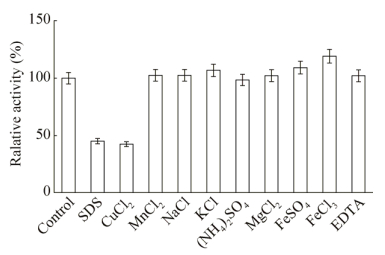
|
| 图 8 离子对重组酶rAlgV2酶活的影响 Figure 8 Effects of reagents on enzymatic activity of recombinant rAlgV3. |
| |
不同NaCl浓度对重组酶rAlgV3活性的影响:由于褐藻胶裂解酶主要产自海洋微生物,NaCl浓度是其发生反应的一个重要影响因素,故考察了高NaCl浓度对该酶的影响。在酶与底物的反应体系中,分别加入终浓度为0、100、300、500、700、900 mmol/L的NaCl溶液,测定相对酶活,结果见图 9。由图可以看出,NaCl浓度为300−500 mmol/L时,酶活性最高,适量的NaCl对酶活具有促进作用。

|
| 图 9 不同NaCl浓度对rAlgV3酶活性的影响 Figure 9 Effects of different NaCl concentration on the rAlgV3 lyase activity. |
| |
重组酶rAlgV3底物偏好性分析:由于古罗糖醛酸的聚合物(Poly-G)能和Ca2+形成白色的胶状络合物,而聚甘露糖醛酸(Poly-M)和Ca2+结合是无色的,因此,如果加入的酶能够降解Poly-G,则滤纸片周围出现透明的圆圈;如果不降解Poly-G,则出现白色的晕圈;如果既降解Poly-G又降解Poly-M,则也是出现透明圈。根据此原理,将2 μL褐藻胶裂解酶液和携带空载的裂解菌液分别加在0.5%海藻酸钠、Poly-G、Poly-M成分的平板上,40 ℃培养24 h后加入CaCl2溶液,结果见图 10。分别以0.5%的海藻酸钠、Poly-G、Poly-M为底物,检测相对酶活,结果见图 11。
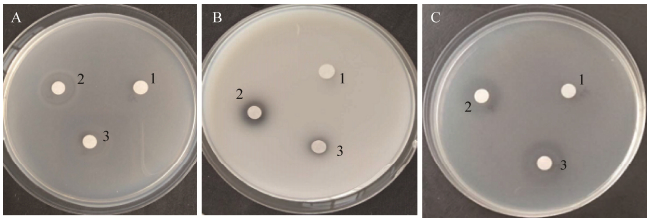
|
| 图 10 褐藻酸酶降解不同成分平板的结果 Figure 10 Results of decomposing sodium alginate (A), Poly-G (B) and Poly-M (C). 1: control; 2, 3: rAlgV3. |
| |

|
| 图 11 重组酶rAlgV3的底物偏好性 Figure 11 The substrate specificity of the recombinant rAlgV3. |
| |
由结果可知,该酶在3个平板上均产生透明圈,且对3种底物都有活性,说明该酶既可以分解Poly-M片段,也可以分解Poly-G片段,具有广泛底物特性,且偏好Poly-M。目前发现的褐藻胶裂解酶普遍具有单一性,能同时分解两种片段的酶比较少,该酶的发现可将褐藻胶有效降解成单糖,为褐藻的生物转化奠定基础。
2.4 褐藻胶裂解酶降解产物ESI-MS分析为了对该酶的降解产物作进一步的分析,采用ESI-MS分析方法,将0.9 mL的0.5%海藻酸钠与0.1 mL重组酶rAlgV3酶液于40 ℃反应1 h,然后煮沸5 min灭活,产物离心脱盐后,进行ESI-MS质谱分析(图 12)。

|
| 图 12 褐藻胶裂解酶降解产物ESI-MS分析 Figure 12 ESI-MS analysis of alginate lyase degradation products. |
| |
从质谱图上可以看出,对于海藻酸盐,酶解后产物主要是聚合度较低的寡糖,其中二糖和三糖为主要降解产物(图 12),可见该酶为内切酶。褐藻胶寡糖应用广泛,该酶可作为生产褐藻胶寡糖的有效工具。
3 结论本研究对解藻酸弧菌上可能的5个褐藻胶裂解酶进行了克隆表达,成功构建了5株重组菌,其中观察到重组酶rAlgV3具有较高酶活。对重组酶rAlgV3进行了诱导条件的优化,该酶的表达量明显提高,且酶活力达到1.68×105 U/L,相比文献报道的rAlgV3酶活性高出168倍[20],可能由于蛋白序列差异引起的,值得进一步探究。对重组酶rAlgV3的酶学性质进行探究发现,其对环境变化高度适应,具有较广的温度范围和很好的pH稳定性,这与大部分报道的褐藻胶裂解酶相比具有明显的优势。对重组酶rAlgV3底物偏好性分析发现,该酶既能分解Poly-M片段,也能分解Poly-G片段,具有底物多样性的特点。ESI-MS分析发现,该酶可将底物主要降解为二糖和三糖,是产生褐藻胶寡糖的有效工具,可广泛应用在医药、食品、能源等领域,是一种在商业上具有应用潜力的酶。
| [1] | Wong TY, Preston LA, Schiller NL. Alginate lyase: review of major sources and enzyme characteristics, structure-function analysis, biological roles, and applications. Ann Rev Microbiol, 2000, 54(1): 289–340. DOI: 10.1146/annurev.micro.54.1.289 |
| [2] | Takeda H, Yoneyama F, Kawai S, et al. Bioethanol production from marine biomass alginate by metabolically engineered bacteria. Energy Environ Sci, 2011, 4(7): 2575–2581. DOI: 10.1039/c1ee01236c |
| [3] | Ravanal MC, Pezoa-Conte R, Von Schoultz S, et al. Comparison of different types of pretreatment and enzymatic saccharification of Macrocystis pyrifera for the production of biofuel. Algal Res, 2016, 13: 141–147. DOI: 10.1016/j.algal.2015.11.023 |
| [4] | Thomas F, Lundqvist LCE, Jam M, et al. Comparative characterization of two marine alginate lyases from Zobellia galactanivorans reveals distinct modes of action and exquisite adaptation to their natural substrate. J Biol Chem, 2013, 288(32): 23021–23037. DOI: 10.1074/jbc.M113.467217 |
| [5] | Kim HS, Lee CG, Lee EY. Alginate lyase: structure, property, and application. Biotechnol Bioprocess Eng, 2011, 16(5): 843–851. DOI: 10.1007/s12257-011-0352-8 |
| [6] | Haug A, Larsen B. Quantitative determination of the uronic acid composition of alginates. Acta Chem Scand, 1962, 16: 1908–1918. DOI: 10.3891/acta.chem.scand.16-1908 |
| [7] | Tømmeraas K, Vårum KM, Christensen BE, et al. Preparation and characterization of oligosaccharides produced by nitrous acid depolymerisation of chitosans. Carbohydr Res, 2001, 333(2): 137–144. DOI: 10.1016/S0008-6215(01)00130-6 |
| [8] | Nagasawa N, Mitomo H, Yoshii F, et al. Radiation-induced degradation of sodium alginate. Polym Degrad Stab, 2000, 69(3): 279–285. DOI: 10.1016/S0141-3910(00)00070-7 |
| [9] |
Zhao JP, Yu DH, Zhu GJ, et al. Study on ultrasound method in the degradation reaction of sodium alginate.
Chin J Spectrosc Lab, 2009, 26(2): 242–245.
(in Chinese). 赵建平, 于大海, 朱光军, 等. 超声方法对于海藻酸钠的降解反应研究. 光谱实验室, 2009, 26(2): 242-245. |
| [10] | Nakada HI, Sweeny PC. Alginc acid degradation by eliminases from abalone hepatopancreas. J Biol Chem, 1967, 242(5): 845–851. |
| [11] | Romeo T, Preston Ⅲ JF. Liquid-chromatographic analysis of the depolymerization of (1→4)-β-D-mannuronan by an extracellular alginate lyase from a marine bacterium. Carbohydr Res, 1986, 153(2): 181–193. DOI: 10.1016/S0008-6215(00)90261-1 |
| [12] | Yamasaki Y, Yokose T, Nishikawa T, et al. Effects of alginate oligosaccharide mixtures on the growth and fatty acid composition of the green alga Chlamydomonas reinhardtii. J Biosci Bioeng, 2012, 113(1): 112–116. DOI: 10.1016/j.jbiosc.2011.09.009 |
| [13] | Tusi SK, Khalaj L, Ashabi G, et al. Alginate oligosaccharide protects against endoplasmic reticulum-and mitochondrial-mediated apoptotic cell death and oxidative stress. Biomaterials, 2011, 32(23): 5438–5458. DOI: 10.1016/j.biomaterials.2011.04.024 |
| [14] | Liu SY, Liu GY, Yi YT. Novel vanadyl complexes of alginate saccharides: synthesis, characterization, and biological activities. Carbohydr Polym, 2015, 121: 86–91. DOI: 10.1016/j.carbpol.2014.11.069 |
| [15] | Xu X, Iwamoto Y, Kitamura Y, et al. Root growth-promoting activity of unsaturated oligomeric uronates from alginate on carrot and rice plants. Biosci Biotechnol Biochem, 2003, 67(9): 2022–2025. DOI: 10.1271/bbb.67.2022 |
| [16] | Zhu BW, Yin H. Alginate lyase: Review of major sources and classification, properties, structure-function analysis and applications. Bioengineered, 2015, 6(3): 125–131. DOI: 10.1080/21655979.2015.1030543 |
| [17] | Falkeborg M, Cheong LZ, Gianfico C, et al. Alginate oligosaccharides: enzymatic preparation and antioxidant property evaluation. Food Chem, 2014, 164: 185–194. DOI: 10.1016/j.foodchem.2014.05.053 |
| [18] | Wargacki AJ, Leonard E, Win MN, et al. An engineered microbial platform for direct biofuel production from brown macroalgae. Science, 2012, 335(6066): 308–313. DOI: 10.1126/science.1214547 |
| [19] | Enquist-Newman M, Faust AME, Bravo DD, et al. Efficient ethanol production from brown macroalgae sugars by a synthetic yeast platform. Nature, 2014, 505(7482): 239–243. DOI: 10.1038/nature12771 |
| [20] |
Xu QQ. The research on alginate lyase genes in Vibrio Alginolyticus ATCC 17749[D]. Shanghai: East China University of Science and Technology, 2012 (in Chinese). 胥晴晴. 解藻酸弧菌ATCC 17749株海藻酸裂解酶基因的相关研究[D]. 上海: 华东理工大学, 2012. |
| [21] | Hurwitz J, Weissbach A. The formation of 2-keto-3-deoxyheptonic acid in extracts of Escherichia coli B. Ⅱ. Enzymic studies. J Biol Chem, 1959, 234(4): 705–709. |
| [22] | Zhu BW, Tan HD, Qin YQ, et al. Characterization of a new endo-type alginate lyase from Vibrio sp. W13. Int J Biol Macromol, 2015, 75: 330–337. DOI: 10.1016/j.ijbiomac.2015.01.053 |
| [23] | Li SY, Wang LN, Hao JH, et al. Purification and characterization of a new alginate lyase from marine bacterium Vibrio sp. SY08. Mar Drugs, 2017, 15(1): 1–12. |
| [24] | Sawabe T, Ohtsuka M, Ezura Y. Novel alginate lyases from marine bacterium Alteromonas sp. strain H-4. Carbohydr Res, 1997, 304(1): 69–76. DOI: 10.1016/S0008-6215(97)00194-8 |
| [25] | Nelson N. A photometric adaptation of the somogyi method for the determination of glucose. J Biol Chem, 1944, 153: 375–380. |
| [26] | Stevens RA, Levin RE. Purification and characteristics of an alginase from Alginovibrio aquatilis. Appl Environ Microbiol, 1977, 33(5): 1156–1161. |
 2018, Vol. 34
2018, Vol. 34