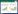中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王思琦, 张昭寰, 穆丽丽, 刘海泉, 潘迎捷, 赵勇
- Wang Siqi, Zhang Zhaohuan, Mu Lili, Liu Haiquan, Pan Yingjie, Zhao Yong
- 人工模拟胃肠道模型在食源性致病菌耐受及致病机理中的应用
- Applications of simulated gastro-intestinal model in foodborne pathogens:tolerance and pathogenesis
- 生物工程学报, 2018, 34(6): 839-851
- Chinese Journal of Biotechnology, 2018, 34(6): 839-851
- 10.13345/j.cjb.180058
-
文章历史
- Received: February 9, 2018
- Accepted: April 8, 2018
2 上海水产品加工及贮藏工程技术研究中心, 上海 201306;
3 农业部水产品贮藏保鲜质量安全风险评估实验室 (上海), 上海 201306;
4 上海海洋大学 食品热加工工程技术研究中心, 上海 201306
2 Shanghai Engineering Research Center of Aquatic-Product Processing & Preservation, Shanghai 201306, China;
3 Laboratory of Quality & Safety Risk Assessment for Aquatic Product on Storage and Preservation (Shanghai), Ministry ofAgriculture, Shanghai 201306, China;
4 Engineering Research Center of Food Thermal-processing Technology, Shanghai Ocean University, Shanghai 201306, China
食源性致病菌引起的食品安全问题是全球性的焦点问题,也是亟待解决的科研难点问题[1]。据世界卫生组织(WHO)研究报道,全世界每年共暴发6亿起食品安全事件,其中28%是由食源性致病菌的污染所导致的[2]。常见食源性致病菌包括大肠杆菌、沙门氏菌、金黄色葡萄球菌、副溶血性弧菌及单增李斯特菌等,均能引起发烧、呕吐、腹泻、流产等症状,严重的可导致原发性败血症甚至致人死亡,对食品安全和公众健康构成了极大的威胁[3-4]。在人体胃肠道中对食源性致病菌的耐受性及致病机理进行研究,有助于揭示其导致人体患病的根本原因,从源头上遏制食源性致病菌的患病风险[5]。但是,由于现阶段科研条件所限,且有悖道德伦理等方面的因素,往往难以对人体胃肠道直接进行取样研究[6]。因此,开发替代人体实验的人工模拟胃肠道模型,具有重要的科研价值和实际意义。
人工模拟胃肠道模型(Stimulated gastro- intestinal tract model)是在实验室条件下,通过模拟人体消化过程中化学、物理及生物作用,进行食源性致病菌的耐受性、致病机理、肠道菌群互作及疫苗开发等研究。一般来说,可将人工模拟胃肠道模型分为体外模型和体内模型。体外模型指的是对人体消化液及胃肠道内壁的蠕动进行模拟,如Wijnands等[7]使用体外静态模型对沙门氏菌在模拟胃液中的存活进行研究,Roussel等[8]则使用体外动态模型对成年人及儿童胃肠道进行模拟,并结合流式细胞仪探究了大肠杆菌在不同人群中致病力的区别;体内模型通常依靠实验动物进行模拟,如Ritchie等[5]使用新西兰幼兔对副溶血性弧菌的致病力及在小肠的定植作用进行了研究,Mathur等[6]首次将人源化小肠上皮细胞的小鼠模型应用于沙门氏菌疫苗的鉴定。人工模拟胃肠道模型的出现弥补了人体试验的不足,随着此类模型的快速发展,已逐步应用于各类食源性致病菌耐受及致病机理的研究,具有广泛的应用前景和巨大的发展潜力。
因此,本文针对食源性致病菌这一重点、难点问题,详细综述了人工模拟胃肠道模型在其耐受性及致病机理研究中的应用,将现存的人工模拟胃肠道模型系统地划分为4类:体外静态模型、体外动态模型、普通动物模型及人源化动物模型,对其概念和特性进行了详细的介绍,总结了其优势与不足,并提出了人工模拟胃肠道模型未来的研究方向,以期提供全面系统的信息,为食源性致病菌致病机理研究的进一步发展奠定坚实的理论基础。
1 体外胃肠模型人体胃肠道是一个非常复杂的内部结构,如图 1所示,包括胃、小肠(十二指肠、空肠、回肠)及大肠(横结肠、升结肠、降结肠、盲肠),还涉及胃肠道内壁蠕动以及胃酸、胆汁、蛋白酶的分泌等一系列物理化学的过程[9]。体外胃肠模型(In-vitro gastro-intestinal system)通常在实验室条件下,运用相关设备结合人工添加的模拟消化液(模拟胃液、模拟肠液等),对胃肠道消化过程中物理和化学作用进行模拟。目前,体外胃肠模型已被广泛应用于微生物学、毒理学及营养学等领域。相较于活体实验,体外胃肠模型能够降低实验成本,具有较高的重复性和准确性,且不受到伦理道德的限制。本文根据体外模型能否模拟人体胃肠道蠕动过程中的物理作用,将其分为体外静态模型(In-vitro static gastro-intestinal model)和体外动态模型(In-vitro dynamic gastro- intestinal model),现综述如下。
体外静态胃肠模型对胃肠道消化液的化学成分进行模拟,其并不涉及复杂物理及生物作用。如图 2所示,该模型将锥形瓶、离心管或烧杯等作为反应器,通过添加模拟胃液、肠液、胆汁等[11-12],在模拟人体正常体温中(37 ℃)进行反应。该模型具有操作简便、重复性好等优点,是目前使用最为广泛的人工模拟胃肠道模型,可应用于研究食源性致病菌的耐受性、毒力表达及对肠上皮细胞的作用等。
|
| 图 2 体外静态胃肠模型模拟示意图 Figure 2 A schematic diagram of in-vitro static gastro-intestinal model simulation. |
| |
表 1综述了现阶段体外静态胃肠模型在食源性致病菌研究中的应用情况,主要为探究致病菌在人体模拟消化中的耐受性。1995年,Arnold等[13]研究了纯培养状态下不同致病性的大肠杆菌在pH 1.5的模拟胃液中的存活能力,结果表明,较肠侵袭性、肠致病性大肠杆菌及痢疾志贺氏菌而言,肠出血性大肠杆菌具有更强的存活能力。1999年Roering等[14]用pH 1.5模拟胃液处理大肠杆菌O157:H7、单增李斯特菌及鼠伤寒沙门氏菌时,发现在模拟胃液中大肠杆菌具有较高的存活率。2008年Tamplin[15]研究了牛肉中大肠杆菌在模拟胃液中的失活特性,证明了pH值是造成大肠杆菌失活的主要因素。2007年Bergholz等[16]探究了果肉基质中耐四环素大肠杆菌的耐酸能力,发现耐四环素大肠杆菌在模拟胃液中耐酸能力高于标准菌株,耐药质粒转移程度是标准菌株的2.3倍。此外,静态模型还可与细胞实验相结合,如2011年Wijnands等[7]将经过模拟胃液和肠液处理的沙门氏菌加入含有Caco-2细胞的12孔板,在5% CO2气体状态下,研究了该菌对人体细胞的黏附能力(ATT)和侵袭能力(ITV)。
| Strain | Research | Enzymes or chemicals | Simulated conditions | Reference |
| Escherichia coli | 1. Adaptation of E. coli O157:H7 to pH alters resistance and verotoxin secretion to simulated gastric fluid acid | Gastric juice: 8.3 g/L proteose- peptone, 3.5 g/L glucose, 0.013 3 g/L pepsin, 0.1 g/L lysozyme, 0.05 g/L bile salt | Temperature: 37 ℃ Time: 0-2 h Treatment: static pH: 1.5 | [17] |
| 2. Survival of E. coli O157:H7 in simulated gastric fluid | Gastric juice: 3 g/L pepsin | Temperature: 37 ℃ Time: 0-2 h Treatment: 100 r/min pH: 2.0 | [18] | |
| 3. Survival of E. coli O157:H7 in simulated intestinal fluid | Intestine juice: 0.1 g/L pancreatin, 8.5 g/L saline | Temperature: 37 ℃ Time: 0-2 h Treatment: 100 r/min pH: 8.0 | [18] | |
| 4. Inactivation rate of E. coli in raw ground beef and saline in simulated gastric fluid | Gastric juice: 8.3 g/L proteose-peptone, 3.5 g/L glucose, 0.05 g/L ox bile, 0.1 g/L lysozyme, 0.013 3 g/L pepsin | Temperature: 37 ℃ Time: 4 h Treatment: static pH: 1.5 | [15] | |
| 5. Effect of storage in juice with or without pulp on the survival of E. coli O157:H7 in simulated gastric fluid | Gastric juice: 8.3 g/L proteose- peptone, 3.5 g/L glucose, 0.013 3 g/L pepsin, 0.1 g/L lysozyme, 0.05 g/L bile | Temperature: 37 ℃ Time: 1.5-4 h Treatment: static pH: 1.5 | [19] | |
| Salmonella | 1. Quantification of Salmonella survival and infection in simulate gastric fluid | Gastric juice: 65.0 g/L glucose, 2.0 g/L glucuronic acid, 33.0 g/L glucosamine, 1.0 g/L bovine serum albumin, 25.0 g/L urea, 3.0 g/L mucin, 1.3 g/L pepsin | Temperature: 37 ℃ Time: 0.5 h Treatment: static pH: 2.5 | [7] |
| 2. Quantification of Salmonella survival and infection in simulate intestinal fluid | Gastric juice: 25.0 g/L urea, 1.0 g/L bovine serum albumin, 0.5 g/L lipase, 3.0 g/L pancreatin, 6.0 g/L bile, Caco-2 cell | Temperature: 37 ℃ Time: 2 h Treatment: 50 r/min pH: 8.0 Gas condition: 5% CO2 | [7] | |
| Listeria monocytogenes | 1. Survival of L. monocytogenes in simulated gastric fluid | Gastric juice: 65 g/L glucose, 1.0 g/L bovine serum albumin, 3.0 g/L mucin, 2.5 g/L pepsin | Temperature: 37 ℃ Time: 2 h Treatment: 120 r/min pH: 3.5 | [20] |
| 2. Survival of L. monocytogenes in simulated intestinal fluid | Intestine juice: 1.0 g/L bovine serum albumin, 1.5 g/L lipase, 9 g/L trypsin, 30 g/L bile | Temperature: 37 ℃ Time: 2 h Treatment: 120 r/min pH: 6.5 | [20] |
除细胞模型外,体外静态模型还可结合流式细胞仪、微流控及生物芯片等前沿技术,对食源性致病菌在胃肠道模拟消化过程中生理生化的变化进行研究。然而体外静态模型难以模拟人体胃肠道消化过程中的动态变化,如胃排空、胃蠕动、pH值变化、酶以及消化液分泌等动态过程,使得该模型在致病菌研究中仍存在一些不足[21]。为了克服这一缺点,研究人员开发了可模拟人体胃肠道蠕动时物理作用的体外动态胃肠模型。现阶段,该类模型主要有3种,分别是荷兰应用科学研究组织Minekus和Havenaar[22]构建的TIM模型(TNO intestinal model)、比利时根特大学Molly等[23]构建的SHIME模型(Simulator of the human intestinal microbial ecosystem)及马德里自治大学Barroso等[24]构建的SIMGI模型(Simulator of the gastro-intestinal tract)。
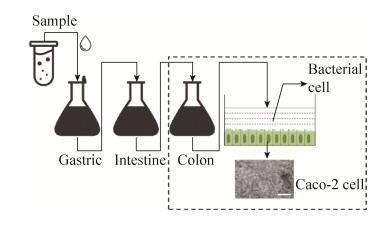
TIM模型是由Minekus和Havenaar[22]研制的,最初用于药物活性的动态测试,是一种基于多单元计算机控制的体外消化模拟系统,也是目前在食源性致病菌研究中应用最广泛、使用频次最高的体外动态胃肠模型系统。TIM模型可以分为两个部分:TIM1可用于模拟人体的胃、十二指肠、空肠、回肠以及结肠(图 3)[25]。TIM2模型可模拟人体大肠(横结肠、升结肠、降结肠、盲肠),如图 4所示,并增加了模拟肠道微生物的功能[26]。

|
| 图 3 TIM 1系统示意图(改自Verwei等[25]) Figure 3 The TIM-1 system (adapted from Verwei et al[25]). A: stomach; B: the compartments are connected by peristaltic valves controlling the gastric emptying; C: duodenum; D and F: intestinal transit; E: jejunum; G: ileum; I: simulated gastric secretion; J: duodenal secretion; K1 and K2: jejunum and ileum secretion; L: pre-filter; M: hollow fiber semi-permeable membrane; N: filtrate collection for analyzing bioaccessible API from jejunum (N1) and ileum (N2); P: pH electrode; Q: pressure sensor; R: temperature sensor (in each compartment); S: liquid level sensor; T: inlet. |
| |
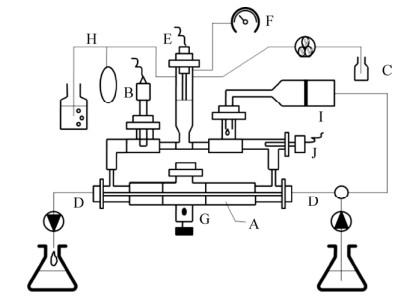
|
| 图 4 TIM 2系统示意图(改自Rehman等[26]) Figure 4 The TIM-2 system(adapted from Rehman et al[26]). A: peristaltic compartments containing fecal matter; B: pH electrode; C: alkali pump; D: dialysis liquid circuit with hollow fibre membrane; E: level sensor; F: N2 gas inlet; G: sampling port; H: gas outlet; I: ileal efflux; J: temperature sensor. |
| |
TIM系统可通过pH电极、温度及液位传感器等实现对模拟室的监控,并通过计算机程序控制对体内胃肠环境的稳定模拟,也可对处理样品随时抽样。2014年,Miszczycha等[27]将大肠杆菌O26:H11和大肠杆菌O157:H7接种于生牛乳,并将其发酵制作为奶酪,随后用TIM1系统研究奶酪中的大肠杆菌在模拟消化过程中的存活率变化。发现大肠杆菌O26:H11存活率比大肠杆菌O157:H7高12倍,同种属菌株间对模拟消化液的耐受性存在巨大差异。TIM系统还可通过回波平面磁共振成像和肠道菌群宏基因组测序,对孕妇、儿童及老人等不同人群胃肠道环境进行精准模拟[8]。2016年,Roussel等[8]利用TIM1系统对儿童和成年人胃肠道中大肠杆菌O157:H7的存活率和毒力基因表达变化进行模拟。虽然在传统平板涂布进行细菌计数的方法中,大肠杆菌O157:H7的存活率在两种模拟模型中没有明显差异,但通过流式细胞仪分析细胞活性并结合毒力基因表达结果,发现大肠杆菌O157:H7在儿童模型中的致病能力显著高于成年人模型。
SHIME系统是由Molly等[23]开发的由计算机控制的五步反应器模拟人体胃肠道微生物生态系统(图 5)。SHIME模型包括胃、小肠、升结肠、横结肠及降结肠五个反应器,反应器为双夹层的玻璃罐,有pH电极、液位及温度传感器,并由蠕动泵实现混合液的转移。该系统常用于研究食物成分与人体内常驻微生物群落的相互作用[8]。SIMGI系统与SHIME系统很相似,也具有小肠、升结肠、模拟罐、横结肠、降结肠五个反应器,微生物菌群结构也与SHIME系统基本一致。不同的是,SIMGI系统的胃部模拟装置并不是用双夹层的玻璃罐,而是由两个丙烯酸甲酯制成的透明塑料模块组成,并且利用覆盖着水的硅胶模对胃内容物的蠕动进行模拟,这种反应器的优点在于可以减少机械力对食源性致病菌的损伤,更加符合真实环境,但SIMGI系统造价昂贵,目前仍处于系统测试阶段,尚未投入应用[24]。
尽管体外模拟胃肠模型已广泛应用于致病菌耐受性的研究,但人体真实胃肠道具有复杂的组织结构、微生物群落及机体免疫等生物学特性,导致体外模型在研究致病菌致病机理中存在一定的局限性,需要运用更符合人体生命特性的模型进行研究。体内胃肠道模型(In-vivo gastro-intestinal model),也可称为动物模型(Animal model),通常利用遗传背景明确的实验动物进行模型的构建,并可对其组织、器官及所携带的微生物进行“人源化”的处理[28]。
常用动物模型包括啮齿目、兔形目、灵长目及偶蹄目等。由于灵长目、偶蹄目和奇蹄目存在培育时间较长、培育环境要求严格等困难,实验室用的动物模型多为啮齿目(小鼠、大鼠、豚鼠)和兔形目(兔、鼠兔)。1906年,Donaldson博士在Wistar研究所建立了第一个标准化的实验动物,迄今所使用的大鼠模型50%都属于Wistar品系[29]。本文根据实验动物模型的不同特性,分为普通动物模型和人源化动物模型分别进行综述。
2.1 普通动物模型普通动物模型(Conventional animal model)广义上是指来源清晰、背景明确且不经过任何“人源化”处理的动物模型。表 2主要综述了普通动物模型在食源性致病菌致病机理研究中的应用。以副溶血性弧菌致病机理研究为例,2012年Ritchie等[5]运用新西兰幼兔构建了副溶血性弧菌感染的动物模型,清晰地重现了该菌侵袭动物胃肠道的全过程:该菌首先吸附在肠上皮绒毛表面形成细胞簇,通过持续消耗肠上皮细胞的细胞质内容物,并对肠上皮细胞进行挤压,促使肠上皮细胞解体,造成幼兔的肠道组织水肿而导致水样腹泻。并通过基因敲除实验,在幼兔模型中进一步证明了三型分泌系统是副溶血性弧菌最为重要的产毒器官。同年,Whitaker等[30]利用C57BL/6小鼠模型对副溶血性弧菌ToxRS操纵子的作用进行了研究,结果表明,ToxRS操纵子的调控是副溶血性弧菌致病力的关键因素,toxRS敲除的菌株不能在小鼠肠道中定植。2014年,Livny等[31]再一次运用新西兰幼兔模型分析了感染时副溶血性弧菌的转录组,从全基因组表达的角度进一步研究了该菌的致病机理。
| Strain | Research | Animal model | Reference |
| Escherichia coli | Critical roles for stx2, eae, and tir in E. coli-induced diarrhea and intestinal inflammation | NZW rabbit | [32] |
| Salmonella | Horizontal transfer of the Salmonella virulence plasmid pESI to the gut microbiota | C57/BL6 mice | [33] |
| Listeria monocytogenes | Inhibition of lutein prevents L. monocytogenes infection | BALB/c mice | [34] |
| Vibrio parahaemolyticus | Mechanism for V. parahaemolyticus induced diarrhea | NZW rabbit | [5] |
| Comparative transcriptome analysis V. parahaemolyticus gene expression during infection | NZW rabbit | [31] | |
| Role of toxRS in V. parahaemolyticus RIMD2210633 during infection | C57BL/6 mice | [30] | |
| Vibrio vulnificus | Infection and metastasis of the MARTX toxin in V. vulnificus | ICR mice | [35] |
| NZW rabbit: New Zealand white rabbit; ICR mice: Institute of Cancer Research mice. | |||
尽管普通动物模型被视为食源性致病菌研究的可靠工具,但此类动物模型与真实的人体仍旧存在不小的差异。首先,与真实人体相比,小鼠等普通动物的肠道菌群仅有4%的相似性[36];第二,由于物种间的差异性,实验动物的免疫反应与人类差距较大,由普通动物模型中得到的结论不能直接推论于人体免疫系统[33]。因此,研究人员对普通动物模型进行了“人源化”改造,开发出了相应的人源化菌群动物模型和人源化免疫系统动物模型。
2.2.1 人源化菌群动物模型人源化菌群动物模型(Human flora-associated animal model,HFAAM),指的是在无菌动物体内接种人类粪便或肠道内容物的菌悬液,使人类肠道微生物在动物的胃肠道内定植,从而构建的肠道菌群“人源化”模型[37]。人源化菌群动物模型可用于研究食源性致病菌与宿主肠道菌群间的相互作用关系,从而揭示食源性致病菌的致病机理、在肠道内的定植作用,并提供可靠的治疗方案。如2008年Barman等[38]用沙门氏菌感染人源化菌群的Fvb小鼠模型,发现感染后的小鼠肠道细菌总数下降,菌群组成发生改变,食源性致病菌与肠道菌群存在相互作用关系,可通过肠道菌群的调整,对食源性致病菌引起的疾病进行治疗。2010年Faure等[39]运用人源化菌群的C3H大鼠模型,研究了沙门氏菌CTX-M耐药基因的转移,结果表明这一抗性基因可从沙门氏菌转移到肠道菌群,增加了耐药基因扩散的风险。2014年Ali等[40]运用人源化菌群的C57BL/6小鼠构建了沙门氏菌感染模型,发现果糖-天冬酰胺复合物对沙门氏菌的肠道定植起关键作用。2016年Bian等[41]运用人源化菌群的BALB/c小鼠构建大肠杆菌感染模型,研究了瑞士乳杆菌KLDS 1.8701和嗜酸乳杆菌KLDS AD1对大肠杆菌的致病作用,发现这两株菌可通过调整小鼠肠道菌群结构,缓解大肠杆菌导致的腹泻症状。
目前,最常用的人源化菌群动物模型构建方法是将粪便菌悬液接种于小鼠肠道,但是这种方法存在一定的缺陷,即并非所有的菌群都成功定植于小鼠肠道内部,研究表明约有20%的菌群会在定植过程中发生种水平的变化[42],使获得模型不能准确反映人体肠道菌群的真实情况,这将成为未来需要解决的一个重要科学问题。
2.2.2 人源化免疫系统动物模型人源化免疫系统动物模型(Humanized immune system animal model),一般通过人工敲除实验动物的免疫细胞,或对实验动物的相关免疫基因进行靶向失活,以获得免疫缺陷的动物,之后通过移植人类细胞、组织、器官或者基因,使普通动物具有人体免疫应答反应。图 6以重症联合免疫缺陷综合征(Severe combined immun deficiency,SCID)的小鼠模型为例,介绍4种不同人源化免疫系统小鼠模型的构建途径。
如图 6所示,Hu-PBL-SCID模型是由Mosier等[44]在1988年构建,将人外周血白细胞(Peripheral blood leukocytes,PBL)通过腹腔或静脉注射的方式,植入SCID小鼠体内,进行模型的构建,主要用于人类成熟免疫细胞功能的研究。Hu-SRC- SCID模型由Lapidot等[45]于1992年构建,从骨髓、脐带血、胎肝及外周血造血干细胞中获取人源造血干细胞(Hematopoietic stem cell,HSC),再经皮下或静脉注射入新生SCID小鼠、或静脉注射入成年SCID小鼠体内所构建的模型,造血干细胞能够在小鼠体内分化发育出造血系统的各系细胞,移植后可以重建免疫缺陷小鼠的造血系统,使其具有人源化的造血功能。2007年,Shultz等[46]在SCID小鼠的肾包膜下植入人类胎肝和胸腺,构建了SCID-Hu模型,可用于人类免疫缺陷病毒HIV-1的研究,但由于该模型缺乏人类造血和免疫干细胞,因此所模拟的免疫系统并不完整。BLT模型(Bone marrow,liver,thymus)综合了上述各类模型的优势,由Shultz等[46]于2007年构建,将人类胎肝和胸腺植入SCID小鼠的肾包膜下,同时静脉注射入同源的胎肝干细胞,使得小鼠不仅拥有完整的人源化免疫系统,而且具备健全的人源化造血功能,是现阶段较为可靠的人源化免疫/造血系统小鼠模型,具有广阔的应用前景。
2012年,哥伦比亚大学的Mathur等[6]通过Hu-PBL-SCID建立了首个鼠伤寒沙门氏菌感染的小鼠模型,研究了Toll样受体11 (TOLL-like receptor 11,TLR11)的免疫功能。TLR11在巨噬细胞、树突状细胞表面表达,能特异地识别病原相关的分子模式,在免疫反应中发挥着重要作用[47]。该研究构建了TLR11受体缺乏小鼠模型,经沙门氏菌感染可知,受体缺乏小鼠出现了伤寒症状,而在TLR11受体不缺乏小鼠感染实验中,TLR11能够在小鼠胃肠道上皮细胞中大量表达,以抵御沙门氏菌的侵袭。这一类模型建立的意义重大,口服感染的人源化模型可以清晰地重现食源性致病菌真实的致病过程(从口腔进入胃肠道),对于其致病机理的研究具有显著的优势[6]。
3 总结与展望体内、外胃肠道模型在食源性致病菌耐受性和致病性的研究中具有不可替代的优势,表 3系统地总结了各模型在食源性致病菌研究应用中的优势、缺陷及应用领域。其中,体外静态模型具有操作简便、重复性好等优点,广泛应用于致病菌耐受性及致病力的研究;但是,此类模型无法模拟胃肠道消化的动态过程,使其与真实的消化过程相去甚远。体外动态模型在体外静态模型的基础之上,结合计算机和蠕动泵等设备,弥补了这一缺陷,并可针对不同人群进行“个性化”模拟;但是,其仍缺乏肠道菌群及肠道组织结构等,难以对人体胃肠道的生物过程进行模拟。较体外模型而言,在动物体内构建的模型,不仅可对致病菌的耐受性及致病力进行研究,还可进一步研究其致病机理及疫苗的开发。普通动物模型可以较为真实地重现致病菌感染的全过程,但是与真实人体相比,实验动物的消化与免疫应答等反应仍存在一定程度上的差距。人源化动物模型可对实验动物的胃肠道或免疫系统进行“人源化”模拟,虽然构建过程相对艰难,但是一旦成功构建,该类模型在食源性致病菌致病机理及疫苗开发的研究中具有独一无二的优势。
| Model | Advantage | Disadvantage | Application |
| In-vitro static gastro-intestinal model | Easy to construct and repeat | No uniform formula; no dynamic simulation | Tolerance |
| In-vitro dynamic gastro-intestinal model | Dynamic gastrointestinal simulation; Adding human cells | No human gastrointestinal tissue; no human gut microbiota | Tolerance; virulence |
| Conventional animal model | Similar process and gastrointestinal tissue of digestion human | Low similarity compared to human gut microbiota; no human immunity stimulation; ethics restriction | Tolerance; virulence; pathogenesis |
| Humanized animal model | Humanized gut microbiota; Humanized immune system | Difficult to construct; ethics restriction | Tolerance; virulence; pathogenesis; vaccine development; interaction with gut microbiota |
综上所述,尽管经过了近一个世纪的发展,体内外胃肠道模型仍存在一定程度上的缺陷,需要进一步完善,使其更加符合研究的需求。因此,本文从以下4个方面对未来人工模拟胃肠道模型的研究方向进行了展望。
1) 开发更符合真实情况的体外胃肠道模型。体外胃肠模型具有操作简单、重复性好、可实时监控等优势,而且与体内实验相比,此类模型不涉及伦理谴责问题。在食源性致病菌研究中的应用前景十分广阔,具有不可取代的地位。但现行各类体外胃肠模型尚缺乏统一的标准,如果不同研究中所采用的模拟消化液配方不尽相同,构成模拟消化液酶类的来源也有所差异,例如人源酶、猪源酶及牛源酶等,甚至消化液的pH值也不尽相同,导致所获得的结果不能进行很好的比较归类,难以进行系统性研究[17-20]。因此,后续研究应对体外胃肠系统模拟条件作进一步修正,使此类模型更符合真实的情况,还可针对不同的人群,建立特异人群的胃肠道模型,同时开发更加简便的装置和更加仿真的模型,使其更好地应用于食源性致病菌的研究。
2) 综合运用系统生物学技术构建新型人源化动物模型。现阶段,人源化动物模型已广泛应用于食源性致病菌的研究,由于此类模型能够较好地模拟人体的肠道菌群或免疫系统,有助于重现致病菌在致病过程中的具体变化,以进行致病菌致病机理与疫苗开发等方面的研究。但是,目前将人源化菌群动物模型和人源化免疫系统动物模型相结合的研究还相对较少。后续研究应运用系统生物学理论,结合不断发展的组学技术,构建一种“1+1”的新型人源化动物模型,即在实验动物体内实现肠道菌群、组织器官的同步人源化研究,充分结合并发挥这两类模型的优势,从而对食源性致病菌的致病机理进行更全面系统的研究,为疾病研究及人类健康作出贡献。
3) 食源性致病菌在模拟胃肠道中耐受异质性研究。研究表明,食源性致病菌存在菌株异质性,指的是同种属的不同菌株之间存在生长、耐受、致病力等方面的差异[48-53]。但是,目前食源性致病菌在体内外模拟胃肠道模型中的研究往往局限于单一菌株,并不能很好地代表所有菌株耐受及致病性的总体情况。菌株异质性的存在,会对体内外模型中致病菌耐受性及致病机理的研究结果造成影响。因此,应运用体内外模型研究大量菌株的耐受性和致病力,总结其耐受异质性及致病异质性的规律,全面揭示其潜在的致病机制。
4) 综合运用体内外模拟胃肠道模型服务于食品安全风险评估。食品安全风险评估是一种科学可靠的工具,可用于评估食源性致病菌的致病风险,有效地控制食源性致病菌的潜在危害[1, 54]。然而,传统的风险评估模型通常将“入口概率”直接视为“致病概率”,忽略了致病菌进入人体之后的命运变化[55]。食源性致病菌在体内外模拟胃肠道中耐受及致病机理的研究,正好能够弥补风险评估的这一不足,对提升风险评估的准确性具有非常重要的意义。此外,目前国内外微生物风险评估中均在使用美国FDA所建立的剂量-反应模型[56],该模型不仅年代久远较为陈旧,而且不一定符合所有人种的身体情况,因此,可综合运用体内外模拟胃肠道模型,研究更符合实际的情况“剂量-反应”关系,以构建更加全面系统的食品安全风险评估体系。
| [1] |
Zhao Y, Wang JJ, Tang XY, et al. Risk assessment research status of food-borne pathogenic microorganism in aquatic products.
J Shanghai Ocean Univ, 2012, 21(5): 899–905.
(in Chinese). 赵勇, 王敬敬, 唐晓阳, 等. 水产品中食源性致病微生物风险评估研究现状. 上海海洋大学学报, 2012, 21(5): 899-905. |
| [2] | 2017 Fact sheet: Food safety. [2017-06-14]. http://www.who.int/mediacentre/factsheets/fs399/en/. |
| [3] | Velusamy V, Arshak K, Korostynska O, et al. An overview of foodborne pathogen detection:in the perspective of biosensors. Biotechnol Adv, 2010, 28(2): 232–254. DOI: 10.1016/j.biotechadv.2009.12.004 |
| [4] | Scallan E, Hoekstra RM, Mahon BE, et al. An assessment of the human health impact of seven leading foodborne pathogens in the United States using disability adjusted life years. Epidemiol Infect, 2015, 143(13): 2795–2804. DOI: 10.1017/S0950268814003185 |
| [5] | Ritchie JM, Rui HP, Zhou XH, et al. Inflammation and disintegration of intestinal villi in an experimental model for Vibrio parahaemolyticus- induced diarrhea. PLoS Pathog, 2012, 8(3): e1002593. DOI: 10.1371/journal.ppat.1002593 |
| [6] | Mathur R, Oh H, Zhang DK, et al. A mouse model of Salmonella typhi infection. Cell, 2012, 151(3): 590–602. DOI: 10.1016/j.cell.2012.08.042 |
| [7] | Wijnands LM, Teunis PFM, Kuijpers AFA, et al. Quantification of Salmonella survival and infection in an in vitro model of the human intestinal tract as proxy for foodborne pathogens. Front Microbiol, 2017, 8: 1139. DOI: 10.3389/fmicb.2017.01139 |
| [8] | Roussel C, Cordonnier C, Galia W, et al. Increased EHEC survival and virulence gene expression indicate an enhanced pathogenicity upon simulated pediatric gastrointestinal conditions. Pediatr Res, 2016, 80(5): 734–743. DOI: 10.1038/pr.2016.144 |
| [9] | Hur SJ, Lim BO, Decker EA, et al. In vitro human digestion models for food applications. Food Chem, 2011, 125(1): 1–12. DOI: 10.1016/j.foodchem.2010.08.036 |
| [10] | Blausen Medical. Medical gallery of Blausen Medical 2014. Wiki J Med, 2014, 1(2): 10. |
| [11] | Versantvoort CHM, Oomen AG, van de Kamp E, et al. Applicability of an in vitro digestion model in assessing the bioaccessibility of mycotoxins from food. Food Chem Toxicol, 2005, 43(1): 31–40. DOI: 10.1016/j.fct.2004.08.007 |
| [12] | Koseki S, Mizuno Y, Sotome I. Modeling of pathogen survival during simulated gastric digestion. Appl Environ Microbiol, 2011, 77(3): 1021–1032. DOI: 10.1128/AEM.02139-10 |
| [13] | Arnold KW, Kaspar CW. Starvation-and stationary-phase-induced acid tolerance in Escherichia coli O157:H7. Appl Environ Microbiol, 1995, 61(5): 2037–2039. |
| [14] | Roering AM, Luchansky JB, Ihnot AM, et al. Comparative survival of Salmonella typhimurium DT 104, Listeria monocytogenes, and Escherichia coli O157:H7 in preservative-free apple cider and simulated gastric fluid. Int J Food Microbiol, 1999, 46(3): 263–269. DOI: 10.1016/S0168-1605(98)00198-6 |
| [15] | Tamplin ML. Inactivation of Escherichia coli O157:H7 in Simulated human gastric fluid. Appl Environ Microbiol, 2005, 71(1): 320–325. DOI: 10.1128/AEM.71.1.320-325.2005 |
| [16] | Bergholz TM, Whittam TS. Variation in acid resistance among enterohaemorrhagic Escherichia coli in a simulated gastric environment. J Appl Microbiol, 2007, 102(2): 352–362. |
| [17] | Yuk HG, Marshall DL. Adaptation of Escherichia coli O157:H7 to pH alters membrane lipid composition, verotoxin secretion, and resistance to simulated gastric fluid acid. Appl Environ Microbiol, 2004, 70(6): 3500–3505. DOI: 10.1128/AEM.70.6.3500-3505.2004 |
| [18] | Mao Y, Doyle MP, Chen J. Role of colanic acid exopolysaccharide in the survival of enterohaemorrhagic Escherichia coli O157:H7 in simulated gastrointestinal fluids. Lett Appl Microbiol, 2006, 42(6): 642–647. |
| [19] | Yuk HG, Jo SC, Seo HK, et al. Effect of storage in juice with or without pulp and/or calcium lactate on the subsequent survival of Escherichia coli O157:H7 in simulated gastric fluid. Int J Food Microbiol, 2008, 123(3): 198–203. DOI: 10.1016/j.ijfoodmicro.2008.01.013 |
| [20] | Jiang LL, Olesen I, Andersen T, et al. Survival of Listeria monocytogenes in simulated gastrointestinal system and transcriptional profiling of stress- and adhesion-related genes. Foodborne Pathog Dis, 2010, 7(3): 267–274. DOI: 10.1089/fpd.2009.0361 |
| [21] |
Chen Z, Jia H. The advances of digestion simulation in vitro.
Farm Prod Pro, 2017(9): 61–64, 68.
(in Chinese). 陈责, 贾慧. 体外消化模型的研究进展. 农产品加工, 2017(9): 61-64, 68. |
| [22] | Minekus M, Havenaar R. In vitro model of an in vivo digestive tract: US, US5525305. 1996-06-11. |
| [23] | Molly K, Vande Woestyne M, Verstraete W. Development of a 5-step multi-chamber reactor as a simulation of the human intestinal microbial ecosystem. Appl Microbiol Biotechnol, 1993, 39(2): 254–258. DOI: 10.1007/BF00228615 |
| [24] | Barroso E, Cueva C, Peláez C, et al. Development of human colonic microbiota in the computer-controlled dynamic SIMulator of the GastroIntestinal tract SIMGI. LWT-Food Sci Technol, 2015, 61(2): 283–289. DOI: 10.1016/j.lwt.2014.12.014 |
| [25] | Verwei M, Minekus M, Zeijdner E, et al. Evaluation of two dynamic in vitro models simulating fasted and fed state conditions in the upper gastrointestinal tract (TIM-1 and tiny-TIM) for investigating the bioaccessibility of pharmaceutical compounds from oral dosage forms. Int J Pharm, 2016, 498(1/2): 178–186. |
| [26] | Rehman A, Heinsen FA, Venema K, et al. Effects of probiotics and antibiotics on the intestinal homeostasis in a computer controlled model of the large intestine. BMC Microbiol, 2012, 12: 47. DOI: 10.1186/1471-2180-12-47 |
| [27] | Miszczycha SD, Thévenot J, Denis S, et al. Survival of Escherichia coli O26:H11 exceeds that of Escherichia coli O157:H7 as assessed by simulated human digestion of contaminated raw milk cheeses. Int J Food Microbiol, 2014, 172: 40–48. DOI: 10.1016/j.ijfoodmicro.2013.11.029 |
| [28] |
Fu HW, He SP, Shi XD, et al. Construction and detection of metastases in animal models.
Chin J Gen Pract, 2017, 15(1): 142–146.
(in Chinese). 付后卫, 何顺朋, 施祥德, 等. 转移瘤动物模型的构建及其检测方法. 中华全科医学, 2017, 15(1): 142-146. |
| [29] | Clause BT. The wistar rat as a right choice:establishing mammalian standards and the ideal of a standardized mammal. J Hist Biol, 1993, 26(2): 329–349. DOI: 10.1007/BF01061973 |
| [30] | Whitaker WB, Parent MA, Boyd A, et al. The Vibrio parahaemolyticus ToxRS regulator is required for stress tolerance and colonization in a novel orogastric streptomycin-induced adult murine model. Infect Immun, 2012, 80(5): 1834–1845. DOI: 10.1128/IAI.06284-11 |
| [31] | Livny J, Zhou XH, Mandlik A, et al. Comparative RNA-Seq based dissection of the regulatory networks and environmental stimuli underlying Vibrio parahaemolyticus gene expression during infection. Nucleic Acids Res, 2014, 42(19): 12212–12223. DOI: 10.1093/nar/gku891 |
| [32] | Ritchie JM, Thorpe CM, Rogers AB, et al. Critical roles for stx2, eae and tir in enterohemorrhagic Escherichia coli-induced diarrhea and intestinal inflammation in infant rabbits. Infect Immun, 2003, 71(12): 7129–7139. DOI: 10.1128/IAI.71.12.7129-7139.2003 |
| [33] | Aviv G, Rahav G, Gal-Mor O. Horizontal transfer of the Salmonella enterica serovar infantis resistance and virulence plasmid pESI to the gut microbiota of warm-blooded Hosts. MBio, 2016, 7(5): e01395–16. |
| [34] | Liu BW, Teng ZH, Wang JF, et al. Inhibition of listeriolysin O oligomerization by lutein prevents Listeria monocytogenes infection. Fitoterapia, 2017, 116: 45–50. DOI: 10.1016/j.fitote.2016.11.012 |
| [35] | Gavin HE, Beubier NT, Satchell KJ. The effector domain region of the Vibrio vulnificus MARTX toxin confers biphasic epithelial barrier disruption and is essential for systemic spread from the intestine. PLoS Pathog, 2017, 13(1): e1006119. DOI: 10.1371/journal.ppat.1006119 |
| [36] | Hugenholtz F, de Vos WM. Mouse models for human intestinal microbiota research:a critical evaluation. Cell Mol Life Sci, 2018, 75(1): 149–160. DOI: 10.1007/s00018-017-2693-8 |
| [37] |
Che CY. Establishment and intestinal mucosal immune study of human flora-associated piglet[D]. Nanjing: Nanjing Agricultural University, 2009 (in Chinese). 车传燕. 人源菌群仔猪模型的建立及其胃肠道黏膜免疫的研究[D]. 南京: 南京农业大学, 2009. |
| [38] | Barman M, Unold D, Shifley K, et al. Enteric salmonellosis disrupts the microbial ecology of the murine gastrointestinal tract. Infect Immun, 2008, 76(3): 907–915. DOI: 10.1128/IAI.01432-07 |
| [39] | Faure S, Perrin-Guyomard A, Delmas JM, et al. Transfer of plasmid-mediated CTX-M-9 from Salmonella enterica serotype Virchow to Enterobacteriaceae in human flora-associated rats treated with cefixime. Antimicrob Agents Chemother, 2010, 54(1): 164–169. DOI: 10.1128/AAC.00310-09 |
| [40] | Ali MM, Newsom DL, González JF, et al. Fructose-asparagine is a primary nutrient during growth of Salmonella in the inflamed intestine. PLoS Pathog, 2014, 10(6): e1004209. DOI: 10.1371/journal.ppat.1004209 |
| [41] | Bian X, Wang TT, Xu M, et al. Effect of Lactobacillus strains on intestinal microflora and mucosa immunity in Escherichia coli O157:H7-induced diarrhea in mice. Curr Microbiol, 2016, 73(1): 65–70. DOI: 10.1007/s00284-016-1010-3 |
| [42] | Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome. Nature, 2012, 486(7402): 207–214. DOI: 10.1038/nature11234 |
| [43] | Brehm MA, Jouvet N, Greiner DL, et al. Humanized mice for the study of infectious diseases. Curr Opin Immunol, 2013, 25(4): 428–435. DOI: 10.1016/j.coi.2013.05.012 |
| [44] | Mosier DE, Gulizia RJ, Baird SM, et al. Transfer of a functional human immune system to mice with severe combined immunodeficiency. Nature, 1988, 335(6187): 256–259. DOI: 10.1038/335256a0 |
| [45] | Lapidot T, Pflumio F, Doedens M, et al. Cytokine stimulation of multilineage hematopoiesis from immature human cells engrafted in SCID mice. Science, 1992, 255(5048): 1137–1141. DOI: 10.1126/science.1372131 |
| [46] | Shultz LD, Ishikawa F, Greiner DL. Humanized mice in translational biomedical research. Nat Rev Immunol, 2007, 7(2): 118–130. DOI: 10.1038/nri2017 |
| [47] | Medzhitov R, Preston-Hurlburt P, Janeway Jr JC. A human homologue of the Drosophila Toll protein signals activation of adaptive immunity. Nature, 1997, 388(6640): 394–397. DOI: 10.1038/41131 |
| [48] | Lianou A, Koutsoumanis KP. Strain variability of the behavior of foodborne bacterial pathogens:a review. Int J Food Microbiol, 2013, 167(3): 310–321. DOI: 10.1016/j.ijfoodmicro.2013.09.016 |
| [49] | Liu BX, Liu HQ, Pan YJ, et al. Comparison of the effects of environmental parameters on the growth variability of Vibrio parahaemolyticus coupled with strain sources and genotypes analyses. Front Microbiol, 2016, 7: 994. |
| [50] |
Tang XY, Guo XB, Guo DF, et al. High throughput and rapid determination of the maximum specific growth rate of pathogenic and non-pathogenic Vibrio parahaemolyticus.
Microbiol China, 2013, 40(11): 2138–2144.
(in Chinese). 唐晓阳, 郭晓滨, 郭丹凤, 等. 高通量快速测定致病性及非致病性副溶血性弧菌最大比生长速率. 微生物学通报, 2013, 40(11): 2138-2144. |
| [51] |
Tang XY, Han T, Xie J, et al. Comparison of growth parameters of different pathogenic Vibrio parahaemolyticus stains on Litopenaeus vannamei.
Sci Technol Food Ind, 2013, 34(2): 78–82.
(in Chinese). 唐晓阳, 韩婷, 谢晶, 等. 不同致病性副溶血性弧菌在南美白对虾中的生长动力学参数比较研究. 食品工业科技, 2013, 34(2): 78-82. |
| [52] | Song XY, Ma YJ, Fu JJ, et al. Effect of temperature on pathogenic and non-pathogenic Vibrio parahaemolyticus biofilm formation. Food Control, 2016, 73: 485–491. |
| [53] | Lou Y, Liu HQ, Zhang ZH, et al. Mismatch between antimicrobial resistance phenotype and genotype of pathogenic Vibrio parahaemolyticus isolated from seafood. Food Control, 2016, 59: 207–211. DOI: 10.1016/j.foodcont.2015.04.039 |
| [54] |
Tang XY, Qiu HL, Ba Q, et al. A review on the microbial risk assessment in foods.
Chin Bull Life Sci, 2015, 27(3): 383–388.
(in Chinese). 唐晓阳, 邱红玲, 巴乾, 等. 食品微生物风险评估概述. 生命科学, 2015, 27(3): 383-388. |
| [55] |
Zhang ZH, Lou Y, Du SP, et al. Application and prospects of molecular biology techniques in predictive microbiology:a review.
Food Sci, 2017, 38(9): 248–257.
(in Chinese). 张昭寰, 娄阳, 杜苏萍, 等. 分子生物学技术在预测微生物学中的应用与展望. 食品科学, 2017, 38(9): 248-257. DOI:10.7506/spkx1002-6630-201709040 |
| [56] |
Dong QL, Wang HM, Malakar PK, et al. Review of progress in quantitative microbiological risk assessment in China.
Food Sci, 2015, 36(11): 221–229.
(in Chinese). 董庆利, 王海梅, MalakarPK, 等. 我国食品微生物定量风险评估的研究进展. 食品科学, 2015, 36(11): 221-229. DOI:10.7506/spkx1002-6630-201511042 |
 2018, Vol. 34
2018, Vol. 34