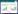中国科学院微生物研究所、中国微生物学会主办
文章信息
- 何展, 王航, 韩笑, 马天天, 杭奕, 俞慧飞, 卫芳芳, 孙静, 杨永春, 程昌勇, 宋厚辉
- He Zhan, Wang Hang, Han Xiao, Ma Tiantian, Hang Yi, Yu Huifei, Wei Fangfang, Sun Jing, Yang Yongchun, Cheng Changyong, Song Houhui
- 单核细胞增多性李斯特菌氨基肽酶Lmo1711体外表达及其酶活分析
- Characterization of a recombinant aminopeptidase Lmo1711 from Listeria monocytogenes
- 生物工程学报, 2018, 34(5): 685-693
- Chinese Journal of Biotechnology, 2018, 34(5): 685-693
- 10.13345/j.cjb.170468
-
文章历史
- Received: November 28, 2017
- Accepted: January 22, 2018
单核细胞增多性李斯特菌(简称单增李斯特菌)是一种人畜共患的食源性胞内寄生菌。在欧美西方国家暴发单增李斯特菌病的概率很高,并且每次都引起高死亡率[1]。单增李斯特菌病在免疫力低下的人群、孕妇及胎儿中多发,主要症状表现为败血症、脑膜炎、单核细胞增多和胎儿流产等[2-3]。单增李斯特菌的感染过程都需要毒力因子的参与,其中关键步骤包括:宿主细胞粘附和侵袭,细胞内增殖和运动以及细胞间传播[4]。在单增李斯特菌中PrfA是毒力因子,可维持细菌胞内环境的稳态;内化素(InlA和InlBv)介导细菌对宿主细胞的黏附、内化;溶血素(LLO)和磷脂酰肌醇磷脂酶(PlcA)裂解吞噬体膜;磷脂酶(PlcA和PlcB)参与细菌的逃逸过程[5-7]。
氨基肽酶作为一种水解酶,可能参与单增李斯特菌毒力因子修饰,进而与细菌感染过程存在某种关联。氨基肽酶是针对寡肽、多肽和蛋白质释放N-末端氨基残基的酶,不仅能够完全降解蛋白质和多肽,使得组分氨基酸被重新利用;而且用于蛋白质翻译后加工,使得蛋白质在到达正确的空间位置或处于必需的生理发育环境前无法被激活[8]。多数氨基肽酶属于金属蛋白酶,其基因中存在H-E-X-X-H (His-Glu-X-X-His)活性位点序列对于催化和配位金属离子是必需的。金属蛋白酶活性由一个或两个二价离子(通常是锌)介导,这些离子激活水分子以进行底物肽键的亲核攻击,进而起到降解、加工、抑制等作用[9]。金属蛋白酶对机体也具有多样性的功能,例如基质金属蛋白酶(MMPs)是参与细胞外基质降解的酶,其生物学功能不仅仅是蛋白水解功能,还包括诱导复合信号传导以及诱导独立于蛋白水解活性的癌细胞迁移等[10-11]。
单增李斯特菌中lmo1603和lmo1711被注释为编码氨基肽酶的基因。此前本实验室已证实Lmo1603具有氨基肽酶的性质,但Lmo1711是否具有氨基肽酶活性尚未知晓。本研究表达并纯化得到了高纯度Lmo1711蛋白,以氨基酸-对硝基苯胺作为底物鉴别出Lmo1711具有氨基肽酶的活性和对于底物的选择性;而加入不同金属离子产生的酶活性差异表明这些金属离子对Lmo1711活性增强效果不同。以上结果证实了Lmo1711属于氨基肽酶M29家族成员,具有高度的金属离子依赖性,为进一步探讨lmo1711在单增李斯特菌体内发挥的作用和功能奠定了基础。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒以及引物本试验中所用到的菌株及质粒包括:单增李斯特菌参考菌株EGD-e、大肠杆菌DH5α和Rosetta、重组蛋白原核表达载体pET30a(+),以上菌株和质粒均为本试验室保存。单增李斯特菌培养于BHI (Brain heart infusion,BHI)培养基,DH5α和Rosetta菌株培养于LB培养基中。本试验所涉及菌株的培养条件均为37 ℃ (静置或振荡)培养,试验涉及引物为(下划线表示酶切位点):lmo1711-fwd: 5′-CGG GGTACCATGACAGTATTTAGTGAAAAGT TAGAAAAGTATGC-3′;lmo1711-rev: 5′-CGCGGATCC TTAGAACGCCCA GTCGCCTTTA-3′。
1.1.2 试剂BHI培养基购自Oxoid公司;LB培养基购自上海生工生物工程有限公司;KOD plus Neo PCR试剂盒购自Toyobo公司;PCR产物纯化和回收试剂盒购自上海莱枫生物科技有限公司;限制性核酸内切酶购自NEB公司;质粒提取试剂盒购自天根生化科技有限公司;BCA蛋白浓度测定试剂盒购自碧云天公司;镍柱亲和层析柱购自北京韦氏博慧色谱科技有限公司;氨基酸-对硝基苯胺均购自Sigma-Aldrich公司;本试验所用化学试剂均为国产分析纯。
1.2 方法 1.2.1 Lmo1711蛋白生物信息学分析根据Uniprot网站预测,单增李斯特菌Lmo1711全长410 aa,分子量45 kDa,无信号肽,属于M29.002家族。而MEROPS网站分析,M29家族氨基肽酶典型特征是钴离子依赖性,并且保守位点是Glu-Glu-His-Tyr-His-Asp。利用CLC Sequence Viewer 6软件将单增李斯特菌的Lmo1711氨基酸序列同其他细菌氨基肽酶序列进行比较,分析关键氨基酸位点或区域,同时构建系统进化树;在Swiss Model Workspace中以肺炎链球菌Streptococcus pneumoniae PepS为模板,对Lmo1711的空间结构进行模拟比对。
1.2.2 Lmo1711重组原核蛋白表达质粒的构建NCBI数据库下载lmo1711基因序列(GenBank登录号NC_003210.1),导入Vector NTI软件设计出针对lmo1711完整ORF引物,命名为lmo1711-fwd和lmo1711-rev。lmo1711与本实验室数据库中的pET30a模拟构建重组质粒。通过PCR对目的基因扩增,然后对载体和目的片段用Kpn Ⅰ和BamH Ⅰ进行双酶切反应,酶切产物以T4 DNA连接酶过夜连接,最后将酶连产物热击转化至大肠杆菌DH5α感受态细胞。通过PCR筛选得到携带重组质粒(根据实验室命名规则,命名为pSL350)的重组菌株,经测序验证正确后用于下一步的蛋白表达。
1.2.3 Lmo1711重组蛋白的原核表达与纯化将测序正确的重组表达质粒pSL350通过热击法转入大肠杆菌Rosetta感受态细胞。取5 mL Rosetta-pSL350菌液转接至500 mL LB (Kana),待OD600为0.6-0.8时,加入IPTG (Isopropyl β-D-1-thiogalactopyranoside,异丙基-β-D-硫代半乳糖苷,工作浓度1 mmoL/L)低温诱导12 h。离心收集菌体,50 mmol/L PBS洗涤菌体2次,置于冰上超声破碎后离心取上清与2.5 mL镍柱4 ℃翻转结合6 h。用30-300 mmol/L咪唑洗脱杂蛋白并收集目标蛋白。收集到的目的蛋白进行SDS-PAGE分析验证蛋白纯度与大小。目的蛋白浓度用BCA试剂盒进行测定。
1.2.4 Lmo1711氨基肽酶活性分析试剂准备:Lmo1711蛋白;25 mmol/L Tris-HCl (pH 7.4);100 mmol/L氨基酸对硝基苯胺aa-pNA,本试验中选用3种底物,分别是Arg-pNA、Leu-pNA、Lys-pNA。将各反应成分混合后以200 μL体积(Lmo1711工作浓度0.1 μmol/L,aa-pNA工作浓度0.1-10 mmol/L)加入96孔板,振荡混匀后,立即放入酶标仪进行检测,游离的对硝基苯胺吸收峰值为405 nm。反应温度37 ℃,间隔时间1 h,连续读数12 h。
1.2.5 Lmo1711氨基肽酶活性的金属离子依赖性分析为了验证Lmo1711是否具有金属依赖性,选取Co2+、Zn2+、Mn2+、Cd2+等氨基肽酶常见辅助金属离子,以相同工作浓度(0.5 mmol/L)加入反应体系,底物为Leu-pNA,测定产物浓度。反应温度37 ℃,间隔时间5 min,连续读数60 min。
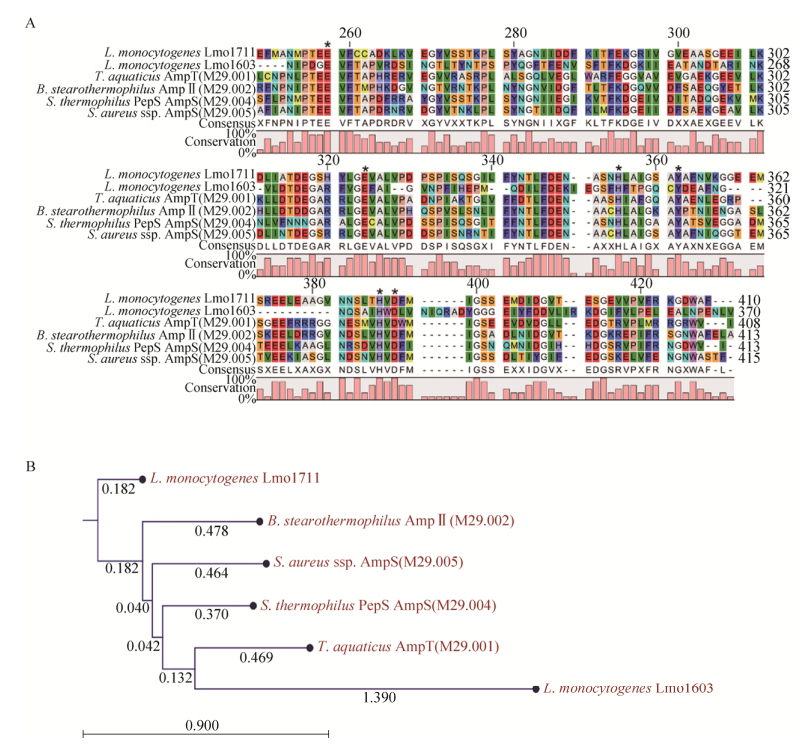
2 结果 2.1 单增李斯特菌Lmo1711属于氨基肽酶M29家族单增李斯特菌M29家族的代表性成员有:氨基肽酶T(AmpT)、氨基肽酶Ⅱ(AmpⅡ)、PepS氨基肽酶、氨基肽酶S(AmpS),它们分别属于001、002、004和005亚族,其中Lmo1603属于AmpT。通过氨基酸序列比对,推测Lmo1711保守活性位点是Glu250、Glu316、His345、Tyr352、His378、Asp380 (图 1A)。进化树分析发现,Lmo1711与之前报道的AmpT Lmo1603亲缘关系较远,而与来自嗜热菌中的氨基肽酶Ⅱ(AmpⅡ M29.002)进化关系最近,与UniProt网站预测结果一致(图 1B)。以PepS (PDB:4ICQ)为模板利用SWISS-MODEL Workspace对Lmo1711蛋白三级结构进行模拟,发现两者的空间折叠结构高度相似,即Lmo1711的催化结构域亦是二聚体的非对称结构。此外,Lmo1711与同属于M29家族的AmpS (PDB:1ZJC)、AmpT (PDB:2AYI)、AmpⅡ等结构非常类似,所有保守活动区域均处于该结构域中(图 2)。根据以上生物信息学分析,推测单增李斯特菌lmo1711基因编码的蛋白质与M29家族其他的氨基肽酶可能存在相似的性质。
|
| 图 1 M29家族部分氨基肽酶氨基酸序列比对(“*”为推测的活性位点) (A)和进化关系分析(B) Figure 1 Amino acid sequence alignment (A) and phylogenetic tree (B) of Lmo1711 and the members of the M29 aminopeptidase family. The predicted active sites are denoted with "*". |
| |
|
| 图 2 以M29家族多种氨基肽酶晶体结构预测模拟Lmo1711空间结构 Figure 2 Predicted protein structure of Lmo1711 based on the templates with known crystal structures from the members of the M29 aminopeptidase. |
| |
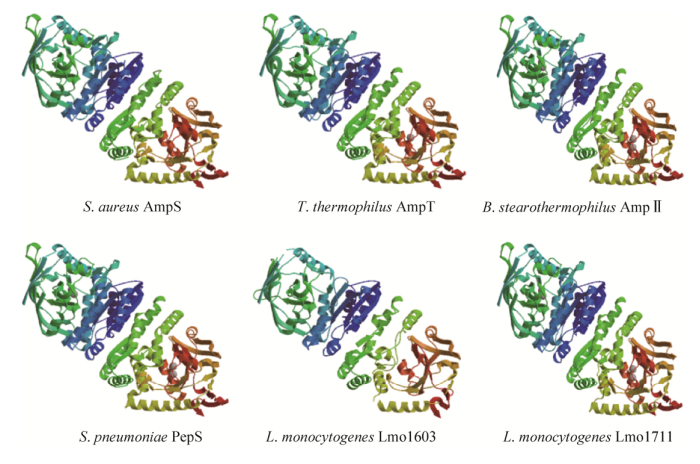
利用PCR扩增出EGD-e lmo1711目的片段,结果显示在1 347 bp处出现了明显条带(图 3A),与理论大小一致。PCR产物与蛋白表达载体pET30a(+)重组。转化后挑取若干单菌落接种于LB Kana培养基。以EGD-e为阳性对照,利用菌液PCR验证得到符合目的条带大小的阳性质粒,目的条带大小仍在1 347 bp (图 3B)。将测序正确的重组质粒转入E. coli Rosetta,得到Lmo1711蛋白表达菌株。
|
| 图 3 lmo1711的全基因序列(A)、阳性克隆验证电泳图(B)及表达产物SDS-PAGE分析(C) Figure 3 Verification of the recombinant Lmo1711 expression strains using PCR (A-B) and protein purification using SDS-PAGE analysis (C). |
| |
通过UniProt网站以及Vector NTI软件分析预测Lmo1711蛋白大小在44.96 kDa,与His-tag融合后的大小在49.3 kDa。采用大肠杆菌表达系统诱导蛋白表达,在蛋白纯化后用SDS-PAGE分析,显示纯化蛋白有明显的单一条带,大小与预测值一致(图 3C)。以上结果证实已成功获得重组Lmo1711蛋白,可用于体外酶活试验。
2.4 Lmo1711具有氨基肽酶的活性Lmo1711能够水解不同种类的氨基酸-对硝基苯胺,而且对不同的底物水解强度也存在差异。Lmo1711水解Leu-pNA的能力要强于水解Arg-pNA和Lys-pNA (图 4A,4B)。证实Lmo1711作为氨基肽酶对底物具有选择性,且优先水解Leu-pNA。
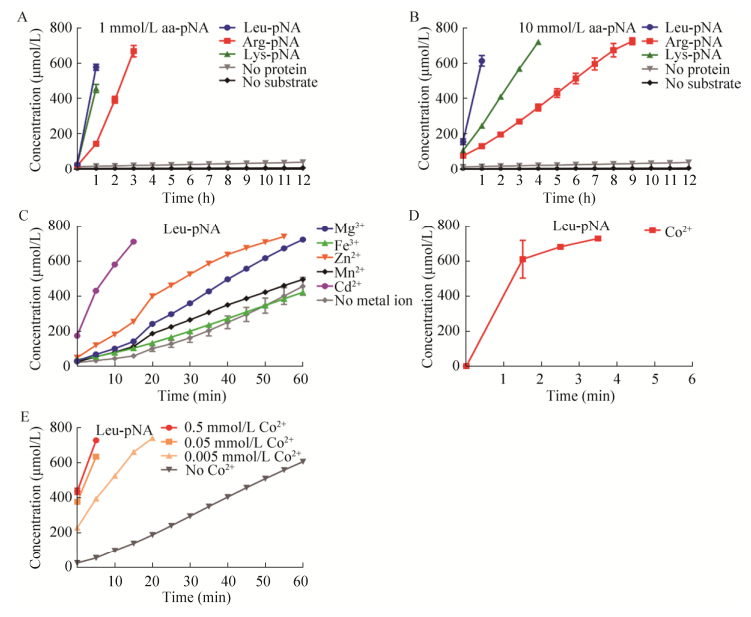
|
| 图 4 Lmo1711对不同底物的催化能力及金属离子对该酶活性的影响 Figure 4 Enzymatic characteristics of the recombinant Lmo1711. (A-B) Kinetic aminopeptidase activity of Lmo1711 using 1 mmol/L or 10 mmol/L Leu-pNA, Arg-Pna or Lys-pNA as the substrate. (C-E) Effects of metal ions (Mg2+, Fe3+, Zn2+, Mn2+ and Co2+) on enzymatic activity of Lmo1711. |
| |
本实验发现,多种金属离子均能够增强Lmo1711的水解能力,且对酶活的增强存在较大的差异。加入Co2+后Lmo1711的活性极显著增强,约4 min后就超出酶标仪测量量程(图 4D);加入其他金属离子Lmo1711的活性也得到不同程度的提升(图 4C)。Co2+在0.5 mmol/L基础上稀释100倍后加入反应体系,仍可以显著增强Lmo1711的水解能力(图 4E)。由此证实Lmo1711属于金属依赖性氨基肽酶,其中Co2+催化Lmo1711氨基肽酶的水解能力最强。
3 讨论本研究证实Lmo1711在体外所具有的氨基肽酶活性和金属依赖性,而M29家族中众多嗜热菌来源的氨基肽酶也具有类似的性质。例如:氨基肽酶T具有广泛的底物特异性,但会优先水解释放Leu、Val、Phe或Tyr,受到EDTA或1, 10-邻菲咯啉抑制剂抑制时,通过添加外源Co2+可恢复其活性。嗜热脂肪芽孢杆菌氨基肽酶Ⅱ同样显示出对亮氨-酸对硝基苯胺(Leu-pNA)的显著偏好[12-13],而PepS氨基肽酶(M29.004)[14]与氨基肽酶S(M29.005)同样也与Lmo1711性质相似[15]。迄今为止,除了细菌外,还在古生菌(Archaeobacteria)和植物中鉴定出编码M29氨基肽酶的基因,然而只有来自水生栖热菌、嗜热栖热菌、嗜热脂肪芽孢杆菌和嗜热链球菌的氨基肽酶才被认为是该家族真正的成员[16]。基于进化树分析,Lmo1711与氨基肽酶S、氨基肽酶Ⅱ、氨基肽酶T和PepS氨基肽酶同属M29家族,其中与嗜热菌中的氨基肽酶Ⅱ (AmpⅡ M29.002)进化关系最为密切,推测Lmo1711属于M29.002家族。氨基肽酶通常具有一些保守氨基酸位点,在结合和催化底物过程中起关键作用,如金黄色葡萄球菌AmpS的活性位点会通过溶剂分子(可能是氢氧根离子)和Glu319桥连后先结合两个金属离子,再借助Glu253和His348的协助结合第3个金属离子,进而暴露酶的活性中心[15]。根据氨基酸序列分析,Lmo1711具有6个保守氨基酸位点,分别为Glu250、Glu316、His345、Tyr352、His378、Asp380,这些位点可能决定了Lmo1711的酶活特性。
分泌型的细菌氨基肽酶都是单体,而胞内酶则是单体或多聚体[17]。除了金属蛋白酶TAPBb外,目前尚未发现M29家族的其他六聚体氨基肽酶,如水生栖热菌和嗜热脂肪芽孢杆菌的氨基肽酶T是二聚体,嗜热链球菌的PepS是单体[16]。根据Biozentrum网站预测,Lmo1711为同源二聚体,三级结构为细长状,其N端结构域包括至少7个α-螺旋片段围绕中心结构,同时存在平行β-折叠片段,C端有2个β-折叠片段。TAPBb是致病微生物中最早发现的M29家族氨基肽酶,其六聚体结构是发挥该酶活性的基础[18]。另外,二硫键不参与TAPBb的低聚组装,因为其单体化不依赖于还原剂的存在,这与M29家族的部分成员以及其他氨基肽酶(例如牛的亮氨酰氨基肽酶晶状体和氨基肽酶A)的结构一致[18]。
本研究首次研究并证实单增李斯特菌Lmo1711能够特异性水解N-末端具有芳香族化合物的多肽,且对Leu-pNA、Arg-pNA、Lys-pNA等底物具有不同亲和力。在金属离子催化下Lmo1711活性得到显著增强,尤其以Co2+的激活效果最明显。Lmo1711的6个保守位点对其催化或底物结合是否起到关键作用以及该蛋白在细菌体内是否参与修饰毒力因子或相关蛋白的合成、加工及成熟尚未得知,因此我们下一步将针对Lmo1711在李斯特菌感染宿主过程中发挥的生物学功能及分子机制开展深入研究。
| [1] | Voetsch AC, Angulo FJ, Jones TF, et al. Reduction in the incidence of invasive listeriosis in foodborne diseases active surveillance network sites, 1996-2003. Clin Infect Dis, 2007, 44(4): 513–520. DOI: 10.1086/511006 |
| [2] | Pagliano P, Arslan f, Ascione T. Epidemiology and treatment of the commol/Lonest form of listeriosis: meningitis and bacteraemia. Infez Med, 2017, 25(3): 210–216. |
| [3] | Jackson KA, Iwamoto M, Swerdlow D. Pregnancy-associated listeriosis. Epidemiol Infect, 2010, 138(10): 1503–1509. DOI: 10.1017/S0950268810000294 |
| [4] | David DJV, Cossart P. Recent advances in understanding Listeria monocytogenes infection: the importance of subcellular and physiological context. F1000Res, 2017, 6(F1000 Faculty Rev): 1126. |
| [5] | Cossart P, Toledo-Arana A. Listeria monocytogenes, a unique model in infection biology: an overview. Microbes Infect, 2008, 10(9): 1041–1050. DOI: 10.1016/j.micinf.2008.07.043 |
| [6] | Pamer EG. Immol/Lune responses to Listeria monocytogenes. Nat Rev Immol, 2004, 4(10): 812–823. DOI: 10.1038/nri1461 |
| [7] | Lin PL, Cheng YM, Wu DW, et al. A combination of anti-PD-L1 mAb plus Lm-LLO-E6 vaccine efficiently suppresses tumor growth and metastasis in HPV-infected cancers. Cancer Med, 2017, 6(9): 2052–2062. DOI: 10.1002/cam4.2017.6.issue-9 |
| [8] | |
| [9] | Hecht KA, Wytiaz VA, Ast T, et al. Characterization of an M28 metalloprotease family member residing in the yeast vacuole. FEMS Yeast Res, 2013, 13(5): 471–484. DOI: 10.1111/femsyr.2013.13.issue-5 |
| [10] | Alford VM, Kamath A, Ren XD, et al. Targeting the hemopexin-like domain of latent matrix metalloproteinase-9 (proMMOL/LP-9) with a small molecule inhibitor prevents the formation of focal adhesion junctions. ACS Chem Biol, 2017, 12(11): 2788–2803. DOI: 10.1021/acschembio.7b00758 |
| [11] | Bedrosian LD, Ferguson KK, Cantonwine DE, et al. Urinary phthalate metabolite concentrations in relation to levels of circulating matrix metalloproteinases in pregnant women. Sci Total Environ, 2017, 613-614: 1349–1352. |
| [12] | Myrin PÂ, Hofsten BV. Purification and metal ion activation of an aminopeptidase (aminopeptidase Ⅱ) from Bacillus stearothermophilus. Biochim Biophys Acta-Enzymol, 1974, 350(1): 13–25. DOI: 10.1016/0005-2744(74)90198-3 |
| [13] | Wang TF, Lin MG, Lo HF, et al. Biophysical characterization of a recombinant aminopeptidase Ⅱ from the thermophilic bacterium Bacillus stearothermophilus. J Biol Phys, 2014, 40(1): 25–40. DOI: 10.1007/s10867-013-9332-x |
| [14] | Ta HM, Bae S, Han S, et al. Structure-based elucidation of the regulatory mechanism for aminopeptidase activity. Acta Crystallogr D, 2013, 69: 1738–1747. DOI: 10.1107/S0907444913012651 |
| [15] | Odintsov SG, Sabała I, Bourenkov G, et al. Staphylococcus aureus aminopeptidase S is a founding member of a new peptidase clan. J Biol Chem, 2005, 280(30): 27792–27799. DOI: 10.1074/jbc.M502023200 |
| [16] | Fernandez-Espla MD, Rul F. PepS from Streptococcus thermophilus. A new member of the aminopeptidase T family of thermophilic bacteria. Eur J Biochem, 1999, 263(2): 502–510. DOI: 10.1046/j.1432-1327.1999.00528.x |
| [17] | Gonzales T, Robert-Baudouy J. Bacterial aminopeptidases: properties and functions. FEMS Microbiol Rev, 1996, 18(4): 319–344. DOI: 10.1111/fmr.1996.18.issue-4 |
| [18] | Bertin PB, Lozzi SP, Howell JK, et al. The thermophilic, homohexameric aminopeptidase of Borrelia burgdorferi is a member of the M29 family of metallopeptidases. Infect Immol, 2005, 73(4): 2253–2261. DOI: 10.1128/IAI.73.4.2253-2261.2005 |
 2018, Vol. 34
2018, Vol. 34