中国科学院微生物研究所、中国微生物学会主办
文章信息
- 蔚德睿, 马洁, 解增言, 白明泽, 朱云平, 舒坤贤
- Derui Yu, Jie Ma, Zengyan Xie, Mingze Bai, Yunping Zhu, Kunxian Shu
- 基于谱图库的蛋白质鉴定策略研究进展
- Progress in the spectral library based protein identification strategy
- 生物工程学报, 2018, 34(4): 525-536
- Chinese Journal of Biotechnology, 2018, 34(4): 525-536
- 10.13345/j.cjb.170321
-
文章历史
- Received: August 14, 2017
- Accepted: September 29, 2017
2 军事医学科学院放射与辐射医学研究所 蛋白质药物国家工程研究中心 北京蛋白质组研究中心 蛋白质组学国家重点实验室 国家蛋白质科学中心 (北京),北京 102206
2 National Center for Protein Sciences, State Key Laboratory of Proteomics, Beijing Proteome Research Center, Engineering Research Center for Protein Drugs, Institute of Radiation Medicine, Beijing 102206, China
蛋白质组学旨在鉴定出一个细胞、组织、器官或个体全部的蛋白质信息[1],而如何快速、准确地鉴定出样本中的蛋白质信息又是其最重要的研究内容。随着质谱技术的发展,鸟枪法目前已成为最主要的蛋白质鉴定方法[2-3]。该方法首先把蛋白酶解为短的肽片段,再用质谱仪对这些短的肽片段进行裂解和分析,最后用一系列信息学方法鉴定出这些图谱文件对应的肽段离子及其含量。鸟枪法蛋白质鉴定的信息学方法主要分为三大类:第一类为序列数据库搜索,该策略根据样本中可能存在的蛋白质序列以一定的酶解和碎裂模式碎裂得到理论图谱,把待鉴定图谱与理论图谱比对,得到可能的肽段-谱图对(Peptide-spectrum matches,PSMs)。另一类是从头测序(de-novo sequencing),该策略不利用蛋白质序列数据库,直接根据二级谱图,利用图论和动态规划等算法推导得到可能的肽段序列。第三类是谱图库搜索,该策略基于已经被鉴定实验图谱构建谱图库,将待鉴定图谱与谱图库中的谱图比对得到可能的PSMs。
序列数据库搜索策略是目前应用最广泛的蛋白质鉴定方法,谱图库搜索策略原理与其类似,都是将待鉴定的实验图谱与参考图谱比对,得到鉴定结果。但不同之处在于,谱图库搜索策略中的参考谱图库基于实际谱图构建,与序列数据库搜索相比具有搜索速度快、鉴定精度高及鉴定率高的优点。首先,谱图库搜索策略只搜索谱图库中包含的谱图,不需要穷尽所有可能的碎裂模式,搜索空间更小,搜索速度也相应的更快。Lam等分别用SpectraST和SEQUEST搜索鉴定同一批数据,前者在1个CPU的机器上运行一天完成搜索,而后者则在80个CPU的集群上运行超过一周[4]。其次,序列数据库搜索策略在生成理论图谱时需要人为设定其碎裂模式,然而到目前为止,人们掌握的肽段的碎裂模式非常有限,因此遗漏了很多不常见的碎裂方式;同时,生成的理论图谱只考虑了离子的质荷比信息,而谱图库中的参考图谱来源于已经被鉴定的真实图谱,包含了具体的峰强度信息以及非常规碎裂模式等,增加了搜索的灵敏度和准确度,有效地提高了谱图的鉴定率。有研究表明谱图库搜索方法相比序列库搜索方法可以将谱图鉴定率提高25%-156%[5]。也有研究表明由于谱图库中的谱图包含更多的信息,使其相似度的计算更加精确[6]。因此在定量蛋白质组学的研究中,谱图库搜索策略可以替代序列数据库搜索策略,且在修饰和共碎裂肽的鉴定方面具有明显优势,可以看作是对序列数据库方法的补充[7-8]。但其只能识别谱图库中包含的肽和蛋白,因此不适用于新蛋白的鉴定。
本文将从以下几个方面介绍基于谱图库搜索的蛋白质鉴定策略:首先介绍基于谱图库搜索的质谱数据蛋白质鉴定流程;接着介绍了公开发表的主要谱图库构建和谱图库搜索工具并阐明了其特点;最后分析谱图库搜索策略存在的问题与挑战。
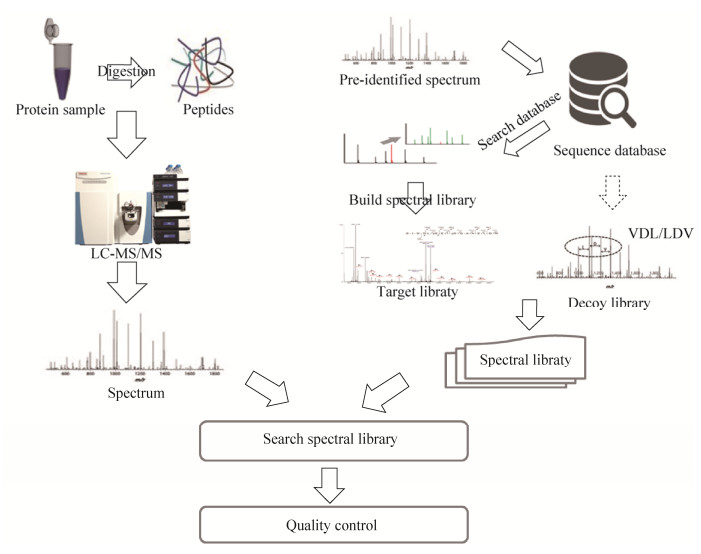
1 谱图库鉴定策略实现流程基于谱图库的蛋白质鉴定策略的通用流程如图 1所示,首先根据待鉴定的实验数据构建搜库需要的参考谱图库,谱图库可以从公共数据库中下载,也可以根据研究需求构建自定义的谱图库;然后选择合适谱图库的搜索工具进行搜库,最后对搜库结果进行质控[9],得到可靠鉴定结果。
|
| 图 1 蛋白质组质谱数据谱图库鉴定策略的实施流程 Figure 1 Workflow chart of the spectra library searching strategy for proteomics mass spectrometry data. |
| |
谱图库的获取有两种途径,从公共数据库中下载或构建自定义谱图库。
1.1.1 公共图谱资源数据库目前,谱图库在蛋白质组学领域的应用还比较有限,但其在挥发性化学小分子领域的应用十分广泛[10]。随着质谱技术的发展,谱图数据爆炸式增长,肽谱图数据库也发展了统一的国际标准[11-12],许多蛋白质组数据库根据数据库中收集的谱图数据构建了不同类型的谱图库,可供下载使用。目前可使用的主要公共谱图库见表 1。
| Library | Format | Link |
| NIST | MSP, splib, NIST binary | http://peptide.nist.gov |
| PeptideAtlas | sptxt, splib | http://www.peptideatlas.org//speclib/ |
| GPM | hlf, MGF | http://ftp://ftp.thegpm.org/projects/xhunter/libs/ |
| PRIDE | MSP | https://www.ebi.ac.uk/pride/cluster/#/libraries |
| B. Raught’s lab | sptxt, splib | http://www.raughtlab.ca/resources/msresources.php |
| Lee et al | sptxt, splib | http://ms-utils.org/zebrafish/ |
| Gunaratne et al | sptxt, splib | ftp://ftp.peptideatlas.org/pub/PeptideAtlas/Repository/PAe003810/ |
NIST (National Institute of Standards and Technology)是全球公认的串联质谱谱图库的黄金准则[10],共收录了380万张谱图,构建了9种不同物种的谱图库,每个物种的谱图库的大小差异很大。NIST在构建谱图库时使用了多种序列库搜索软件进行搜库,再综合其搜库结果以保证用于构库的PSMs准确可靠。
GPM (Global Proteome Machine)数据库[13]是第二大谱图数据库,也是物种覆盖最广泛的谱图数据库,包含了28个真核生物、115个原核生物和7个病毒,此外,还提供了常见污染蛋白谱图库(Common repository of adventitious proteins,cRAP)。GPM构建谱图库时利用X!Tandem搜索鉴定GPMDB数据库中的谱图数据,筛选出可信的PSM,再通过X!Hunter构建谱图库。因此,GPM提供的谱图库中的所有谱图均只保留了丰度强度最大的20个峰。
PeptideAtlas[14]数据库包含了17个不同的物种,有些物种还针对特定的组织或磷酸化肽段构建了特殊的谱图库。PeptideAtlas构建谱图库时整合了多种蛋白质组学分析流程,并通过使用SpectraST构建一致性谱图库为NIST提供数据支持。
2013年PRIDE[8-15]开始利用谱图聚类的方法构建谱图库,截至目前,PRIDE构建的谱图库包含了16个物种以及一个包含54 000谱图的污染物谱图库,虽然谱图数不及GPM的多,但是PRIDE没有限制谱图中离子峰的数量,提高了谱图搜索的灵敏度。且PRIDE构建的谱图库利用了部分未鉴定谱图的信息,在一定程度上校正了仅基于正确鉴定谱图构建谱图库的偏性。
除了上述4个谱图库外,还有一些实验室构建了特殊的谱图库。Raught等[16]使用SpectraST构建了Ubiquitin、NEDD8、SUMO-1、SUMO-2和SUMO-3蛋白的谱图库;Lee等[17]构建了成年斑马鱼器官和组织的谱图库;Gunaratne等[18]构建了磷酸肽谱图库;Hu等建立了人类和4种模式生物(酿酒酵母、黑腹果蝇、秀丽隐杆线虫和小鼠)中磷酸化肽谱图库[19]。
1.1.2 构建自定义谱图库如果公共可获得的谱图库不能满足研究需求,还可以构建自定义谱图库。构建自定义谱图库是根据已经被可靠鉴定的自产或者公共质谱数据构建参考谱图库。谱图库的构建一般分为3个步骤:一、原始谱图数据初步筛选;二、生成特征谱图;三、谱图库加工和质量控制。
原始谱图数据初筛指在构建谱图库之前需要对谱图数据进行初步筛选。原始质谱数据来源于公共数据库中收集的或自产的已被鉴定的质谱数据,这些质谱数据含有仪器、样本、操作人员等带来的实验误差,以及数据分析过程中引入的错误鉴定。低质量或未被正确鉴定的谱图会降低谱图库的精确度,从而增加谱图库搜索的错误率,因此在构建谱图库时,需要通过设置严格的置信阈值筛选出高可信的谱图,再根据信噪比去除低质量的谱图。
生成特征谱图是指用一张标志性谱图代表同一肽段离子对应的多张谱图。当某一个肽段离子对应多张谱图时,需要定义一张特征谱唯一对应该肽段离子。特征谱图可以通过寻找最优替代谱图和生成一致性谱图两种方式获得。寻找最优替代谱图是指从来源于同一肽段离子的一组谱图中挑选出最具代表性的谱图作为该肽段离子的特征谱图;生成一致性谱图是指从该组谱图中产生新的谱图作为该肽段离子的特征谱。
谱图库加工和质量控制。谱图库加工指在生成谱图库中加入实验元信息和谱图注释信息,使之成为完整的试验记录,可以根据其信息进行重复验证;然后根据注释离子数量、信噪比卡值去除部分低质量谱图以及特征谱中的部分背景峰,从而减小搜索空间,提高搜索速度,同时也增加了搜索的准确度[20-21]。
1.1.3 构建诱饵谱图库诱饵谱图库是根据目标谱图库生成的一种虚假的谱图库。目标-诱饵策略是应用最广泛的质控方法,通过数据整体置信水平来评估匹配的可信度。其原理是同时搜索目标和诱饵谱图库,假设目标数据库中错误匹配的数目与诱饵数据库中正确匹配的数目相等,从而估计数据集的假阳性率[22, 23],因此需要根据目标数据库构建合适的诱饵库。也有研究指出实际的错误发现率(False discovery rates,FDR)是不可知的,所有的计算FDR的方法都是建立在一定的假设的基础之上。因此,许多谱图库搜索工具改进了谱图相似性的计算方法,使得其分值可以很好地反映谱图匹配的可信度,而不需要加入诱饵谱图库计算FDR[24]。因此,构建诱饵谱图库不是谱图库搜索策略的必需步骤。
1.2 谱图库搜索谱图库搜索是指以待鉴定谱图搜索参考谱图库进行图谱鉴定。搜索过程中直接把实验图谱与谱图库中的参考谱图进行对齐比对,计算谱图之间的相似度与该匹配统计学可信度,并对这一匹配进行综合打分,得分高的高质量谱图被认为是最佳匹配。谱图库搜索一般分为3个步骤:首先对实验谱图和谱图库中的谱图进行预处理,去除噪音和区分度低的离子,降低可能干扰匹配打分的因素,并根据某一函数转化实际峰强度值以降低丰度值对打分的影响;然后根据实验谱图从谱图库中筛选出一组候选谱图,比较实验谱图与候选集中的每一张谱图,计算实验谱图与谱图库中的谱图之间的相似度;最后根据匹配的相似度计算每一对匹配的综合分值,作为判断鉴定结果是否正确的依据。
1.3 谱图库搜索结果质量控制与序列库鉴定策略相同,谱图库搜库结果并非完全准确,因此需要筛选搜索结果,保证输出的PSMs是可信的匹配。最常用的方法是根据肽段匹配数目计算FDR,当FDR在一定的阈值内,则认为PSM可信;也存在一些软件根据一定的模型对PSM重新评估,计算某一PSM可能是随机匹配的概率,随机匹配概率小于一定的分值则为可信的PSM。
2 谱图库构建算法和工具谱图库可以看作是已经可靠鉴定的谱图的索引,从而可以通过搜索谱图库找到与实验谱图匹配的参考谱图及其鉴定信息。在谱图库搜索策略中存在目标谱图库和诱饵谱图库两种谱图库,其构建方法很多,下面将介绍几种常见的谱图库构建的工具及其实现方法,见表 2。
| Software | Format | Link | |
| Target library | SpectraST | splib sptxt | Part of the TPP (http://sourceforge.net/projects/sashimi/) |
| Bibliospec | blib ms2 | https://skyline.gs.washington.edu/labkey/project/home/software/BiblioSpec/begin.view | |
| pMatch | plib | http://pfind.ict.ac.cn/pmatch/ | |
| Liberator | sptxt | http://javaprotlib.sourceforge.net/packages/tools/liber/index.html | |
| PRIDE Cluster | msp | https://www.ebi.ac.uk/pride/cluster/#/libraries | |
| Decoy library | SpectraST | splib sptxt | Part of the TPP (http://sourceforge.net/projects/sashimi/) |
| DeLiberator | sptxt MSP | http://javaprotlib.sourceforge.net/packages/tools/delib2/index.html | |
| PSDG | msp | http://ms.iis.sinica.edu.tw/COmics/Software.html (available upon request) |
Bibliospec[25]工具包是通过其中的BlibBuild和BlibFilter根据序列数据库搜库结果构建谱图库。BlibBuild从鉴定结果中获取谱图及其鉴定信息,构建肽段与谱图的索引,并以二进制格式存储;谱图库中存在一个肽段离子对应多张谱图,BlibFilter通过计算同一肽段离子对应的多张谱图两两之间的相似度,并求其平均相似度,用平均相似度最高的谱图作为该肽段离子的特征谱图,并删除其他重复的谱图,使得谱图库中的肽段离子唯一对应一张谱图,同时删除平均相似度都很低的所有肽段离子及其对应的谱图,以保证谱图库中的PSMs都是可信的。
2.1.2 生成一致性图谱法2009年Lam等发表的SpectraST[26]可以根据已有的谱图库或序列数据库搜索结果构建谱图库,与Bibliospec不同的是SpectraST采用了通过生成一致性谱图的方式构建谱图库。但SpectraST要求序列数据库搜索结果必须经过PeptideProphet验证,从而根据PeptideProphet的分值筛选PSMs。生成一致性谱图的方法是通过对齐的方法筛选出稳定出现的离子组成新的谱图。SpectraST首先计算所有重复谱图间的相似度,过滤掉与其他谱图相似度都低的谱图,保留下的谱图按信噪比降序排列,为每一个碎片离子在一定的误差范围内统计其出现次数,保留下出现次数超过谱图数60%的离子作为一致性谱图中的离子,计算质荷比和丰度的加权平均值作为特征谱中离子的质荷比和丰度,且碎片离子质量误差范围随着离子丰度变化,每一个碎片离子具有不同的误差范围,以优化丰度对鉴定的影响。生成的特征谱更具有代表性。SpectraST同时严格控制谱图库的质量,每张一致性谱仅保留丰度最大的20个离子,并通过自搜索的方法排除具有争议的谱图。
2010年Ye等发表了pMatch[27],同样通过生成一致性谱图的方式构建谱图库,但其充分利用谱图原始信息和序列信息生成优化的一致性谱。生成一致性谱的同时根据序列产生该肽段离子的理论谱图,结合一致性谱与理论谱生成优化的一致性谱,一致性谱中的离子丰度值归一化后乘以1-θ (0≤θ≤1),理论谱中相应离子丰度乘以θ (0≤θ≤1),两者求和作为优化后的一致性谱的丰度值。pMatch可以更好地识别由于修饰引起的未知肽碎裂模式,能够识别大量非常规修饰信息。pMatch同时产生与目标谱图库大小相同的诱饵谱图库用于后续的质量控制。
2012年Oliver Horlacher等发表了Liberator工具构建谱图库,并于2015年更新至Licerator2.0[28]。Liberator以输入的鉴定结果中分值的高低排序,再计算谱图间的余弦距离,生成最小生成树,只保留最高层的谱图,保留每张谱图中在该分支中20%的谱图中都出现了的离子峰或具有B、Y离子峰注释的离子作为一致性谱图中的离子峰,最后筛除与丰度最高的离子质量误差超过10 Da的离子以缩小谱图库,同时用平方根替换原始丰度值。
2013年Griss等发表了PRIDE Cluster[8, 15]工具用于谱图聚类,改善了MS-Cluster算法[29],每一个类生成可以代表该类的一致性谱图,结合序列数据库搜索结果构建谱图库。该方法生成的一致性谱是通过对原始谱图聚类实现的,其中包含了很多未鉴定的谱图,而这一部分谱图中含有部分高质量的谱图,PRIDE Cluster利用了这部分有意义数据,对只依赖于具有鉴定结果的谱图构建的谱图库具有一定的修正和补充作用,也使得未鉴定的谱图得到重新的鉴定。
2.2 构建诱饵谱图库 2.2.1 肽段层面SpectraST是最早出现的也是运用最广泛的生成诱饵库的工具[30]。其原理是使用“随机-重定位”的方法,在不改变背景噪音的情况下把匹配到的峰进行重新定位。这一方法的问题是,生成的诱饵谱图库与实验谱图库相似,然而诱饵库与目标库过于相似不利于谱图鉴定,基于这一问题,Ahrne等[31]对这一方法进行改进,DeLiberator比较了生成的诱饵谱图库与实验谱图库的相似度,如果相似度过高则不断循环“随机-重定位”的方法。
2.2.2 谱图层面Precursor-Swap-Decoy-Generation(PSDG)[32]生成诱饵谱图库的原理则与前面介绍的两个软件不同,PSDG不生成诱饵序列,使用precursor-swap方法交换两个谱图的前体离子质量值直接根据实验谱图生成诱饵谱图。这一方法不需要任何鉴定信息,不需要考虑离子类型、碎裂方式以及未匹配上的离子,而保留了大量的谱图的特性,生成的诱饵谱图更接近真实的谱图。
3 谱图库搜索算法和工具谱图库搜索是该策略中至关重要的一步,其性能直接影响鉴定结果的优劣。谱图库搜索是将待检测的实验谱图与谱图库中的候选谱图一一比较,搜索引擎根据一定的评分方法对每对谱图-谱图对(SSM)评估。点积(Dot-product,DP)是谱图库搜索算法的基础。点积计算中把每个图谱根据质荷比分为n个单元,每个单元赋予一个权重值,从而转化成一个n维向量。其中,n可以根据碎片离子的质量误差范围设定,权重值根据该单元内的分值强度设定。但对于谱峰密集谱图或者当谱图被少数高丰度谱峰主导时,点积的结果将不准确[33-34]。因此,很多研究团队基于点积法进行修改和改进,发展了大量的谱图库搜索算法和工具,常见的工具见表 3。
Frewen团队发表的Bibliospec工具和Stephen E.Steind等发表的MSPepSearch[35]都是典型的基于点积的谱图搜索工具,以点积的结果作为评判依据,通过以丰度值的平方根置换原始丰度值降低丰度对点积的影响。随后NIST更新了算法,谱图库中具有修饰的谱图根据质荷比移位,从而增加了MSPepSearch检索修饰肽的精确度[36]。
Craig团队发表的X!Hunter[37]以平方根置换原始丰度值,并根据点积的分数计算期望值表征SSM的可信度。
Lam团队2007年发布的SpectraST[4]是目前最流行的谱图库搜索工具。SpectraST对离子丰度取平方根,谱图库中未匹配的离子的丰度乘0.2,以此突出主要离子的贡献。并应用点偏差表征SSM的特异性。为了避免谱图本身质量对结果的影响,最新版本的SpectraST在搜索时为每一对谱图都建立一个不同的相似度分布模型,最终转换为SSM的概率。为了增加搜索速度,SpectraST推出基于GPU的版本(FastPaSS)[38],该版本要比基于CPU的版本要快得多。另外,Mohammed等开发了云计算环境的SpectraST[39]。2014年Manson等又提出了分层打分的方法[40],对每对匹配在不同的电荷状态进行独立打分,最后的结果综合各层打分结果来识别修饰。
Haomin Li等发表的COPaKBClient[41]对点积进行了改进,引入了滑动点积和噪音点积的概念,计算相邻单元和噪音数据的点积,使得COPaKBClient可以适用于所有的仪器类型的数据,并反应了噪音数据对整体结果的影响。作者表明其打分值还可以用来判断谱图是否被正确鉴定。
Wang等2010年发表的M-SPLIT[2, 42-43]使用修正的点积的方法计算谱图的相似度,即余弦距离。其特点是通过把混合光谱看作多个独立的光谱的线性组合以识别混合光谱。
Oliver Horlacher等2015年发表了MzMod[28],基于MzJava库[44]和Apache Spark框架实现为大规模的谱图库构建OMS (Open modification searche)工作流,为肽段离子的每一个修饰分别计算FDR,过滤所有的匹配结果,从而识别蛋白质修饰。
3.2 基于概率模型的谱图库搜索算法2010年Ye等发表的pMatch[27]是出现最早的可以识别未知修饰的谱图库搜索算法。pMatch同样用丰度的平方根代替丰度值,但其打分系统结合了点积和概率函数,以所有候选匹配为背景来评估特定匹配的显著性。Yen等发表的Spec2Spec[45]使用丰度的排序代替离子的真实丰度,并应用了一种类似于MyriMatch的基于概率的算法修正点积的结果,为实验谱图与参考谱图随机匹配建立超几何分布模型。这一算法较点积算法更适合于大规模的蛋白质组数据的谱图库搜索。Cannon等发表的MR-MSPolygraph[46-47]则采用了MSPolygraph算法进行修正。2013年Wang等发表的Tremolo[48]则把点积的结果转换为期望值作为谱图匹配的评估,以谱图匹配低得分的概率来代替在随机匹配高得分的概率。
2012年Dasari等发表的Pepitome[49]应用了一种完全基于概率打分的方法,该方法综合了3种分值:运用超几何检验计算在随机匹配零假设下给定匹配的最终概率、匹配峰之间的Kendall-Tau秩相关系数和误差来源于仪器精度的概率。用Fisher的方法综合前两种分值得到谱图匹配的P值,同时对质量误差进行评估用于当P值相同时选取最佳匹配依据。
3.3 基于机器学习模型的谱图库搜索算法除了传统的计算谱图相似度的方法评估谱图的匹配,一些机器学习的方法同样被应用到谱图分析中。2007年Wu等发表的HMMatch[50]是出现最早的不基于点积的谱图库搜索工具,HMMatch利用图谱中质荷比的分布及其峰值强度训练隐马尔科夫模型,用于对实验谱图的分析。2011年Ahrne等发表的QuickMod[51]使用支持向量机(Support vector machine,SVM)对实验图谱尽可能地利用图谱的所有信息进行分析,并使用了一种特殊的算法获取翻译后修饰的位置信息,而且QuickMod可以识别未知的修饰。
4 谱图库搜索策略存在的问题和挑战谱图库搜索策略相较于序列数据库搜索策略,速度更快,准确度更高。但是谱图库搜索策略本身仍然存在很多问题和挑战。
4.1 谱图库工具缺乏友好的使用界面基于谱图库搜索的蛋白质策略逐渐兴起并流行,且发展了许多谱图库搜索工具,但是这些工具多数只有命令行版本,缺乏友好的使用界面,有些甚至需要用户进行编译和编程,这不便于大多数用户的使用。
4.2 谱图库缺乏统一的国际标准目前出现的谱图库搜索相关的工具都是各实验室独立开发的,其输入与输出文件格式各异,没有统一的标准,使得工具间的衔接差。目前,蛋白质组标准组织(Proteomics standards initiative,PSI)正在积极准备构建谱图库的标准格式,该问题有望解决。
4.3 谱图库不能增量式更新截至2017年7月,本文所调研的谱图库构建方法均不能支持增量式更新,谱图库的更新只能通过重新构建来实现,对时间和资源造成不必要的浪费,同时限制了用户构建特殊的谱图库用于个人研究。
4.4 谱图库中蛋白的覆盖率较低使用谱图库搜索策略需要有比较完整的谱图库作为参考谱图库,才能尽可能多地检索到样本中的蛋白;可供下载的公共谱图库一般基于某一蛋白质数据库的资源构建,蛋白覆盖率仍有待提高,例如NIST最新版(2016-9-23)的人类谱图库蛋白质组的覆盖率仅有27.51%[52];因此,整合多平台数据,构建较完整的谱图库是谱图库构建的一大挑战。也有研究表明通过整合谱图库和序列库可以在一定程度上缓解谱图库覆盖度低的问题[53]。
4.5 谱图库的准确度可进一步提高谱图库构建方法需要序列数据库搜索结果作为基础,因此为谱图库引入了一定的错误鉴定结果,同时忽略了许多未被鉴定的高质量谱图。近年来有研究采用谱图聚类的方法构建谱图库,考虑到错误鉴定和未被鉴定谱图,从而增加谱图库的准确度。因此,如何改进谱图库构建的方法,进一步提高谱图聚类准确率是一重大挑战。
5 总结质谱技术是蛋白质组学的最主要的研究方法,但有研究指出质谱的鉴定效率仍然较低[54],因此提高谱图的鉴定率是蛋白质组学的重要研究方向。而谱图库搜索策略可以有效地提高谱图的鉴定率,近年来发展迅速,已经成为蛋白质鉴定领域最重要的方法之一。
谱图库中特定肽段离子唯一对应一张谱图,搜索谱图库时仅搜索谱图库中包含的谱图,而不需要穷尽某一肽段的所有碎裂模式,搜索速度快;谱图库中的谱图包含了实际离子丰度等信息,使得搜索更加灵敏,打分算法也更加可信;谱图库构建时结合了多个实验数据,库中的谱图可靠且含有许多非常规的碎裂模式及修饰,鉴定结果更加准确,且在共碎裂肽的鉴定方面也具有优势。但目前谱图库搜索相关的工具多数缺乏友好的操作界面和统一的文件格式,需要发展谱图库相关文件格式的国际标准以及格式转换工具,开发蛋白质分析工具框对谱图库搜索相关工具进行包装。谱图库相关算法众多,需要对其进行统一的测试评估并不断改进,以最大程度地提高谱图的鉴定效率。
| [1] | Aebersold R, Mann M. Mass spectrometry-based proteomics. Nature, 2003, 422(6928): 198–207. DOI: 10.1038/nature01511 |
| [2] | Edwards NJ. Protein identification from tandem mass spectra by database searching//Wu C, Arighi C, Ross K, Eds. Protein Bioinformatics. New York: Humana Press, 2017, 1558: 357–380. |
| [3] | Wither MJ, Hansen KC, Reisz JA. Mass spectrometry-based bottom-up proteomics: sample preparation, LC-MS/MS analysis, and database query strategies. Curr Protoc Protein Sci, 2016, 86: 16.4.1–16.4.20. |
| [4] | Lam H, Deutsch EW, Eddes JS, et al. Development and validation of a spectral library searching method for peptide identification from MS/MS. Proteomics, 2007, 7(5): 655–667. DOI: 10.1002/(ISSN)1615-9861 |
| [5] | Ahrné E, Masselot A, Binz PA, et al. A simple workflow to increase MS2 identification rate by subsequent spectral library search. Proteomics, 2009, 9(6): 1731–1736. DOI: 10.1002/pmic.v9:6 |
| [6] | Zhang X, Li YZ, Shao WG, et al. Understanding the improved sensitivity of spectral library searching over sequence database searching in proteomics data analysis. Proteomics, 2011, 11(6): 1075–1085. DOI: 10.1002/pmic.v11.6 |
| [7] | Yilmaz S, Vandermarliere E, Martens L. Methods to calculate spectrum similarity//Keerthikumar S, Mathivanan S, Eds. Proteome Bioinformatics. New York: Humana Press, 2017, 1549: 75–100. |
| [8] | Griss J, Perez-Riverol Y, Lewis S, et al. Recognizing millions of consistently unidentified spectra across hundreds of shotgun proteomics datasets. Nat Methods, 2016, 13(8): 651–656. DOI: 10.1038/nmeth.3902 |
| [9] | Elias JE, Gygi SP. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods, 2007, 4(3): 207–214. DOI: 10.1038/nmeth1019 |
| [10] | NIST Atomic Spectra Database (Version 3.1.0).[EB/OL]. [2017-05-20]. http://physics.nist.gov/asd3. |
| [11] | Vizcaíno JA, Deutsch EW, Wang R, et al. ProteomeXchange provides globally coordinated proteomics data submission and dissemination. Nat Biotechnol, 2014, 32(3): 223–226. DOI: 10.1038/nbt.2839 |
| [12] | Hoopmann MR, Mendoza L, Deutsch EW, et al. An open data format for visualization and analysis of cross-linked mass spectrometry results. J Am Soc Mass Spectrom, 2016, 27(11): 1728–1734. DOI: 10.1007/s13361-016-1435-8 |
| [13] | Craig R, Cortens JP, Beavis RC. Open source system for analyzing, validating, and storing protein identification data. J Proteome Res, 2004, 3(6): 1234–1242. DOI: 10.1021/pr049882h |
| [14] | Deutsch EW. The peptideatlas project//Hubbard S, Jones A, Eds. Proteome Bioinformatics. New York: Humana Press, 2010, 604: 285–296. |
| [15] | Griss J, Foster JM, Hermjakob H, et al. PRIDE Cluster: building a consensus of proteomics data. Nat Methods, 2013, 10(2): 95–96. DOI: 10.1038/nmeth.2343 |
| [16] | Srikumar T, Jeram SM, Lam H, et al. A ubiquitin and ubiquitin-like protein spectral library. Proteomics, 2010, 10(2): 337–342. DOI: 10.1002/pmic.v10:2 |
| [17] | van Steendam K, de Wulf O, Dhaenens M, et al. Species identification from hair by means of spectral library searches. Int J Legal Med, 2014, 128(5): 873–878. DOI: 10.1007/s00414-014-1008-y |
| [18] | Gunaratne J, Schmidt A, Quandt A, et al. Extensive mass spectrometry-based analysis of the fission yeast proteome: the Schizosaccharomyces pombe PeptideAtlas. Mol Cell Proteomics, 2013, 12(6): 1741–1751. DOI: 10.1074/mcp.M112.023754 |
| [19] | Hu YW, Lam H. Expanding tandem mass spectral libraries of phosphorylated peptides: advances and applications. J Proteome Res, 2013, 12(12): 5971–5977. DOI: 10.1021/pr4007443 |
| [20] | Yang XY, Neta P, Stein SE. Quality control for building libraries from electrospray ionization tandem mass spectra. Anal Chem, 2014, 86(13): 6393–6400. DOI: 10.1021/ac500711m |
| [21] | Haynes PA, Stein SE, Washburn MP. Data quality issues in proteomics-there are many paths to enlightenment. Proteomics, 2016, 16(18): 2433–2434. DOI: 10.1002/pmic.201600277 |
| [22] | Feng XD, Li LW, Zhang JH, et al. Using the entrapment sequence method as a standard to evaluate key steps of proteomics data analysis process. BMC Genomics, 2017, 18(Suppl 2): 143. |
| [23] |
Feng XD, Ma J, Chang C, et al. The application and progress of target-decoy database search strategy in identification and quality control of tandem mass spectrometry data in shotgun proteomics.
Prog Biochem Biophys, 2016, 43(7): 661–672.
(in Chinese). 冯晓东, 马洁, 常乘, 等. 目标-诱饵库搜索策略在蛋白质组质谱鉴定和质控中的应用及研究进展. 生物化学与生物物理进展, 2016, 43(7): 661-672. |
| [24] | Shao WG, Zhu K, Lam H. Refining similarity scoring to enable decoy-free validation in spectral library searching. Proteomics, 2013, 13(22): 3273–3283. DOI: 10.1002/pmic.v13.22 |
| [25] | Frewen BE, Merrihew GE, Wu CC, et al. Analysis of peptide MS/MS spectra from large-scale proteomics experiments using spectrum libraries. Anal Chem, 2006, 78(16): 5678–5684. DOI: 10.1021/ac060279n |
| [26] | Lam H, Deutsch EW, Eddes JS, et al. Building consensus spectral libraries for peptide identification in proteomics. Nat Methods, 2008, 5(10): 873–875. DOI: 10.1038/nmeth.1254 |
| [27] | Ye D, Fu Y, Sun RX, et al. Open MS/MS spectral library search to identify unanticipated post-translational modifications and increase spectral identification rate. Bioinformatics, 2010, 26(12): i399–i406. DOI: 10.1093/bioinformatics/btq185 |
| [28] | Horlacher O, Lisacek F, Müller M. Mining large scale tandem mass spectrometry data for protein modifications using spectral libraries. J Proteome Res, 2016, 15(3): 721–731. DOI: 10.1021/acs.jproteome.5b00877 |
| [29] | Frank AM, Monroe ME, Shah AR, et al. Spectral archives: extending spectral libraries to analyze both identified and unidentified spectra. Nat Methods, 2011, 8(7): 587–591. DOI: 10.1038/nmeth.1609 |
| [30] | Lam H, Deutsch EW, Aebersold R. Artificial decoy spectral libraries for false discovery rate estimation in spectral library searching in proteomics. J Proteome Res, 2010, 9(1): 605–610. DOI: 10.1021/pr900947u |
| [31] | Ahrné E, Ohta Y, Nikitin F, et al. An improved method for the construction of decoy peptide MS/MS spectra suitable for the accurate estimation of false discovery rates. Proteomics, 2011, 11(20): 4085–4095. DOI: 10.1002/pmic.201000665 |
| [32] | Cheng CY, Tsai CF, Chen YJ, et al. Spectrum-based method to generate good decoy libraries for spectral library searching in peptide identifications. J Proteome Res, 2013, 12(5): 2305–2310. DOI: 10.1021/pr301039b |
| [33] | Shao WG, Lam H. Tandem mass spectral libraries of peptides and their roles in proteomics research. Mass Spectrom Rev, 2017, 36(5): 634–648. DOI: 10.1002/mas.21512 |
| [34] | Lee S, Kwon MS, Lee HJ, et al. Enhanced peptide quantification using spectral count clustering and cluster abundance. BMC Bioinform, 2011, 12: 423. DOI: 10.1186/1471-2105-12-423 |
| [35] | Stein SE, Scott DR. Optimization and testing of mass spectral library search algorithms for compound identification. J Am Soc Mass Spectrom, 1994, 5(9): 859–866. DOI: 10.1016/1044-0305(94)87009-8 |
| [36] | Burke MC, Mirokhin YA, Tchekhovskoi DV, et al. The hybrid search: a mass spectral library search method for discovery of modifications in proteomics. J Proteome Res, 2017, 16(5): 1924–1935. DOI: 10.1021/acs.jproteome.6b00988 |
| [37] | Craig R, Cortens JC, Fenyo D, et al. Using annotated peptide mass spectrum libraries for protein identification. J Proteome Res, 2006, 5(8): 1843–1849. DOI: 10.1021/pr0602085 |
| [38] | Baumgardner LA, Shanmugam AK, Lam H, et al. Fast parallel tandem mass spectral library searching using GPU hardware acceleration. J Proteome Res, 2011, 10(6): 2882–2888. DOI: 10.1021/pr200074h |
| [39] | Mohammed Y, Mostovenko E, Henneman AA, et al. Cloud parallel processing of tandem mass spectrometry based proteomics data. J Proteome Res, 2012, 11(10): 5101–5108. DOI: 10.1021/pr300561q |
| [40] | Ma CWM, Lam H. Hunting for unexpected post-translational modifications by spectral library searching with tier-wise scoring. J Proteome Res, 2014, 13(5): 2262–2271. DOI: 10.1021/pr401006g |
| [41] | Li HM, Zong NC, Liang XB, et al. A novel spectral library workflow to enhance protein identifications. J Proteomics, 2013, 81: 173–184. DOI: 10.1016/j.jprot.2013.01.026 |
| [42] | Edwards NJ. Protein identification from tandem mass spectra by database searching//Wu C, Chen C, Eds. Bioinformatics for Comparative Proteomics. New York: Humana Press, 2011, 694: 119–138. |
| [43] | Wang J, Tucholska M, Knight JDR, et al. MSPLIT-DIA: sensitive peptide identification for data-independent acquisition. Nat Methods, 2015, 12(12): 1106–1108. DOI: 10.1038/nmeth.3655 |
| [44] | Horlacher O, Nikitin F, Alocci D, et al. MzJava: an open source library for mass spectrometry data processing. J Proteomics, 2015, 129: 63–70. DOI: 10.1016/j.jprot.2015.06.013 |
| [45] | Yen CY, Houel S, Ahn NG, et al. Spectrum-to-spectrum searching using a proteome-wide spectral library. Mol Cell Proteomics, 2011, 10(7): M111.007666. DOI: 10.1074/mcp.M111.007666 |
| [46] | Kalyanaraman A, Cannon WR, Latt B, et al. MapReduce implementation of a hybrid spectral library-database search method for large-scale peptide identification. Bioinformatics, 2011, 27(21): 3072–3073. DOI: 10.1093/bioinformatics/btr523 |
| [47] | Cannon WR, Rawlins MM, Baxter DJ, et al. Large improvements in MS/MS-based peptide identification rates using a hybrid analysis. J Proteome Res, 2011, 10(5): 2306–2317. DOI: 10.1021/pr101130b |
| [48] | Wang MX, Bandeira N. Spectral library generating function for assessing spectrum-spectrum match significance. J Proteome Res, 2013, 12(9): 3944–3951. DOI: 10.1021/pr400230p |
| [49] | Dasari S, Chambers MC, Martinez MA, et al. Pepitome: evaluating improved spectral library search for identification complementarity and quality assessment. J Proteome Res, 2012, 11(3): 1686–1695. DOI: 10.1021/pr200874e |
| [50] | Wu X, Tseng CW, Edwards N. HMMatch: peptide identification by spectral matching of tandem mass spectra using hidden Markov models. J Comput Biol: A J Comput Mol Cell Biol, 2007, 14(8): 1025–1043. DOI: 10.1089/cmb.2007.0071 |
| [51] | Ahrné E, Nikitin F, Lisacek F, et al. QuickMod: a tool for open modification spectrum library searches. J Proteome Res, 2011, 10(7): 2913–2921. DOI: 10.1021/pr200152g |
| [52] | Cho JY, Lee HJ, Jeong SK, et al. Epsilon-Q: an automated analyzer interface for mass spectral library search and label-free protein quantification. J Proteome Res, 2017. DOI: 10.1021/acs.jproteome.6b01019 |
| [53] | Cho JY, Lee HJ, Jeong SK, et al. Combination of multiple spectral libraries improves the current search methods used to identify missing proteins in the chromosome-centric human proteome project. J Proteome Res, 2015, 14(12): 4959–4966. DOI: 10.1021/acs.jproteome.5b00578 |
| [54] | Kimhofer T, Fye H, Taylor-Robinson S, et al. Proteomic and metabonomic biomarkers for hepatocellular carcinoma: a comprehensive review. Br J Cancer, 2015, 112(7): 1141–1156. DOI: 10.1038/bjc.2015.38 |
 2018, Vol. 34
2018, Vol. 34