中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王媛, 贾鹏, 李学俊, 李玉红, 陈鹏
- Wang Yuan, Jia Peng, Li Xuejun, Li Yuhong, Chen Peng
- 水稻N-糖酰胺酶基因(OsPNGase A)的克隆及在毕赤酵母中的分泌表达
- Cloning of Oryza sativa N-glycanase gene (OsPNGase A) and its expression in Pichia pastoris
- 生物工程学报, 2018, 34(3): 421-428
- Chinese Journal of Biotechnology, 2018, 34(3): 421-428
- 10.13345/j.cjb.170254
-
文章历史
- Received: June 24, 2017
- Accepted: September 11, 2017
2 西北农林科技大学 园艺学院,陕西 杨凌 712100
2 Horticulture College, Northwest A & F University, Yangling 712100, Shaanxi, China
糖基化修饰是蛋白质翻译后最复杂的修饰之一,其对于维持蛋白质的稳定性和生物学功能至关重要[1]。蛋白质的去糖基化主要通过化学法和酶法。由于酶法去糖基化具有操作简单,条件温和,能够提供糖链的残基组成、排列顺序和糖苷键的α或β构型信息等优点,是目前应用最为广泛的去糖基化方式[2]。
N-糖酰胺酶(Peptide-N4-(N-acetyl-β-D-glucosaminyl) asparagine amidase,PNGase;EC 3.5.1.52)是分布最为广泛的糖基化修饰类型——N-聚糖结构和功能分析中的关键工具酶,其能够催化N-聚糖和Asn (天冬酰胺)残基之间的β-天冬氨酰基葡糖胺键的水解,生成完整的糖链和蛋白质或肽。N-糖酰胺酶主要分为PNGase F和PNGase A。PNGase F首次发现于脑膜炎败血黄杆菌属Flavobacterium meningosepticum,其主要水解高甘露糖型、复合型和杂合型的N-聚糖[3]。在大肠杆菌中重组表达的PNGase F广泛应用于动物来源的糖蛋白的去糖基化,但对于含有核心α-1, 3岩藻糖/木糖的糖蛋白或糖肽糖链水解效率极低[4]。PNGase A首次发现于杏仁Prunus amygdalus var. Dulcis,故此命名。研究发现植物体内PNGase A在释放N-聚糖以及调节N-聚糖修饰蛋白质的生物活性中具有重要的功能,也可能参与酸性细胞器(如液泡)中丧失生物学作用的糖蛋白的转运[5-6]。PNGase A作用底物范围广,其不仅能够水解与PNGase F相同的底物,也能够水解含有核心α-1, 3岩藻糖/木糖的糖蛋白或糖肽N-聚糖[7],这是植物和无脊椎动物聚糖修饰的典型特征[8-9]。因此,PNGase A更适用于植物和无脊椎动物中糖基化的研究用酶。
随着糖组学与蛋白组学研究的深入,解析多糖结构与功能之间的关系日益受到关注和重视。研发新的去糖基化酶成为核心的技术需求。目前商业化的PNGase A主要来自杏仁[10]。Takahashi首先发现杏仁中存在N-聚糖酶活性的糖苷酶[11],Altmann F对杏PNGase A分析显示其由55 kDa和27 kDa两个亚基组成的异源二聚体,且自身存在糖基化[10, 12]。异源二聚体的结构特征在一定程度上限制了其通过重组表达进行制备。Kimura等从大豆Glycine max粉中纯化得到单一亚基的PNGase A[13]。从天然原料中纯化上述两种酶步骤繁琐,耗时费力,并且产率较低,每100 g杏仁仅可纯化获得280 μg酶蛋白。迄今为止,仅有番茄Lycopersicum esculentum的PNGase A基因克隆并进行重组表达的报道[5]。
本研究从水稻中克隆OsPNGase A基因,利用无缝克隆的方式构建毕赤酵母表达载体,探索OsPNGase A在毕赤酵母SMD1168H中的分泌表达和纯化方法,并对重组酶的催化特性进行分析,以期为糖蛋白的去糖基化研究提供一个新的工具酶,同时为OsPNGase A的高效制备奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒水稻(Oryza sativa Japonica Group)总RNA由西北农林科技大学生命科学学院陈坤明教授惠赠。大肠杆菌菌株TOP10、毕赤酵母SMD1168H和毕赤酵母表达载体pPICZ(α)A均为本实验室保存。
1.1.2 工具酶与主要试剂2× iProof™ HF Master Mix为Bio-Rad公司产品。pCloneEZ克隆载体为CloneSmart公司产品。一步法克隆试剂NEBuilder® HiFi DNA Assembly为NEB公司产品。阴离子层析柱DEAE-Sepharose Fast Flow阴离子交换柱和金属离子螯合层析柱HisTrap HP为GE Healthcare公司产品。Glycopeptidase F (PNGase F)购自TaKaRa公司。水稻重组表达的转铁蛋白(Transferrin,TRF)、辣根中纯化的辣根过氧化物酶(Horse radish peroxidase,HRP)和玉米中重组表达的抗生物素蛋白(Avidin)均为Sigma公司产品。其他生物化学及分子生物学试剂均为国产或进口分析纯。引物合成及测序由北京奥科生物技术有限责任公司完成。
1.2 方法 1.2.1 水稻OsPNGase A基因的克隆以水稻总RNA (2 μg)为模板,20 μL体系进行反转录得到cDNA。根据NCBI数据库报道的序列(登录号XM_015775832)设计引物(OsP-F/ OsP-R,表 1),从水稻cDNA中扩增OsPNGase A编码序列。PCR反应体系(25 μL):cDNA 1 μL、引物OsP-F (10 μmol/L)和OsP-R (10 μmol/L)各1 μL、2× iProof mix 12.5 μL、ddH2O 2.5 μL,5× GC增强剂7 μL。PCR反应程序为:95 ℃预变性2 min;95 ℃变性10 s,65 ℃退火10 s,72 ℃延伸1 min,10个循环;95 ℃变性10 s,60 ℃退火15 s,72 ℃延伸1 min,32个循环;72 ℃终延伸10 min。PCR产物经1%琼脂糖凝胶电泳检测后将目的条带割胶回收,连接至pCloneEZ克隆载体进行测序。
| Primer name | Primer sequence (5'-3') |
| OsP-F | ATGGCGGAGTCGTGCTACGTGCG |
| OsP-R | TCACGAGCTCAGCCGTGCCACT |
| POsP-F | GAAAAGAGAGGCTGAAGCTTCACCTCTCAAGCTTCGGCTATCTGCCT |
| POsP-R | TCAATGATGATGATGATGATGCGAGCTCAGCCGTGCCACT |
| piLinearSac-F | CGCTCATTCCAATTCCTTCTATTAGGC |
| piLinearSac-R | AGCTCCAATCAAGCCCAATAACTG |
| 5'AOX | GACTGGTTCCAATTGACAAGC |
| 3'AOX | GCAAATGGCATTCTGACATCC |
| The homologous arm sequences were underlined. | |
设计带有同源臂的引物(POsP-F/POsP-R,表 1),从测序正确的克隆载体pCloneEZ-OsPNGase A上扩增OsPNGase A成熟蛋白的编码区序列,参照NEBuilder® HiFi DNA Assembly试剂盒说明书构建酵母分泌表达载体pPICZ(α)A-OsPNGase A。
1.2.3 毕赤酵母转化及转化子鉴定以重组质粒pPICZ(α)A-OsPNGase A为模板,用线性化引物piLinearSac-F和piLinearSac-R (表 1)引物进行PCR扩增制备线性化重组载体,电转化毕赤酵母SMD1168H,涂布于YPD固体培养基上,zeocin筛选浓度为100 μg/mL,28 ℃培养2 d。AOX引物(表 1)对转化子进行菌落PCR鉴定。
1.2.4 重组OsPNGase A的诱导表达及纯化将单克隆接种于YPD,28 ℃、220 r/m培养至OD600=2.0时,取50 μL菌液,3 000 r/min收集菌体,用BMGY培养基悬浮菌体,转接于5 mL BMGY,28 ℃、220 r/min培养。当OD600为2.0时,以同样方式转接于20 mL BMMY中进行诱导。每隔24 h补加甲醇至终浓度为1% (V/V),诱导96 h后,离心收集发酵液。
发酵液经截留分子量为10 kDa超滤管浓缩,浓缩样品依次通过DEAE-Sepharose阴离子交换层析和HisTrap金属离子螯合层析对OsPNGase A进行纯化。SDS-PAGE电泳检测纯化产物的纯度,Bradford法测定蛋白浓度[14]。
1.2.5 不同反应温度、pH、还原剂、表面活性剂对酶活性的影响分析不同反应时间温度(0 ℃、4 ℃、25 ℃、37 ℃、50 ℃、60 ℃)和pH (4.0、5.0、6.0、7.0、8.0、9.0、10.0)对水解活性的影响,以确定OsPNGAase A的最适反应条件。反应体系与条件为:取1 μL Avidin (5 mg/mL)溶于15 μL不同pH的缓冲液中,95 ℃加热变性10 min后迅速置于冰上。然后加入2 μL 10% NP-40和0.1 μg酶液。不同温度保温5 h。15% SDS-PAGE检测酶切结果。参考上述体系,在最适条件下分析10、50、100 mmol/L还原剂DTT/β-巯基乙醇以及0.1%、0.5%、1%的表面活性剂Triton X-100对酶活性的影响。
1.2.6 OsPNGase A对不同糖蛋白底物的去糖基化参照1.2.5反应体系,分别以TRF、HRP和Avidin为底物检测OsPNGase A的去糖基化活性。12.5% SDS-PAGE检测重组OsPNGase A对不同类型糖蛋白的去糖基化效果。酶活力单位定义为,在上述反应条件下,每分钟水解1 μg Avidin糖蛋白所需要的酶量为1个活力单位(U)。
1.2.7 重组OsPNGase A与PNGase F去糖基化作用的比较参考1.2.5反应体系,两组反应分别加入1 mU的PNGase A和商业化PNGase F,37 ℃反应5 h,15% SDS-PAGE检测。
2 结果与分析 2.1 OsPNGase A基因扩增与序列分析以水稻cDNA为模板,成功克隆得到OsPNGase A编码基因(图 1)。其ORF大小为1 851 bp,编码616个氨基酸;预测含有25个氨基酸的信号肽序列,成熟蛋白的分子量为63.8 kDa,等电点为4.97。
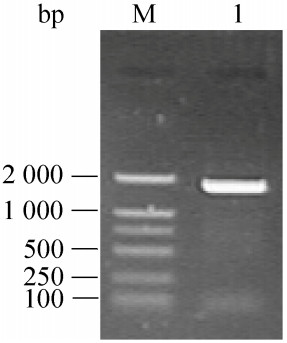
|
| 图 1 OsPNGase A基因的PCR扩增 Figure 1 PCR amplification of the OsPNGase A gene. M: DL2000 DNA marker; 1: PCR product. |
| |
pPICZ (α) A-OsPNGase A转化的毕赤酵母SMD1168H经含有100 μg/mL Zeocin的YPD筛选后,抗性菌落进一步通过5′ AOX和3′ AOX引物PCR鉴定(图 2A)。阳性克隆用以分泌表达OsPNGase A。SDS-PAGE检测结果显示(图 2B),浓缩、透析后的发酵液经DEAE-Sepharose阴离子交换层析(图 2B,泳道5)和HisTrap金属离子螯合层析(图 2B,泳道6)纯化获得单一条带的OsPNGase A,分子大小与预期一致。蛋白含量测定结果显示OsPNGase A的产量达到12.3 mg/L。

|
| 图 2 OsPNGase A转基因酵母的PCR检测(A)和OsPNGase A重组蛋白的分泌表达及纯化(B) Figure 2 PCR screening of P. pastoris clones with OsPNGase A integrated and purification of recombinant OsPNGase A. (A) PCR detection of Zeocin-resistant P. pastoris. Lane 1 and 2: PCR products of two separate Zeocin-resistant P. pastoris clones. M: DL2000 DNA molecular weight marker. (B) Secretory expression and purification of recombinant OsPNGase A. Lane 1 and 2: medium of P. pastoris after induction with methanol; lane 3 and 4: medium of P. pastoris before induction; lane 5 and 6: purified product of OsPNGase A with DEAE-Sepharose and HisTrap immobilized nickel chromatography, respectively. The black arrows indicate the target gene and protein band, respectively. |
| |
在pH 4-10范围内分析OsPNGase A对Avidin的水解活性,结果如图 3A所示,OsPNGase A在pH 6.0-10.0的酸碱范围内都保持一定的活性,最适pH为6.0 (图 3A,泳道3)。在0-70 ℃范围内测定温度对OsPNGase A活性的影响,如图 3B所示,在0-50 ℃范围内OsPNGase A均具有水解活性,在40 ℃时酶活最高。
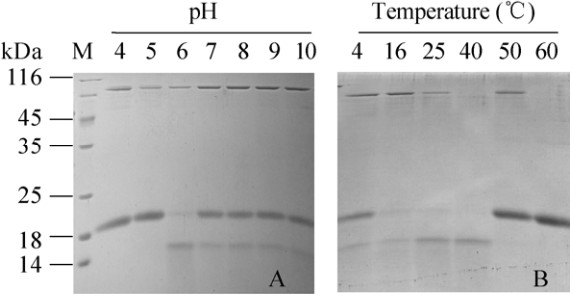
|
| 图 3 pH (A)和温度(B)对OsPNGase A酶活性的影响 Figure 3 Effects of pH (A) and temperature (B) on OsPNGase A activity. |
| |
不同底物的去糖基化结果表明,OsPNGase A对TRF (图 4A)、HRP (图 4B)以及Avidin (图 4C)都具有去糖基化的能力,表明OsPNGase A在植物来源的糖蛋白的研究中具有潜在应用价值。在相同的反应条件下,3种底物相比,OsPNGase A对Avidin的去糖基化效率最高。选择Avidin作为底物进行酶活力的确定,其比活力为258 U/mg。
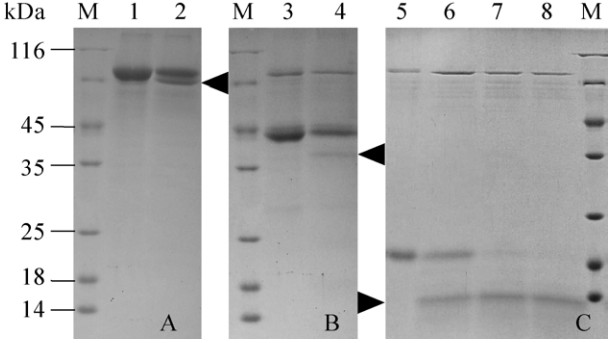
|
| 图 4 12.5% SDS-PAGE检测OsPNGase A对不同糖蛋白底物的去糖基化 Figure 4 12.5% SDS-PAGE analyzed the deglycosylation of different glycoproteins with OsPNGase A. (A) Recombinant human transferrin from rice was incubated for 5 h at 37 ℃ with heat-inactivated OsPNGase A (lane 1) or active OsPNGase A (lane 2), respectively. (B) HRP from horseradish was incubated for 5 h at 37 ℃ with heat-inactivated OsPNGase A (lane 3) or active OsPNGase A (lane 4), respectively. (C) Recombinant egg white Avidin from corn was incubated at 37 ℃ with heat-inactivated OsPNGase A for 5 h (lane 5) or with active OsPNGase A for 3 h (lane 6), 4 h (lane 7), or 5 h (lane 8), respectively. After enzymatic digestion, samples were analyzed by 12.5% SDS-PAGE. The black arrows indicate the deglycosylated protein products. |
| |
OsPNGase A的活性受到表面活性剂Triton X-100的抑制,随浓度的增加而抑制增强,在终浓度1%的Triton X-100下其活力基本丧失(图 5A)。图 5B显示,在10-100 mmol/L还原剂DTT和β-巯基乙醇的浓度范围内,OsPNGase A都具有活性。
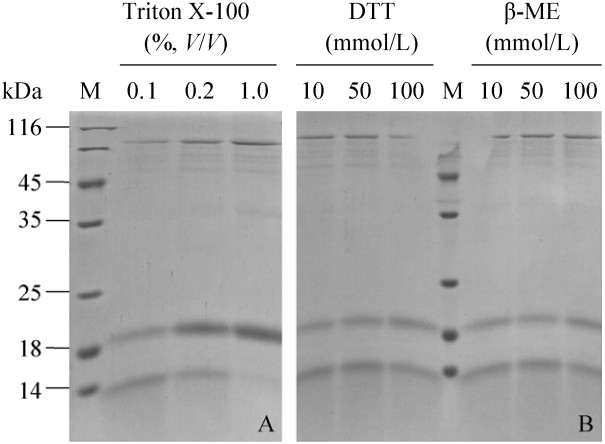
|
| 图 5 Triton X-100 (A),DTT和β-巯基乙醇(B)对OsPNGase A活性的影响 Figure 5 Effects of Triton X-100 (A, lane 1-3), DTT (B, lane 4-6) and β-mercaptoethanol (B, lane 7-9) on OsPNGase A activity. |
| |
以本研究重组表达的OsPNGase A和商业化的PNGase F对Avidin进行酶切,结果显示OsPNGase A对底物的去糖基化效果明显(图 6,泳道3)。在相同的反应条件下,PNGase F酶切后的Avidin分子量并未改变(图 6,泳道1)。结果表明OsPNGase A更适用于该糖蛋白的去糖基化。

|
| 图 6 15% SDS-PAGE检测OsPNGase A和商业化的PNGase F对Avidin的去糖基化 Figure 6 15% SDS-PAGE analyzed the deglycosylation of Avidin with OsPNGase A and PNGase F. Avidin was incubated for 5 h at 37 ℃ with the PNGase F (lane 1), heat-inactivated PNGase F (lane 2), OsPNGase A (lane 3) and heat-inactivated OsPNGase A (lane 4), respectively. After enzymatic digestion, samples were analyzed by 15% SDS-PAGE. |
| |
N-糖基化是真核生物蛋白质最重要的翻译后修饰之一,对于蛋白的结构、酶学活性以及稳定性具有重要意义[15]。目前在蛋白质N-糖基化修饰类型的研究中,使用广泛的工具酶为PNGase F和PNGase A[7]。对于PNGase F基因的克隆及重组表达研究较多,并已有蛋白结构信息的报道[16];对于PNGase A,目前主要从杏仁中提取获得。本研究根据NCBI数据库中预测的水稻OsPNGase A序列(XM_015775832),对其进行了基因克隆。由于OsPNGase A序列具有较高的GC含量(69.48%),进行了多次反转录及扩增,电泳检测均未得到预期大小的产物。因此在反转录及PCR体系中添加GC增强剂以提高解链效率,最终实现了OsPNGase A基因的克隆。使用NEB的一步法克隆试剂盒构建重组表达载体pPICZ (α)A-OsPNGase A,克服了传统的酶切连接方法构建载体时基因内部酶切位点的限制以及连接时间长、连接效率低等缺点。此外,根据pPICZ (α) A载体上SacⅠ限制性酶切位点两侧的载体序列设计引物,使用Bio-Rad高保真的iProof DNA聚合酶进行重组载体的扩增来实现线性化,避免了重组质粒大量制备过程中昂贵的Zeocin抗生素的使用以及质粒酶切、回收等繁琐的操作。从方法学上,本研究的体系对于真核生物高GC含量基因的克隆以及毕赤酵母高通量表达体系的构建有重要的参考价值。
对于商业化的PNGase A,主要从杏仁中提取获得,产率低下,目前尚未有重组表达杏仁PNGase A的报道。异源二聚体的结构特征可能是限制其通过重组表达进行制备的原因。虽已有番茄来源的LePNGase A在毕赤酵母中实现了重组表达,但表达水平极低,电泳检测仅显示微弱的条带。毕赤酵母中重组表达的LePNGase A最适pH为4.5,在中性及碱性pH条件下活力完全丧失[5]。本研究实现了OsPNGase A在毕赤酵母中的高效分泌表达,1 L发酵液中可纯化获得12.3 mg目的蛋白。对OsPNGase A催化特性分析显示其在较为宽泛的pH条件下均具有活性,这为OsPNGase A的实践应用奠定了良好的酶学性质基础。OsPNGase A的最适反应温度为40 ℃,与杏来源的PNGase A基本相同[10]。在4 ℃时OsPNGase A仍然具有一定的活性,暗示其可用于在常温下不稳定的糖蛋白底物的去糖基化。在蛋白提取制备及长期保存过程中,常会用到一些还原剂,如还原剂DTT和β-ME以保护“-SH”基团,防止蛋白氧化[17-18],表面活性剂Triton X-100增加蛋白的稳定性和溶解度[19]。迄今为止,尚未见这些试剂对PNGase A活性影响的分析报道。本研究较为系统地分析了不同浓度的还原剂和Triton X-100对OsPNGase A活性的影响,如图 5所示,一定浓度范围内的上述3种蛋白稳定化试剂可用于OsPNGase A的去糖基化反应。
毕赤酵母重组表达的番茄LePNGase A仅以来自人的转铁蛋白为底物,检测该酶的去糖基化能力,并没有以植物来源的糖蛋白为底物进行去糖基化反应。本研究中OsPNGase A对于3种植物来源的糖蛋白都具有去糖基化的能力,但去糖基化后的蛋白在SDS-PAGE后着色较浅。可能是由于底物蛋白N-聚糖修饰的异质性,使得去糖基化后的蛋白产物不均一,进而影响主产物考马斯亮蓝R250的染色强度[20]。Yang等和Gray等用杏仁中纯化的PNGase A对HRP进行酶切,对释放出的聚糖进行MALDI-TOF/MS研究,结果表明,HRP中存在“(Xyl) Man3 (Fuc) GlcNAc2”、“(Xyl) Man2 (Fuc) GlcNAc2”、“Man3 (Fuc) GlcNAc2”的N-糖基化修饰[21-22]。本研究表明OsPNGase A能够作用含有核心[α-Fuc-(1→3)] (α-1→3岩藻糖)的N-聚糖。但相比于Avidin,OsPNGase A对于分子量较大的HRP和TRF底物的去糖基化效果欠佳,可能是由于这两种底物分子量较大而产生的空间位阻所导致[23]。
已有研究表明,PNGase F不能用于含有核心α-1.3岩藻糖的N-聚糖的去除,且该糖基化修饰类型的糖蛋白多来自植物[9]。本研究与商业化的PNGase F比较结果显示,OsPNGase A更适用于植物来源的Avidin去糖基化,说明OsPNGase A在植物糖蛋白的研究中具有应用潜能。水稻OsPNGase A的成功表达为植物糖蛋白的研究提供一个新的工具酶,酵母分泌表达体系的建立为PNGase A的大量制备奠定了基础。
| [1] | Spiro RG. Protein glycosylation: nature, distribution, enzymatic formation, and disease implications of glycopeptide bonds. Glycobiology, 2002, 12(4): 43R–56R. DOI: 10.1093/glycob/12.4.43R |
| [2] |
Guo H, Deng WX, Zhang Y. Research progression of glycoprotein.
Biotechnol Bull, 2009(3): 16–19.
(in Chinese). 郭慧, 邓文星, 张映. 糖蛋白的研究进展. 生物技术通报, 2009(3): 16-19. |
| [3] | Barsomian GD, Johnson TL, Borowski M, et al. Cloning and expression of peptide-N4-(N-acetyl-β-D-glucosaminyl) asparagine amidase F in Escherichia coli. J Biol Chem, 1990, 265(12): 6967–6972. |
| [4] | Tretter V, Altmann F, M rz L. Peptide-N4-(N-acetyl-β-glucosaminyl) asparagine amidase F cannot release glycans with fucose attached α1→3 to the asparagine-linked N-acetylglucosamine residue. Eur J Biochem, 1991, 199(3): 647–652. DOI: 10.1111/ejb.1991.199.issue-3 |
| [5] | Hossain MA, Nakano R, Nakamura K, et al. Molecular identification and characterization of an acidic peptide: N-glycanase from tomato (Lycopersicum esculentum) fruits. J Biochem, 2010, 147(2): 157–165. DOI: 10.1093/jb/mvp157 |
| [6] | Berger S, Menudier A, Julien R, et al. Do de-N-glycosylation enzymes have an important role in plant cells?. Biochimie, 1995, 77(9): 751–760. DOI: 10.1016/0300-9084(96)88193-4 |
| [7] | Ishimizu T. Plant N-glycans and their degrading enzymes// Taniguchi N, Endo T, Hart GW, et al, Eds. Glycoscience: Biology and Medicine. Japan: Springer, 2015: 1111-1117. |
| [8] | Staudacher E, Altmann F, Wilson IBH, et al. Fucose in N-glycans: from plant to man. Biochim Biophys Acta, 1999, 1473(1): 216–236. DOI: 10.1016/S0304-4165(99)00181-6 |
| [9] | Wilson IBH, Zeleny R, Kolarich D, et al. Analysis of Asn-linked glycans from vegetable foodstuffs: widespread occurrence of Lewis a, core α1, 3-linked fucose and xylose substitutions. Glycobiology, 2001, 11(4): 261–274. DOI: 10.1093/glycob/11.4.261 |
| [10] | Altmann F, Paschinger K, Dalik T, et al. Characterisation of peptide-N4-(N-acetyl-β-glucosaminyl) asparagine amidase A and its N-glycans. Eur J Biochem, 1998, 252(1): 118–123. DOI: 10.1046/j.1432-1327.1998.2520118.x |
| [11] | Takahashi N. Demonstration of a new amidase acting on glycopeptides. Biochem Biophys Res Commun, 1977, 76(4): 1194–1201. DOI: 10.1016/0006-291X(77)90982-2 |
| [12] | Taga EM, Waheed A, van Etten RL. Structural and chemical characterization of a homogeneous peptide N-glycosidase from almond. Biochemistry, 1984, 23(5): 815–822. DOI: 10.1021/bi00300a006 |
| [13] | Kimura Y, Ohno A. A new peptide-N4-(acetyl-β-glucosaminyl) asparagine amidase from soybean (Glycine max) seeds: purification and substrate specificity. Biosci Biotechnol Biochem, 1998, 62(2): 412–418. DOI: 10.1271/bbb.62.412 |
| [14] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72(1/2): 248–254. |
| [15] | Imperiali B, O'Connor SE. Effect of N-linked glycosylation on glycopeptide and glycoprotein structure. Curr Opin Chem Biol, 1999, 3(6): 643–649. DOI: 10.1016/S1367-5931(99)00021-6 |
| [16] | Norris GE, Stillman TJ, Anderson BF, et al. The three-dimensional structure of PNGase F, a glycosyl asparaginase from Flavobacterium meningosepticum. Structure, 1994, 2(11): 1049–1059. DOI: 10.1016/S0969-2126(94)00108-1 |
| [17] | Cleland WW. Dithiothreitol, a new protective reagent for SH groups. Biochemistry, 1964, 3(4): 480–482. DOI: 10.1021/bi00892a002 |
| [18] | Erel O, Neselioglu S. A novel and automated assay for thiol/disulphide homeostasis. Clin Biochem, 2014, 47(18): 326–332. DOI: 10.1016/j.clinbiochem.2014.09.026 |
| [19] | Chae PS, Wander MJ, Cho KH, et al. Carbohydrate-containing Triton X-100 analogues for membrane protein solubilization and stabilization. Mol Biosyst, 2013, 9(4): 626–629. DOI: 10.1039/c3mb25584k |
| [20] | Hanisch FG, Jovanovic M, Peter-Katalinic J. Glycoprotein identification and localization of O-glycosylation sites by mass spectrometric analysis of deglycosylated/alkylaminylated peptide fragments. Anal Biochem, 2001, 290(1): 47–59. DOI: 10.1006/abio.2000.4955 |
| [21] | Yang BY, Gray JSS, Montgomery R. The glycans of horseradish peroxidase. Carbohydr Res, 1996, 287(2): 203–212. DOI: 10.1016/0008-6215(96)00073-0 |
| [22] | Gray JSS, Yang BY, Montgomery R. Heterogeneity of glycans at each N-glycosylation site of horseradish peroxidase. Carbohydr Res, 1998, 311(1/2): 61–69. |
| [23] | Bager R, Johansen JS, Jensen JK, et al. Protein conformational change delayed by steric hindrance from an N-linked glycan. J Mol Biol, 2013, 425(16): 2867–2877. DOI: 10.1016/j.jmb.2013.05.007 |
 2018, Vol. 34
2018, Vol. 34




