中国科学院微生物研究所、中国微生物学会主办
文章信息
- 马子玉, 任亚辉, 凌敏, 王华岩
- Ma Ziyu, Ren Yahui, Ling Min, Wang Huayan
- METTL3对猪干细胞多能基因表达的调控作用
- METTL3 regulates expression of pluripotent genes in porcine pluripotent stem cells
- 生物工程学报, 2018, 34(3): 369-378
- Chinese Journal of Biotechnology, 2018, 34(3): 369-378
- 10.13345/j.cjb.170247
-
文章历史
- Received: June 21, 2017
- Accepted: September 4, 2017
2 西北农林科技大学 创新实验学院,陕西 杨凌 712100
2 Innovation Experimental College, Northwest A & F University, Yangling 712100, Shaanxi, China
将外源多能转录因子Oct4、Sox2、Klf4、cMyc导入成体细胞,经过细胞重编程,能够获得一种具有自我更新和多向分化潜能的诱导性多能干细胞(即iPS细胞)[1],可以在体内外分化为内、中、外三个胚层细胞和各种组织、器官[2]。小鼠的iPS建系后,许多大动物的iPS细胞系也相继获得[3-5]。本实验室在猪胚胎成纤维细胞中表达OSKM四因子,并在培养基中添加外源白血病抑制因子LIF和碱性成纤维细胞生长因子bFGF,获得原始态(naïve)样的猪iPS细胞,能经历30代次以上的传代并保持多能性[6]。然而,猪iPS细胞的建系仍然进展缓慢,目前未能获得真正naïve状态的细胞,缺少稳定的体外诱导培养体系,不能实现生殖系嵌合[7-8],因此,优化体外培养体系、获得真正naïve状态的猪iPSCs具有十分重要的意义。m6A甲基化作为重要的表观遗传修饰方式,对小鼠和人的胚胎干细胞和诱导多能干细胞的干性维持与逆转、细胞重编程以及胚胎发育等生物学过程均起着重要的调节作用。
m6A修饰是指腺嘌呤第6位N原子上的甲基化修饰,是真核生物mRNA中最常见的转录后修饰[9],影响mRNA的剪接、出核转运、翻译和稳定性等[10]。mRNA的m6A修饰由甲基转移酶复合体(WTAP/METTL3/METTL14)催化形成[11-12],由去甲基化酶ALKBH5和FTO催化去甲基化[13-14],并被YTH结构域家族识别[15-16],结合蛋白YTHDF2招募CCR4-NOT复合体降解被m6A修饰的mRNA[17]。METTL3亚基是m6A甲基转移酶复合体中的核心组分[18],定位于核散斑[19],Mettl3失活使m6A水平降低后促进小鼠ESC细胞自我更新,并阻碍分化[20]。有报道表明,Mettl3可以作为小鼠naïve状态的分子开关,确保多能性因子正确下调,为细胞分化做准备[21]。m6A甲基转移酶将甲基供体S-腺苷甲硫氨酸(SAM)上的甲基转移到mRNA腺嘌呤的第6位N原子上[22],环亮氨酸是SAM转移酶的竞争性抑制剂。有研究表明,在培养体系中添加环亮氨酸可以调控小鼠成纤维细胞的重编程进程[23]。由此可见,m6A修饰可以影响mRNA的稳定性,调控多能因子的转录水平,以及调控小鼠多能干细胞的表观遗传状态与细胞重编程。因此,我们推测METTL3能够影响猪多能干细胞中多能因子mRNA的稳定性,调节这些因子的表达水平,从而对猪多能干细胞的多能性产生影响。
我们克隆了猪m6A甲基化修饰相关基因METTL3,构建了过表达和干扰载体,并通过在猪多能干细胞培养体系中添加甲基化抑制剂环亮氨酸,研究其对猪多能干细胞状态转变和多能基因的影响,为优化猪iPSCs的诱导培养体系提供新的方向和理论依据。
1 材料与方法 1.1 材料大肠杆菌Stellar感受态细胞、质粒pEGFP-C1、pCDH、pSIH1、人胚肾细胞(HEK293T)、小鼠胎儿成纤维细胞(MEF)、猪胎儿成纤维细胞(PEF)、猪肾上皮细胞(PK-15)、猪诱导多能干细胞(DOX-iPS、PS11、PS23)由陕西省干细胞工程技术研究中心保存。pGEM-T Easy购自Promega公司。猪各组织器官采集于陕西万盛肉类加工有限公司的屠宰场。
1.2 试剂基因组提取试剂盒、质粒提取试剂盒、DNA纯化回收试剂盒、总RNA试剂盒、DNA marker购自天根生化科技(北京)有限公司;限制性内切酶、T4 DNA连接酶、反转录试剂盒购自Thermo Fisher Scientific;高保真DNA聚合酶和CCK-8试剂盒购自南京诺唯赞生物科技有限公司;Taq聚合酶购自康为世纪生物科技有限公司;荧光定量PCR试剂盒购自北京全式金生物技术有限公司;DMEM购自Hyclone;Opti-MEM、DPBS、Trypsin、胎牛血清(FBS)购自Gibco;琼脂糖、脂质体(Lipofectamine 2000)、非必需氨基酸(NEAA)、谷氨酰胺(GlutaMAX)、β-巯基乙醇(β-ME)、购自Invitrogen;SB431542、CHIR99021购自Stem RD;明胶、Doxycycline、DMSO、环亮氨酸、α-磷酸萘酚钠、Fast Red TR购自Sigma-Aldrich;多聚甲醛、Tris-HCl购自北京索莱宝科技有限公司;引物由华大基因合成(表 1)。
| Name | Sequence (5′–3′) | Size (bp) |
| METTL3-F | CTAGTCTAGAATGTCGGACACGTGGAGCTCT | 1 859 |
| METTL3-R | TTCGAACCCAACTACAATACAAACTT | |
| METTL3-F | CCACTTCTGGTGGCCCTAAG | 502 |
| METTL3-R | AGGTCAACATCTGAGGCAGC | |
| GAPDH-F | AAGGTCGGAGTGAACGGATT | 549 |
| GAPDH-R | AGTCTTCTGGGTGGCAGTGAT | |
| qMETTL3-F | CCTATGCTGATCATTCCAAG | 260 |
| qMETTL3-R | TTGTTGGTTCAGAAGGCTCTC | |
| qOCT4-F | TGAGGCTTTGCAGCTCAGTT | 179 |
| qOCT4-R | TCTCCAGGTTGCCTCTCACT | |
| qLIN28A-F | TGCCGGCATCTGTAAATGGT | 246 |
| qLIN28A-R | CTCTCGCTCCCAATGCAGAA | |
| qNANOG-F | AGGGCTCAGCCAGTACAGAA | 101 |
| qNANOG-R | CCAGCTCTGATTACCCCACA | |
| qβ-ACTIN-F | GTGACAGCAGTCGGTTGGAT | 161 |
| qβ-ACTIN-R | TTTTGGGAAGGCAGGGACTT | |
| shRNA-F | GATCCGCAGTTCCTGAGCTAGCTACATCAAGA GTGTAGCTAGCTCAGGAACTGCTTTTTG |
55 |
| shRNA-R | AATTCAAAAAGCAGTTCCTGAGCTAGCTACAC TCTTGATGTAGCTAGCTCAGGAACTGCG |
登录NCBI下载不同物种METTL3氨基酸序列,使用在线软件phylogeny (http://www.phylogeny.fr/index.cgi)构建METTL3蛋白进化树。使用Primer Blast设计引物,通过RT-PCR检测METTL3在猪的不同器官(肺、脾、脑、肾等)、不同细胞(PEF、PK-15)和不同多能干细胞(DOX-iPS、PS11、PS23)中的表达情况。使用软件ImageJ对DNA琼脂糖电泳结果进行灰度分析。
1.4 猪METTL3的克隆和载体构建从PEF细胞中提取总RNA,反转录得到cDNA。设计合成猪METTL3上下游引物,以得到的cDNA为模板克隆METTL3 (CDS + polyA)片段,连接pGEM-T Easy载体,送华大基因测序鉴定,并将测序结果与NCBI公布的猪METTL3序列进行对比。
用XhoⅠ和Hind Ⅲ将目的基因METTL3和载体pEGFP-C1进行酶切,电泳、回收后将METTL3连接到载体上,获得过表达载体pEGFP-METTL3;重新设计带XbaⅠ和BstbⅠ酶切位点的METTL3上下游引物,以构建好的pEGFP-METTL3为模板进行PCR扩增,电泳、回收后重新酶切、连接,构建慢病毒载体pCDH-METTL3。设计并合成METTL3的shRNA序列(表 1),用BamH Ⅰ和EcoRⅠ将pSIH1-copGFP、pSIH1-PURO进行酶切、电泳、回收,将退火后的干扰序列连接到载体上,获得慢病毒干扰载体pSIH1-copGFP-sh-METTL3和pSIH1-Puro-sh-METTL3。将以上构建的载体转化到大肠杆菌中,挑取阳性菌落,提取质粒,进行酶切鉴定,选取鉴定正确的质粒送华大基因测序鉴定。
1.5 细胞培养、转染和感染HEK293T、PK-15细胞培养在10% FBS的DMEM中;MEF、PEF细胞培养在15% FBS的DMEM中,并添加1% NEAA;DOX-iPS细胞培养在15% FBS的DMEM中,并添加1% NEAA、1% GlutaMAX、0.1 mmol/L β-ME、2 μmol/L SB431542、3 μmol/L CHIR99021、4 μg/mL Doxycycline。称取不同重量的环亮氨酸溶解在DOX-iPS培养基中,配制成不同浓度的环亮氨酸培养基,用于竞争性抑制猪piPS细胞内源METTL3的活性。
进行细胞转染时,将HEK293T细胞接种于直径为60 mm的培养皿中培养,按照脂质体2000转染说明书,将构建的过表达和干扰载体分别转染HEK293T细胞包装病毒,48 h后收集上清毒液,经0.45 μm滤器过滤后加入浓度为4 μg/mL的polybrene混匀,分别感染DOX-iPS,12 h后换液。重复感染2次。
1.6 生长曲线测定MEF feeder细胞接种于0.1%明胶包被过的48孔板中,过夜培养后,每孔接种2×103个DOX-iPS细胞,加入含有环亮氨酸的培养基200 μL,设置空白组(未接种DOX-iPS细胞)和对照组(0 mmol/L环亮氨酸培养基)。每组设3个重复。每孔加入20 μL CCK-8溶液,37 ℃孵育1 h,取100 μL上清液转移到96孔板中,测定450 nm各孔吸光值,连续测定7 d。
1.7 碱性磷酸酶(AP)染色称取5 mg Fast Red TR和2 mg α-磷酸萘酚钠盐,溶解于5 mL的0.1 mol/L Tris-HCl (pH 9.0)中,配制成AP染色液。吸去DOX-iPS细胞培养液,用PBS清洗1遍;加入适量的4%多聚甲醛,常温固定20 min;吸去固定液,用PBS清洗3遍,加入AP染色液洗1遍,加入AP染色液常温染色10−20 min,吸去染色液,用PBS清洗3遍之后,置于显微镜下进行观察。
1.8 荧光定量RT-PCR按照总RNA提取和反转录试剂盒说明书,提取RNA并反转录获得cDNA,设计METTL3、OCT4、LIN28A、NANOG、β-ACTIN等引物(表 1),按照荧光定量试剂盒说明书检测基因的表达水平,并用2–ΔΔCt法计算表达变化倍数。
1.9 统计学分析所有数据均用平均值±标准差表示,并经过t-test或ANOVA检验差异是否具有统计学意义。*表示P < 0.05,**表示P < 0.01。
2 结果与分析 2.1 猪METTL3的表达谱和分子克隆比对不同物种METTL3蛋白序列,构建出分子进化树,由进化树可以看出,猪METTL3蛋白与人、鼠的进化距离较远,与牛、羊的进化距离较近(图 1A)。通过RT-PCR对猪胃、皮肤、心脏、脂肪、肌肉、小肠、大肠、子宫、肝脏、肺、脾脏、肾脏、脑各组织和器官中的METTL3表达情况进行检测,并对凝胶电泳结果进行了灰度分析,结果表明METTL3在不同组织中广泛表达(图 1B)。用RT-PCR检测了猪体细胞PEF和PK-15、多能干细胞DOX-iPS、PS11和PS23中METTL3的表达情况,结果表明,METTL3在猪不同细胞中都有表达(图 1C)。从猪的基因组中克隆获得1 859 bp的METTL3基因的编码序列片段(图 1D),构建了过表达载体pEGFP-METTL3,经酶切鉴定正确(图 1E)。将质粒瞬时转染HEK293T细胞,与对照组EGFP蛋白相比,绿色荧光和DAPI细胞核染色显示了EGFP-METTL3融合蛋白定位在细胞核中,证明该融合蛋白具备核定位功能特性(图 1F)。

|
| 图 1 猪METTL3表达谱和分子克隆 Figure 1 Porcine METTL3 expression profile and cloning. (A) Phylogenetic tree of METTL3. (B–C) RT-PCR and densitometry analysis of porcine METTL3 expression in different tissues (B) and cells (C). M3: METTL3. GAP: GAPDH. (D) Cloning of porcine METTL3 (1 859 bp). M: DNA marker. (E) Enzyme digestion analysis. 1: uncut pEGEP-METTL3. 2: pEGEP-METTL3 digested with XhoⅠ/Hind Ⅲ. 3: pEGEP-METTL3 digested with BglⅡ/NdeⅠ. (F) Translocalization of EGFP-METTL3 fusion protein in HEK293T cells. Scale bar=50 μm. |
| |
将pCDH-METTL3过表达载体转染HEK293T细胞包装病毒,感染多能干细胞DOX-iPS,设定感染了空载体pCDH的多能干细胞为对照组。在荧光显微镜下观察到过表达载体pCDH-METTL3成功感染DOX-iPS (图 2A)。与对照组相比,RT-PCR检测到METTL3在DOX-iPS中表达量升高(图 2B),经qRT-PCR确认METTL3表达升高8.41倍(图 2C)。显微镜观察结果显示,细胞形态未发生显著变化,但是,AP染色结果显示,实验组细胞碱性磷酸酶活性比对照组低(图 2D)。qRT-PCR结果显示,实验组OCT4和LIN28A的表达水平变化不显著(图 2E)。

|
| 图 2 过表达METTL3对多能基因表达的调控 Figure 2 Expression of pluripotent genes in piPS with METTL3 overexpression. (A) Infection of pCDH (Ctrl) and pCDH-METTL3 (OE) in HEK293T and DOX-iPS. RT-PCR (B) and qRT-PCR (C) analysis of METTL3 expression in DOX-iPS. (D) Morphology and AP staining of DOX-iPS. (E) qRT-PCR analysis of pluripotent genes expression in DOX-iPS. **P < 0.01; n=3. Scale bar=100 μm. |
| |
将pSIH1-copGFP-sh-METTL3干扰载体转染HEK293T细胞包装病毒,感染PEF细胞,设定感染了空载体pSIH1-copGFP的细胞为对照组。72 h时后观察证明有较高的感染效率(图 3A),qRT-PCR检测结果显示,72 h干扰效率达到49%,96 h干扰效率达到了72% (图 3B)。随后,通过HEK293T细胞将pSIH1-Puro、pSIH1-Puro-sh-METTL3包装成病毒,感染DOX-iPS细胞。与感染空载体的对照组相比,RT-PCR结果显示METTL3在DOX-iPS中表达量降低(图 3C),qRT-PCR结果显示METTL3干扰效率达到88% (图 3D)。显微镜观察结果显示,干扰METTL3后细胞形态变得更紧凑、立体,边缘整齐,其碱性磷酸酶活性明显高于对照组(图 3E)。qRT-PCR检测干扰METTL3的DOX-iPS中多能基因的表达,结果显示,NANOG、OCT4和LIN28A的表达水平显著高于对照组(图 3F)。

|
| 图 3 干扰METTL3对多能基因表达的调控 Figure 3 Knockdown of METTL3 regulates the expression of pluripotent genes in piPS. (A) Infecting of PEF with pSIH1 (Ctrl) and shRNA (KD) for 72 h. Scale bar=200 μm. (B) qRT-PCR analysis of METTL3 expression in PEF. (C–D) RT-PCR (C) and qRT-PCR (D) analysis of METTL3 expression in DOX-iPS. (E) Morphology and AP staining of DOX-iPS. Scale bar=100 μm. (F) qRT-PCR analysis of pluripotent genes expression in DOX-iPS. *P < 0.05; **P < 0.01; n=3. |
| |
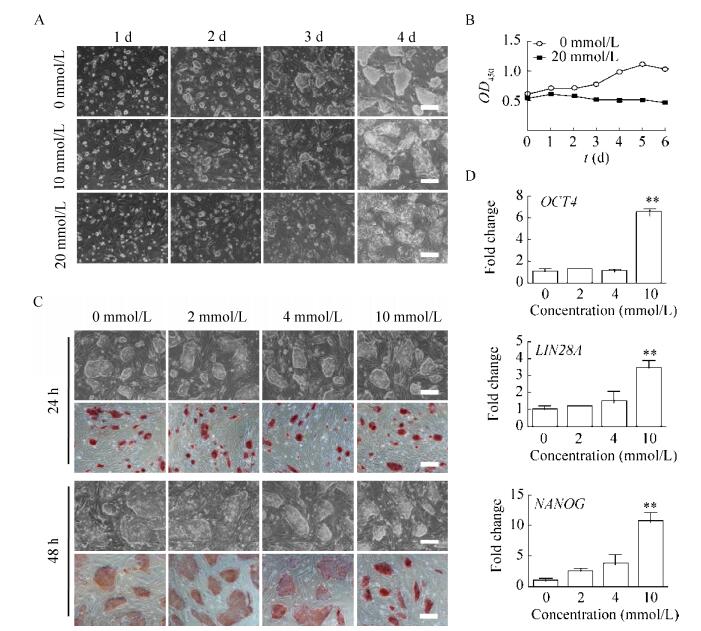
参考甲基化抑制剂环亮氨酸在小鼠ES细胞上的用量[23],分别配制了10 mmol/L和20 mmol/L的环亮氨酸培养基,用于培养DOX-iPS细胞,并观察其在接种后1–4 d的形态学变化。结果显示,DOX-iPS在20 mmol/L环亮氨酸培养基中培养,细胞克隆形态松散(图 4A),并且生长速度缓慢(图 4B)。因此,我们又配制了2 mmol/L、4 mmol/L、10 mmol/L三个浓度的环亮氨酸培养基,分别培养DOX-iPS 24 h和48 h,观察细胞形态学变化。结果显示,24 h和48 h的细胞克隆形态均未出现明显改变,其中10 mmol/L实验组的碱性磷酸酶活性强于其他组(图 4C)。qRT-PCR检测48 h处理组的多能基因表达结果显示,在10 mmol/L培养基中NANOG、OCT4和LIN28A的表达量显著升高于其他处理组(图 4D)。这一结果与干扰METTL3的实验结果相印证,添加一定浓度(10 mmol/L)环亮氨酸,使猪多能干细胞内源性多能基因的表达水平显著提高。
|
| 图 4 添加环亮氨酸对多能基因表达的调控 Figure 4 Expression of pluripotent genes in piPS treated with cycloleucine. (A) Morphology of DOX-iPS. (B) Growth curve of DOX-iPS. (C) Morphology and AP staining of DOX-iPS. (D) qRT-PCR analysis of pluripotent genes expression in DOX-iPS. **P < 0.01, n=3. Scale bar=200 μm. |
| |
目前,发现真核生物mRNA的m6A修饰是由甲基转移酶复合体催化形成的,其中,甲基转移酶METTL3广泛分布于各种组织中,与METTL14/WTAP共定位于细胞核中的核散斑,甲基转移酶METTL3、METTL14和WTAP的表达降低都会导致m6A水平的下降[11-12, 24]。
在小鼠(或人)的胚胎干细胞中,干扰Mettl3基因后,m6A水平降低,干细胞多能性相关基因Nanog、Sox2、Myc等的半衰期延长,促进干性维持[20]。Mettl3基因敲除小鼠的胚胎干细胞会始终维持在naïve阶段,naïve状态的多能因子表达水平显著升高,维持超多能的状态,在体外虽然能形成类胚体,但在随后发育过程中的分化潜力受到限制[21]。METTL3蛋白在物种之间有很强的保守性,本实验通过序列比对,也证实了这种保守性。有文献报道,在猪的细胞中,METTL3和环亮氨酸引起的m6A水平变化,与在小鼠和人的细胞中是一致的,即过表达METTL3使m6A水平升高,敲低METTL3或者添加环亮氨酸,使m6A水平下降[25]。本研究中,用构建的METTL3过表达载体和干扰载体感染piPSCs,发现过表达METTL3后,对细胞形态和多能基因(OCT4和LIN28A)表达无显著影响,我们推测,可能是由于目前获得的猪iPSCs多能基因调控网络与小鼠和人有所不同,多能基因的表达水平本身较低[26]。敲低METTL3表达后,出现类似naïve样克隆,碱性磷酸酶活性升高,多能基因NANOG、OCT4、LIN28A的表达显著升高。此结果表明,降低METTL3表达有助于维持piPSCs细胞的多能性状态。
环亮氨酸是SAM转移酶的竞争性抑制剂,猪脂肪细胞经环亮氨酸处理后显著降低mRNA的m6A甲基化水平,从而显著提高了成脂分化相关基因和脂肪酸从头合成相关基因的表达[27]。环亮氨酸还能通过降低m6A水平调控小鼠成纤维细胞的重编程过程[23]。本实验中,piPSCs在10 mmol/L环亮氨酸培养基中培养48 h后,碱性磷酸酶活性升高,多能基因NANOG、OCT4和LIN28A的表达显著升高。此结果表明,环亮氨酸可以作为辅助成分添加到培养基中,促进和维持piPSCs细胞的多能性状态。
为研究METTL3对猪干细胞中重要多能基因表达的影响,我们建立了一套表达调控检测体系。首先,克隆了猪METTL3编码区序列,设计合成了METTL3干扰片段;将上述序列和片段构建相应的慢病毒过表达载体和干扰载体,并通过PCR鉴定和测序进行确认;利用HEK293T细胞制备慢病毒颗粒,并感染多能干细胞;最后,通过过表达和敲低METTL3,观察其对多能基因表达和干细胞多能状态的影响。本研究建立的这一体系为进一步探索METTL3及m6A甲基化对猪多能干细胞的调控作用提供了基础研究手段。
综上所述,本研究表明降低METTL3表达或添加环亮氨酸能够显著提高猪干细胞内源性多能基因的表达水平,为优化猪多能干细胞培养体系提供了新的方向和实验依据,为进一步研究m6A修饰对表观遗传状态的影响建立了基础。
| [1] | Takahashi K, Yamanaka S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell, 2006, 126(4): 663–676. DOI: 10.1016/j.cell.2006.07.024 |
| [2] | Nichols J, Smith A, et al. Naive and primed pluripotent states. Cell Stem Cell, 2009, 4(6): 487–492. |
| [3] | Takahashi K, Tanabe K, Ohnuki M, et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell, 2007, 131(5): 861–872. DOI: 10.1016/j.cell.2007.11.019 |
| [4] | Yu JY, Vodyanik MA, Smuga-Otto K, et al. Induced pluripotent stem cell lines derived from human somatic cells. Science, 2007, 318(5858): 1917–1920. DOI: 10.1126/science.1151526 |
| [5] | Liu HS, Zhu FF, Yong J, et al. Generation of induced pluripotent stem cells from adult rhesus monkey fibroblasts. Cell Stem Cell, 2008, 3(6): 587–590. DOI: 10.1016/j.stem.2008.10.014 |
| [6] | Cheng D, Guo YJ, Li ZZ, et al. Porcine induced pluripotent stem cells require LIF and maintain their developmental potential in early stage of embryos. PLoS ONE, 2012, 7(12): e5177. |
| [7] | Ezashi T, Telugu BPVL, Roberts RM. Induced pluripotent stem cells from pigs and other ungulate species: an alternative to embryonic stem cells?. Reprod Domest Anim, 2012, 47(Suppl 4): 92–97. |
| [8] | Brevini T, Pennarossa G, Maffei S, et al. Pluripotency network in porcine embryos and derived cell lines. Reprod Domest Anim, 2012, 47(Suppl 4): 86–91. |
| [9] | Narayan P, Ayers DF, Rottman FM, et al. Unequal distribution of N6-methyladenosine in influenza virus mRNAs. Mol Cell Biol, 1987, 7(4): 1572–1575. DOI: 10.1128/MCB.7.4.1572 |
| [10] | Yue YN, Liu JZ, He C. RNA N6-methyladenosine methylation in post-transcriptional gene expression regulation. Genes Dev, 2015, 29(13): 1343–1355. DOI: 10.1101/gad.262766.115 |
| [11] | Liu JZ, Yue YN, Han DL, et al. A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation. Nat Chem Biol, 2014, 10(2): 93–95. DOI: 10.1038/nchembio.1432 |
| [12] | Ping XL, Sun BF, Wang L, et al. Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase. Cell Res, 2014, 24(2): 177–189. DOI: 10.1038/cr.2014.3 |
| [13] | Jia GF, Fu Y, Zhao X, et al. N6-methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO. Nat Chem Biol, 2011, 7(12): 885–887. DOI: 10.1038/nchembio.687 |
| [14] | Zheng GQ, Dahl JA, Niu YM, et al. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility. Mol Cell, 2013, 49(1): 18–29. DOI: 10.1016/j.molcel.2012.10.015 |
| [15] | Wang X, Lu ZK, Gomez A, et al. N6-methyladenosine-dependent regulation of messenger RNA stability. Nature, 2014, 505(7481): 117–120. DOI: 10.1038/nature12730 |
| [16] | Cao GC, Li HB, Yin ZN, et al. Recent advances in dynamic m6A RNA modification. Open Biol, 2016, 6(4): 160003. DOI: 10.1098/rsob.160003 |
| [17] | Du H, Zhao Y, He JQ, et al. YTHDF2 destabilizes m6A-containing RNA through direct recruitment of the CCR4-NOT deadenylase complex. Nat Commun, 2016, 7: 12626. DOI: 10.1038/ncomms12626 |
| [18] | Wang X, Feng J, Xue Y, et al. Structural basis of N6-adenosine methylation by the METTL3-METTL14 complex. Nature, 2016, 534(7608): 575–578. DOI: 10.1038/nature18298 |
| [19] | Bokar JA, Rath-Shambaugh ME, Ludwiczak R, et al. Characterization and partial purification of mRNA N6-adenosine methyltransferase from HeLa cell nuclei. Internal mRNA methylation requires a multisubunit complex. J Biol Chem, 1994, 269(26): 17697–17704. |
| [20] | Batista PJ, Molinie B, Wang JK, et al. m6A RNA modification controls cell fate transition in mammalian embryonic stem cells. Cell Stem Cell, 2014, 15(6): 707–719. DOI: 10.1016/j.stem.2014.09.019 |
| [21] | Geula S, Moshitch-Moshkovitz S, Dominissini D, et al. m6A mRNA methylation facilitates resolution of naïve pluripotency toward differentiation. Science, 2015, 347(6225): 1002–1006. DOI: 10.1126/science.1261417 |
| [22] | Rottman FM, Bokar JA, Narayan P, et al. N6-adenosine methylation in mRNA: substrate specificity and enzyme complexity. Biochimie, 1994, 76(12): 1109–1114. DOI: 10.1016/0300-9084(94)90038-8 |
| [23] | Chen T, Hao YJ, Zhang Y, et al. m6A RNA methylation is regulated by microRNAs and promotes reprogramming to pluripotency. Cell Stem Cell, 2015, 16(3): 289–301. DOI: 10.1016/j.stem.2015.01.016 |
| [24] | Dominissini D, Moshitch-Moshkovitz S, Schwartz S, et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature, 2012, 485(7397): 201–206. DOI: 10.1038/nature11112 |
| [25] | Wang XX, Zhu LN, Chen JQ, et al. mRNA m6A methylation downregulates adipogenesis in porcine adipocytes. Biochem Biophys Res Commun, 2015, 459(2): 201–207. DOI: 10.1016/j.bbrc.2015.02.048 |
| [26] | Liu YJ, Ma YY, Yang JY, et al. Comparative gene expression signature of pig, human and mouse induced pluripotent stem cell lines reveals insight into pig pluripotency gene networks. Stem Cell Rev Rep, 2014, 10(2): 162–176. DOI: 10.1007/s12015-013-9485-9 |
| [27] |
Zhu LN. Effect of FTO, METTL3 gene expression on mRNA m6A methylation and lipid metabolism in porcine subcutaneous adipocytes[D]. Hangzhou: Zhejiang University, 2014(in Chinese). 朱琳娜. FTO、METTL3基因表达对猪脂肪细胞mRNA N6-甲基腺苷水平及脂肪沉积的影响研究[D]. 杭州: 浙江大学, 2014. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2559210 |
 2018, Vol. 34
2018, Vol. 34




