中国科学院微生物研究所、中国微生物学会主办
文章信息
- 孙小宝, 万嘉欣, 曹佳雯, 斯越秀, 王谦
- Sun Xiaobao, Wan Jiaxin, Cao Jiawen, Si Yuexiu, Wang Qian
- 溶解性多糖单加氧酶的研究进展
- Progress in lytic polysaccharide monooxygenase
- 生物工程学报, 2018, 34(2): 177-187
- Chinese Journal of Biotechnology, 2018, 34(2): 177-187
- 10.13345/j.cjb.170197
-
文章历史
- Received: May 14, 2017
- Accepted: July 31, 2017
碳水化合物是自然界中重要的化学物质,是地球上生命存在的重要物质基础。木质纤维素是植物细胞壁的主要组成部分(占90%以上),主要包括纤维素、半纤维素和木质素,是自然界中分布广泛且数量极多的生物质资源。由于缺乏有效的利用方式,植物茎杆等废弃物绝大部分被焚烧,不仅造成资源浪费,而且加剧环境污染,亟需寻找对其进行综合利用的途径。然而,纤维素等多聚糖需要被降解成寡糖或单糖后才能被有效利用。目前,木质纤维素的降解方法主要有化学水解法和酶水解法。与化学法相比,酶水解法具有反应条件温和、能耗低、环境友好等优点,因而受到广泛关注。利用微生物产生的糖苷水解酶(Glycosyl hydrolase, GH)分解和转化木质纤维素,是生物质利用的有效途径,近年来对于糖苷水解酶的研究取得了很大进展[1]。从化学反应过程来看,GH水解纤维素生成葡萄糖的过程主要涉及α/β-1, 4-糖苷键的断裂,很难对结构更加复杂的木质纤维素进行有效降解。一方面,由于木质纤维素物理结构复杂,相邻的多糖链之间可通过氢键形成高度有序的结晶结构,许多天然的纤维素还包埋在木质素和半纤维素中,这些复杂的结构使木质纤维素的降解难度增加;另一方面,由于GH酶活性低下、不耐高温、不耐酸碱或反应过程中存在抑制剂等因素,极大制约了其规模化的应用。此外,植物细胞壁中的纤维素、半纤维素、木质素等组分呈复杂的交联结构,单一水解酶很难有效分解。
最新研究表明,最初被碳水化合物活性酶数据库(Carbonhydrate-activity enzymes database,CAZy)划分为糖苷水解酶61家族(GH61)和碳水化合物结合域33家族(Carbohydrate-binding module family 33,CBM33)的一些成员具有多糖单加氧酶的活性[2-3]。这一重大发现改变了传统酶法降解结晶多糖的模式,也使得GH61成为了木质纤维素资源开发研究的新热点,这些基因被称为溶解性多糖单加氧酶(Lytic polysaccharide monooxygenase, LPMO/PMO)。
传统的木质纤维素降解过程主要是在外切葡聚糖酶、内切葡聚糖酶、纤维二糖酶等一系列水解酶的作用下,通过打断糖苷键的方式来降解木质纤维素。与传统GH水解多聚糖链的方式不同,LPMO能够通过氧化作用断裂糖苷键产生寡糖链,暴露更多糖苷水解酶结合的位点,从而加快反应进程,提高寡糖或单糖的生成量(图 1)[4]。因此,LPMO是一种全新的生物质降解酶,改变并进一步丰富了生物质的降解模式。

|
| 图 1 LPMO参与纤维素降解的工作模型 Figure 1 LPMO participates in a working model of the degradation of cellulose |
| |
LPMO广泛存在于细菌与真菌中。早在20世纪80年代,研究人员在粘质沙雷氏菌Serratia marcescens中发现了一种分子量较小(约21 kDa)的CBM33家族蛋白[5]。到了90年代,Suzuki等[6]发现它对β-几丁质有很强的结合力,因此命名为几丁质结合蛋白(Chitin binding protein,CBP21)。2010年,Vaaje-Kolstad等[7-8]进一步通过对粘质沙雷氏菌来源的CBP21研究发现,该蛋白是一种氧化酶,在氧和电子供体的存在下,能够随机氧化裂解几丁质的多糖链。并且,CBP21是一种金属依赖性酶,通过添加金属螯合剂EDTA和突变金属结合位点,会引起蛋白失活。进一步研究显示,CBP21活性可能与铜离子等二价金属离子密切相关[9]。由于其具有一定的内切葡聚糖酶活性,CBP21最初被划分为GH61家族。
2011年,研究人员发现来自太瑞斯梭壳孢霉Thielavia terrestris的GH61E和嗜热子囊菌Thermoascus aurantiacus的GH61A对木质纤维素和传统的糖苷酶底物均无水解作用。但将太瑞斯梭壳孢霉的发酵液与等量的瑞氏木霉Trichoderma reesei复合纤维素酶混合后,纤维素降解效果较好,糖转化的效率明显提高。即使复合纤维素酶的总量减少一半,仍能保证相同的水解效果。进一步通过比较太瑞斯梭壳孢霉和瑞氏木霉胞外纤维素酶系的差别,发现3个可能与糖转化相关的胞外酶GH61B、GH61E和GH61G。它们并不属于GH61家族,但可以显著提高纤维素酶对经过预处理的玉米秸秆的水解作用[10]。直到2011年底,这些酶才被正式命名为LPMO/PMO。此后,更多的研究证明这些酶并不是真正的糖苷水解酶,而属于氧化酶类[11]。
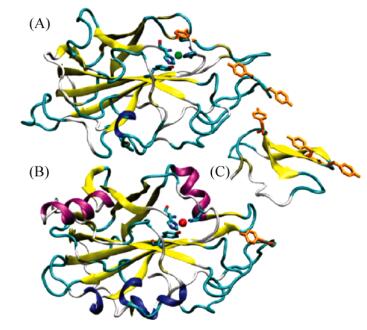
2 LPMO的结构与催化机制研究人员利用多重同晶置换(Multiple isomorphous replacement,MIR)获得太瑞斯梭壳孢霉的GH61E的三维结构(图 2)。GH61E是一个结构致密、含有两个β折叠所组成的三明治结构,同时具有金属离子的结合位点。在金属离子存在的情况下,LPMO才能发挥它的氧化功能。GH61E和TrGH61B蛋白结构在靠近蛋白N末端都有3个高度保守的组氨酸,这些氨基酸可能与金属离子结合、传递电子过程有着重要联系[10]。
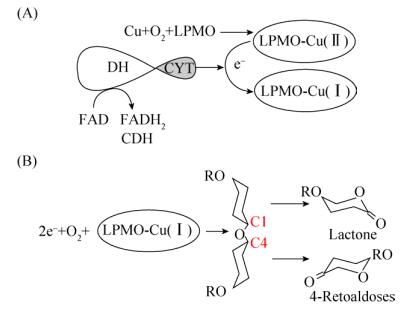
目前,对于LPMO确切的作用机制仍不清楚。通常认为,在氧分子和电子供体存在时,LPMO可氧化断裂多聚糖主链的β-1, 4糖苷键,释放出寡糖,用于后续的进一步降解。电子供体通常为小分子还原剂,如抗坏血酸和没食子酸或纤维二糖脱氢酶(Cellobiose dehydrogenase,CDH)等酶类还原剂[12]。研究人员利用电子顺磁共振(Electron paramagnetic resonance,EPR)对LPMO的功能位点构象和动力学特性进行分析,发现LPMO的催化过程需要二价金属离子(如Cu2+、Co2+、Fe2+等)才能发挥作用释放出寡糖[7, 13-15]。进一步研究显示,LPMO是通过铜离子在Cu+与Cu2+之间的转变来催化底物(图 3)[16-18]。CDH蛋白含有两个结构域,分别是结合黄素腺嘌呤二核苷酸(Flavin adenine dinucleotide,FAD)的脱氢酶结构域(Dehydrogenase domain,DH)和结合细胞色素结构域(Cytochrome domain,CYT)。催化过程中,FAD被还原成FADH2产生电子,与此同时,在有氧环境中LPMO与Cu离子结合,形成LPMO-Cu(Ⅱ)复合物。CDH产生的电子可将LPMO-Cu(Ⅱ)复合物转化成LPMO-Cu(Ⅰ)复合物(图 3A),该复合物随后与氧分子结合,氧分子的电子转移给Cu从而形成Cu的过氧化物中间体。该中间体能够从多糖链内部的葡萄糖单体C1或C4位置夺取1个氢原子,形成碳水化合物多糖的自由基和过氧化氢铜的中间体。CDH的电子会使中间体的O-O键发生断裂,释放出水分子并形成铜氧自由基,与之前形成的自由基发生偶合,在C1或C4位置生成羟基化的多糖,最终生成醛酮糖或者糖内酯(图 3B)[12-13, 19]。
|
| 图 3 LPMO反应过程中经电子供体介导Cu+与Cu2+互相转化的催化机制 Figure 3 Bioconversion between Cu+ and Cu2+ during LPMO catalysis via electron donor |
| |
LPMO的氧化作用可发生在多聚糖糖环上C1、C4和C6位,其中以C1和(或) C4较为常见。2014年,Forsberg等[20]发现来自天蓝色链霉菌Streptomyces coelicolor的ScLPMO10B的氧化作用发生在C1和C4位。因此,根据LPMO催化机制的不同,可以将其大致分为PMO-1型、PMO-2型和PMO-3型3类。其中PMO-1型作用于C1,从多糖链葡萄糖单体的C1夺取氧原子产生内酯型糖,接着内酯型的糖被转化为醛糖酸,该醛酮糖被磷酸化之后,通过磷酸戊糖途径被代谢;PMO-2型作用于C4,从多糖链葡萄糖单体的C4夺取氢原子生产非还原端被氧化的醛酮糖;PMO-3型作用于C1或C4位[21],但由于C4和C6催化产物具有相同的分子量,多数研究者对于是否发生C6氧化仍存在争议,需要进一步实验验证。
基于LPMO的氧化作用和糖苷水解酶的水解作用,LPMO与不同糖苷水解酶(如葡聚糖酶、木聚糖酶、纤维二糖脱氢酶等)协同作用时,可显著提高木质纤维素的转化效果。前期工作中,我们将来自枯草芽孢杆菌Bacillus subtilis 的LPMO与甘露聚糖酶协同催化底物,发现可提高甘露聚糖底物的转化效率(数据尚未发表)。反应过程中,LPMO以氧化方式打断多糖长链,释放出更多还原端,与糖苷水解酶协同、发挥加成作用,大幅度提高酶解效率[22]。如图 1所示,内切葡聚糖和外切葡聚糖酶单独作用时,很难水解纤维素的结晶区;当与LPMO协同作用时,LPMO在电子供体CDH及金属离子存在下,随机断裂纤维素结晶区间的糖苷键,暴露出更多还原端,有助于内切葡聚糖和外切葡聚糖酶结合在纤维素链的结晶区上,从而加快纤维素的降解速率(图 1)。
3 LPMO的分类根据LPMO催化糖环上的位点不同,可大致将其分为3类:PMO-1、PMO-2和PMO-3型。但这种方式不能对LPMO进行合理分类,甚至有些酶可同时归为PMO-1型和PMO-3型,从而产生混淆。随着更多深入研究的开展,研究人员发现不同家族的LPMO不仅来源不同,其对底物的选择性也不相同。部分LPMO同时还具有纤维素的降解能力[23]、几丁质的降解能力[24]或淀粉的降解能力[22]。
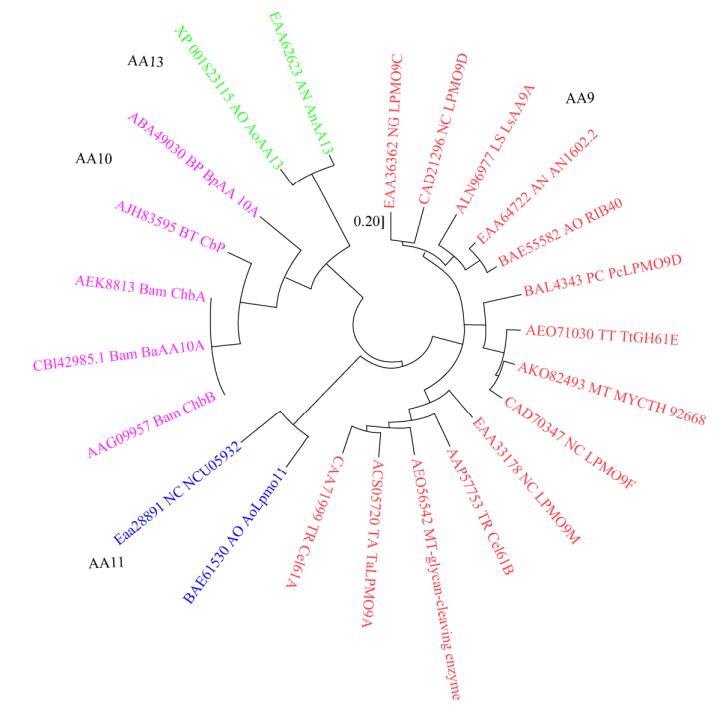
2013年,Levasseur等[3]通过系统分析CAZy数据库中的LPMO蛋白来源和底物类型等信息,将LPMO重新划分为辅助酶类家族(Auxiliary activity,AA)。系统发育树分析也表明,总体聚类形成4个基因簇,分别为AA9、AA10、AA11和AA13家族(图 4)。其中,AA9、AA11和AA13家族LPMO主要在真菌中发现。AA10家族则是以细菌为主,但也含有少量真核生物和病毒。Meera等[25]从烟曲霉MKU1克隆得到的AA9家族蛋白对羧甲基纤维素钠底物催化活性最高,达到0.549 IU/mg。而来自奇迹束丝放线菌Actinosynnema mirum DSM 43827的Am5属于AA10家族,对几丁质有氧化活性且吸附作用较强,但对纤维素无催化能力且吸附能力较弱[26]。研究显示,AA10家族的LPMO对不同形态的同一底物,结合能力也不相同。如来自苏云金芽孢杆菌Bacillus thuringiensis的BtLPMO10A[27]、灰色链霉菌Streptomyces griseus的SgLPMO10F[28]以及李斯特氏菌Listeria monocytogenes的LmLPMO10[29]对α和β-几丁质均具有很强的吸附能力,对微晶纤维素、胶体几丁质(Colloidal chitin)和几丁质珠(Chitin beads)吸附能力较弱。值得注意的是,LmLPMO10对纤维素的吸附能力也很强。
|
| 图 4 LPMOs系统发育树分析 Figure 4 Phylogenetic tree analysis of LPMOs |
| |
综上所述,AA9家族LPMO作用的底物主要为纤维素。AA10和AA11家族LPMO作用的底物主要为几丁质,也有少数成员可催化纤维素[30]。AA13家族LPMO可催化淀粉降解(表 1)。此外,原来的GH61家族和CBM33家族蛋白分别被划为AA9和AA10家族。氨基酸序列比对发现,所有LPMO成熟肽序列第一个氨基酸都含有一个完全保守的组氨酸。同时,第二个组氨酸结合中心附近存在相对保守的序列排布规律。其中,AA9家族为H-X8-Q/E-X-Y,AA10家族为R-X4-E-X-F或H-X2-Q-X-Y,AA11家族为N-X-E-X-Y,AA13家族为Q-X2-Q-X-Y (图 5)[19]。
| Subfamily | Organism | Names | Substrates | Positions oxidized |
PDB | References |
| AA9 | Aspergillus fumigatus | Endoglucanase/CMCase (Eng61) |
Cellulose; CMC |
ND | / | [25] |
| Aspergillus nidulans | Endo-β-1, 4-glucanase (AN1602.2) |
/ | ND | / | [31] | |
| Aspergillus oryzae | Aspergillus oryzae RIB40 | / | ND | / | [32] | |
| Myceliophthora thermophila |
β-glycan-cleaving enzyme(StCel61a; MYCTH_46583)(Cel61A) |
/ | ND | / | [32] | |
| MYCTH_92668 | Cellulose | ND | / | [33] | ||
| Neurospora crassa | LPMO9D; LPMO9M | Cellulose | ND | 4EIR[A, B], 4EIS[A, B] |
[15] | |
| LPMO9C | Cellulose; cellooligosaccharides |
C1, C4 or both |
4D7U[A, B] 4D7V[A, B] |
[23] | ||
| Trichoderma reesei | GH61D; PcGH61D; PcLPMO9D |
Cellulose | C1 | 4B5Q[A, B] | [34-35] | |
| Cel61B=GH61B | Cellulose | C1, C4 | 2VTC[A, B] | [36] | ||
| AA10 | Bacillus | ChbB | Chitin | ND | / | [37] |
| amyloliquefaciens | BAXH7_01677 (ChbA) | Chitin | ND | / | [38] | |
| ChbB; BaAA10A; BaCBM33; Rbam17540 |
Cellulose | C1 | 2YOW[A, B] 2YOX[A, B] 2YOY[A, B] |
[39] | ||
| Bacillus thuringiensis | BF36_2236 (CbP) | GlcNAc | ND | / | [40] | |
| AA11 | Aspergillus oryzae | AoLpmo11 | Chitin | ND | 4MAH[A] 4MAI[A] |
[41] |
| AA13 | Aspergillus nidulans | AnAA13; AN5463.2 | Starch | ND | / | [31] |
| Aspergillus oryzae | AoAA13 | Starch | C1 | 4OPB[A] | [42] | |
| ND: not determined. | ||||||

|
| 图 5 LPMOs氨基酸序列比对 Figure 5 Amino acid sequence alignment of LPMOs |
| |
糖苷水解酶活性测定的方法主要通过测定产物的生量成以及底物的消耗量来实现的。然而,LPMO是具有氧化活性的酶,直接催化底物降解的效率并不高,因此很难实现直接测定寡糖生成量来实现酶活测定。目前,测定LPMO活性的方法主要包括以下几种。
4.1 DNS法将LPMO与糖苷水解酶协同作用纤维素底物,与DNS试剂混合后加热显色,测定还原糖的OD540吸光值,通过对比单独测活以及协同测活以产物的提高量来判定LPMO活性。该方法操作简便,成本低,但灵敏度较低,只能检测到还原性糖,对于部分产物如一些非还原性的寡糖则无法检测。此外,LPMO微弱的活性容易被背景干扰。
4.2 色谱/质谱法采用电喷雾-飞行时间/质谱(Electrospray ionization-time-of-flight-mass spectrometry,ESI-TOF/MS)正离子模式对产物进行分析鉴定,可与糖苷水解酶协同反应后检测寡糖生成量。如LPMO (Am5)、4 μmol/L抗坏血酸与几丁质酶协同降解几丁质,在37 ℃下反应24 h,产物为几丁四糖酸、几丁五糖酸和几丁六糖酸,可提高几丁质酶60%的水解效率[26]。Forsberg等[43]将ScLPMO10B在2 mmol/L抗坏血酸、20 mmol/L醋酸铵(pH 6)缓冲液中单独反应以及与纤维素酶CelS2协同作用水解纤维素底物,反应8-16 h,与单独反应相比,协同作用的水解效率明显提高6倍左右,并通过高效离子色谱(High performance anion exchange chromatography,HPAEC)对产物进行分析,产物中成功分离出聚合度为2-8的纤维寡糖,并通过质谱成功鉴定其氧化位点是C4位。Westereng等[24]通过超高效液相色谱(Ultra-high-performance liquid chromatography,UHPLC),在紫外检测器下测得产物聚合度为4-10的纤维寡糖。该方法可以高效且快速地对产物含量、结构进行分析,适用于对极微量寡糖的检测,分辨率与灵敏度高。虽然色谱/质谱法操作较为复杂,且需要专门的工作站,但目前仍是最为有效、广泛采用的方法。
4.3 荧光分析法针对LPMO测活较为困难的现状,Kittl等[44]提出了一种基于Amplex Red试剂的荧光分析法。Amplex Red是一种良好的过氧化物酶荧光底物,是高度灵敏和稳定的H2O2探针。LPMO与O2在CDH、抗坏血酸或纤维二糖提供电子的情况下形成H2O2,当反应体系中存在辣根过氧化物酶(Horse radish peroxidase,HRP)时,Amplex Red试剂与H2O2以1:1定量比反应,形成高荧光的试卤灵(Resorufin),可用于定量分析。该方法灵敏度很高,但稳定性较差,所以在测定LPMO活性上并没有被广泛应用。
5 LPMO的应用 5.1 饲料添加剂谷实类籽粒细胞壁主要由非淀粉多糖(Non-starch polysaccharide, NSP)组成。NSP是除淀粉以外的多糖类物质,是谷物类饲料中一种主要的抗营养因子,其抗营养作用主要体现在溶于水后具有高度粘性。在牛、羊等反刍动物的瘤胃中存在大量能催化木质纤维素降解的瘤胃细菌、原虫和真菌,通过酶的催化使纤维素和半纤维素分解为瘤胃能够吸收的小分子物质[45]。然而,家禽等单胃动物体内不能产生降解NSP的酶类,动物采食后,会增加肠道食糜的粘度,降低胃肠道运动对食糜的混合效率,从而影响消化酶与底物接触和消化产物向小肠上皮绒毛渗透扩散,阻碍饲料消化和养分吸收[46]。此外,NSP还会引起肠粘膜形态和功能的变化,导致雏禽胰腺肿大[47]。因此,在饲料中添加LPMO与糖苷水解酶可有效降解NSP,消除其抗营养作用。同时,生成功能性低聚寡糖等益生元,减少家畜胃肠道病害,促进动物健康生长。
5.2 功能性食品低聚寡糖是指含有2-10个单糖通过糖苷键聚合而成的化合物,广泛参与生命体的生理生化反应,如促进机体的健康发育、降低龋齿,能部分或全部替代口香糖、糕点和糖果中的糖类[48]。大多数功能性寡糖不被人体消化吸收,因此糖尿病和高血压等慢性疾病患者均可以食用。
寡糖可作为底物被肠道内的微生物发酵利用从而降低了肠道内pH值,促进肠道屏障功能、营养吸收和免疫能力,减少肠道病原微生物。在饲粮中添加0.2%纤维寡糖可使生长猪平均日重提高7.51% (P < 0.05),料重降低5.93% (P < 0.05)[49]。研究表明,壳寡糖能显著促进生长期大鼠胰岛β细胞的生长和胰岛素的释放,为糖尿病患者的治疗提供新的途径[50]。糖苷水解酶可通过水解多聚糖生成低聚寡糖,额外加入LPMO与糖苷水解酶协同作用,可进一步提升寡糖的生产效率,在功能性食品领域具有较好的应用前景。
5.3 生物能源木质纤维素乙醇是指以林业如灌木、乔木枝叶干等森林废弃物或农副产品秸秆、玉米芯、中药渣等为原料,通过木质纤维素的降解发酵等工艺生产生物乙醇[51-52]。生物乙醇的转化主要包括纤维素降解成单糖和单糖生物发酵两个阶段。其中,催化多聚糖生成可发酵单糖是生物乙醇制备的关键步骤,利用高效的纤维素降解菌或添加高效纤维素酶是提高木质纤维素水解效率的常用手段[53]。将LPMO应用于单糖的生物转化,发挥LPMO的氧化作用,协同糖苷水解酶的水解作用,是高效降解木质纤维素生成单糖、进一步糖酵解途径生产生物乙醇的有效途径。
6 结语木质纤维素因其组分的异质性和结构的复杂性,只有将其降解成寡糖或单糖才能更好地发挥作用。LPMO以氧化方式作用于多聚糖长链,丰富了木质纤维素的降解模式。当与糖苷水解酶协同作用时,可加速降解底物的降解效率,如何最大程度地开发其商业应用潜力成为新的挑战。目前,丹麦诺维信公司尝试将LPMO加入到复合纤维素酶Ce11icCTec3中,使纤维素乙醇生产成本降到约2美元/加仑,接近淀粉酒精和汽油的生产成本。这为利用LPMO高效降解木质纤维素、进一步开发生物乙醇提供了良好示范。
| [1] | Shimizu M, Kaneko Y, Ishihara S, et al. Novel β-1, 4-mannanase belonging to a new glycoside hydrolase family in Aspergillus nidulans. J Biol Chem, 2015, 290(46): 27914–27927. DOI: 10.1074/jbc.M115.661645 |
| [2] | Cantarel BL, Coutinho PM, Rancurel C, et al. The Carbohydrate-Active EnZymes database (CAZy): an expert resource for glycogenomics. Nucleic Acids Res, 2009, 37(S1): D233–D238. |
| [3] | Levasseur A, Drula E, Lombard V, et al. Expansion of the enzymatic repertoire of the CAZy database to integrate auxiliary redox enzymes. Biotechnol Biofuels, 2013, 6: 41. DOI: 10.1186/1754-6834-6-41 |
| [4] | Hemsworth GR, Johnston EM, Davies GJ, et al. Lytic polysaccharide monooxygenases in biomass conversion. Trends Biotechnol, 2015, 33(12): 747–761. DOI: 10.1016/j.tibtech.2015.09.006 |
| [5] | Fuchs RL, McPherson SA, Drahos DJ. Cloning of a Serratia marcescens gene encoding chitinase. Appl Environ Microb, 1986, 51(3): 504–509. |
| [6] | Suzuki K, Suzuki M, Taiyoji M, et al. Chitin binding protein (CBP21) in the culture supernatant of Serratia marcescens 2170. Biosci Biotech Biochem, 1998, 62(1): 128–135. DOI: 10.1271/bbb.62.128 |
| [7] | Vaaje-Kolstad G, Westereng B, Horn SJ, et al. An oxidative enzyme boosting the enzymatic conversion of recalcitrant polysaccharides. Science, 2010, 330(6001): 219–222. DOI: 10.1126/science.1192231 |
| [8] | Vaaje-Kolstad G, Bøhle LA, Gåseidnes S, et al. Characterization of the chitinolytic machinery of Enterococcus faecalis V583 and high-resolution structure of its oxidative CBM33 enzyme. J Mol Biol, 2012, 416(2): 239–254. DOI: 10.1016/j.jmb.2011.12.033 |
| [9] | Aachmann FL, Sørlie M, Skjåk-Bræk G, et al. NMR structure of a lytic polysaccharide monooxygenase provides insight into copper binding, protein dynamics, and substrate interactions. Proc Natl Acad Sci USA, 2012, 109(46): 18779–18784. DOI: 10.1073/pnas.1208822109 |
| [10] | Harris PV, Welner D, McFarland KC, et al. Stimulation of lignocellulosic biomass hydrolysis by proteins of glycoside hydrolase family 61: structure and function of a large, enigmatic family. Biochemistry, 2010, 49(15): 3305–3316. DOI: 10.1021/bi100009p |
| [11] | Moses V, Hatherley R, Bishop ÖT. Bioinformatic characterization of type-specific sequence and structural features in auxiliary activity family 9 proteins. Biotechnol Biofuels, 2016, 9: 239. DOI: 10.1186/s13068-016-0655-2 |
| [12] | Beeson WT, Vu VV, Span EA, et al. Cellulose degradation by polysaccharide monooxygenases. Annu Rev Biochem, 2015, 84: 923–946. DOI: 10.1146/annurev-biochem-060614-034439 |
| [13] | Tan TC, Kracher D, Gandini R, et al. Structural basis for cellobiose dehydrogenase action during oxidative cellulose degradation. Nat Commun, 2015, 6: 7542. DOI: 10.1038/ncomms8542 |
| [14] | Gudmundsson M, Kim S, Wu M, et al. Structural and electronic snapshots during the transition from a Cu(Ⅱ) to Cu(Ⅰ) metal center of a lytic polysaccharide monooxygenase by X-ray photoreduction. J Biol Chem, 2014, 289(27): 18782–18792. DOI: 10.1074/jbc.M114.563494 |
| [15] | Quinlan RJ, Sweeney MD, Leggio LL, et al. Insights into the oxidative degradation of cellulose by a copper metalloenzyme that exploits biomass components. Proc Natl Acad Sci USA, 2011, 108(37): 15079–15084. DOI: 10.1073/pnas.1105776108 |
| [16] | Kjaergaard CH, Qayyum MF, Wong SD, et al. Spectroscopic and computational insight into the activation of O2 by the mononuclear Cu center in polysaccharide monooxygenases. Proc Natl Acad Sci USA, 2014, 111(24): 8797–8802. DOI: 10.1073/pnas.1408115111 |
| [17] | Phillips CM, Beeson IV WT, Cate JH, et al. Cellobiose dehydrogenase and a copper-dependent polysaccharide monooxygenase potentiate cellulose degradation by Neurospora crassa. ACS Chem Biol, 2011, 6(12): 1399–1406. DOI: 10.1021/cb200351y |
| [18] | Beeson WT, Phillips CM, Cate JHD, et al. Oxidative cleavage of cellulose by fungal copper-dependent polysaccharide monooxygenases. J Am Chem Soc, 2012, 134(2): 890–892. DOI: 10.1021/ja210657t |
| [19] | Eibinger M, Ganner T, Bubner P, et al. Cellulose surface degradation by a lytic polysaccharide monooxygenase and its effect on cellulase hydrolytic efficiency. J Biol Chem, 2014, 289(52): 35929–35938. DOI: 10.1074/jbc.M114.602227 |
| [20] | Forsberg Z, Røhr ÅK, Mekasha S, et al. Comparative study of two chitin-active and two cellulose-active AA10-type lytic polysaccharide monooxygenases. Biochemistry, 2014, 53(10): 1647–1656. DOI: 10.1021/bi5000433 |
| [21] | Li X, Beeson IV WT, Phillips CM, et al. Structural basis for substrate targeting and catalysis by fungal polysaccharide monooxygenases. Structure, 2012, 20(6): 1051–1061. DOI: 10.1016/j.str.2012.04.002 |
| [22] | Vu VV, Beeson WT, Phillips CM, et al. Determinants of regioselective hydroxylation in the fungal polysaccharide monooxygenases. J Am Chem Soc, 2014, 136(2): 562–565. DOI: 10.1021/ja409384b |
| [23] | Borisova AS, Isaksen T, Dimarogona M, et al. Structural and functional characterization of a lytic polysaccharide monooxygenase with broad substrate specificity. J Biol Chem, 2015, 290(38): 22955–22969. DOI: 10.1074/jbc.M115.660183 |
| [24] | Westereng B, Arntzen MØ, Agger JW, et al. Analyzing activities of lytic polysaccharide monooxygenases by liquid chromatography and mass spectrometry//Abbott D, Lammerts van Bueren A, Eds. Protein-Carbohydrate Interactions: Methods in Molecular Biology. New York, NY; Humana Press, 2017: 71-92. |
| [25] | Meera B, Vanitha MC, Ramani G, et al. Cloning and expression of an intronless gene encoding endoglucanase from Aspergillus fumigatus MKU1. Indian J Biotechnol, 2011, 10(4): 480–486. |
| [26] |
Shi XW, Zhang WT, Zhang XF, et al. The heterologous expression and characterization of lytic polysaccharide monooxygenase from Actinosynnema mirum DSM 43827.
China Biotechnol, 2014, 34(7): 17–23.
(in Chinese). 施贤卫, 张伟涛, 张小飞, 等. Actinosynnema mirum DSM43827溶解性多糖单加氧酶的异源表达和酶学性质表征. 中国生物工程杂志, 2014, 34(7): 17-23. |
| [27] | Zhang HY, Zhao Y, Cao HL, et al. Expression and characterization of a lytic polysaccharide monooxygenase from Bacillus thuringiensis. Int J Biol Macromol, 2015, 79: 72–75. DOI: 10.1016/j.ijbiomac.2015.04.054 |
| [28] | Nakagawa YS, Kudo M, Loose JSM, et al. A small lytic polysaccharide monooxygenase from Streptomyces griseus targeting α-and β-chitin. FEBS J, 2015, 282(6): 1065–1079. DOI: 10.1111/febs.13203 |
| [29] | Paspaliari DK, Loose JSM, Larsen MH, et al. Listeria monocytogenes has a functional chitinolytic system and an active lytic polysaccharide monooxygenase. FEBS J, 2015, 282(5): 921–936. DOI: 10.1111/febs.13191 |
| [30] |
Zhang HY. Expression and characterization of a novel lytic polysaccharide monooxygenases[D]. Dalian: Dalian Polytechnic University, 2015 (in Chinese). 张卉妍. 一种新型多糖裂解单加氧酶的克隆表达及其性质研究[D]. 大连: 大连工业大学, 2015. |
| [31] | Galagan JE, Calvo SE, Cuomo C, et al. Sequencing of Aspergillus nidulans and comparative analysis with A. fumigatus and A. oryzae. Nature, 2005, 438(7071): 1105–1115. DOI: 10.1038/nature04341 |
| [32] | Machida M, Asai K, Sano M, et al. Genome sequencing and analysis of Aspergillus oryzae. Nature, 2005, 438(7071): 1157–1161. DOI: 10.1038/nature04300 |
| [33] | Berka RM, Grigoriev IV, Otillar R, et al. Comparative genomic analysis of the thermophilic biomass-degrading fungi Myceliophthora thermophila and Thielavia terrestris. Nat Biotechnol, 2011, 29(10): 922–927. DOI: 10.1038/nbt.1976 |
| [34] | Westereng B, Ishida T, Vaaje-Kolstad G, et al. The putative endoglucanase PcGH61D from Phanerochaete chrysosporium is a metal-dependent oxidative enzyme that cleaves cellulose. PLoS ONE, 2011, 6(11): e27807. DOI: 10.1371/journal.pone.0027807 |
| [35] | Wu M, Beckham GT, Larsson AM, et al. Crystal structure and computational characterization of the lytic polysaccharide monooxygenase GH61D from the Basidiomycota fungus Phanerochaete chrysosporium. J Biol Chem, 2013, 288(18): 12828–12839. DOI: 10.1074/jbc.M113.459396 |
| [36] | Karkehabadi S, Hansson H, Kim S, et al. The first structure of a glycoside hydrolase family 61 member, Cel61B from Hypocrea jecorina, at 1.6 Åresolution. J Mol Biol, 2008, 383(1): 144–154. DOI: 10.1016/j.jmb.2008.08.016 |
| [37] | Chu HH, Hoang V, Hofemeister J, et al. A Bacillus amyloliquefaciens ChbB protein binds β-and α-chitin and has homologues in related strains. Microbiology, 2001, 147(7): 1793–1803. DOI: 10.1099/00221287-147-7-1793 |
| [38] | Yang HL, Liao YL, Wang B, et al. Complete genome sequence of Bacillus amyloliquefaciens XH7, which exhibits production of purine nucleosides. J Bacteriol, 2011, 193(19): 5593–5594. DOI: 10.1128/JB.05880-11 |
| [39] | Hemsworth GR, Taylor EJ, Kim RQ, et al. The copper active site of CBM33 polysaccharide oxygenases. J Am Chem Soc, 2013, 135(16): 6069–6077. DOI: 10.1021/ja402106e |
| [40] | Johnson SL, Daligault HE, Davenport KW, et al. Complete genome sequences for 35 biothreat assay-relevant Bacillus species. Genome Announc, 2015, 3(2): e00151–15. |
| [41] | Hemsworth GR, Henrissat B, Davies GJ, et al. Discovery and characterization of a new family of lytic polysaccharide monooxygenases. Nat Chem Biol, 2014, 10(2): 122–126. DOI: 10.1038/nchembio.1417 |
| [42] | Leggio LL, Simmons TJ, Poulsen JCN, et al. Structure and boosting activity of a starch-degrading lytic polysaccharide monooxygenase. Nat Commun, 2015, 6: 5961. DOI: 10.1038/ncomms6961 |
| [43] | Forsberg Z, Mackenzie AK, Sørlie M, et al. Structural and functional characterization of a conserved pair of bacterial cellulose-oxidizing lytic polysaccharide monooxygenases. Proc Natl Acad Sci USA, 2014, 111(23): 8446–8451. DOI: 10.1073/pnas.1402771111 |
| [44] | Kittl R, Kracher D, Burgstaller D, et al. Production of four Neurospora crassa lytic polysaccharide monooxygenases in Pichia pastoris monitored by a fluorimetric assay. Biotechnol Biofuels, 2012, 5: 79. DOI: 10.1186/1754-6834-5-79 |
| [45] | Zebeli Q, Aschenbach JR, Tafaj M, et al. Invited review: role of physically effective fiber and estimation of dietary fiber adequacy in high-producing dairy cattle. J Dairy Sci, 2012, 95(3): 1041–1056. DOI: 10.3168/jds.2011-4421 |
| [46] |
Gong M. Effect of NSP in wheat on intestinal microflora and sodium/glucose cotransporters in weaned piglets[D]. Nanchang: Nanchang University, 2011 (in Chinese). 龚敏. 小麦非淀粉多糖对断奶仔猪肠道菌群区系及糖转运载体的影响研究[D]. 南昌: 南昌大学, 2011. http://cdmd.cnki.com.cn/Article/CDMD-11902-1012290859.htm |
| [47] |
Wang DQ. Effects of different non-starch polysaccharides enzymes preparations on performance and digestive physiology of wheat-based or corn-based Diets for Broilers[D]. Ya'an: Sichuan Agricultural University, 2013 (in Chinese). 王冬群. 玉米型和小麦型饲粮中添加非淀粉多糖复合酶对肉鸡生产性能和消化道生理的影响[D]. 雅安: 四川农业大学, 2013. |
| [48] |
Li XT, Qiu H, Yin SL, et al. The improvement of vifidus milk powder contatining Bifidobacterium Breve on intestinal environment of children.
Chin J Microecol, 2000, 12(3): 180–183.
(in Chinese). 李雪驼, 邱华, 殷素兰, 等. 双歧杆菌奶粉对幼儿肠道内环境影响. 中国微生态学杂志, 2000, 12(3): 180-183. |
| [49] |
Xu LR, Luan ZS, Hu CH, et al. Effects of dietary cello-oligosaccharide on growth performance, colonic microflora and intestinal mucosal permeability of growing pigs.
Chin J Anim Nutr, 2013, 25(6): 1293–1298.
(in Chinese). 徐露蓉, 栾兆双, 胡彩虹, 等. 饲粮中添加纤维寡糖对生长猪生长性能、结肠菌群和肠黏膜通透性的影响. 动物营养学报, 2013, 25(6): 1293-1298. |
| [50] |
Liu B. Antidiabetic effects of chitooligosaccharides and it's derivatives[D]. Qingdao: Ocean University of China, 2007 (in Chinese). 刘冰. 壳寡糖及其配合物对糖尿病的作用研究[D]. 青岛: 中国海洋大学, 2007. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y1112174 |
| [51] |
Xiong XK. Energy potential of cellulosic ethanol for Robinia pseudoacacia plantation in Henan Province[D]. Beijing: Beijing Forestry University, 2016 (in Chinese). 熊小康. 河南洛宁刺槐人工林纤维素乙醇能源潜力研究[D]. 北京: 北京林业大学, 2016. |
| [52] |
Jiang N. Catalytic conversion of lignocellulosic biomass into biofuels and high-value chemicals[D]. Tianjin: Tianjin University, 2014 (in Chinese). 姜楠. 木质纤维素制备生物质能源与生物基化学品的研究[D]. 天津: 天津大学, 2014. |
| [53] |
Ma J. Screening efficient degradation of cellulose strains and preliminary study on utilizing straw to ferment ethanol[D]. Wuhan: Huazhong Agricultural University, 2008 (in Chinese). 马洁. 筛选高效纤维素降解菌及利用秸秆发酵酒精初步研究[D]. 武汉: 华中农业大学, 2008. |
 2018, Vol. 34
2018, Vol. 34