中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李晓丽, 涂涛, 姚斌, 谢响明, 罗会颖
- Li Xiaoli, Tu Tao, Yao Bin, Xie Xiangming, Luo Huiying
- 嗜热子囊菌JCM12803来源的双功能木聚糖/纤维素酶
- A novel bifunctional xylanase/cellulase TcXyn10A from Thermoascus crustaceus JCM12803
- 生物工程学报, 2018, 34(12): 1996-2006
- Chinese Journal of Biotechnology, 2018, 34(12): 1996-2006
- 10.13345/j.cjb.180067
-
文章历史
- Received: February 22, 2018
- Accepted: May 18, 2018
2 中国农业科学院 饲料研究所,北京 100081
2 Feed Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081, China
近年来,可持续发展的可再生能源和生物质降解受到了人们的广泛关注,利用酶促反应将生物质转换成第二代生物燃料是目前的新兴产业[1]。植物细胞壁多糖主要是由纤维素和半纤维等组成[2],将其完全降解需要一系列相关酶的协同作用。为了更好地利用植物细胞壁多糖这块巨大的可再生生物资源,有关纤维素降解酶或者木聚糖降解相关酶之间的协同作用已有大量的研究报道[3]。例如,当第45家族内切葡聚糖酶和纤维二糖水解酶的配比为1:4的时候,协同效果最好[4];木聚糖酶、木糖苷酶和乙酰木糖酯酶协同水解乙酰木聚糖,效果显著[5]。尽管如此,与双功能酶相比,这种不同催化反应之间承接与互作的内部紧密程度要远远高于由不同单功能酶组成的酶系[6]。
在自然进化的过程中,微生物会根据其生长的需要进化出可降解多种底物的非特异性酶,即多功能酶[7]。其中,双功能酶的研究相对较多。根据酶蛋白结构域与功能之间的对应关系,双功能酶大致可分为两大类:1)单结构域双功能酶,这类酶催化不同底物的活性中心共用同一个催化结构域。以来源于热解纤维素菌Caldicellulosiruptor bescii的双功能木聚糖酶/纤维素酶CbXyn10C为例[8],该酶仅含有一个催化结构域,能容纳、结合和催化具有不同组成和空间结构的纤维素和木聚糖底物,但这类酶的酶活性通常都不会太高。2)双结构域双功能酶,这类催化中心分别位于同一多肽链上的不同结构域里,这类酶多见于细菌来源的酶中,两个不同的催化结构域分别负责降解植物细胞壁多糖的主链和侧链,从而发挥协同降解的作用。如来源于黄化瘤胃球菌Ruminococcus flavefaciens 17的木聚糖酶/葡聚糖酶,N端为木聚糖酶结构域,C端为葡聚糖酶结构域,中间为Linker部分[9]。基于以上理论,研究者逐步建立了基于双功能酶活的融合酶。例如,将葡聚糖酶和木聚糖酶融合表达,成功获得了同时具有这两种酶活性的双结构域双功能酶[10]。由此可见,作为复合酶制剂的补充,大力开发和利用双功能酶具有很好的应用前景。
根据碳水化合物活性酶数据库(http://www.cazy.org)的分类,绝大多数的木聚糖酶被归类到糖苷水解酶(Glucoside hydrolase, GH)第10和11家族[11]。人们普遍认为,GH11家族木聚糖酶才是真正的木聚糖酶,因为其特异水解木聚糖[12]。而GH10家族木聚糖酶表现出一定的杂泛性,除了降解木聚糖之外,还可催化水解番茄苷[13]、大麦葡聚糖[14]和微晶纤维素[8]等。本研究从嗜热子囊菌Thermoascus crustaceus JCM12803中克隆了一个同时具有木聚糖酶和纤维素酶酶活的双功能酶基因,构建了高效表达的毕赤酵母基因工程菌株,并对其酶学性质进行系统的研究。利用同源建模和分子对接等生物信息学技术,讨论了其催化机制。
1 材料与方法 1.1 材料 1.1.1 菌株和载体嗜热子囊菌T. crustaceus JCM12803购自日本微生物保藏中心,可用马铃薯葡萄糖(PDB)培养基活化和培养;大肠杆菌质粒pEASY-T3和感受态Escherichia coli Trans1-T1购自北京全式金生物技术有限公司,用于基因片段的克隆和测序。质粒pPIC9和毕赤酵母Pichia pastoris GS115由本实验室保存,用于重组载体的构建和表达。
1.1.2 试剂底物榉木木聚糖购自Sigma公司;质粒小提中量试剂盒购自天根生化科技有限公司;DNA纯化试剂盒购自OMEGA公司;限制性内切酶EcoRⅠ和NotⅠ购自TaKaRa有限公司;总RNA提取试剂盒以及T4 DNA连接酶购自Promega (Madison, WI);FastPfu DNA聚合酶和EasyScript First-Strand cDNA Synthesis Super MIX购自全式金生物技术有限公司;蛋白Marker购自Genestar生物公司。其他所用化学试剂如乙醇、甲醇、乙酸等,若无特殊说明,均为国产分析纯。
1.1.3 主要培养基LB液体培养基:胰蛋白胨1% (W/V,下同),酵母提取物0.5%,NaCl 1%,LB固体培养基在液体培养基的基础上添加2%的琼脂。
YPD培养基:酵母提取物1%,蛋白胨2%,葡萄糖2%。
BMGY培养基:蛋白胨2%,酵母提取物1%,甘油1% (V/V),生物素0.000 04%,无氨基酵母氮源(YNB) 1.34%。
BMMY培养基:蛋白胨2%,酵母提取物1%,生物素0.000 04%,YNB 1.34%,甲醇0.5%。
MD培养基:葡萄糖2%,琼脂糖2%,YNB 1.34%,生物素0.000 04%。
1.2 双功能酶基因Tcxyn10a的克隆嗜热子囊菌T. crustaceus JCM12803的全基因组序列已由上海美吉公司完成测序。经过对注释序列进行生物信息学分析,Tcxyn10a是一个典型的木聚糖酶基因。利用SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)预测该基因的信号肽序列[15],Softberry Server (http://inux1.softberry.com/berry.phtml)分析其内含子和外显子,Vector NTI Advance 10.0软件分析Tcxyn10a基因编码蛋白的理论分子量和等电点等信息后,设计该基因特异引物Tcxyn10a-F:5′-ACAGAATTCAACCCTCTTCTTGCGGGACGCCAA-3′,Tcxyn10a-R:5′-AAGCGGCCGCTTAGCG CAGTGCATCGAC-3′ (下划线部分代表酶切位点EcoRⅠ和NotⅠ)。
将嗜热子囊菌T. crustaceus JCM12803转接至诱导培养基中培养3 d后,用SV总RNA纯化系统提取总RNA,采用Reverse Tra Ace-α-TM kit反转录试剂盒(TOYOBO)合成cDNA单链。以反转录的cDNA为模板,用高保真FastPfu DNA聚合酶、引物Tcxyn10a-F和Tcxyn10a-R进行PCR扩增,获得目的基因Tcxyn10a。PCR扩增产物已去除信号肽序列,将其切胶回收后,连接至克隆载体pEASY-T3上,并热击转化至克隆菌株Trans-T1用于测序验证。
1.3 重组表达载体的构建提取载体pPIC9的质粒,与本研究中提取的基因Tcxyn10a同时用EcoRⅠ和NotⅠ进行双酶切。将酶切产物进行核酸凝胶电泳分析,切胶回收后,用T4 DNA连接酶将它们连接。连接产物采用热击法转入大肠杆菌TransI-T1感受态细胞。次日,挑取转化子进行菌落PCR验证和酶切验证后,获得重组表达载体pPIC9-Tcxyn10a。
1.4 重组表达载体在毕赤酵母中的表达和纯化大量提取重组表达载体pPIC9-Tcxyn10a质粒,将其进行SalⅠ单酶切线性化处理,酶切产物回收后采用电击法转化到GS115感受态细胞中,并涂布于MD板上。30 ℃条件下倒置培养2 d后,分别挑取96个MD板上的单克隆于BMGY培养基中。30 ℃、200 r/min摇床振荡培养48 h后,4 500 r/min离心5 min,弃上清,换BMMY培养基,在0.5%甲醇的条件下诱导培养2 d。将获得的培养液在4 000 r/min离心10 min,进行酶液的收集和小管酶活的测定。
将筛选出的酶活最高的转化子进行摇瓶放大培养和诱导。同样用BMGY培养基进行菌体富集和BMMY培养基诱导产酶液。获得的发酵液于12 000 r/min离心10 min,得到粗酶液。粗酶液首先用10 kDa截留量的膜包进行浓缩处理,去除培养基残留和细胞碎片。随后,用0.02 mol/L柠檬酸-磷酸-氢二钠缓冲液(pH 7.5)进行脱盐处理,一方面去除培养基中的盐分,另一方面对酶液进行缓冲液置换。最后,选用HiTrap Q XL阴离子层析柱进行分离纯化,平衡的A液为0.02 mol/L柠檬酸-磷酸-氢二钠缓冲液(pH 7.5),B液在A液的基础上,添加1 mol/L的NaCl,用于梯度洗脱目的蛋白。经过色谱峰的酶活性检测和十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE,12%聚丙烯酰胺)检测目的蛋白的纯度和分子量。目的条带送中国农业科学院蜜蜂研究所进行液相色谱-电喷雾离子化-质谱联用(Liquid chromatography- electrospray ionization-tandem mass spectrometry, LC-ESI-MS)进行鉴定。
1.5 酶活力的测定木聚糖酶酶活的测定采用二硝基水杨酸(DNS)法检测还原糖的生成量[16-17]。配制1% (W/V)终浓度的榉木木聚糖溶于pH 5.0、0.1 mol/L柠檬酸-磷酸氢二钠缓冲液中作为底物。反应体系包括900 μL的底物和100 μL恰当稀释倍数的酶液,在pH 5.0、65 ℃下反应10 min,加入1.5 mL DNS试剂终止反应。沸水浴处理5 min后,快速冷却。通过酶标仪读取在540 nm波长下OD值,计算还原糖的产生量。
酶活定义:在pH 5.0、65 ℃条件下,每分钟降解榉木木聚糖生成1 μmoL木糖所需要的酶量定义为一个酶活单位。蛋白浓度采用Bradford法[18]测定。
1.6 酶学性质分析纯化后的TcXyn10A在65 ℃、不同pH (pH 3.0–8.0,0.1 mol/L柠檬酸-磷酸氢二钠缓冲液)的条件下与底物进行酶促反应(10 min),以测定其最适pH值。将TcXyn10A在不同pH值的缓冲液(pH 2.2–12.0;其中pH 2.2–8.0是0.1 mol/L柠檬酸-磷酸氢二钠缓冲液,pH 9.0–12.0是0.1 mol/L NaOH-甘氨酸缓冲液)中置于37 ℃处理1 h,待酶液用适当倍数的pH 5.0缓冲液校正之后,于pH 5.0、65 ℃条件下测定剩余酶活,未作处理的酶活为100%对照,以研究TcXyn10A的pH稳定性。
在pH 5.0的条件下,取恰当稀释倍数的TcXyn10A在不同温度(30–80 ℃)下与底物进行酶促反应(10 min),以测定其最适温度。将50 μg/mL的TcXyn10A分别在60 ℃和70 ℃下保温处理不同时间,在间隔时间点取样后迅速置于冰上,调整稀释倍数后于pH 5.0、65 ℃条件下测定剩余酶活,未作处理的酶活为100%对照,以研究TcXyn10A的温度稳定性。
分别配制不同浓度(0.5–10 mg/mL)的榉木木聚糖底物溶于pH 5.0、0.1 mol/L柠檬酸-磷酸氢二钠缓冲液中,与适当稀释倍数的TcXyn10A在65 ℃下进行酶促反应,一级反应时间定为5 min。利用GraphPad Prism 5.01软件分析得到TcXyn10A以榉木木聚糖为底物时的Vmax和Km值。
1.7 金属离子和化学试剂对木聚糖酶活性的影响在木聚糖酶反应体系中,分别加入0.005 mol/L的金属离子或化学试剂(Na+、K+、Ca2+、Cu2+、Mn2+、Zn2+、Fe3+、Cu2+、Mg2+、Ni2+、Cr3+、Co2+、SDS、EDTA和β-巯基乙醇),在pH 5.0、65 ℃条件下测定酶活。以未添加离子的酶液反应体系作为100%对照,每个实验组设置3个平行。根据剩余酶活的大小分析不同金属离子和化学试剂对酶活的影响。
1.8 底物特异性分析分别配制1% (W/V)的榉木木聚糖、小麦阿拉伯木聚糖、滤纸、羧甲基纤维素钠(CMC)、角豆胶、微晶纤维素、地衣多糖等底物于pH 5.0、0.1 mol/L柠檬酸-磷酸氢二钠缓冲液中,加入适当稀释倍数的TcXyn10A后,于65 ℃下反应,检测各还原糖的生成量。
根据底物特异性的分析结果,对榉木木聚糖、小麦阿拉伯木聚糖和CMC等底物在40 ℃下过夜反应。反应产物在经过高速离心和小分子去除之后,进行高效离子色谱(High Performance Anion Exchange Chromatography, HPAEC)分析[19]。以未添加酶液的底物作为对照。流动相为1 mol/L NaOH,流速为0.45 mL/min。标准对照品分别为木糖、木二糖、木三糖、木四糖、木五糖和木六糖,以及葡萄糖、纤维二糖、纤维三糖、纤维四糖、纤维五糖和纤维六糖。
1.9 同源建模及分子对接为了从分子层面研究TcXyn10A具有纤维素酶活性的分子机制,利用同源建模的方法构建了其三维模型。以Thermoascus aurantiacus来源的木聚糖酶(PDB: 1K6A; 1.14 Å, 序列一致性为82.5%)为模板[20],利用Discovery Studio 2017软件进行三维结构的建模。构建好的模型经过能量优化和评估之后,用于分子对接的受体。小分子配体木七糖和纤维六糖分别取自晶体复合物结构,PDB注册号分别为5OFK和5OFL[21]。利用Discovery Studio 2017软件中柔性对接(Flexible docking)模块,分别对TcXyn10A与木七糖、TcXyn10A与纤维六糖进行分子对接试验,获得酶-底物复合物结构。
2 结果与分析 2.1 木聚糖酶基因Tcxyn10a的克隆与序列分析分别以T. crustaceus JCM12803的基因组DNA和cDNA单链为模板,利用特异引物Tcxyn10a-F和Tcxyn10a-R进行PCR扩增,获得木聚糖酶基因Tcxyn10a的全长序列和cDNA序列,基因全长序列与基因组测序序列完全一致。该基因全长为1 762 bp,含有10个内含子序列。cDNA序列为984 bp,编码327个氨基酸和一个终止密码子。基因Tcxyn10a的相关信息提交至GenBank数据库中,序列注册号为MG879231。
经过序列分析,该基因的编码蛋白N端18个氨基酸为信号肽序列(图 1)。基因Tcxyn10a所编码的成熟蛋白预测理论分子量为33.6 kDa,等电点为5.36,无潜在的N-糖基化修饰位点。在氨基酸的组成方面,酸性氨基酸(天冬氨酸和谷氨酸)占9.71%,碱性氨基酸(赖氨酸和精氨酸)占7.77%,极性氨基酸(天冬酰胺、半胱氨酸、谷氨酰胺、丝氨酸、苏氨酸和酪氨酸)占28.8%,疏水氨基酸(丙氨酸、异亮氨酸、亮氨酸、苯丙氨酸、色氨酸和缬氨酸)占39.16%。将Tcxyn10a基因序列及其推测的氨基酸序列在NCBI数据库中进行序列比对,该基因与T. aurantiacus来源的木聚糖酶基因有最高的一致性,为82%。通过结构域分析,木聚糖酶TcXyn10A是一个单结构域蛋白,无碳水化合物结合结构域。

|
| 图 1 木聚糖酶TcXyn10A的氨基酸序列 Figure 1 The amino acid sequence of xylanase TcXyn10A. The signal peptide was marked with black line, the sequences of internal peptides identified by LC-ESI-MS are marked as colors. |
| |
重组表达载体pPIC9-Tcxyn10a在毕赤酵母中成功实现高效表达,摇瓶表达水平为(9.2±0.2) U/mL。通过小管初筛,挑选出酶活最高的转化子并进行摇瓶水平放大培养。获得的发酵液作为粗酶液进行后续纯化处理。经过蛋白浓缩、脱盐和阴离子交换色谱柱等纯化步骤,获得单一洗脱峰。经SDS-PAGE鉴定达到了蛋白电泳纯(图 2),条带大小与理论分子量一致。LC-ESI-MS鉴定结果如图 2所示,鉴定得到的4条肽段与木聚糖酶TcXyn10A的氨基酸序列高度吻合,进一步证明所获重组蛋白即为木聚糖酶TcXyn10A。

|
| 图 2 木聚糖酶TcXyn10A的蛋白电泳分析 Figure 2 SDS-PAGE analysis of purified recombinant TcXyn10A. |
| |
重组TcXyn10A的最适pH为5.0,在pH 4.0– 6.5之间保持60%以上的酶活(图 3A),是一个酸性木聚糖酶。TcXyn10A的pH稳定范围非常宽泛,在pH 3.0–11.0的条件下处理1 h后,残存酶活均能保持80%以上的酶活性(图 3B),说明其在弱酸至强碱性环境中均表现出很强的稳定性。TcXyn10A的最适温度为65–70 ℃ (图 3C),在50–75 ℃下都能保持较高酶活,属于中高温酶;在60 ℃保持稳定,热处理1 h后仍保持80%的酶活,在70 ℃下处理5 min,保持50%左右的酶活(图 3D)。

|
| 图 3 重组TcXyn10A的酶学性质 Figure 3 Enzymatic properties of recombinant TcXyn10A. (A) pH optima. (B) pH stability. (C) Temperature optima. (D) Temperature stability. |
| |
以榉木木聚糖为底物,在pH 5.0、65 ℃的条件下,重组木聚糖酶TcXyn10A的比活为(1 480±26) U/mg。动力学参数Km和Vmax分别为(2.6± 0.19) mg/mL和(1 943±63) μmol/(min·mg)。
不同金属离子和化学试剂对TcXyn10A酶活的影响如表 1所示。绝大多数的离子对其酶活无明显影响,Ni2+、K+和EDTA对其具有明显的促进作用,而Cu2+和SDS对其有明显的抑制作用。
| Ions or chemicals | Relative activity (%) |
| Control | 100.0±1.0 |
| Mg2+ | 87.9±2.6 |
| Mn2+ | 81.4±3.0 |
| Co2+ | 81.5±2.1 |
| Ni2+ | 122.8±3.4 |
| Zn2+ | 80.3±0.8 |
| Ca2+ | 67.3±1.7 |
| Cr3+ | 69.8±0.7 |
| Na+ | 80.8±0.5 |
| K+ | 115.3±1.7 |
| Fe3+ | 87.3±1.3 |
| Cu2+ | 51.8±1.7 |
| EDTA | 110.8±1.7 |
| SDS | 32.4±1.4 |
| β-mercaptoethanol | 99.6±1.5 |
重组木聚糖酶TcXyn10A底物特异性非常广泛。通过对不同底物降解试验,当酶促反应为10 min时,TcXyn10A对小麦阿拉伯木聚糖、CMC和地衣多糖有明显的降解活性,比活分别为(2 055±28) U/mg、(7.4±0.2) U/mg和(10.9±0.4) U/mg。相对于榉木木聚糖酶活,TcXyn10A对小麦阿拉伯木聚糖、CMC和地衣多糖的酶活分别为138.8%、0.5%和0.7%。而TcXyn10A对滤纸和微晶纤维素无降解活性。
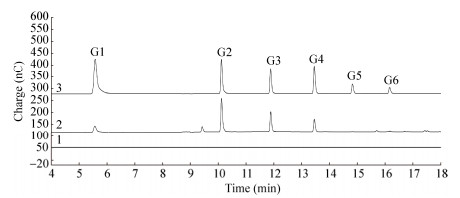
以小麦阿拉伯木聚糖、榉木木聚糖和CMC为底物反应10 h后,利用HPAEC分析水解终产物。TcXyn10A对小麦阿拉伯木聚糖和榉木木聚糖的主要产物是木糖和木二糖(图 4)。其中,TcXyn10A以小麦阿拉伯木聚糖为底物时生成木糖和木二糖的量分别为695 μmol和1.98 mmol,以榉木木聚糖为底物时生成木糖和木二糖的量分别为558 μmol和1.34 mmol,木二糖的产量是木糖的2.4倍。除此之外,小麦阿拉伯木聚糖和榉木木聚糖的水解终产物中还检测到其他的糖组分。TcXyn10A对CMC的主要产物是葡萄糖、纤维二糖、纤维三糖和纤维四糖(图 5),产量分别为0.21、3.4、1.73和0.76 μmol,呈典型的内切纤维素酶酶活。

|
| 图 4 TcXyn10A对小麦阿拉伯木聚糖和榉木木聚糖的水解产物分析 Figure 4 Products analysis of TcXyn10A toward wheat arabinoxylan and beechwood xylan. 1: the control of substrate incubated without enzyme; 2: beechwood xylan; 3: wheat arabinoxylan; 4: the xylooligosaccharide standards. X1, xylose; X2, xylobiose; X3, xylotriose; X4, xylotetraose; X5, xylopentaose; X6, xylohexaose. |
| |
|
| 图 5 TcXyn10A对CMC的水解产物分析 Figure 5 Products analysis of TcXyn10A toward CMC. 1: the control of substrate incubated without enzyme; 2: CMC; 3: the cellooligosaccharide standards. G1, glucose; G2, cellobiose; G3, cellotriose; G4, cellotetraose; G5, cellopentaose; G6, cellohexaose. |
| |
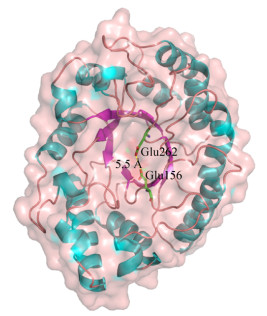
以1K6A为模板,对TcXyn10A进行了同源建模。通过模型评估分析与优化之后,对TcXyn10A的结构特征进行分析。如图 6所示,TcXyn10A呈现典型的糖苷水解酶第10家族木聚糖酶(β/α) 8桶状结构,可见TcXyn10A的双功能酶活性由该单一结构域实现。与其他GH10家族木聚糖酶相似,8个β折叠片相互连成氢键网,排列成内桶;8个α-螺旋则在其间沿着相同的方向排列在β旋折叠桶的外侧,成为外桶。无规则卷曲结构(loop区)连接β接折叠片和α叠螺旋,不同长度的loop区,其构象不同。两个严格保守的催化残基(Glu156和Glu262)处于对立的位置,侧链间的距离为5.5 Å。
|
| 图 6 TcXyn10A的同源建模模型 Figure 6 Homologous model of TcXyn10A. |
| |
纤维素和木聚糖是植物细胞壁中主要的非淀粉多糖,二者的充分利用对于人类的可持续发展有着极为重要的现实意义,相关的研究报道也从未间断[22-24]。但有意思的是,尽管有许多纤维素酶中发现了木聚糖酶活性,只有很少部分的木聚糖酶被检测到纤维素酶活性[25]。本研究中,我们从嗜热子囊菌T. crustaceus JCM12803中克隆得到了一个GH10家族双功能木聚糖酶TcXyn10A。底物特异性分析结果显示,TcXyn10A不仅对榉木木聚糖和小麦阿拉伯木聚糖有较高的酶活,而且也可降解CMC和地衣多糖底物,是一个典型的拥有纤维素酶活性的木聚糖酶。将TcXyn10A与同类双功能酶进行比较(表 2),TcXyn10A的木聚糖酶活性要高于牦牛瘤胃、德米奎纳菌Demequina sp.和热解纤维素菌C. bescii来源的双功能酶,但TcXyn10A的纤维素酶活要低于牦牛瘤胃来源的双功能酶。经过序列和同源建模结构分析,TcXyn10A只含有单一催化结构域,两个严格保守的催化残基Glu156和Glu262分别位于催化口袋内部的两个loop区上,是典型的GH10家族木聚糖酶[26]。双功能酶TcXyn10A的获得,一方面加深了我们对单结构域双功能木聚糖酶/纤维素酶的认识,另一方面也为双功能酶结构与其功能关系的研究提供了很好的素材。
本课题组对植物细胞壁多糖的高效酶学降解和利用进行了较长时间的研究[29]。最近,我们还获得了嗜热厌氧细菌C. bescii来源的第10家族木聚糖酶CbXyn10C及其分别与木七糖和纤维六糖的复合物晶体结构[21]。为了探究TcXyn10A降解木聚糖和纤维素的催化机制,在TcXyn10A同源建模的基础上,采用分子对接的方法分别获得了TcXyn10A与木七糖和纤维六糖的复合物结构模型。如图 7A所示,木七糖位于TcXyn10A狭窄的催化通道中,处于–3到+4位的构象位置。–1位和+1位木糖单元连接的糖苷氧原子和Glu156的OE2原子间形成氢键(3.0Å),而Glu262的侧链与–1位木糖单元的C1原子(异头碳)间隔3.5Å的距离。根据糖苷水解酶的催化机制分析,催化残基Glu156为质子供体,Glu262为亲核试剂[30]。在这种情况下,位于–1位的木糖单元将会扭曲成B3, O船型构象[31]。Trp112、Trp292和Trp300分别对–2位和–1位木糖单元提供了疏水作用力。–1位木糖单元的O2原子与Asn155的ND2原子,O3原子与Lys75的NZ原子、His108的NE2原子,O5原子与His234的NE2原子间形成氢键,进一步增强了TcXyn10A与–1位木糖单元之间的结合力。除此之外,–2位木糖单元的O2原子与Trp292的NE1原子、Glu71的OE1原子,O3原子与Asn72的ND2原子,O5原子与Lys75的NZ原子形成氢键网络。在木聚糖酶与木寡糖复合物的晶体结构中,–2位和–1位木糖单元与木聚糖酶之间的氢键相互作用数目最多。例如,来源于嗜热土脂肪芽胞杆菌Geobacillus stearothermophilus的木聚糖酶与木五糖的晶体复合物(PDB ID: 1R87)中,–2位和–1位木糖单元与酶之间形成的氢键数目分别为6个和9个[32]。从这一方面讲,双功能酶TcXyn10A与其他已报道的木聚糖酶一致。双功能酶TcXyn10A与纤维六糖的结合模式如图 7B所示。纤维六糖与木聚糖底物共用催化通道,处于–2到+4位的构象位置。位于–1位的葡萄糖单元与同样位置的木糖单元构象类似,糖苷氧原子和Glu156的OE2原子间形成氢键(3.1 Å),而Glu262的侧链与异头碳的间隔距离3.1 Å,这意味着双功能酶TcXyn10A对木聚糖和纤维素底物的降解,是通过共用一对催化残基(Glu156和Glu262)来进行酸碱催化的。与木糖相比,葡萄糖要多出C6和O6两个原子。而相同的疏水作用力(Trp112、Trp292和Trp300)同样在–2位和–1位葡萄糖单元的位置呈现,氢键作用网络也与木七糖相似。两个结合模型中,木七糖与纤维六糖的整体构象差异非常小(图 7C),这说明双功能酶TcXyn10A的催化口袋完全可以结合纤维寡糖,进而将其降解。

|
| 图 7 TcXyn10A分别与木七糖(A)和纤维六糖(B)的结合模式图及两种底物的叠合图(C) Figure 7 Illustration of the substrate xyloheptaose (A) and cellohexaose (B) docked to the TcXyn10A catalytic pocket, and the superimposition of them (C). |
| |
双功能酶TcXyn10A对榉木木聚糖和小麦阿拉伯木聚糖有较高的活性,分别为(1 480±26) U/mg和(2 055±28) U/mg;而对CMC和地衣多糖的降解活性相对较低,比活分别为(7.4±0.2) U/mg和(10.9±0.4) U/mg,提升空间较大。Ichinose等[33]对链霉菌Streptomyces olivaceoviridis E-86来源的木聚糖酶底物特异性进行了改造,通过将底物–2位和–1位糖单元处的Gln88和Arg275突变成Ala,消除了空间位阻,使得葡萄糖的C6和O6原子能够完全结合,对纤维素底物的亲和力提高。这些成功案例的结果,结合TcXyn10A与木七糖和纤维六糖结合模式的分析,对于双功能酶的结构与其功能的关系的研究有一定的参考价值。
| [1] | Wang JH, Feng JJ, Jia WT, et al. Lignin engineering through laccase modification: a promising field for energy plant improvement. Biotechnol Biofuels, 2015, 8(1): 145. |
| [2] | Scheller HV, Ulvskov P. Hemicelluloses. Annu Rev Plant Biol, 2010, 61(1): 263–289. DOI: 10.1146/annurev-arplant-042809-112315 |
| [3] | Gao DH, Uppugundla N, Chundawat SP, et al. Hemicellulases and auxiliary enzymes for improved conversion of lignocellulosic biomass to monosaccharides. Biotechnol Biofuels, 2011, 4(1): 5. |
| [4] | Toda H, Nagahata N, Amano Y, et al. Gene cloning of cellobiohydrolase Ⅱ from the white rot fungus Irpex lacteus MC-2 and its expression in Pichia pastoris. Biosci Biotechnol Biochem, 2008, 72(12): 3142–3147. DOI: 10.1271/bbb.80316 |
| [5] | Bachmann SL, McCarthy AJ. Purification and cooperative activity of enzymes constituting the xylan-degrading system of Thermomonospora fusca. Appl Environ Microbiol, 1991, 57(8): 2121–2130. |
| [6] | Vrzheshch PV. Steady-state kinetics of bifunctional enzymes. Taking into account kinetic hierarchy of fast and slow catalytic cycles in a generalized model. Biochemistry (Moscow), 2007, 72(9): 936–943. DOI: 10.1134/S0006297907090039 |
| [7] | Khandeparker R, Numan MT. Bifunctional xylanases and their potential use in biotechnology. J Ind Microbiol Biotechnol, 2008, 35(7): 635–644. DOI: 10.1007/s10295-008-0342-9 |
| [8] | Xue XL, Wang R, Tu T, et al. The N-terminal GH10 domain of a multimodular protein from Caldicellulosiruptor bescii is a versatile xylanase/ β-glucanase that can degrade crystalline cellulose. Appl Environ Microbiol, 2015, 81(11): 3823–3833. DOI: 10.1128/AEM.00432-15 |
| [9] | Flint HJ, Martin J, McPherson CA, et al. A bifunctional enzyme, with separate xylanase and beta (1, 3-1, 4)-glucanase domains, encoded by the xynD gene of Ruminococcus flavefaciens. J Bacteriol, 1993, 175(10): 2943–2951. DOI: 10.1128/jb.175.10.2943-2951.1993 |
| [10] | Lu P, Feng MG. Bifunctional enhancement of a β-glucanase-xylanase fusion enzyme by optimization of peptide linkers. Appl Microbiol Biotechnol, 2008, 79(4): 579–587. DOI: 10.1007/s00253-008-1468-4 |
| [11] |
Bai WQ, Wang QH, Ma YH. Progress in the thermophilic and alkalophilic xylanases.
Chin J Biotech, 2014, 30(6): 828–837.
(in Chinese). 柏文琴, 王钦宏, 马延和. 嗜热和嗜碱木聚糖酶研究进展. 生物工程学报, 2014, 30(6): 828-837. |
| [12] | Cheng YS, Chen CC, Huang JW, et al. Improving the catalytic performance of a GH11 xylanase by rational protein engineering. Appl Microbiol Biotechnol, 2015, 99(22): 9503–9510. DOI: 10.1007/s00253-015-6712-0 |
| [13] | Roldán-Arjona T, Pérez-Espinosa A, Ruiz-Rubio M. Tomatinase from Fusarium oxysporum f. sp. lycopersici defines a new class of saponinases. Mol Plant Microbe Interact, 1999, 12(10): 852–861. DOI: 10.1094/MPMI.1999.12.10.852 |
| [14] | Shi PJ, Tian J, Yuan TZ, et al. Paenibacillus sp. strain E18 bifunctional xylanase-glucanase with a single catalytic domain. Appl Environ Microbiol, 2010, 76(11): 3620–3624. DOI: 10.1128/AEM.00345-10 |
| [15] | Petersen TN, Brunak S, Von Heijne G, et al. SignalP 4.0: discriminating signal peptides from transmembrane regions. Nat Methods, 2011, 8(10): 785–786. DOI: 10.1038/nmeth.1701 |
| [16] | Miller GL. Use of dinitrosalicylic acid reagent for determination of reducing sugar. Anal Chem, 1959, 31(3): 426–428. DOI: 10.1021/ac60147a030 |
| [17] |
Wang XY, Liu WN, Xie XM, et al. A highly active GH11 xylanase from Penicillium sp. L1 with potential applications in xylo-oligosaccharide production.
Chin J Biotech, 2018, 34(1): 68–77.
(in Chinese). 王晓宇, 刘伟娜, 谢响明, 等. 青霉L1来源具有生产木寡糖应用潜力的高比活GH11木聚糖酶. 生物工程学报, 2018, 34(1): 68-77. |
| [18] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976, 72: 248–254. DOI: 10.1016/0003-2697(76)90527-3 |
| [19] | Yang WX, Bai YG, Yang PL, et al. A novel bifunctional GH51 exo-α-l-arabinofuranosidase/endo-xylanase from Alicyclobacillus sp. A4 with significant biomass- degrading capacity. Biotechnol Biofuels, 2015, 8(1): 197. DOI: 10.1186/s13068-015-0366-0 |
| [20] | Lo Leggio L, Kalogiannis S, Eckert K, et al. Substrate specificity and subsite mobility in T. aurantiacus xylanase 10A. FEBS Lett, 2001, 509(2): 303–308. DOI: 10.1016/S0014-5793(01)03177-5 |
| [21] | Chu YD, Tu T, Penttinen L, et al. Insights into the roles of non-catalytic residues in the active site of a GH10 xylanase with activity on cellulose. J Biol Chem, 2017, 292(47): 19315–19327. DOI: 10.1074/jbc.M117.807768 |
| [22] | Hu JG, Arantes V, Saddler JN. The enhancement of enzymatic hydrolysis of lignocellulosic substrates by the addition of accessory enzymes such as xylanase: is it an additive or synergistic effect?. Biotechnol Biofuels, 2011, 4(1): 36. DOI: 10.1186/1754-6834-4-36 |
| [23] | Shi PJ, Chen XY, Meng K, et al. Distinct actions by Paenibacillus sp. strain E18 α-l-arabinofuranosidases and xylanase in xylan degradation. Appl Environ Microbiol, 2013, 79(6): 1990–1995. DOI: 10.1128/AEM.03276-12 |
| [24] | Ji L, Yang JS, Fan H, et al. Synergy of crude enzyme cocktail from cold-adapted Cladosporium cladosporioides Ch2-2 with commercial xylanase achieving high sugars yield at low cost. Biotechnol Biofuels, 2014, 7(1): 130. |
| [25] | Nakazawa H, Okada K, Kobayashi R, et al. Characterization of the catalytic domains of Trichoderma reesei endoglucanase Ⅰ, Ⅱ, and Ⅲ, expressed in Escherichia coli. Appl Microbiol Biotechnol, 2008, 81(4): 681–689. DOI: 10.1007/s00253-008-1667-z |
| [26] | Henrissat B, Davies GJ. Structural and sequence-based classification of glycoside hydrolases. Curr Opin Struct Biol, 1997, 7(5): 637–644. DOI: 10.1016/S0959-440X(97)80072-3 |
| [27] | Chang L, Ding MZ, Bao L, et al. Characterization of a bifunctional xylanase/endoglucanase from yak rumen microorganisms. Appl Microbiol Biotechnol, 2011, 90(6): 1933–1942. DOI: 10.1007/s00253-011-3182-x |
| [28] | Meng X, Shao ZZ, Hong YZ, et al. A novel pH-stable, bifunctional xylanase isolated from a deep-sea microorganism, Demequina sp. JK4. J Microbiol Biotechnol, 2009, 19(10): 1077–1084. |
| [29] | Tu T, Bai YG, Luo HY, et al. A novel bifunctional pectinase from Penicillium oxalicum SX6 with separate pectin methylesterase and polygalacturonase catalytic domains. Appl Microbiol Biotechnol, 2014, 98(11): 5019–5028. DOI: 10.1007/s00253-014-5533-x |
| [30] | Rye CS, Withers SG. Glycosidase mechanisms. Curr Opin Chem Biol, 2000, 4(5): 573–580. DOI: 10.1016/S1367-5931(00)00135-6 |
| [31] | Speciale G, Thompson AJ, Davies GJ, et al. Dissecting conformational contributions to glycosidase catalysis and inhibition. Curr Opin Struct Biol, 2014, 28(28C): 1–13. |
| [32] | Zolotnitsky G, Cogan U, Adir N, et al. Mapping glycoside hydrolase substrate subsites by isothermal titration calorimetry. Proc Natl Acad Sci USA, 2004, 101(31): 11275–11280. DOI: 10.1073/pnas.0404311101 |
| [33] | Ichinose H, Diertavitian S, Fujimoto Z, et al. Structure-based engineering of glucose specificity in a family 10 xylanase from Streptomyces olivaceoviridis E-86. Process Biochem, 2012, 47(3): 358–365. DOI: 10.1016/j.procbio.2011.06.002 |
 2018, Vol. 34
2018, Vol. 34