中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张海灵, 李颜颜, 王小元
- Zhang Hailing, Li Yanyan, Wang Xiaoyuan
- 代谢工程改造谷氨酸棒状杆菌合成及分泌途径生产L-缬氨酸
- Metabolic engineering of L-valine synthesis and secretory pathways in Corynebacterium glutamicum for higher production
- 生物工程学报, 2018, 34(10): 1606-1619
- Chinese Journal of Biotechnology, 2018, 34(10): 1606-1619
- 10.13345/j.cjb.180112
-
文章历史
- Received: March 27, 2018
- Accepted: April 28, 2018
2 江南大学 生物工程学院,江苏 无锡 214122
2 School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China
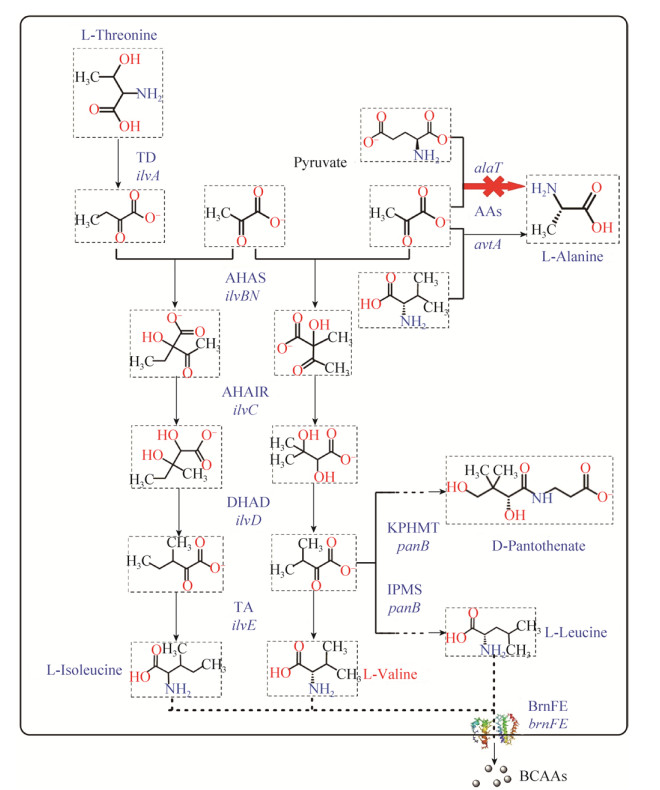
L-缬氨酸(L-valine)是一种重要的支链氨基酸,是人体8种必需氨基酸之一,广泛应用于食品、医药、化妆品、饲料等领域[1]。目前工业上主要通过微生物发酵法生产L-缬氨酸,生产菌株主要包括谷氨酸棒状杆菌Corynebacterium glutamicum、大肠杆菌Escherichia coli以及酿酒酵母Saccharomyces cerevisiae,谷氨酸棒状杆菌由于产率高、生物安全等特性而被广泛应用[2-4]。传统诱变育种是L-缬氨酸工业生产菌主要来源[5],但是传统的育种方式获得的菌株遗传背景不明确、菌株遗传特性不稳定,随着谷氨酸棒状杆菌生理学和遗传学知识的积累,基于理性设计的代谢工程正在成为选育氨基酸高产菌株的主要方式,更多的谷氨酸棒杆菌生产特性得以改良,根据已经获得的遗传学信息,系统分析菌株L-缬氨酸代谢网络,通过基因工程技术改造谷氨酸棒杆菌以提高L-缬氨酸的产量[6-7]。近年来,一些谷氨酸棒杆菌的基因敲除、表达系统已经被构建[8-13],针对谷氨酸棒状杆菌中的启动子工程也已经开展[11, 14]。围绕L-缬氨酸的生物合成途径,谷氨酸棒状杆菌中的理性代谢工程设计从DNA层次上的定点突变以消除转录弱化调控和改变启动子活性、蛋白层次上改造酶的催化结构域和调节结构域、强化通往L-缬氨酸的碳代谢流以及加强L-缬氨酸的胞外转运等几个方面展开[15]。
在前期的研究中,我们选育了一株L-缬氨酸产量较高的谷氨酸棒状杆菌VWB-1,通过对其进行转录组学和蛋白质组学的分析研究其遗传背景,发现菌株的产酸水平有待进一步提高[16]。本研究中,借助代谢工程改造的技术手段,我们首先在谷氨酸棒状杆菌L-缬氨酸高产菌株VWB-1中对丙氨酸合成途径丙氨酸氨基转移酶编码基因alaT进行敲除,发现丙氨酸合成途径的阻断能够降低细胞内杂酸丙氨酸的生成。其次,对L-缬氨酸合成代谢限速酶乙酰羟酸合酶(AHAS)进行抗反馈抑制的定点突变,解除L-缬氨酸对该酶的反馈抑制,进而通过过量表达L-缬氨酸合成途径基因加强通往L-缬氨酸的碳代谢流。最后,表达与L-缬氨酸转运相关的基因加强菌株的L-缬氨酸转运能力。通过摇瓶发酵和5 L罐发酵实验,检测重组工程菌株的产L-缬氨酸水平。
1 材料与方法 1.1 菌株和质粒本研究所采用的菌株和质粒见表 1。pEC- XK99E载体为E. coli和C. glutamicum穿梭表达载体[17]。
| Name | Relevant genotype | Reference |
| Plasmids | ||
| pEC-XK99E | E. coli and C. glutamicum shuttle vector | Laboratory preservation |
| pk19mobsacBalaT | C. glutamicum alaT knocking out vector | [20] |
| pEC-XK99E-ilvBN | pEC-XK99E with the insertion of ilvBN | This research |
| pEC-XK99E-ilvBNM13 | pEC-XK99E with the insertion of ilvBN1M13 | This research |
| pEC-XK99E-ilvBN1 | pEC-XK99E with the insertion of ilvBN1 | This research |
| pEC-XK99E-ilvBN1M13 | pEC-XK99E with the insertion of ilvBN1M13 | This research |
| pEC-XK99E-ilvBN1M13CD | pEC-XK99E with the insertion of ilvBN1M13CD | This research |
| pEC-XK99E-ilvBN1M13CE | pEC-XK99E with the insertion of ilvBN1M13CE | This research |
| pEC-XK99E-ilvBN1M13CD-lrp1-brnFE | pEC-XK99E with the insertion of ilvBN1M13CD-lrp1-brnFE | This research |
| pEC-XK99E-ilvBN1M13CE-lrp1-brnFE | pEC-XK99E with the insertion of ilvBN1M13CE-lrp1-brnFE | This research |
| Strains | ||
| E. coli JM109 | Laboratory preservation | |
| C. glutamicum VWB-1 | [16] | |
| C. glutamicum VWB-2 | VWB-1 with the deletion of alaT | This research |
| ATCC 13869/pJYW-ilvBNC1-lrp1-brnFE | ATCC 13869 harboring pJYW-ilvBN1C-lrp1-brnFE | [21] |
| VWB-1/pEC-XK99E | VWB-1 harboring pEC-XK99E | This research |
| VWB-1/pEC-XK99E-ilvBN | VWB-1 harboring pEC-XK99E-ilvBN | This research |
| VWB-1/pEC-XK99E-ilvBN1M13 | VWB-1 barboring pEC-XK99E-ilvBN1M13 | This research |
| VWB-1/pEC-XK99E-ilvBN1 | VWB-1 harboring pEC-XK99E-ilvBN1 | This research |
| VWB-1/pEC-XK99E-ilvBN1M13 | VWB-1 harboring pEC-XK99E-ilvBN1M13 | This research |
| VWB-1/pEC-XK99E-ilvBN1M13CD | VWB-1 harboring pEC-XK99E-ilvBN1VM13CD | This research |
| VWB-1/pEC-XK99E-ilvBN1M13CE | VWB-1 harboring pEC-XK99E-ilvBN1M13CE | This research |
| VWB-1/pEC-XK99E-ilvBN1M13CD-lrp1-brnFE | VWB-1 harboring pEC-XK99E-ilvBN1M13CD-lrp1-brnFE | This research |
| VWB-1/pEC-XK99E-ilvBN1M13CE-lrp1-brnFE | VWB-1 harboring pEC-XK99E-ilvBN1M13CE-lrp1-brnFE | This research |
| VWB-2/pEC-XK99E-ilvBN1M13CD-lrp1-brnFE | VWB-2 harboring pEC-XK99E-ilvBN1M13CD-lrp1-brnFE | This research |
| VWB-2/pEC-XK99E-ilvBN1M13CE-lrp1-brnFE | VWB-2 harboring pEC-XK99E-ilvBN1M13CE-lrp1-brnFE | This research |
大肠杆菌E. coli JM109使用LB培养基(酵母浸提物5 g/L,蛋白胨10 g/L,NaCl 10 g/L;固体培养基加入琼脂粉20 g/L)进行培养,培养温度为37 ℃。谷氨酸棒状杆菌C. glutamicum ATCC 13869和VWB-1使用LBGB培养基(LB培养基中添加5 g/L葡萄糖以及18.5 g/L脑心浸液),培养温度为30 ℃。C. glutamicum感受态制备培养基(酵母浸提物0.5 g/L,蛋白胨1 g/L,NaCl 1 g/L,甘氨酸3%,吐温800.1%),C. glutamicum电转化恢复培养基LBHIS (酵母浸提物2.5 g/L,蛋白胨5 g/L,NaCl 5 g/L,D-山梨醇91 g/L,脑心浸液18.5 g/L;固体培养基加入琼脂粉20 g/L)。必要时添加质量浓度为30 μg/mL的卡那霉素。大肠杆菌感受态制备及转化方法参照文献[18],谷氨酸棒状杆菌感受态制备及转化方法参照文献[12]。
1.3 突变菌株、定点突变、重组载体和基因重组菌构建本研究所用引物序列及相关信息见表 2。引物主要依据C. glutamicum ATCC 13032基因组序列设计[19],ilvN1M13突变相关引物ilvNM13-F/ilvNM13-R根据VWB-1相关基因测序结果设计。
| Primer name | Sequence (5'–3') |
| ilvBN-F (EcoRⅠ) | CGGAATTCAAAGGAGGAGAATCAGTAAAGGAGCCAGAAAGTCGTG |
| ilvBN-R (SmaⅠ) | GCACCCGGGGATTAATTGCTGTTTAGATCTTGG |
| ilvNM13-F | GTCCGTACTCGTTCAGGACGTAGACGATGACTTTTCCCGCGTATC |
| ilvNM13-R | CGGGTGAACATACCTGATACGCGGGAAAAGTCATCGTCTACGTCC |
| ilvBNC-F (EcoRⅠ) | CGGAATTCAAAGGAGGAGAATCAGTAAAGGAGCCAGAAAGTCGTG |
| ilvBNC-R (SmaⅠ) | GATCCCGGGTATGTACAAAGTGCACAGCAGGT |
| ilvD-F (XbaⅠ) | CGTCTAGAAAAAGCGCATCATGATCCCACTTCGT |
| ilvD-R (XbaⅠ) | CATCTAGACCAAAGTAGGGAACCGGTGCTCAAACA |
| ilvE-F (XbaⅠ) | CGTCTAGAGTGTATCTGTCAGGTAGCAGGT |
| ilvE-R (XbaⅠ) | GCTCTAGAGTTTAATGCAGCGGGGTCTTAA |
| Ptac-lrp-brnFE-F(NdeⅠ) | GGTATTTCACACCGCATATGACTCGACAACTGTTAATT |
| Ptac-lrp-brnFE-R(NdeⅠ) | TACTGAGAGTGCACCATATGTTAGAAAAGATTCACCAGTC |
| alaT-veri-F | GACTACAGACAAGCGCAAAACCTCTAAGAC |
| alaT-veri-R | GGAAGTTACCCAGGCGCTCAATTGC |
| “_” represents the cleavage site of restriction endonuclease, “=” represents the mutation sites of ilvNM13. | |
应用sacB基因的条件致死性原理,进行VWB-1丙氨酸氨基转移酶编码基因alaT的敲除。利用基于sacB的整合型敲除载体pK19mobsacBalaT (德国尤里希生物技术研究中心Lothar Eggeling教授提供)[20],对菌株VWB-1进行alaT敲除,谷氨酸棒状杆菌alaT基因敲除方法参照文献[12]。获得了不含有kan抗性的无痕基因敲除突变菌株VWB-1/ΔalaT (命名为VWB-2)。
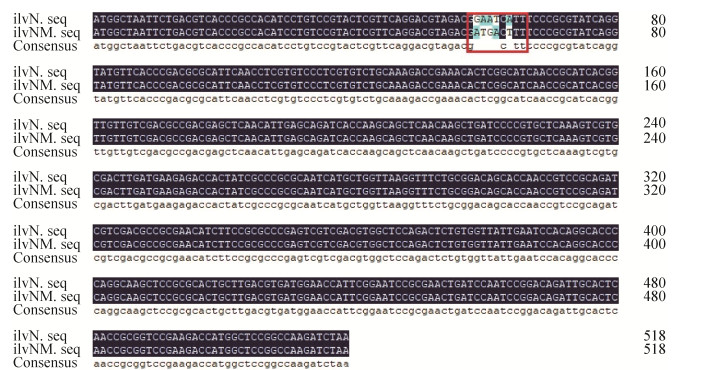
1.3.2 乙酰羟酸合酶调节亚基IlvN定点突变前期测序结果表明,VWB-1来源的ilvBN (标记为ilvBN1)编码的乙酰羟酸合酶(标记为AHAS1)蛋白相比于ATCC 13869来源的ilvBN编码的乙酰羟酸合酶蛋白,氨基酸序列并非完全一致。如图 1所示,分别以C. glutamicum ATCC 13869和VWB-1的基因组为模板扩增ilvBN相关基因。以ilvBN-F和ilvBN-R为引物分别扩增ilvBN和ilvBN1,将获得的PCR片段与载体pEC-XK99E共同进行EcoRⅠ和SmaⅠ双酶切并纯化,在T4 DNA连接酶作用下进行连接,构建得到载体pEC-XK99E-ilvBN和pEC-XK99E-ilvBN1。分别以构建好的重组载体pEC-XK99E-ilvBN和pEC- XK99E-ilvBN1为模板,以ilvNM13-F/ilvNM13-R为引物,进行全质粒扩增,PCR反应结束,直接在PCR反应体系中加入1 μL DpnⅠ,混匀后30 ℃孵育1 h。DpnⅠ消化结束后直接转化E. coli JM109感受态细胞,将转化后的细胞全部涂布到含有适当抗生素的平板上,过夜培养。挑取生长后的转化子提取质粒进行酶切验证,确认得到的质粒大小正确以后,对该质粒进行测序,比对测序结果,鉴定突变是否成功。突变成功后的载体命名为pEC-XK99E-ilvBNM13和pEC-XK99E-ilvBN1M13。

|
| 图 1 重组质粒的构建流程 Figure 1 Construction procedure of the recombinant plasmids. |
| |
如图 1所示,以C. glutamicum VWB-1基因组为模板,以ilvC-F/ilvC-R为引物,扩增获得目的基因ilvC,将ilvC片段和pEC-XK99E-ilvBN1M13分别进行双酶切,在T4 DNA酶的作用下进行连接,得到新载体pEC-XK99E-ilvBN1M13C。继而分别以C. glutamicum VWB-1基因组ilvD-F/ilvD-R、ilvE-F/ilvE-R为引物,克隆得到ilvD和ilvE,将ilvD和ilvE各自进行XbaⅠ酶切,与同样经XbaⅠ酶切的载体pEC-XK99E-ilvBN1M13C进行连接。连接反应产物转入到E. coli JM109感受态细胞,提取转化子质粒酶切验证,同时测序验证片段的连接顺序。构建分别得到pEC-XK99E-ilvBN1M13CD和pEC-XK99E-ilvBN1M13CE。
1.3.4 L-缬氨酸转运系统基因的串联表达如图 1所示,以陈诚等[21]构建的载体pJYW-4- lrp1-brnFE为模板,以Ptac-lrp1-brnFE-F/Ptac-lrp1- brnFE-R为引物,扩增得到lrp1和brnFE上游均含有PtacM启动子序列的片段PtacM-lrp1-brnFE,将该片段用NdeⅠ进行酶切,在T4 DNA连接酶的作用下,与同样经NdeⅠ酶切的载体pEC- XK99E-ilvBN1M13CD和pEC-XK99E-ilvBN1M13CE进行连接,转入到E. coli JM109感受态细胞。提取转化子质粒酶切验证,同时测序验证片段的连接顺序。构建得到新载体命名为pEC-XK99E- ilvBN1M13CD-lrp1-brnFE和pEC-XK99E-ilvBN1M13 CE-lrp1-brnFE。
1.4 谷氨酸棒状杆菌发酵用培养基和发酵条件 1.4.1 种子培养基30 g/L葡萄糖,1 g/L KH2PO4,0.4 g/L MgSO4,10 mg/L FeSO4,10 mg/L MnSO4,20 g/L (NH4)2SO4,40 g/L尿素,60 ml/L大豆浸出液,0.1 g/L DL-甲硫氨酸,50 μg/L生物素,200 μg/L维生素B1。NaOH调pH为6.3,121 ℃灭菌20 min。
1.4.2 发酵培养基120 g/L葡萄糖,1 g/L KH2PO4,0.4 g/L MgSO4,10 mg/L FeSO4,10 mg/L MnSO4,40 g/L (NH4)2SO4,10 mL/L大豆浸出液,0.7 g/L DL-甲硫氨酸,50 μg/L生物素,300 μg/L维生素B1,0.25 g/L L-异亮氨酸。NaOH调pH为7.2,摇瓶发酵需预先加入4 g/100 mL CaCO3以中和发酵过程产酸,115 ℃灭菌15 min。
1.4.3 摇瓶发酵菌种活化:将冻存于–70 ℃的甘油管保藏菌株于固体培养基上划线,30 ℃培养36 h。种子培养:利用接种环挑取活化好的新鲜菌苔,接种于装有25 mL种子培养基的250 mL三角瓶中,30 ℃、200 r/min培养16 h。摇瓶发酵:将培养好的种子液体按照初始OD562控制为1.0,接种入装有50 mL发酵培养基的500 mL三角瓶,必要时添加30 μg/mL卡那霉素维持菌株抗性,同样置于30 ℃、200 r/min条件下培养。含有质粒的重组菌株,在发酵进行到8 h时,添加1 mmol/L IPTG诱导重组载体上携带的基因进行表达。
1.4.4 5 L罐发酵菌种活化和种子培养参照摇瓶发酵,培养200 mL种子液,接种入装有2 L发酵培养基的5 L发酵罐。发酵过程中,通过流加50%的氨水的方式自动控制发酵培养基pH为7.2,通过夹套加热控制温度为30 ℃,通过发酵罐搅拌转速(200–800 r/min)和通气量(1.5 vvm)的偶联控制溶氧水平保持30%。发酵至8 h时,添加1 mmol/L IPTG诱导重组载体上携带的基因进行表达。当测得发酵液中葡萄糖质量浓度低于20 g/L时,流加质量浓度为80%的葡萄糖溶液,以保证菌株生长和产酸所必需的葡萄糖供应。每隔12 h取样,发酵培养96 h。
1.5 发酵过程参数分析发酵液中的葡萄糖浓度采用SBA-40C生物传感分析仪(山东省科学院生物研究所)测定;发酵液菌体OD562采用紫外可见分光光度计(UV-1800,日本岛津公司)测定;基于细胞OD562与细胞干重(Dry cell weight,DCW)的计算公式为:DCW=0.649 5×OD562–2.792 5,乘以稀释倍数计算获得[22];L-缬氨酸、丙氨酸的含量通过高效液相色谱(Agilient Technoligies 1200 series,USA)测定。
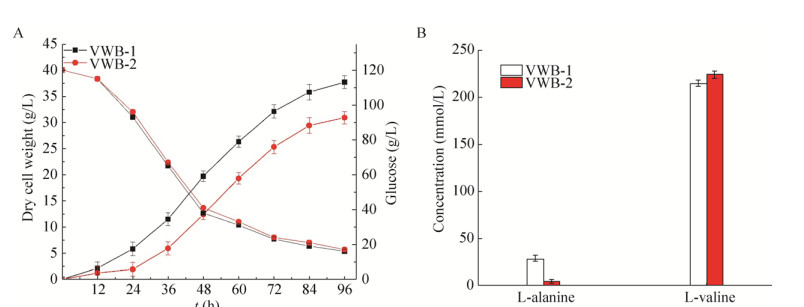
2 结果与分析 2.1 敲除alaT以降低胞内杂酸在前期的实验研究中我们发现,VWB-1在5 L发酵罐上的L-缬氨酸产量可以达到29.85 g/L,远高于ATCC 13869的1.42 g/L。通过HPLC对VWB-1发酵液中的氨基酸含量进行分析发现,VWB-1发酵过程中主要杂酸为丙氨酸[16]。根据文献报道,谷氨酸棒状杆菌中存在两条L-丙氨酸合成途径[20] (图 2)。丙氨酸氨基转移酶AlaT和AvtA催化丙酮酸生产L-丙氨酸的氨基供体分别为L-谷氨酸和L-缬氨酸。由AlaT催化下生成L-丙氨酸的途径为细胞内产生L-丙氨酸的主要途径,由AvtA催化生成L-丙氨酸的途径为次级途径。为了研究谷氨酸棒状杆菌中alaT在L-缬氨酸和L-丙氨酸合成中所起到的作用,我们对VWB-1中的丙氨酸氨基转移酶编码基因alaT进行敲除,得到突变菌株VWB-2。以L-缬氨酸高产菌株VWB-1作为对照菌株,对菌株VWB-2进行摇瓶发酵实验,并检测菌株的生长以及L-缬氨酸和L-丙氨酸合成情况(图 3)。
|
| 图 2 谷氨酸棒状杆菌中L-缬氨酸合成代谢途径 Figure 2 The biosynthetic pathway of L-valine in C. glutamicum. |
| |
|
| 图 3 alaT敲除突变株VWB-2生长(A)及产L-丙氨酸和L-缬氨酸水平检测(B) Figure 3 Growth curve of VWB-2 (A) and its L-Alanine and L-Valine producing level (B). |
| |
从图 3A中可以看出,alaT敲除后菌株的生长有轻微下调。如图 3B所示,比较VWB-1与VWB-2的L-丙氨酸产生情况可以发现,alaT缺失菌株的L-丙氨酸产量出现大幅度的下调,同时L-缬氨酸产量有轻微的上调。由此表明,alaT的缺失能够使谷氨酸棒状杆菌L-丙氨酸合成能力大幅下降,但同时菌体仍有一定的丙氨酸合成能力,以保证细胞正常生长,这可能与细胞内存在的另外一个丙氨酸氨基转移酶编码基因avtA有关。由于丙氨酸途径同时也是L-缬氨酸合成代谢的底物竞争途径(图 2),两条途径有共同的关键底物丙酮酸。对谷棒细胞中的丙氨酸途径进行阻断,一方面有利于减少发酵液中丙氨酸含量,降低下游分离纯化的成本,另一方面减少了L-缬氨酸途径的碳代谢流损耗,对L-缬氨酸合成有利,因此VWB-2可以作为L-缬氨酸代谢工程改造出发菌株。
2.2 ilvBN定点突变及其表达对谷氨酸棒状杆菌发酵产L-缬氨酸的影响谷氨酸棒状杆菌中的乙酰羟酸合酶(AHAS)是L-缬氨酸合成代谢中的关键酶,由2个亚基组成,分别是ilvB编码的催化亚基IlvB和ilvN编码的调节亚基IlvN[23]。AHAS同时也是细胞中3种支链氨基酸(L-缬氨酸、L-亮氨酸、L-异亮氨酸)合成的共用酶(图 2),且受到这3种支链氨基酸的反馈抑制,ilvBN基因的表达受3种支链氨基酸的调控。在L-缬氨酸生产中,乙酰羟酸合酶(AHAS)是L-缬氨酸合成代谢中的关键酶。根据文献报道,当乙酰羟酸合酶调节亚基IlvN 20–22位氨基酸由Gly-Ile-Ile突变为Asp-Asp-Phe时,能够解除这3种支链氨基酸对该酶的反馈抑制,从而有利L-缬氨酸的生产[24]。为了比较来源于ATCC 13869和VWB-1的ilvBN点突变前后AHAS对在L-缬氨酸代谢中作用程度的不同,我们将不同来源的ilvBN连接到表达载体pEC-XK99E上,构建了重组载体pEC-XK99E-ilvBN和pEC- XK99E-ilvBN1,并以此为模板构建了含有抗反馈抑制突变位点的重组载体pEC-XK99E-ilvBNM13和pEC-XK99E-ilvBN1M13,从图 4中的测序结果可以看出,ilvN点突变构建成功。将上述重组质粒转入到VWB-1中并进行摇瓶发酵,以比较不同的AHAS对菌株L-缬氨酸产量的影响(图 5)。如图 5A所示,ilvBN的表达对菌株生长均没有影响,进入到对数生长后期以及稳定期,含有重组质粒的菌株对葡萄糖的消耗逐渐增加,这是因为ilvBN的表达促进细胞合成L-缬氨酸,对底物葡萄糖的需求能力加强。分析菌株发酵液中的L-缬氨酸含量,如图 5B所示,ATCC 13869和VWB-1来源的ilvBN的过量表达均能够提高菌株的L-缬氨酸产量,而且在同等发酵条件下,表达含有突变位点M13的ilvBN基因的重组质粒对菌株L-缬氨酸产量的提升效果更为明显。由此表明,IlvN 20–22位氨基酸的突变能够更好地促进细胞代谢产L-缬氨酸,对L-缬氨酸生产有利。在之前的测序比对中,来源于VWB-1的AHAS与野生型AHAS相比存在9个氨基酸位点的突变,其中8个突变位点位于该酶催化亚基IlvB,分别为K30Q、G128S、A139V、H213R、A226S、Y252H、T362S、V562A,另外有一个突变位点H47L位于调节亚基IlvN[16]。相较于野生型来源的ilvBN和ilvBNM13,VWB-1来源的ilvBN1和ilvBN1M13的表达对L-缬氨酸产量提升的效果更为明显,推测原因可能是由于AHAS活性中心存在的突变位点加强了该酶与底物的结合能力[24],从而更利于发挥酶的催化功能。选用含有该重组质粒的菌株VWB-1/pEC-XK99E-ilvBN1M13作为进一步代谢改造的出发菌株。
|
| 图 4 AHAS调节亚基编码基因ilvN突变构建测序结果 Figure 4 The sequencing results of the mutated AHAS regulatory subunit coding gene ilvN. |
| |

|
| 图 5 抗反馈抑制AHAS的表达对L-缬氨酸产量影响(A:菌株生长;B:L-缬氨酸产量) Figure 5 Influence of the feedback resistant AHAS overexpression over the production of L-valine. (A) Growth curve and glucose consumption. (B) L-valine production level. |
| |
为了进一步提高菌株的L-缬氨酸生产能力,对L-缬氨酸合成代谢相关的一系列基因进行过量表达。谷氨酸棒状杆菌中L-缬氨酸的合成途径如图 2所示,从丙酮酸出发,L-缬氨酸的合成代谢主要经由乙酰羟酸合酶(AHAS)、乙酰羟酸同分异构酶(AHAIR)、二羟酸脱水酶(DHAD)和支链氨基酸转氨酶(TA)四个主要酶的催化下完成。已经有报道表明,相关基因的过量表达能够提高谷氨酸棒状杆菌的产L-缬氨酸水平[25-27]。本研究将AHAIR编码基因ilvC、DHAD的编码基因ilvD、TA编码基因ilvE通过酶切连接,串联入载体pEC-XK99E-ilvBN1M13,构建得到pEC- XK99E-ilvBN1M13CD和pEC-XK99E-ilvBN1M13CE,将上述质粒转入VWB-1中,进行摇瓶发酵实验。结果如图 6所示,在消耗相同葡萄糖的情况下,菌株VWB-1/pEC-XK99E-ilvBN1M13C的L-缬氨酸产量达到了321.1 mmol/L,相较于VWB-1/pEC-XK99E (214.3 mmol/L)提高了49.8%,而菌株VWB-1/pEC- XK99E-ilvBN1M13CD以及VWB-1/pEC-XK99E- ilvBN1M13CE的L-缬氨酸产量则分别为353.4 mmol/L和370.5 mmol/L,相较于VWB-1/pEC-XK99E对比分别提高了64.9%和72.9%。由此证明,VWB-1 L-缬氨酸合成途径关键基因ilvC、ilvD和ilvE的过量表达,均能够有效地提高谷棒菌株产L-缬氨酸的能力。

|
| 图 6 L-缬氨酸合成途径和转运途径关键基因的表达对产量的影响 Figure 6 Effect of the biosynthesis and transport related genes overexpression over the production of L-valine. (A) Growth curve and glucose consumption. (B) L-valine production level. |
| |
细胞内L-缬氨酸的积累会抑制相关酶的活性,对L-缬氨酸生产不利,因此L-缬氨酸在细胞体内合成后需要迅速地分泌到细胞外。在谷氨酸棒状杆菌中,负责L-缬氨酸胞外分泌的蛋白为分别由brnF和brnE编码的膜转运酶BrnF和BrnE,BrnFE同时也是细胞内L-亮氨酸、L-异亮氨酸和L-甲硫氨酸的转运蛋白,且brnFE的表达需要调控因子lrp的存在[28]。陈诚等在前期测序研究发现,VWB-1来源的lrp1基因编码的Lrp1蛋白相比于ATCC 13869来源的Lrp有1个点突变Arg39Trp,lrp和brnFE的共表达能够促进细胞L‑异亮氨酸、L-缬氨酸等的生产[21, 29]。将来源于ATCC 13869和VWB-1的lrp1基因分别在VWB-1中进行表达,发现两者均能提高细胞产L-缬氨酸的能力,且后者的作用更为明显。另外,Lrp1的表达能促进细胞内与L-缬氨酸合成和转运相关的基因ilvBC、ilvD、lrp和brnFE等的上调。为了提高细胞的L-缬氨酸胞外运输能力,我们以陈诚等[29]构建的质粒pJYW-4-lrp1-brnFE为模板,扩增得到lrp1-brnFE,与前述构建的重组载体pEC-XK99E‑ilvBN1M13CD以及pEC-XK99E- ilvBN1M13CE进行串联,构建得到pEC-XK99E- ilvBN1M13CD-lrp1-brnFE以及pEC-XK99E-ilvBN1M13 CE-lrp1-brnFE,将重组质粒转入到VWB-1中,进行摇瓶发酵实验。图 6B是重组菌株产L-缬氨酸能力比较,可以看出,VWB-1/pEC-XK99E-ilvBN1M13 CD-lrp1-brnFE以及VWB-1/pEC-XK99E-ilvBN1M13 CE-lrp1-brnFE的L-缬氨酸产量相较于没有lrp1-brnFE过表达菌株均有所提高,分别达到390.1 mmol/L和403.8 mmol/L,表明lrp1和brnFE的共表达能够提高谷棒菌株的产L-缬氨酸水平。通过与lrp和brnF的启动子的转录融合表达分析,3种支链氨基酸和L-甲硫氨酸刺激brnFE表达时需要调控因子Lrp的存在[28]。由于BrnFE负责细胞内L-缬氨酸的胞外转运,且Lrp1对brnFE的表达有正向的调控作用,因此,lrp1和brnFE的共表达能够提高谷棒菌株VWB-1胞内的BrnFE蛋白水平,从而提高细胞支链氨基酸转运能力。细胞内合成的过量的L-缬氨酸被BrnFE大量的转移到胞外,同时加快了L-缬氨酸的合成速度,从而更有利于L-缬氨酸生产。
2.5 L-缬氨酸合成及转运途径相关基因的共表达以提高L-缬氨酸产量作者通过基因敲除获得了VWB-1的丙氨酸氨基转移酶编码基因alaT的敲除突变株VWB-2,降低杂酸丙氨酸生成的同时减少了丙氨酸途径对L-缬氨酸合成底物丙酮酸的竞争。并且通过构建重组质粒的方式,将细胞中参与L-缬氨酸代谢合成相关基因和转运相关基因进行过量表达,显著提高了细胞了L-缬氨酸合成能力和转运水平。为了更好地提高细胞产L-缬氨酸的水平,同时降低杂酸丙氨酸的含量,我们将对L-缬氨酸产量提高作用最为显著的质粒pEC-XK99E-ilvBN1M13CE- lrp1-brnFE转入到VWB-2中,构建得到VWB-2/ pEC-XK99E-ilvBN1M13CE-lrp1-brnFE。摇瓶水平发酵表明,上述菌株的L-缬氨酸产量相较于VWB-1/ pEC-XK99E-ilvBN1M13CE-lrp1-brnFE有进一步提高,且菌株发酵液中的杂酸丙氨酸含量由30.5 mmol/L下降为5.3 mmol/L,降幅达到82.6%。进一步对该菌株进行5 L罐发酵实验,检测菌株在发酵过程中各个时期的生长特性、糖耗水平以及产L-缬氨酸能力。如图 7所示,当发酵进行到60 h时,由于葡萄糖即将耗尽,因此进行糖液的补充,提供碳源以使菌株继续生长和产酸。发酵96 h后,菌株的细胞干重达到47.2 g/L,L-缬氨酸产量达到461.4 mmol/L,相较于原始菌株提高了115.3%,产率0.564 g/(L·h),比产量1.162 g/(L·g DCW),糖酸转化率达到0.312 g/g葡萄糖。当发酵进入到96 h时,菌株仍然维持比较高的生长和耗糖状态,且产L-缬氨酸水平仍有上升趋势。考虑到发酵周期过度延长可能降低整个发酵过程的产物得率,所以可选择在96 h时结束发酵。当然,该菌株产L-缬氨酸的水平仍有望进一步提高,后期可以进行更深入的代谢工程改造,并对发酵培养基的成分以及发酵策略进行优化,进一步提高菌株产量。

|
| 图 7 5 L罐水平检测菌株生长及生产性能 Figure 7 Growth and L-valine production performance detection of the recombinant strain in 5 L fermentator. |
| |
作为一种重要的支链氨基酸,L-缬氨酸的工业生产一直备受关注。目前,微生物发酵法正取代直接提取法和化学合成法而成为L-缬氨酸生产的主要方法。但是许多工业上应用的L-缬氨酸生产菌株为多次诱变筛选获得,带来的二次有害突变会导致菌株生长缓慢、糖耗降低、杂酸等副产物增多,具有盲目性和不确定性。因此,合理、高效的设计在谷氨酸棒状杆菌代谢工程改造中便显得尤为重要,生产菌株不仅应该有较高的L-缬氨酸生产能力,还要有最少化的副产物积累,从而简化随后的分离和纯化过程,减少生产成本。例如,溶氧速率对生物反应过程有较大影响,进行菌株改造的时候,需要对此加以考虑[30]。微生物分子生物学的发展使人们重新认识了L-缬氨酸生产菌株,DNA重组技术和诱导技术的出现促使代谢工程取代传统育种成为主流的菌株改造策略。近年来,利用创新的基因编辑技术如CRISPR/Cas9系统,研究人员可以在谷氨酸棒状杆菌中快速高效地进行基因编辑,包括基因敲除、插入和替换等,为代谢途径的改造提供了有力的工具,以此促进谷氨酸棒状杆菌的代谢工程育种进程[31-33]。深入了解L-缬氨酸生物合成的代谢和调节机制对谷氨酸棒状杆菌整体代谢工程策略的设计至关重要,未来的研究需要更多的关注谷氨酸棒状杆菌细胞的代谢网络。随着系统生物学和合成生物学技术的快速发展,合理利用转录组学、蛋白组学、代谢组学和计算机建模等现代生物技术,解析L-缬氨酸生产菌株繁杂的合成代谢途径以及多重的调控机制,由点到面、从局部到整体对生产菌株进行改造,提升生产性能[3, 34]。
本研究发现谷氨酸棒状杆菌中丙氨酸氨基转移酶基因alaT的缺失能够降低L-缬氨酸生产菌株发酵液丙氨酸的含量,以此削弱细胞内丙氨酸途径对L-缬氨酸代谢途径关键前体丙酮酸的竞争,同时降低下游分离纯化的成本。Wada等[35]敲除了L-缬氨酸生产菌株ATCC13032ΔilvAΔpanBC pJC1ilvBNCD中的alaT基因,同样极大地降低了细胞内L-丙氨酸的积累。串联表达L-缬氨酸合成途径关键基因ilvBN1M13、ilvC、ilvD、ilvE,能够将细胞内的碳代谢流引入L-缬氨酸途径,在L‑缬氨酸生产菌株代谢改造中非常关键[36]。在减少副产物、加强主通路碳代谢流的基础上,本研究在谷氨酸棒状杆菌中过表达L-缬氨酸转运途径相关基因brnFE及其调控蛋白lrp1,能够显著提高细胞产L-缬氨酸的能力及其转运能力,从而提高菌株L-缬氨酸产量。Chen等[21]在ATCC 13869过表达lrp1-brnFE,同样提高了菌株的L-缬氨酸生产性能。对菌株VWB-2/pEC-XK99E-ilvBN1M13CE- lrp1-brnFE进行补料分批发酵实验,发酵96 h时L-缬氨酸产量达到461.4 mmol/L,糖酸转化率达到0.312 g/g葡萄糖,使L-缬氨酸产量相较于原始菌株大幅度提高,同时菌株发酵液中的杂酸液降到较低的水平。通过与已经报道的L-缬氨酸高产菌株进行比较,该菌株L-缬氨酸产量处于中游水平,仍有较大提升空间[37]。另外,本研究构建的重组菌株均通过导入外源质粒的方法来提高关键基因的表达水平,后期还可以通过染色体重组技术,例如将野生型基因进行突变使其能够抗支链氨基酸反馈抑制、增加重要基因在染色体上的拷贝数、加强野生型启动子的活性等,进一步提高菌株生产性能。
致谢: 感谢Institute of Biotechnology (Forschungszentrum Julich) Lothar Eggeling教授为本研究提供alaT的敲除载体pK19mobsacBalaT。| [1] | Karau A, Grayson I. Amino acids in human and animal nutrition//Zorn H, Czermak P, Eds. Biotechnology of Food and Feed Additives. Berlin: Springer, 2014: 189-228. |
| [2] | Takpho N, Watanabe D, Takagi H. High-level production of valine by expression of the feedback inhibition-insensitive acetohydroxyacid synthase in Saccharomyces cerevisiae. Metab Eng, 2018, 46: 60–67. DOI: 10.1016/j.ymben.2018.02.011 |
| [3] | Park JH, Lee KH, Kim TY, et al. Metabolic engineering of Escherichia coli for the production of L-valine based on transcriptome analysis and in silico gene knockout simulation. Proc Natl Acad Sci USA, 2007, 104(19): 7797–7802. DOI: 10.1073/pnas.0702609104 |
| [4] | Park Y, Park JH, Park S, et al. Enhanced cellular uptake and pharmacokinetic characteristics of doxorubicin-valine amide prodrug. Molecules, 2016, 21(10): E1272. DOI: 10.3390/molecules21101272 |
| [5] | Hermann T. Industrial production of amino acids by coryneform bacteria. J Biotechnol, 2003, 104(1/3): 155–172. |
| [6] | Nielsen J. Metabolic engineering: techniques for analysis of targets for genetic manipulations. Biotechnol Bioeng, 1998, 58(2/3): 125–132. |
| [7] | Bailey JE. Toward a science of metabolic engineering. Science, 1991, 252(5013): 1668–1675. DOI: 10.1126/science.2047876 |
| [8] | Radmacher E, Vaitsikova A, Burger U, et al. Linking central metabolism with increased pathway flux: L-valine accumulation by Corynebacterium glutamicum. Appl Environ Microbiol, 2002, 68(5): 2246–2250. DOI: 10.1128/AEM.68.5.2246-2250.2002 |
| [9] | Xu DQ, Tan YZ, Huan XJ, et al. Construction of a novel shuttle vector for use in Brevibacterium flavum, an industrial amino acid producer. J Microbiol Methods, 2010, 80(1): 86–92. DOI: 10.1016/j.mimet.2009.11.003 |
| [10] | Xu DQ, Tan YZ, Shi F, et al. An improved shuttle vector constructed for metabolic engineering research in Corynebacterium glutamicum. Plasmid, 2010, 64(2): 85–91. DOI: 10.1016/j.plasmid.2010.05.004 |
| [11] | Xu DQ, Tan YZ, Li Y, et al. Construction of a novel promoter-probe vector and its application for screening strong promoter for Brevibacterium flavum metabolic engineering. World J Microbiol Biotechnol, 2011, 27(4): 961–968. DOI: 10.1007/s11274-010-0539-8 |
| [12] | Tan YZ, Xu DQ, Li Y, et al. Construction of a novel sacB-based system for marker-free gene deletion in Corynebacterium glutamicum. Plasmid, 2012, 67(1): 44–52. DOI: 10.1016/j.plasmid.2011.11.001 |
| [13] | Hu JY, Tan YZ, Li YY, et al. Construction and application of an efficient multiple-gene-deletion system in Corynebacterium glutamicum. Plasmid, 2013, 70(3): 303–313. DOI: 10.1016/j.plasmid.2013.07.001 |
| [14] | Holátko J, Elišáková V, Prouza M, et al. Metabolic engineering of the L-valine biosynthesis pathway in Corynebacterium glutamicum using promoter activity modulation. J Biotechnol, 2009, 139(3): 203–210. DOI: 10.1016/j.jbiotec.2008.12.005 |
| [15] |
Wang XY. Metabolic engineering in corynebacterium glutamicum to increase L-valine production.
J Food Sci Biotechnol, 2012, 31(3): 225–231.
(in Chinese). 王小元. 谷氨酸棒杆菌生产缬氨酸的代谢工程研究进展. 食品与生物技术学报, 2012, 31(3): 225-231. DOI:10.3969/j.issn.1673-1689.2012.03.001 |
| [16] | Zhang HL, Li YY, Wang CH, et al. Understanding the high L-valine production in Corynebacterium glutamicum VWB-1 using transcriptomics and proteomics. Sci Rep, 2018, 8: 3632. DOI: 10.1038/s41598-018-21926-5 |
| [17] | Liu Q, Ouyang SP, Kim J, et al. The impact of PHB accumulation on L-glutamate production by recombinant Corynebacterium glutamicum. J Biotechnol, 2007, 132(3): 273–279. DOI: 10.1016/j.jbiotec.2007.03.014 |
| [18] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. 3rd ed. New York: CSH Laboratroy Press, 2001. |
| [19] | Kalinowski J, Bathe B, Bartels D, et al. The complete Corynebacterium glutamicum ATCC 13032 genome sequence and its impact on the production of L-aspartate-derived amino acids and vitamins. J Biotechnol, 2003, 104(1/3): 5–25. |
| [20] | Marienhagen J, Eggeling L. Metabolic function of Corynebacterium glutamicum aminotransferases AlaT and AvtA and impact on L-valine production. Appl Environ Microbiol, 2008, 74(24): 7457–7462. DOI: 10.1128/AEM.01025-08 |
| [21] | Chen C, Li YY, Hu JY, et al. Metabolic engineering of Corynebacterium glutamicum ATCC13869 for L-valine production. Metab Eng, 2015, 29: 66–75. DOI: 10.1016/j.ymben.2015.03.004 |
| [22] | Yin L, Shi F, Hu X, et al. Increasing l-isoleucine production in Corynebacterium glutamicum by overexpressing global regulator Lrp and two-component export system BrnFE. J Appl Microbiol, 2013, 114(5): 1369–1377. DOI: 10.1111/jam.2013.114.issue-5 |
| [23] | Blombach B, Hans S, Bathe B, et al. Acetohydroxyacid synthase, a novel target for improvement of L-lysine production by Corynebacterium glutamicum. Appl Environ Microbiol, 2009, 75(2): 419–427. DOI: 10.1128/AEM.01844-08 |
| [24] | Elišáková V, Pátek M, Holátko J, et al. Feedback-resistant acetohydroxy acid synthase increases valine production in Corynebacterium glutamicum. Appl Environ Microbiol, 2005, 71(1): 207–213. DOI: 10.1128/AEM.71.1.207-213.2005 |
| [25] | Blombach B, Arndt A, Auchter M, et al. L-valine production during growth of pyruvate dehydrogenase complex-deficient Corynebacterium glutamicum in the presence of ethanol or by inactivation of the transcriptional regulator SugR. Appl Environ Microbiol, 2009, 75(4): 1197–1200. DOI: 10.1128/AEM.02351-08 |
| [26] | Bartek T, Znnchen E, Klein B, et al. Analysing overexpression of L-valine biosynthesis genes in pyruvate-dehydrogenase-deficient Corynebacterium glutamicum. J Ind Microbiol Biotechnol, 2010, 37(3): 263–270. DOI: 10.1007/s10295-009-0669-x |
| [27] | Hou XH, Ge XY, Wu D, et al. Improvement of L-valine production at high temperature in Brevibacterium flavum by overexpressing ilvEBNrC genes. J Ind Microbiol Biotechnol, 2012, 39(1): 63–72. DOI: 10.1007/s10295-011-1000-1 |
| [28] | Lange CT, Mustafi N, Frunzke J, et al. Lrp of Corynebacterium glutamicum controls expression of the brnFE operon encoding the export system for L-methionine and branched-chain amino acids. J Biotechnol, 2012, 158(4): 231–241. DOI: 10.1016/j.jbiotec.2011.06.003 |
| [29] |
Chen C, Li YY, Yin LH, et al. Expression of global regulator Lrp facilitates L-valine production in corynebacterium glutamicum.
J Food Sci Biotechnol, 2016, 35(9): 920–927.
(in Chinese). 陈诚, 李颜颜, 尹良鸿, 等. 全局调控因子Lrp的表达强化谷氨酸棒状杆菌发酵生产L-缬氨酸. 食品与生物技术学报, 2016, 35(9): 920-927. DOI:10.3969/j.issn.1673-1689.2016.09.004 |
| [30] | Ka F, Prasad A, Tillack J, et al. Rapid assessment of oxygen transfer impact for Corynebacterium glutamicum. Bioprocess Biosyst Eng, 2014, 37(12): 2567–2577. DOI: 10.1007/s00449-014-1234-1 |
| [31] | Cleto S, Jensen JV, Wendisch VF, et al. Corynebacterium glutamicum Metabolic Engineering with CRISPR Interference (CRISPRi). ACS Synth Biol, 2016, 5(5): 375–385. DOI: 10.1021/acssynbio.5b00216 |
| [32] | Cho JS, Choi KR, Prabowo CS, et al. CRISPR/Cas9-coupled recombineering for metabolic engineering of Corynebacterium glutamicum. Metab Eng, 2017, 42: 157–167. DOI: 10.1016/j.ymben.2017.06.010 |
| [33] | Peng F, Wang XY, Sun Y, et al. Efficient gene editing in Corynebacterium glutamicum using the CRISPR/Cas9 system. Microb Cell Fact, 2017, 16: 201. DOI: 10.1186/s12934-017-0814-6 |
| [34] | Poetsch A, Haumann U, Burkovski A. Proteomics of corynebacteria: from biotechnology workhorses to pathogens. Proteomics, 2011, 11(15): 3244–3255. DOI: 10.1002/pmic.201000786 |
| [35] | Wada M, Hijikata N, Aoki R, et al. Enhanced valine production in Corynebacterium glutamicum with defective H+-ATPase and C-terminal truncated acetohydroxyacid synthase. Biosci Biotechnol Biochem, 2008, 72(11): 2959–2965. DOI: 10.1271/bbb.80434 |
| [36] | Hasegawa S, Suda M, Uematsu K, et al. Engineering of Corynebacterium glutamicum for high-yield L-valine production under oxygen deprivation conditions. Appl Environ Microbiol, 2013, 79(4): 1250–1257. |
| [37] | Wang X, Zhang H, Quinn PJ. Production of L-valine from metabolically engineered Corynebacterium glutamicum. Appl Microbiol Biotechnol, 2018(1): 1–12. |
 2018, Vol. 34
2018, Vol. 34




