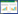中国科学院微生物研究所、中国微生物学会主办
文章信息
- 倪磊, 金震宇, 杨帅, 金帆
- Lei Ni, Zhenyu Jin, Shuai Yang, Fan Jin
- 铜绿假单胞菌蹭行运动单细胞分析方法的建立及应用
- Single-cell analysis method for twitching motility of Pseudomonas aeruginosa
- 生物工程学报, 2017, 33(9): 1611-1624
- Chinese Journal of Biotechnology, 2017, 33(9): 1611-1624
- 10.13345/j.cjb.170131
-
文章历史
- Received: March 31, 2017
- Accepted: May 19, 2017
2 中国科学技术大学 中国科学院软物质化学重点实验室, 安徽 合肥 230026
2 CAS Key Laboratory of Soft Matter Chemistry, University of Science and Technology of China, Hefei 230026, Anhui, China
蹭行运动特指细菌利用四型菌毛在固体表面上爬行,在假单胞菌和奈瑟菌属中十分常见[1]。虽然发现的早,但对蹭行运动早年研究不多,直到发现其与生物被膜的紧密联系后才获得广泛关注。细菌生物被膜是细菌粘附于表面,通过分泌多种多糖基质、蛋白、DNA等胞外聚合物,将其自身包裹起来而形成的大量细菌聚集膜状物。生物被膜会在人体器官和医疗移植的器官表面形成而造成感染和移植手术失败[2],在工业管道内部产生后易导致管道堵塞而大大增加能耗成本。某些细菌产生的生物被膜已经成为危害人类健康的重要杀手,据估计人体中约80%的细菌感染由生物被膜造成[3],其中铜绿假单胞菌的感染尤为常见,常引起肺炎、尿道炎和中耳炎等疾病。铜绿假单胞菌形成的生物被膜也是许多院内感染和术后感染的病原,由于其对很多抗生素天生抗药且易在治疗中产生抗药性,很多感染难以治愈[4]。欲防治生物被膜,需首先理解生物被膜形成的关键因素以及生物被膜难以铲除的原因,部分研究者把目光聚焦于细菌的蹭行运动。
近两年,不断出现蹭行运动研究领域的突破性工作[5-8]。细菌蹭行运动与生物被膜形成的关系主要体现在3个方面:1) 细菌通过四型菌毛的收缩和释放,增强与表面之间的粘附并促进细菌之间的相互接触。Comolli等发现拥有四型菌毛但菌毛无法收缩的pilT、pilU突变株对人上皮细胞和小鼠组织表面的粘附均明显减弱[9];2) 如何进行蹭行运动决定了细菌的表面扩张能力和生物被膜的三维结构。低流场下的成熟生物被膜通常以蘑菇状的形态存在于表面,O’Toole等发现细菌粘附到表面之后会首先形成微菌落,这些微菌落对最终的生物被膜结构具有决定性作用,而对细菌基因组进行大范围筛选发现无法合成四型菌毛的相关突变株无法在表面形成小菌落,最后只能形成薄薄的单层[10]。Klausen等利用荧光共聚焦的方法对生物被膜进行长时间跟踪观测,发现蹭行运动突变株丧失了从微菌落向外继续扩散的能力,因此无法完成整个生物被膜的动态演化过程,最终无法形成成熟生物被膜[11]。Zhao等通过细菌追踪和Psl染色的方法证明,铜绿假单胞菌的蹭行运动协助细菌胞外多糖Psl在表面上的分布并反过来引导细菌的运动、改变粘附行为,从而导致微菌落的形成[12]。3) 蹭行运动主导了细菌在表面上粘附之后的表型转变。Persat等发现铜绿假单胞菌粘附表面后其四型菌毛的伸缩过程启动了甲基化蛋白PilJ,进而经过cAMP、Vfr通路完成了从表面机械信号到细菌内部基因表达信号的转化[5]。Luo等证明PilY1蛋白可以调节环鸟苷二磷酸(c-di-GMP)的合成,同时环腺苷一磷酸(cAMP)、Vfr、AlgR路径可调节PilY1的表达,以此将整个菌毛机械感应机制引向c-di-GMP调节系统[13]。由此可见,蹭行运动既决定了生物被膜成熟结构的形成,又在细菌粘附表面后介导表型转变,对生物被膜的形成十分关键。有学者已经开始尝试通过加入诱导剂和合成不同微结构的材料来改变细菌的蹭行行为,从而达到抗菌的目的[14-15]。因此对蹭行运动的系统性表征在生物被膜构成原理的理解及防治方法的探索中都至关重要。
传统的表征方法是将细菌戳到琼脂板的底部,通过测量细菌群落边界的大小来描述其蹭行运动的能力[16]。这种方法只能从宏观上粗略地定量,无法得到蹭行运动的细节信息,且观察到的菌斑是由多细菌集群化协同运动形成,与细致观测的目标相去甚远。欲深入理解蹭行运动如何决定生物被膜的结构以及介导细菌表型转变,必须从单细菌层面出发,对表面上细菌从粘附到长成生物被膜的过程进行长期细致的跟踪观测和定量分析。另一种方法是选取少数细菌用显微镜记录其运动过程,然后引入粒子追踪算法进行运动轨迹的分析[14, 16]。这种方法可以在单细菌水平上定量研究蹭行运动,但只能进行少量细菌的统计且低效费时,更无法进行推广。且已有的研究发现蹭行运动具有多样性的特点,在表面上细菌之间运动表型差距非常大,运动有快有慢,有站立行走有贴伏爬行,弹射运动或频繁或稀少[17-19],因此对少数细菌的观测无法真正全面理解蹭行运动的本质,必须进行高通量统计分析。
我们在此建立一套集成自动化数据采集和图像处理、图形化输出的方法,系统地描述细菌运动速度、运动轨迹、方向以及转动、弹射运动等情况。同时,改变以往的实验方法以实现大量运动数据的高速采集,从而最终完成对细菌在固体表面蹭行运动的全面统计分析,并利用这一方法对群体感知机制与蹭行运动的关系进行了定量表征。
1 方法 1.1 对铜绿假单胞菌在固体表面的蹭行运动行为进行原位、高通量、自动化的数据采集 1.1.1 细菌培养实验中使用的菌种为铜绿假单胞菌(ATCC15692) ΔfliM和ΔfliCΔpilA菌株,均由美国圣母大学Joshua D. Shrout实验室提供,敲除fliM基因是为了消除细菌鞭毛对蹭行运动的干扰。首先从-80 ℃冰箱中取出冻存的菌种,在含有1.5%琼脂的LB培养板上划线,放在恒温培养箱中37 ℃孵育18 h。待长出菌斑后,挑一个单克隆菌斑接种于1 mL限制性培养基FAB+30 mmol/L谷氨酸钠,在37 ℃恒温条件下、200 r/min摇菌培养10 h。在对数期OD600约0.8收集细菌,按体积比1:30稀释到1 mL新鲜的FAB中。LB培养基成分:氯化钠10 g/L,酵母提取物5 g/L,蛋白胨10 g/L。FAB培养基成分:硫酸铵2 g/L,十二水合磷酸氢二钠12 g/L,磷酸二氢钾3 g/L,氯化钠3 g/L,氯化镁93 mg/L,二水合氯化钙14 mg/L,二水合硫酸钙200 μg/L,七水合硫酸亚铁200 μg/L,一水合硫酸锰20 μg/L,五水合硫酸铜20 μg/L,七水合硫酸锌20 μg/L,七水合硫酸钴10 μg/L,钼酸钠Na2MoO4 10 μg/L,硼酸5 μg/L。
连接好装置后,向flow cell (丹麦科技大学)中提供FAB+0.6 mmol/L谷氨酸钠培养基。在群体感知实验中则首先使用纯化后的DMSO分别配制一级(3-oxo-C12-HSL)、二级(C4-HSL)信号分子(购自Sigma Aldrich公司)的10 mmol/L母液,按终浓度10 μmol/L加入到FAB+0.6 mmol/L培养基中。先除去装置中的气泡,再将稀释好的菌液用1 mL无菌注射器注入通道中,用玻璃胶黏住注菌孔。将整个装置放在显微镜载物台上静置约20 min,待玻片表面上粘附足够数目的细菌后,快速冲走上方悬浮的细菌,然后将注射器泵流速设置为3 mL/h。
1.1.2 细菌蹭行运动视频的自动化采集在显微镜上加载ZDC2连续自动对焦防漂系统,保证高速拍照时焦平面的稳定;加载Ludl XY反馈载物台,防止样品在水平方向的漂移;加载Tokai Hit快速反馈加热台以最大限度减小温度涨落;亮场照明采用稳定、可控的LED灯作为光源,并加入长波滤光片以减轻光照对细菌的损伤。图像采集使用Andor sCMOS相机,像素2 560×2 160,可高速地采集图像。显微镜机架为Olympus IX81光学系统,配备100×Olympus油镜,数值孔径1.4。使用Andor IQ 3.1软件来集成以上设备,使各设备同步运行。实验中控温30 ℃,图像采集帧率为10帧/s,先将焦面移到远离细菌的位置,拍摄一张照片作为背景图像,用作后期的图像校正。选定一个细菌数目适中的视野拍摄20 000张照片,然后移动到下一个视野再拍一张背景图像,继续拍摄20 000张照片。如此重复拍照8个视野,实验结束。每次实验共计可获得10 000张照片约1 000个细菌运动轨迹,将近1 000万个瞬时速度点。
1.2 基于MATLAB图像处理平台编写程序包,对蹭行运动原始图像进行高速、自动化的定量分析 1.2.1 原始图像的背景校正在每个视野观测前,首先将焦面向下方调节200 μm,拍摄一张没有细菌的照片作为背景照片,然后把所有拍摄的照片用单个视野的背景照片做除法校正。
1.2.2 细菌的识别第一步,对背景校正后的图像做高斯滤波,使图像更加平滑,降低图像噪音;第二步,用边缘滤波器使图像增强,细菌边缘的明暗区分更显著;第三步,用高斯滤波减小边缘滤波造成的噪音;第四步,调节图像的亮度阈值,把第三步得到的图像转化为二值图像;第五步,根据细菌的大小,把明显小于细菌通常尺寸的二值连通区域删除掉,再补全细菌轮廓上可能的空洞。
1.2.3 图像基本信息的获取MATLAB使用的是椭圆拟合的方法,根据所得的椭圆给出待测连通域的各个参数,细菌可以看作是一个长圆形,椭圆拟合之后,椭圆的长轴、短轴的长度代表细菌的长度和宽度,椭圆两个顶点AB即为细菌的两个极端的位置,椭圆的中心就是细菌的投影中心位置。椭圆的长轴与水平方向的夹角(-90°-90°)即为细菌在平面上的取向角。
1.2.4 细菌的追踪将二值平面图像时间序列在空间重组,生成三维图像矩阵,再逐层扫描矩阵,确定三维空间中的连通域和连通域中的节点。根据三维连通性重组、拆分原始图像,自动生成分叉的拓扑数据结构。
1.2.5 数据的初步筛选和减噪首先删除追踪时间低于200 s的数据,这些短的数据多是由于接触碰撞导致某些细菌被反复重新识别,或是细菌在将要分裂又未能完全分离时反复被识别,对统计分析的贡献不大;然后删除0.1 s内细菌长度变化超过10个像素的数据,这些数据是由于拍照时焦面发生波动或者细菌之间的相互接触。筛选后记录的位置和长度数据都包含着巨大的噪音,采用小波减噪,减噪后的结果很好保留了高频和低频的信号。先对细菌的长度、方向角、细菌中心的横纵坐标做小波减噪,再根据以上结果算出细菌两端的位置,进而完成对运动速度、轨迹的计算。
1.2.6 生长、运动等各参数的计算对端点轨迹坐标做差分可得细菌两端点横纵坐标的速度分量,合成得到速度矢量。根据细菌主轴所在的方向,将速度矢量在平行和垂直于主轴的两个方向分解得切向速度分量和法向速度分量。对其中一个端点所有时刻的切向位移求和可得该细菌在观测时间窗口内沿着主轴方向的总位移。若该值大于零,则将该端定为头端,反之则将另一端定为头端。切向速度分量沿着头端方向定为正,法向速度分量自主轴顺时针方向定为正。
细菌生长速率分两类计算,对于贴伏于表面的细菌对不同时间菌长值进行线性拟合得到生长速率,这种情况下倾角定为0°。对于翘起的细菌先估计其实际长度,得到估计的生长速率。根据细菌的长度和宽度算出翘起的倾角:α=acos((le-b)/(l-b)),其中le为估计菌长,l, b为实际观测所得菌长和菌宽。
1.2.7 对单细菌蹭行运动的轨迹和速度时间序列进行分析首先作运动轨迹均方末端距(MSD)与时间间隔τ的关系图,各取对数后线性拟合得到均方末端距斜率kMSD值,作为评价细菌运动轨迹曲折程度的参量。对细菌运动速度矢量进行相关性分析,计算细菌在蹭行运动过程中速度矢量在时间上的自相关函数数值,corrV(τ)≡ < v(t)·v(t+τ) > ,速度自相关函数用双指数拟合之后可得特征衰减时间。
1.2.8 建立蹭行运动数据库将所有采集到的蹭行运动参数抽提出来,包括运动速度模量、kMSD、弹射频率、生长速率、转动速度、行走细菌比例等,生成总数据列表。获得数据库之后,对各运动参数进行两两作图,具体做法是将限制条件之内的数据点画在二维图上,每个数据点代表一个细菌的平均坐标位置。然后对各数据点做二维高斯卷积,得到较为平滑的热图。此外,将所有符合条件的细菌样本中的分布数据提取出来并加以合并,归一化之后作速度模量分布、角度分布、速度自相关函数图等。增加筛选功能,以比较实验中不同细菌群体的运动情况。
2 结果与分析 2.1 蹭行运动速度分布的柯西拟合首先将细菌的运动速度在切向和法向进行分解,如图 1A所示。然后分别计算切向速度和法向速度的概率密度分布,用柯西分布拟合。细菌在蹭行运动时处于小雷诺数的力学环境中,菌毛输出功W基本上完全消耗于细菌与表面摩擦中,运动速度为菌毛输出功率P与摩擦力f的比值。P和f均有符合正态分布的特性,因此运动速度呈柯西分布。柯西分布关系式:f(x, a, b)=b/(π(x-a)2+b2)。
|
| 图 1 细菌蹭行运动速度分布的柯西分布拟合(对单一示例细菌的切向速度分布(A)和法向速度分布(B)原始数据的柯西分布拟合;3个不同细菌的切向速度分布(C)和柯西分布拟合参数比较(D). (A)图右上方插图为细菌运动速度矢量分解示意图. (A)、(C)图横坐标为细菌运动速度切向分量大小,纵坐标为对应切向速度分量的概率密度值. (B)图横坐标为细菌运动速度法向分量大小,纵坐标为对应法向速度分量的概率密度值. (D)图横坐标为细菌的序号,纵坐标为切向速度分量分布的中值或半峰宽) Figure 1 Cauchy distribution fitting of the velocity distribution of bacterial twitching motility. Cauchy distribution fitting of the distribution of tangential velocity (A) and normal velocity (B) of a example bacterial cell. Campararion of the tangential velocity distribution (C) and fitting parameters (D) of three bacteria with different twitching motility type. The illustration of velocity vector decomposion was shown upright to caption (A). The X axis of (A) and (C) is the value of tangential velocity, Y axis is the probability density. The X axis of (B) is the value of normal velocity, Y axis is the probability density. The X axis of (D) is the cell serial number and Y axis is the medium value or pick width at half height of the distribution of tangential velocity. |
| |
实验中我们用柯西分布中值(v||, lead, a)来描述细菌运动的快慢程度,柯西分布半峰宽(v||, lead, b)来描述细菌运动速度的波动程度。图 1A、1B为分别对切向速度和法向速度分布进行柯西分布拟合,图 1C、1D所示为实验中采集到的3个不同细菌的速度分布图及柯西分布拟合参数的比较。由图 1D可知,1号、3号细菌速度波动较为接近,但1号细菌明显运动更快;2号、3号细菌运动速度大小接近,但2号细菌运动速度的波动性更大。
2.2 蹭行运动运动速度矢量的自相关函数细菌蹭行运动速度时间自相关函数corrV(τ)≡ < v(t)·v(t+τ) > 服从双指数衰减(corrV(τ)=A1·exp(-τ/τ1)+A2·exp(-τ/τ2)+A3),做拟合之后可得特征衰减时间。如图 2A所示,该细菌运动速度的快衰减特征时间为0.48 s,预示着单根菌毛拉伸的持续时间。慢衰减特征时间为4.07 s,预示着多根菌毛协同拉伸的持续时间。两衰减幅值(A1, A2)较为接近。图 2B比较了不同细菌的速度时间自相关函数,可见不同细菌速度衰减过程有着显著差别。值得注意的是,细菌的自速度相关函数曲线上常出现箭头所指的突出部分,在后面弹射运动分析时再做讨论。

|
| 图 2 细菌蹭行运动速度矢量的时间自相关函数以及双指数拟合. (A)单一细菌蹭行运动速度矢量的自相关函数以及双指数拟合;(B)比较3个不同细菌的自相关函数,右上小图为3个细菌各自的快衰减过程特征衰减时间(τ1). (A)、(B)图横坐标为时间间隔,纵坐标为相应时间间隔下的速度相关函数值 Figure 2 Temporal autocorrelation function of the velocity vector of bacteria twitching motility. (A) Temporal autocorrelation function and doulbe-exponential fitting of velocity vector of one single bacterial cell. (B) Comparation of temporal autocorrelation function of three differert bacteria, top right graph is the comparation of the characteristic time of fast decay process (τ1). The X axis of (A) and (B) is the time interval, Y axis is the corresponding value of autocorrelation function. |
| |
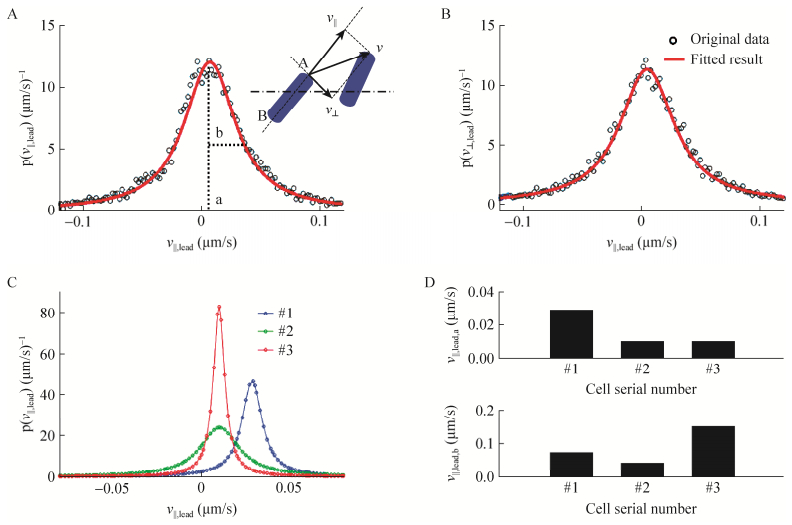
建立蹭行运动数据库之后,设定不同的筛选条件可以实现对不同细菌群体的蹭行运动情况进行比较。图 3A比较了细菌粘附表面之后在不同时间段的运动速度和运动轨迹情况。kMSD基本一致,说明不同时间段细菌的运动轨迹无明显区别。但在粘附表面之初的1.6 h内,细菌蹭行运动速度明显小于后期,提示在初始粘附阶段可能存在着表面适应的过程。图 3B给出了细菌蹭行运动在高速区域(弹射)和低速区域(平移)的分布情况,可见弹射运动主要沿着细菌的两侧法向方向,而平移则主要沿着细菌长轴方向。
|
| 图 3 多细菌蹭行运动参数的统计分析. (A)对1 000个细菌在粘附表面后不同时间段的蹭行运动速度和轨迹的统计分析;横坐标为对运动速度统计后进行柯西分布拟合所得切向速度分量的中值,纵坐标为对运动轨迹均方末端距随时间变化在双对数坐标下线性拟合所得斜率值. (B)对1 000个细菌蹭行运动速度矢量在高速部分(左图)和低速部分(右图)的统计热图.横坐标为细菌运动速度的法向分量值,纵坐标为细菌运动速度的切向分量值.该热图是首先把所有细菌每一个时刻的运动速度矢量画在平面上,然后逐个做二维高斯卷积后再累加,数值越大代表出现频率越高.中间白色斑点为(0, 0) 位置 Figure 3 Statistical analysis of paramers of multicellular twitching motility. (A) Statistical analysis of twitching velocity and motional tracks of 1 000 bacterial cells at different stage after adhesion to surface. The X axis is the medium value of Cauchy distribution fitting of tangential velocity, Y axis is the linear fitting slope of MSD at bi-logarithmic coordinate. (B) Statistical heat map of twitching velocity of 1 000 bacterial cells at high speed scale (left map) and low speed scale (right map). The X axis is the normal velocity value and Y axis is the tangential velocity value. The heat map were created by first plot velocity vector at all time points of all bacteria on the map, then do Gaussian convolution for all points and accumulation, bigger value on the map represent a bigger probability of occurrence. The white spot in the center represent coordinate position (0, 0). |
| |
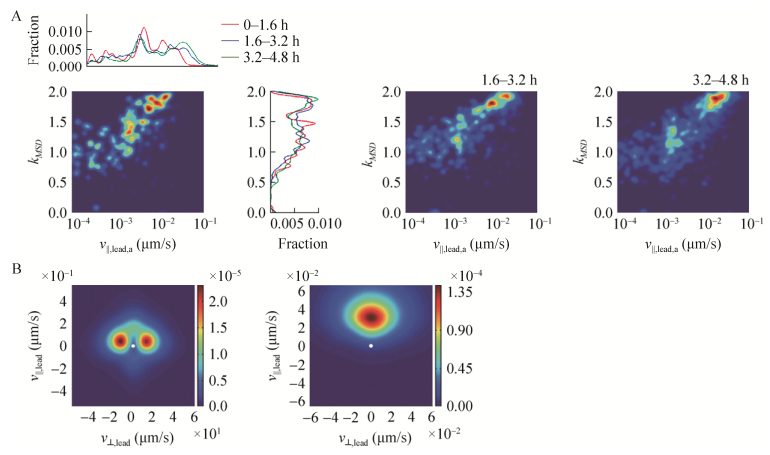
细菌行走是指细菌其中一极上的菌毛完全释放,另外一极的菌毛将细菌拉起来在表面上运动,细菌行走对生物被膜后期成熟结构的形成有重要意义[18]。我们在实验中详细记录了细菌行走过程以及平移和行走间的切换,如图 4A所示。细菌先是微微翘起在表面上快速平移,到2处后发生了快速的转变,直立起来。从3到5一直处于直立状态,6到7过程中经历了上下晃动。图 4B中记录了该过程细菌倾角的变化情况。细菌整体的运动速率比较大,在一个很宽的速度范围内波动(图 4C)。细菌行走过程中投影长度随着细菌立起来反复摇晃而有较大波动,在直立时投影长度接近于细菌的横截面直径(图 4D)。
|
| 图 4 细菌行走过程的示例分析. (A)单细菌行走过程的展示. (B)细菌行走过程中菌身与表面夹角随时间变化图.横坐标为时间轴,纵坐标为细菌菌身与表面之间的夹角,数字标注的红色圆圈对应(A)图中的各个时刻. (C)行走细菌的切向速度分布(左图)和法向速度分布(右图)及柯西分布拟合;横坐标为细菌运动速度切向或法向分量大小,纵坐标为对应切向或法向速度分量的概率密度值. (D)细菌行走过程中投影长度随时间变化图. (E)细菌行走过程中其端点在二维平面上的投影轨迹 Figure 4 Analysis of bacterial walking of a example cell. (A) photo presentation of the walking process of an example bacteria. (B) Time series of the cell-to-surface tilt angle during walking process. (C) Cauchy distribution fitting of the tangential velocity (left map) and normal velocity (right map) of a walking cell. The X axis is the value of tangential velocity or normal velocity, Y axis is the probability density. (D) Time series of cell projection lengh during walking. (E) The 2-D projection trajectory of bacterial leading point during walking motility. |
| |
细菌的弹射运动最早由Jin等发现[17],表面上的细菌会在两种极为不同的运动模式间切换——长时间的低速运动和短时间的高速运动。高速过程在几秒内完成,线速度高于低速模式几十倍,并且伴随着细菌主轴转动和后续运动方向的改变。Jin等认为低速过程是多根菌毛合力的结果,而高速过程则是由于单根菌毛的瞬间释放而失去力学平衡所致。实验中,我们将速度模量高于0.1μm/s的非翘起运动过程划定为弹射运动。我们将弹射事件发生次数对弹射模量在双对数坐标系中作图,发现一个明显的指数衰减。如图 5A所示,随着弹射速度模量的增加,该种弹射的出现次数逐渐减少,线性拟合得衰减级数为2.6。对多个细菌蹭行运动统计后发现弹射行为都有这一指数衰减,衰减级数在2.5到3之间。这是一个典型断裂噪音(Crackling noise)过程,在地震和铁磁相变研究中十分常见。图 5B所示是将500个细菌的弹射过程合并后作出的速度方向分布图。细菌倾向于向垂直于菌身方向或者后方弹射运动,从累计时间权重(左图)和累计速度模量权重(右图)两张图对比可知细菌向后弹射的幅度相对较大。以上结果还提示细菌运动方向后方的菌毛不易释放。
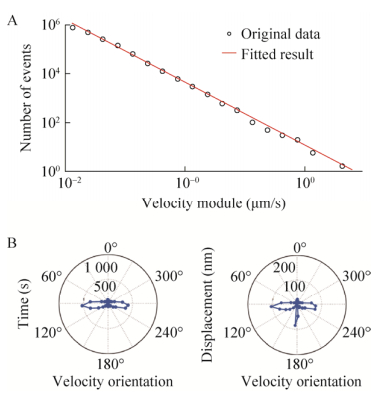
|
| 图 5 弹射运动的表征. (A)双对数坐标下的细菌蹭行运动过程中弹射频率与弹射速度模量的关系图,红线为线性拟合结果;(B)在极坐标下显示对500个细菌蹭行运动过程中弹射运动方向的累计时间权重统计(左图)和累计速度模量权重统计(右图),幅角数值标注在坐标系外圈,对应细菌运动引导点的速度方向.极径数值标记在坐标系半径上,对应相应的累计时间或累计速度模量值 Figure 5 Characterization of slingshot motility. (A) bi-logarithmic coordinate plot of slingshot frequency to slingshot velocity module in twitching process, red line is the linear fitting result. (B), Statistical analysis of the slingshot velocity direction with duration time weight (left) or displacement weight (right) of 500 cells at polar coordinates plain. The value of arguments were labelled outside the outer ring of the coordinates, corresponding to the velocity orientation of the cell motility leading points. The value of polar radii were labelled at the radius line, corresponding to the cumulative duration time or cumulative displacement. |
| |
细菌的蹭行运动速度时间自相关函数常出现图 2B中箭头所指的突出部分。突出部分出现的位置常在τ为0.9 s、1.8 s等位置,说明蹭行运动速度有一定的周期性。我们仔细观察导致突出部分出现的速度序列,发现其中包含很多弹射运动。而且正好以0.9 s为周期,且大致沿着一个方向。把弹射运动区域速度序列删除后,突出部分基本消失(图 6A)。这说明一次大的弹射过程可能包含了若干个子过程。如图 6B所示为一个细菌在200 s内速度模量的变化情况,该细菌共经历了10次跳弹射过程,每次速度模量也不一致,但都有一定的周期性弛豫,持续时间约3 s。对其中一个弹射事件的速度模量放大观察,可清楚看到其中确实包含着若干子过程,如图 6C和6D所示。
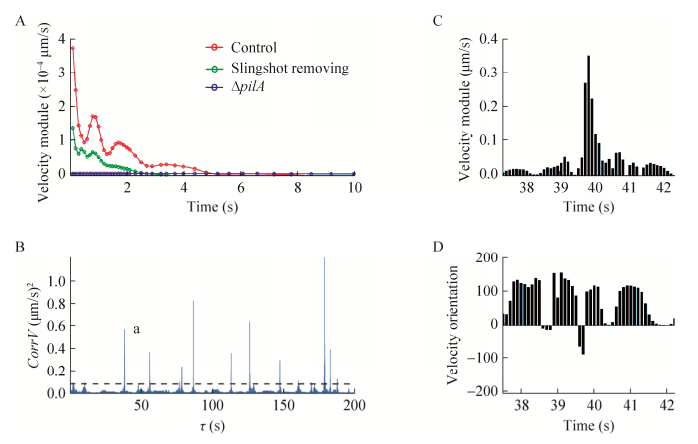
|
| 图 6 细菌弹射运动过程的周期性驰豫. (A)删除弹射过程前后细菌蹭行运动速度时间自相关函数的变化情况,横坐标为时间间隔,纵坐标为相应时间间隔下的速度相关函数值. (B) 400 s内某一细菌的蹭行运动速度模量随时间变化图,其中速度模量大于0.1 μm/s的过程被定为弹射运动;(C)和(D),(B)图中a弹射运动事件中速度模量和方向的详细变化过程 Figure 6 Periodic relaxation of slingshot motility. (A) Comparation of the temporal autocorrelation function of twitching velocity before and after slingshot processes removing. The X axis is the time interval, Y axis is the corresponding value of autocorrelation function. (B) Time series of velocity modulus twitching motility of one single bacterial cell, the velocity modulus bigger than 0.1 μm/s were decided as slingshot motility. (C) and (D), detail presentation of changes of the velocity module and orientation of the slingshot event a in (B). |
| |
Fuqua等首先发现细菌中的群体感知通讯机制[20],1998年首先发现群体感知信号分子合成基因缺失突变株无法产生正常结构的生物被膜,且形成的平整生物被膜对十二烷基硫酸钠的耐受性明显减弱[21]。Bjarnsholt等进一步发现群体感知机制大大增强了生物被膜对妥布霉素、过氧化氢及白细胞的耐受型[22]。此后大量研究者开始尝试设计针对群体感知信号分子的抑制剂来对抗生物被膜[23],因此细菌群体感知机制的阐明对生物被膜的防治非常重要。
然而,当前对群体感知机制与蹭行运动之间的关系尚不清楚,Beatson等认为群体感知与蹭行运动并无关联,他们观察铜绿假单胞菌群体感知系统的多个突变株发现细菌蹭行运动行为与野生型基本一致[24]。而Patriquin等在研究铁元素对细菌蹭行运动的关联时发现铁元素的缺乏促进蹭行运动,并且是通过二级群体感知系统rhlIR完成的[25]。后来Glick等进一步发现rhlIR系统控制的鼠李糖脂的分泌促进了细菌的蹭行运动[26]。由此可见群体感知系统在某些条件下会影响蹭行运动。我们以铜绿假单胞菌为模型,原位观测并定量分析细菌在过量加入一级、二级群体感知信号分子下的蹭行运动行为。
图 7为加入不同群体感知信号分子后细菌的弹射速度分布情况,图A显示加入一级、二级信号分子均可增强细菌的弹射速度模量,图B显示加入二级信号情况下细菌弹射运动频率有所增加。图C给出了细菌运动的速率分布,加入两种信号分子均使得细菌运动加快。总体来看,一级、二级群体感知系统对蹭行运动的影响相似,这提示群体感知可能是通过二级信号系统来调节蹭行运动的,一级系统通过开启二级系统而促进蹭行运动,这与Glick等的观点一致。

|
| 图 7 外源加入群体感知信号分子条件下细菌蹭行运动比较. (A)分别加入10 μmol/L一级和二级群体感知信号分子后对细菌蹭行运动在高速区域的统计分析.横坐标为细菌运动速度的法向分量值,纵坐标为细菌运动速度的切向分量值.该热图是首先把所有细菌每一个时刻的运动速度矢量画在平面上,然后逐个做二维高斯卷积后再累加,数值越大代表出现频率越高. (B)统计加入群体感知信号分子后细菌蹭行运动的弹射频率;(C)统计加入群体感知信号分子后细菌在高速区域的速度模量分布 Figure 7 Comparation of bacterial twitching motility under addition of quorum sensing signaling molecules. (A) Statistical analysis of twitching velocity at high speed scale after addition of 10 μmol/L 3-oxo-C12 HSL or C4-HSL. The X axis is the normal velocity value and Y axis is the tangential velocity value. The heat map were created by first plot velocity vector at all time points of all bacteria on the map, then do Gaussian convolution for all points and accumulation, bigger value on the map represent a bigger probability of occurrence. (B) Statistical analysis of bacterial slingshot frequency after addition of quorum sensing signaling molecules. (C) Statistical analysis of twitching velocity modulus distribution at high speed scale after addition of quorum sensing signaling molecules. |
| |
细菌的蹭行运动行为对其形成生物被膜具有重要的作用,对蹭行运动进行统计分析是当前这一领域的重要问题。本文通过对多种显微镜硬件的集成以及自主开发图像处理程序实现了对细菌蹭行运动行为的高通量、自动化的表征,建立了一套标准化的蹭行运动分析工具并利用这一工具对细菌行走、弹射运动进行了细致的表征,并以群体感知机制为例比较了不同条件下细菌蹭行运动情况。相信这一方法将为系统地研究蹭行运动分子机制以及调控原理打开新的局面。
| [1] | Burrows LL. Pseudomonas aeruginosa twitching motility:type IV pili in action. Annu Rev Microbiol, 2012, 66(1): 493–520. DOI: 10.1146/annurev-micro-092611-150055 |
| [2] | Bruellhoff K, Fiedler J, Möller M, et al. Surface coating strategies to prevent biofilm formation on implant surfaces. Int J Artif Organs, 2010, 33(9): 646–653. |
| [3] | Parsek MR, Singh PK. Bacterial biofilms:an emerging link to disease pathogenesis. Annu Rev Microbiol, 2003, 57(1): 677–701. DOI: 10.1146/annurev.micro.57.030502.090720 |
| [4] | Wolcott RD, Ehrlich GD. Biofilms and chronic infections. JAMA, 2008, 299(22): 2682–2684. DOI: 10.1001/jama.299.22.2682 |
| [5] | Persat A, Inclan YF, Engel JN, et al. Type IV pili mechanochemically regulate virulence factors in Pseudomonas aeruginosa. Proc Natl Acad Sci USA, 2015, 112(24): 7563–7568. DOI: 10.1073/pnas.1502025112 |
| [6] | Kilmury SLN, Burrows LL. Type IV pilins regulate their own expression via direct intramembrane interactions with the sensor kinase PilS. Proc Natl Acad Sci USA, 2016, 113(21): 6017–6022. DOI: 10.1073/pnas.1512947113 |
| [7] | Anyan ME, Amiri A, Harvey CW, et al. Type IV pili interactions promote intercellular association and moderate swarming of Pseudomonas aeruginosa. Proc Natl Acad Sci USA, 2014, 111(50): 18013–18018. DOI: 10.1073/pnas.1414661111 |
| [8] | Oliveira NM, Foster KR, Durham WM. Single-cell twitching chemotaxis in developing biofilms. Proc Natl Acad Sci USA, 2016, 113(23): 6532–6537. DOI: 10.1073/pnas.1600760113 |
| [9] | Comolli JC, Hauser AR, Waite L, et al. Pseudomonas aeruginosa gene products PilT and PilU are required for cytotoxicity in vitro and virulence in a mouse model of acute pneumonia. Infect Immun, 1999, 67(7): 3625–3630. |
| [10] | O'Toole GA, Kolter R. Flagellar and twitching motility are necessary for Pseudomonas aeruginosa biofilm development. Mol Microbiol, 1998, 30(2): 295–304. DOI: 10.1046/j.1365-2958.1998.01062.x |
| [11] | Klausen M, Heydorn A, Ragas P, et al. Biofilm formation by Pseudomonas aeruginosa wild type, flagella and type IV pili mutants. Mol Microbiol, 2003, 48(6): 1511–1524. DOI: 10.1046/j.1365-2958.2003.03525.x |
| [12] | Zhao K, Tseng BS, Beckerman B, et al. Psl trails guide exploration and microcolony formation in Pseudomonas aeruginosa biofilms. Nature, 2013, 497(7449): 388–391. DOI: 10.1038/nature12155 |
| [13] | Luo Y, Zhao K, Baker AE, et al. A hierarchical cascade of second messengers regulates Pseudomonas aeruginosa surface behaviors. mBio, 2015, 6(1): e02456–14. |
| [14] | Haley CL, Kruczek C, Qaisar U, et al. Mucin inhibits Pseudomonas aeruginosa biofilm formation by significantly enhancing twitching motility. Can J Microbiol, 2014, 60(3): 155–166. DOI: 10.1139/cjm-2013-0570 |
| [15] | Singh PK, Parsek MR, Greenberg EP, et al. A component of innate immunity prevents bacterial biofilm development. Nature, 2002, 417(6888): 552–555. DOI: 10.1038/417552a |
| [16] | Oura H, Tashiro Y, Toyofuku M, et al. Inhibition of Pseudomonas aeruginosa swarming motility by 1-naphthol and other bicyclic compounds bearing hydroxyl groups. Appl Environ Microbiol, 2015, 81(8): 2808–2818. DOI: 10.1128/AEM.04220-14 |
| [17] | Jin F, Conrad JC, Gibiansky ML, et al. Bacteria use type-IV pili to slingshot on surfaces. Proc Natl Acad Sci USA, 2011, 108(31): 12617–12622. DOI: 10.1073/pnas.1105073108 |
| [18] | Gibiansky ML, Conrad JC, Jin F, et al. Bacteria use type IV pili to walk upright and detach from surfaces. Science, 2010, 330(6001): 197. DOI: 10.1126/science.1194238 |
| [19] | Ni L, Yang S, Zhang RR, et al. Bacteria differently deploy type-IV pili on surfaces to adapt to nutrient availability. npj Biof Microb, 2016, 2: 15029. DOI: 10.1038/npjbiofilms.2015.29 |
| [20] | Fuqua WC, Winans SC, Greenberg EP. Quorum sensing in bacteria:the LuxR-LuxI family of cell density-responsive transcriptional regulators. J Bacteriol, 1994, 176(2): 269–275. DOI: 10.1128/jb.176.2.269-275.1994 |
| [21] | Davies DG, Parsek MR, Pearson JP, et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science, 1998, 280(5361): 295–298. DOI: 10.1126/science.280.5361.295 |
| [22] | Bjarnsholt T, Jensen PO, Burmølle M, et al. Pseudomonas aeruginosa tolerance to tobramycin, hydrogen peroxide and polymorphonuclear leukocytes is quorum-sensing dependent. Microbiology, 2005, 151(2): 373–383. DOI: 10.1099/mic.0.27463-0 |
| [23] | O'Loughlin CT, Miller LC, Siryaporn A, et al. A quorum-sensing inhibitor blocks Pseudomonas aeruginosa virulence and biofilm formation. Proc Natl Acad Sci USA, 2013, 110(44): 17981–17986. DOI: 10.1073/pnas.1316981110 |
| [24] | Beatson SA, Whitchurch CB, Semmler ABT, et al. Quorum sensing is not required for twitching motility in Pseudomonas aeruginosa. J Bacteriol, 2002, 184(13): 3598–3604. DOI: 10.1128/JB.184.13.3598-3604.2002 |
| [25] | Patriquin GM, Banin E, Gilmour C, et al. Influence of quorum sensing and iron on twitching motility and biofilm formation in Pseudomonas aeruginosa. J Bacteriol, 2008, 190(2): 662–671. DOI: 10.1128/JB.01473-07 |
| [26] | Glick R, Gilmour C, Tremblay J, et al. Increase in rhamnolipid synthesis under iron-limiting conditions influences surface motility and biofilm formation in Pseudomonas aeruginosa. J Bacteriol, 2010, 192(12): 2973–2980. DOI: 10.1128/JB.01601-09 |
 2017, Vol. 33
2017, Vol. 33