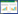中国科学院微生物研究所、中国微生物学会主办
文章信息
- 朱莹, 柴树茂, 曹明明, 王绍琛, 冯治洋
- Ying Zhu, Shumao Chai, Mingming Cao, Shaochen Wang, Zhiyang Feng
- 基于功能宏基因组学技术的金黄色葡萄球菌生物被膜抑制物的发现与活性分析
- Discovery and analysis of Staphylococcus aureus biofilm inhibitors using functional metagenomics approach
- 生物工程学报, 2017, 33(9): 1525-1532
- Chinese Journal of Biotechnology, 2017, 33(9): 1525-1532
- 10.13345/j.cjb.170058
-
文章历史
- Received: February 20, 2017
- Accepted: May 2, 2017
金黄色葡萄球菌Staphylococcus aureus广泛存在于空气、水、灰尘及人和动物的排泄物中,作为一种典型的革兰氏阳性致病菌,在世界范围内造成了大量感染性疾病[1-2]。生物被膜是金黄色葡萄球菌等致病菌的浮游菌株在特定生长条件下经附着、聚集、聚丛和生产胞外多糖,逐渐形成的生物膜结构[3-4]。该结构可显著增加金黄色葡萄球菌对宿主免疫应答和抗生素的抵抗力,使被膜菌较浮游菌的耐药性增大1 000倍,且容易造成二次感染[5-7]。微生物天然产物一直是抗感染药物的重要来源[8],并且通过基因重组[9]、调节微生物代谢[10]等方式可以获得一定的微生物代谢产物。但是,由于只有1%的微生物可以在现有的条件下进行培养研究,从而导致了天然产物重复发现,新结构的天然产物的发现越来越困难[11]。因此,研究新的方法和策略来发掘微生物天然产物资源势在必行。
功能宏基因组学技术直接从环境样品中提取总DNA (Environmental DNA,eDNA),通过构建宏基因组文库并功能表达文库中的eDNA来获得新的生物活性产物,从而有效克服了传统方法对微生物次级代谢产物发现的局限性[12-14]。目前,功能宏基因组技术大多应用大肠杆菌作为筛选宿主,而作为次级代谢产物表达的模式宿主链霉菌,由于技术上的限制,直到最近才见报道[15]。本研究以四川省峨眉山土壤为样品,以链霉菌为宿主,通过功能宏基因组学的方法获得了抗菌活性产物,并分析其对生物膜态金黄色葡萄球菌的抑制作用,以期为抗菌物质发现提供新的技术方法。
1 材料与方法 1.1 材料与试剂土壤样品采自四川省峨眉山市黄湾乡龙门村;克隆宿主菌JTU007/pUZ8002及穿梭载体pJTU2554[16]由上海交通大学陶美凤研究员馈赠;卡那霉素、安普霉素购自上海捷瑞生物有限公司;T4 DNA ligase购自Thermo公司;质粒提取试剂盒和琼脂糖胶回收试剂盒购自TIANGEN有限公司;pWEB-TNCTM Cosmid Cloning Kit为EPICENTRE公司产品;胰蛋白胨、酵母提取物、氯化钠、琼脂粉等试剂购自上海生工生物有限公司。白色链霉菌S. albus、金黄色葡萄球菌S. aureus由本实验室保存。
1.2 仪器与设备Gel X1520凝胶成像仪,购自上海欧翔科学仪器有限公司;CT15RT高速冷冻离心机,购自上海天美;UV-2600紫外可见分光光度计,购自日本岛津公司;GYY-6C稳压稳流电泳仪,购自北京六一生物科技有限公司;LDZM-40KOS立式压力蒸汽灭菌锅、DNP-9162电热恒温培养箱,购自上海申安医疗器械有限公司;SW-CJ-1FD超净工作台,购自苏州苏洁净化设备有限公司;HYG-C型摇床,购自太仓实验设备厂;SL2001N型电子天平,购自上海民桥精密科学仪器有限公司。岛津LC-20A高效液相色谱仪,购自日本岛津公司。
1.3 方法 1.3.1 土壤宏基因组文库的构建将采集的土壤样品先后通过不同孔径的筛网筛理后,经过裂解液和SDS缓冲液直接裂解抽提其中微生物的总DNA,然后通过电泳确定其浓度和纯度大小后,使用pWEB-TNCTM Cosmid Cloning Kit试剂盒中的末端修复酶对DNA末端修饰补平后,与制备的平末端穿梭表达载体pJTU2554连接,连接产物经噬菌体蛋白包装后侵染文库宿主菌,取10 μL侵染混合液铺在含50 μg/mL安普霉素的LB平板上,37 ℃培养过夜,计数计算滴度,按所计算滴度,将侵染获得的文库菌分装到培养试管,使每管含约500克隆,37 ℃过夜培养后备用。
1.3.2 阳性克隆筛选将上述过夜培养的文库菌接种到新的LB培养基培养至OD600达到0.4-0.8 (生长状态)后,与热激促萌发的白色链霉菌孢子混合。随后将混合菌液均匀涂布在含10 mmol/L MgCl2的MS固体平板上,置于30 ℃培养16-18 h。随后涂布含有萘啶酮酸(0.5 mg/mL)和安普霉素(0.4-0.5 mg/mL)的水溶液,继续培养3-5 d。用灭菌纱布将接合子分别影印至R5、MS、ISP4固体培养基上,继续培养5-7 d后,在上层覆盖含金黄色葡萄球菌的琼脂含量为0.6%-0.7%软固体培养基。待上层琼脂凝固后,继续置于30 ℃培养1-2 d后观察上层琼脂有无出现透明抑菌圈的区域,如果出现透明抑菌圈,挑出此克隆划线再次验证其抗菌活性。
1.3.3 阳性克隆发酵及产物提取采用不同的培养基对阳性克隆进行发酵,通过对培养基种类和发酵时间进行优化,最终决定采用MS作为种子培养基、ISP4 (含树脂)作为发酵培养基,使发酵产物活性最高。称取一定量的豆粉加入纯净水煮沸2 h后用纱布过滤,收集滤液,再加入D-mannitol溶解,配制成MS液体培养基,定容后进行分装,每个250 mL锥形瓶中分装50 mL培养基,用8层纱布封口后高温灭菌。按照终浓度50 μg/mL加入安普霉素、接种孢子后,于225 r/min、28 ℃条件下培养3 d。培养基变为棕褐色,按照1%比例转接ISP4 (树脂含量3%-5%)培养基,225 r/min、28 ℃继续培养12 d。树脂吸附发酵产物,洗涤培养基,回收树脂,晾干后,用甲醇溶解吸附树脂上的产物,旋蒸后收集产物,备用。其中,以插入空载体的白色链霉菌的发酵产物为对照组。
1.3.4 粗提物对金黄色葡萄球菌生物被膜的影响菌株的预处理:吸取5 μL金黄色葡萄球菌原始菌液,置于5 mL含2.5% (质量分数)葡萄糖的TSB培养基中,37 ℃培养24 h后,涂布100 μL菌液于倒有20 mL LB培养基的9 cm平板上,37 ℃条件下培养24 h,挑取单个菌落接种于刚果红培养基,37 ℃培养48 h,筛选出菌落变为黑色带晶体且生长状况良好的菌株用于后续实验。
最小抑菌浓度MIC值的测定:采用肉汤常量稀释法[17-19]测定粗提物对金黄色葡萄球菌的MIC值。
粗提物对金黄色葡萄球菌生物被膜形成的抑制作用:参照文献中的方法[20],具体操作如下。用含有2.5% (质量分数)葡萄糖的TSB培养基配制浓度为A600=0.05的金黄色葡萄球菌菌液,分别添加浓度为2、1、1/2、1/4、1/8、1/16 MIC的样品液,各取200 μL接种入96孔板中,每个样品浓度设6个复孔,空白对照孔6个复孔(只加培养基),另设阴性对照孔6个复孔(未加粗提物的生物被膜生长对照),37 ℃培养48 h后,弃去上清液并用磷酸钾缓冲盐(PBS,pH 7.4) 清洗3次。用0.1%结晶紫染色30 min,弃去结晶紫染液,用蒸馏水洗至澄清;加入无水乙醇溶液200 μL溶解,酶标仪测定各孔于波长570 nm处的吸光度。
粗提物对成熟金黄色葡萄球菌生物被膜的清除作用:根据文献中的方法进行测定[20]。向96孔板中加入200 μL用含有2.5% (质量分数)葡萄糖的TSB培养基配制浓度为A600=0.05的金黄色葡萄球菌菌液,37 ℃培养24 h,弃去培养基及悬浮菌,再用PBS清洗3次,用含有2.5% (质量分数)葡萄糖的TSB培养基配制浓度分别为2、1、1/2、1/4、1/8、1/16 MIC的粗提液样品,依次每孔加入200 μL,相同浓度的样品设6个复孔,空白对照孔6个复孔(只加培养基),另设阴性对照孔6个复孔(未加粗提物的生物被膜生长对照),37 ℃培养48 h后,弃上清液并用PBS清洗3次。用0.1%结晶紫染色30 min,弃结晶紫染液,用蒸馏水洗至澄清;加入无水乙醇溶液200 μL溶解,酶标仪测定各孔于波长570 nm处的吸光度。
粗提物对金黄色葡萄球菌生物被膜的抑制率或清除率按下式计算:
抑制率或清除率(%)=(A2-A1)/(A2-A0)×100
式中:A0为空白对照组A570的吸光值;A1为样品作用组A570的吸光值;A2为阴性对照组A570的吸光值。
1.3.5 数据统计分析所有数据利用Microsoft Excel进行统计处理,用SAS 9.2进行ANOVA分析,不同平均值之间利用LSD法进行差异显著性检验。
1.3.6 粗提物的高效液相法(High-performance liquid chromatography,HPLC)分析称取少量发酵粗提物,加入一定量体积的甲醇,配制成浓度为1 mg/mL的溶液,取10 μL用于HPLC分析。色谱条件:Diamonsil C18 (5 μm,4.6 mm×250 mm)色谱柱,柱温25 ℃,检测波长254 nm,流动相甲醇:水由10:90到100:0梯度洗脱1 h,流速1 mL/min。
2 结果与分析 2.1 表型筛选到的阳性克隆鉴定通过筛选80 000个转化子,获得了2个对金黄色葡萄球菌有较强抑制作用(抑菌圈半径约为0.2 mm)的阳性克隆EM110和EM123 (图 1)。为避免假阳性的存在,将EM110和EM123克隆在相应抗性MS平板划线纯化后,对其抗菌活性进行确定,EM110和EM123克隆仍然有抑制金黄色葡萄球菌作用,检测平板上出现抑菌圈。
|
| 图 1 筛选后的阳性克隆 Figure 1 Positive clones obtained by functional based screening. |
| |
随后,以含空载体的S. albus为对照,对EM110和EM123进行发酵,并对发酵产物进行了活性分析(图 2)。结果表明,发酵产物样品能明显抑制金黄色葡萄球菌的生长(抑菌圈半径约为0.3 mm)。该结果说明样品中含有抑菌活性物质。

|
| 图 2 克隆发酵产物的抗菌活性 Figure 2 Anti-bacterial activity of the fermentation extracts from two positive clones. |
| |
阳性克隆EM110与EM123发酵产物的活性分析证明了其发酵产物对金黄色葡萄球菌的抑制作用。利用“肉汤常量稀释法”对EM110与EM123的发酵粗提物进行了最小抑菌浓度分析。结果表明,样品EM110和EM123对金黄色葡萄球菌的MIC值分别为1.30 mg/mL和1.00 mg/mL,显著低于对照组的18 mg/mL。说明两种样品对金黄色葡萄球菌有良好的抑制作用。
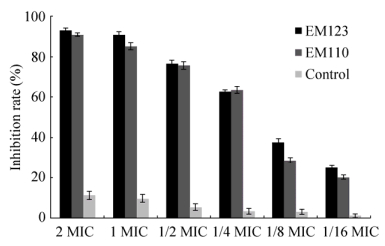
2.2.2 粗提物对金黄色葡萄球菌生物被膜形成的抑制作用浮游态金黄色葡萄球菌在不同基质中形成生物被膜存在复杂的生长过程,在该过程中,浮游态菌体不断被细胞外基质和基质网所包被,形成完整的具有群体效应的生物被膜[21]。这些细胞外基质包含了水、胞外多糖、胞外核酸、胞外蛋白质等成分。因此,金黄色葡萄球菌形成生物被膜的过程并不是简单的致病菌细胞集合,而是具有类似于高等生物组织功能的组合体。并且研究表明,致病菌生物被膜的形成需要不同菌体之间的群体行为,该行为与菌体的浓度及菌体外的密度信号感应(Quorum sensing)有关,类似于高等生物的发育周期[22-23]。因此,研究抑菌剂对金黄色葡萄球菌生物被膜形成过程的抑制作用可以为抑菌剂的有效抑菌提供理论支持。图 3为实验中不同提取物浓度对金黄色葡萄球菌生物被膜形成的抑制作用结果。
|
| 图 3 粗提物对金黄色葡萄球菌生物被膜的抑制作用 Figure 3 Inhibitory effect of the fermentation extracts on the formation of S. aureus biofilm. |
| |
图 3结果显示,不同浓度的样品对金黄色葡萄球菌生物被膜的形成均具有良好抑制作用。与对照相比,两种样品的不同浓度对金黄色葡萄球菌生物被膜的抑制率均显著高于对照组(P < 0.05)。另外,随着样品浓度的增大,两种样品对金黄色葡萄球菌生物被膜形成的抑制作用显著增强;在2 MIC浓度下,两种样品对生物被膜的抑制率都超过了90%,在1/4 MIC浓度下,两种样品对生物被膜的抑制率仍达到60%以上,在1/4-2 MIC浓度范围内,两种样品对金黄色葡萄球菌生物被膜的抑制作用无显著差异(P > 0.05)。
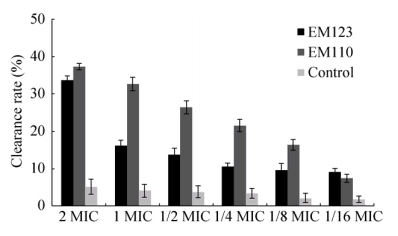
2.2.3 粗提物对金黄色葡萄球菌生物被膜的清除作用研究表明,在成熟的金黄色葡萄球菌生物被膜中可以形成具有三维结构的微菌落(Microcolony)[24-25],这些三维结构将单个菌体构成一个复杂的“群居体”,从而增加了致病菌的耐药性及抵抗宿主自身免疫系统的能力。其耐药性具体表现为生物被膜的渗透限制作用和生物被膜的主动外排机制,这些机制增加了抑菌剂的使用要求[26]。在本研究中,通过功能宏基因组学技术筛选获得的活性成分对金黄色葡萄球菌生物被膜具有有效的清除作用(图 4)。
|
| 图 4 粗提物对金黄色葡萄球菌生物被膜的清除作用 Figure 4 Effect of fermentation extracts on S. aureus biofilm dispersal. |
| |
图 4结果表明,与对照组相比,不同浓度的两种样品对金黄色葡萄球菌生物被膜均有显著的清除作用(P < 0.05),并且随着样品浓度的增加,清除作用显著增加;在1 MIC浓度时,EM110样品对生物被膜清除率超过30%,EM123样品在2 MIC浓度时对生物被膜的清除率达到33.56%±1.24%。另外,当样品浓度增加至大于1/8 MIC时,EM110样品对金黄色葡萄球菌生物被膜的清除率显著高于EM123样品(P < 0.05)。
2.3 阳性克隆发酵粗提物的HPLC检测为检测阳性克隆表达的活性产物,对阳性克隆发酵粗提物进行了HPLC (图 5)。与对照相比,EM123样品可检测到微弱的特异峰,但产量较低。虽然EM110的发酵粗提物的生物被膜活性要高于EM123,但EM110样品没有检测到明显的克隆特异峰(结果未显示),原因可能是EM110中的活性物质产量很低,未来通过发酵优化提高产量和改进检测方法的手段将可能发现新的活性单体化合物。

|
| 图 5 阳性克隆EM123发酵粗提物的HPLC分析 Figure 5 HPLC UV (254 nm) traces of EM110 and EM123 fermentation extracts. Star markers indicate the clone specific peaks. |
| |
以链霉菌为表达宿主,利用功能宏基因组学技术获得了两个对金黄色葡萄球菌均有良好的抑制作用的克隆,其发酵产物对金黄色葡萄球菌的MIC值分别为1.30 mg/mL和1.00 mg/mL。克隆发酵产物对金黄色葡萄球菌生物被膜的抑制作用显著,并且在1/4-2 MIC浓度范围内,两种样品对金黄色葡萄球菌生物被膜的抑制作用无显著差异。同时,两种样品对金黄色葡萄球菌生物被膜也存在显著的清除作用(P < 0.05),当样品浓度大于1/8 MIC时,EM110样品对金黄色葡萄球菌的清除率高于EM123样品。
| [1] |
Luo ZY, Xu LQ, Liu SC, et al. Detection of contamination of enterotoxin of Staphylococcus aureus in foods.
China Trop Med, 2014, 14(3): 296–298.
(in Chinese). 罗泽燕, 徐励琴, 刘思超, 等. 食品中金黄色葡萄球菌污染状况及其毒素检测. 中国热带医学, 2014, 14(3): 296-298. |
| [2] |
Zhou L, Wang Y, Wang FY, et al. Recent advance of general situation and novel detection technologies of Staphylococcus aureus in food.
China Brew, 2016, 35(2): 1–4.
(in Chinese). 周莉, 王永, 王法云, 等. 食品中金黄色葡萄球菌概况及新型检测技术研究进展. 中国酿造, 2016, 35(2): 1-4. DOI:10.11882/j.issn.0254-5071.2016.02.001 |
| [3] |
Wang Q, Tang JN, Chen J, et al. Research progress in detection and control methods of Staphylococcus aureus biofilm formation.
Sci Technol Food Ind, 2014, 35(22): 371–375.
(in Chinese). 王琼, 唐俊妮, 陈娟, 等. 金黄色葡萄球菌生物被膜检测及控制方法研究进展. 食品工业科技, 2014, 35(22): 371-375. |
| [4] |
Chen XW, Cui YD, Zhang H, et al. Advances on Staphylococcus aureus biofilm formation.
Heilongjiang Anim Sci Vet Med, 2011(11): 34–36.
(in Chinese). 陈曦伟, 崔玉东, 张华, 等. 金黄色葡萄球菌生物膜形成的研究进展. 黑龙江畜牧兽医, 2011(11): 34-36. |
| [5] |
Liu MH, Li JK, Liu L. Perspectives on the related genes and detection methods of Staphylococcus aureus biofilm formation.
Food and Ferment Industr, 2016, 42(2): 265–269.
(in Chinese). 刘美慧, 李建科, 刘柳. 金黄色葡萄球菌生物膜形成相关基因及检测方法的研究进展. 食品与发酵工业, 2016, 42(2): 265-269. |
| [6] | Lauderdale KJ, Malone CL, Boles BR, et al. Biofilm dispersal of community-associated methicillin-resistant Staphylococcus aureus on orthopedic implant material. Orthop Res, 2010, 28(1): 55–61. |
| [7] | Kokai-Kun JF, Chanturiya T, Mond JJ. Lysostaphin eradicates established Staphylococcus aureus biofilms in jugular vein catheterized mice. J Antimicrob Chemother, 2009, 64(1): 94–100. DOI: 10.1093/jac/dkp145 |
| [8] | Gillespie DE, Brady SF, Bettermann AD, et al. Isolation of antibiotics turbomycin A and B from a metagenomic library of soil microbial DNA. Appl Environ Microbiol, 2002, 68(9): 4301–4306. DOI: 10.1128/AEM.68.9.4301-4306.2002 |
| [9] | Kim SY, Lee HR, Park KS, et al. Metabolic engineering of Escherichia coli for the biosynthesis of flavonoid-O-glucuronides and flavonoid-O-galactoside. Appl Microbiol Biotechnol, 2015, 99(5): 2233–2242. DOI: 10.1007/s00253-014-6282-6 |
| [10] | Derewacz DK, Covington BC, McLean JA, et al. Mapping microbial response metabolomes for induced natural product discovery. ACS Chem Biol, 2015, 10(9): 1998–2006. DOI: 10.1021/acschembio.5b00001 |
| [11] | Ongley SE, Bian XY, Neilan BA, et al. Recent advances in the heterologous expression of microbial natural product biosynthetic pathways. Nat Prod Rep, 2013, 30(8): 1121–1138. DOI: 10.1039/c3np70034h |
| [12] | Buyer JS, Drinkwater LE. Comparison of substrate utilization assay and fatty acid analysis of soil microbial communities. J Microbiol Methods, 1997, 30(1): 3–11. DOI: 10.1016/S0167-7012(97)00038-9 |
| [13] | Riesenfeld CS, Schloss PD, Handelsman J. Metagenomics:genomic analysis of microbial communities. Annu Rev Genet, 2004, 38(1): 525–552. DOI: 10.1146/annurev.genet.38.072902.091216 |
| [14] | Bittner L, Halary S, Payri C, et al. Some considerations for analyzing biodiversity using integrative metagenomics and gene networks. Biol Direct, 2010, 5(1): 47. DOI: 10.1186/1745-6150-5-47 |
| [15] | Iqbal HA, Low-Beinart L, Obiajulu JU, et al. Natural product discovery through improved functional metagenomics in Streptomyces. J Am Chem Soc, 2016, 138(30): 9341–9344. DOI: 10.1021/jacs.6b02921 |
| [16] | Zhou H, Wang YM, Yu YF, et al. A non-restricting and non-methylating Escherichia coli strain for DNA cloning and high-throughput conjugation to Streptomyces coelicolor. Curr Microbiol, 2012, 64(2): 185–190. DOI: 10.1007/s00284-011-0048-5 |
| [17] | Melchior MB, van Osch MHJ, Lam TJGM, et al. Extended biofilm susceptibility assay for Staphylococcus aureus bovine mastitis isolates:evidence for association between genetic makeup and biofilm susceptibility. J Dairy Sci, 2011, 94(12): 5926–5937. DOI: 10.3168/jds.2011-4243 |
| [18] | Sudagidan M, Yemenicioğlu A. Effects of nisin and lysozyme on growth inhibition and biofilm formation capacity of Staphylococcus aureus strains isolated from raw milk and cheese samples. J Food Protect, 2012, 75(9): 1627–1633. DOI: 10.4315/0362-028X.JFP-12-001 |
| [19] | Tang JN, Chen J, Li HH, et al. Characterization of adhesin genes, Staphylococcal nuclease, hemolysis, and biofilm formation among Staphylococcus aureus strains isolated from different sources. Foodborne Pathog Dis, 2013, 10(9): 757–763. DOI: 10.1089/fpd.2012.1474 |
| [20] | Wang Y, Wang T, Hu JJ, et al. Anti-biofilm activity of Tanreqing, a traditional Chinese medicine used for the treatment of acute pneumonia. J Ethnopharmacol, 2011, 134(1): 165–170. DOI: 10.1016/j.jep.2010.11.066 |
| [21] | Costerton JW. Introduction to biofilm. Int J Antimicrob Agents, 1999, 11(3): 217–221. |
| [22] | Iwase T, Uehara Y, Shinji H, et al. Staphylococcus epidermidis Esp inhibits Staphylococcus aureus biofilm formation and nasal colonization. Nature, 2010, 465(7296): 346–349. DOI: 10.1038/nature09074 |
| [23] | Rode TM, Langsrud S, Holck A, et al. Different patterns of biofilm formation in Staphylococcus aureus under food-related stress conditions. Int J Food Microbiol, 2007, 116(3): 372–383. DOI: 10.1016/j.ijfoodmicro.2007.02.017 |
| [24] | Petrelli D, Zampaloni C, D'Ercole S, et al. Analysis of different genetic traits and their association with biofilm formation in Staphylococcus epidermidis isolates from central venous catheter infections. Eur J Clin Microbiol Infect Dis, 2006, 25(12): 773–781. DOI: 10.1007/s10096-006-0226-8 |
| [25] | Gunduz GT, Tuncel G. Biofilm formation in an ice cream plant. Antonie van Leeuwenhoek, 2006, 89(3): 329–336. |
| [26] | da Silva Meira QG, de Medeiros Barbosa I, Alves Aguiar Athayde AJ, et al. Influence of temperature and surface kind on biofilm formation by Staphylococcus aureus from food-contact surfaces and sensitivity to sanitizers. Food Control, 2012, 25(2): 469–475. DOI: 10.1016/j.foodcont.2011.11.030 |
 2017, Vol. 33
2017, Vol. 33