中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张文超, 张静超, 赵坤
- Wenchao Zhang, Jingchao Zhang, Kun Zhao
- 细菌显微追踪技术在生物被膜中的应用
- Application of bacterial tracking techniques in biofilms
- 生物工程学报, 2017, 33(9): 1411-1432
- Chinese Journal of Biotechnology, 2017, 33(9): 1411-1432
- 10.13345/j.cjb.170137
-
文章历史
- Received: April 2, 2017
- Accepted: May 31, 2017
2 天津大学化工学院, 天津 300350;
3 天津化学化工协同创新中心, 天津 300350
2 School of Chemical Engineering and Technology, Tianjin University, Tianjin 300350, China;
3 Collaborative Innovation Centre of Chemical Science and Engineering, Tianjin University, Tianjin 300350, China
生物被膜(Biofilm)是指细菌粘附于接触表面,通过其分泌的多糖、蛋白和胞外DNA等胞外物质,将细菌自身包绕其中而形成的大量细菌聚集膜样物[1-4]。其形成过程是一个非平衡、非线性的复杂的细菌自组装过程。细菌可以形成多种形式的生物被膜,既能够附着在固体表面形成固体生物被膜,也能聚集在气液界面形成液体生物被膜(Pellicle)。细菌生物被膜是引起细菌持续性感染的常见致病机制,大约60%的临床感染病例都与细菌生物被膜的形成有关[1],比如变形链球菌Streptococcus mutans在口腔形成生物被膜导致龋齿,粘液型铜绿假单胞菌Pseudomonas aeruginosa形成生物被膜会引起慢性肺感染[5]。与自由浮游细菌相比,生物被膜内的细菌提高了其抗药性和免疫逃逸能力,在食品、医疗、工业、军事等诸多领域给人类社会带来严重危害[6],因此理解并设法控制生物被膜是对抗细菌感染的一条重要途径。
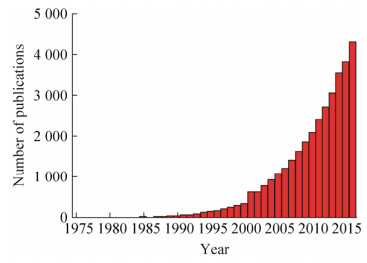
早在1676年,列文虎克就用自制的显微镜观察到了牙菌斑中大量微生物的存在,这被认为是关于生物被膜的首次发现[7]。然而,在随后的300多年时间里,只有零星的关于生物被膜的文献报道[1, 8-11]。究其原因,主要是因为两方面的限制:首先受限于对于生物被膜的认知不够充分。在微生物学的发展历史上,绝大部分研究都集中在对自由运动的浮游细菌的表征上,人们当时还没有认识到生物被膜才是细菌在自然界中的普遍存在方式;其次是受限于当时的技术水平,还不能对生物被膜在更细致的微观水平上(比如生物被膜的微观结构、生物被膜中细菌细胞的表征等)进行研究。直到20世纪80年代,科学家们发现自然界的细菌99%以上都是以生物被膜的方式存在[12],而且生物被膜对于人类的生活、健康和经济活动有着重要的影响[1, 5-6, 13],从此生物被膜的重要性才逐渐被人们所了解。同时,先进的显微成像技术、基因操控技术以及荧光染色技术等的发展,也极大地推动了生物被膜在微观水平上的研究,并吸引了不同学科背景的科研人员加入到该领域中,促使生物被膜逐渐发展为一个涉及微生物学、免疫学、化学、物理学、数学等多学科交叉的前沿热点研究领域,其结果之一就是跟生物被膜相关的文献数量自20世纪90年代后期开始呈现井喷式增长(图 1)。
|
| 图 1 1975–2016年PubMed收录的关于生物被膜的论文数量分布(搜索关键词:biofilm) Figure 1 Number of yearly published papers on biofilms indexed in PubMed from 1975 to 2016 (Searching keyword: biofilm). |
| |
Costerton等于1978年提出了生物被膜的最初理论模型,用于解释细菌粘附的机制[1, 8]。经过几十年的研究,人们对于生物被膜的理解已经获得了长足的进步。生物被膜的发展过程一般可分为5个阶段:可逆吸附阶段,不可逆吸附阶段,微菌落阶段,成熟阶段和从生物被膜解离阶段[14-16]。许多影响生物被膜的因素也逐渐被揭示,比如鞭毛、菌毛等细菌附属物对细菌的附着有非常大的影响[17-18];细菌分泌的多种多糖是构成生物被膜骨架的主要成分,与生物被膜的力学特性紧密相关[19-21];细菌在生物被膜的形成过程中利用小分子信号进行交流,也就是所谓的群体感应(Quorum sensing),来调控生物被膜的结构发展[22-23]。这些研究的实现在很大程度上得益于新技术的发展和应用。传统的方法像染色法、试管法等可以对生物被膜进行宏观观察,但无法做到单细胞水平上的观察。电子显微镜可以直接观测细菌细胞的表面形态,但制样过程对样品损伤较大,且无法实时原位观察活细胞的状态和生物被膜的发展过程。进入20世纪80、90年代后,随着光学和电子技术的发展,新工具和新技术不断涌现和完善,比如先进的显微镜成像技术(如共聚焦显微镜[24]、超分辨成像技术[25-27]和细菌显微追踪技术[28-32])、微流控技术[33-36]、三维成像技术[37]和高精度的荧光染色技术等,这使得人们能够从基因调控、代谢水平、运动机制等多角度,在单细胞水平上对生物被膜进行原位实时观察,极大地推动了生物被膜的研究[38-40]。本文着重介绍近几年发展的细菌显微追踪技术在生物被膜研究中的应用,从细胞的运动方式以及生物被膜的形成过程两个方面,综述了利用该技术进行的生物被膜研究的进展,以期在单细胞水平上阐释生物被膜的结构特征、形成机制和调控机理,并探讨了该技术的发展前景及在相关微生物研究领域中的应用前景。
1 细菌显微追踪技术 1.1 发展历史细菌显微追踪技术是在显微镜和相机等光学系统或电子光学系统设备的基础上发展起来,针对肉眼所不能分辨的微生物进行研究分析的技术。主要步骤包括:首先对通过显微镜延时拍摄采集的图像数据进行微生物图像识别,提取微生物的几何及相关信息,例如微生物的位置、大小、取向以及可能的荧光标识信号等信息;再利用追踪技术将不同时间点得到的微生物信息串联,构建微生物信息数据库,从而得到微生物的时间演变全过程;最后,在前面得到的数据库基础上,经过数据筛选和减噪等处理,利用现代数据分析技术,可以实现对微生物特定行为的研究(图 2)。
在细菌显微追踪技术发展之前,粒子显微追踪技术在胶体科学领域的研究已相对成熟。在胶体科学领域,通过光学显微镜对胶体颗粒的动力学进行实时观察分析,是研究胶体动力学的一种简洁高效的标准方法。早期开发的胶体显微追踪技术[41-43]都是围绕球形胶体系统进行的,后来随着非球形胶体系统的应用,新的非球形胶体的显微追踪技术也被开发出来[44-46]。而细菌显微追踪技术正是在借鉴了胶体粒子显微追踪技术的基础上发展而来的。从某一角度讲,细菌系统可以看做是胶体系统的一个特例,二者在很多方面具有高度相似性:比如细菌细胞的尺度在微米级,属于胶体的尺度范围;细菌细胞和胶体颗粒均会在溶液中做布朗运动(只不过在绝大多数情况下细菌的主动运动行为占主导);胶体颗粒的形状可以是球形、杆形和星形等,种类非常丰富;而细菌的形状也可以是球形、杆形和其他不规则形状。基于此,人们在胶体系统中开发的新技术和分析方法,完全可以尝试应用在微生物系统的研究上,其中细菌显微追踪技术就是胶体科学技术在微生物领域应用的一个成功案例。这也为生物被膜的微观研究提供了新的研究思路和研究手段。
国内外学者也积极开展了利用细菌显微追踪技术研究细菌群体行为的工作。哈佛大学的Howard C. Berg教授课题组和David Weitz教授课题组,普林斯顿大学的Howard A. Stone教授课题组和Joshua W. Shaevitz教授课题组,加州大学洛杉矶分校的Gerard C. L. Wong教授课题组,天津大学赵坤教授课题组,中国科学技术大学的金帆教授课题组以及澳大利亚悉尼科技大学的Cynthia Winchurch教授课题组等都发展了针对不同研究对象的细菌显微追踪技术,并结合先进的基因操控技术在单细胞水平上对细菌的群体行为比如生物被膜、细菌群集和子实体的形成等开展了研究,加深了人们对生物被膜和其他群体行为的理解[28, 30-32, 47-50]。
1.2 系统配备及要求利用细菌显微追踪技术进行细菌行为研究的平台包括硬件和软件两部分。
硬件方面主要是光学显微镜和相机,具体的硬件性能参数看需求而定。以研究生物被膜为例,光学显微镜需要具备焦点漂移自动补偿和全自动功能。因为生物被膜的数据采集周期相对较长,可以从几小时到几天不等,因而利用全自动功能自动采集、存储数据,并利用焦点漂移自动补偿保持长时间的焦平面稳定非常必要,而且相机需要有较高的分辨率和拍摄速率。如果使用荧光,相机应有较高的量子效率,可以探测弱荧光信号。有条件的实验室可以考虑配备共聚焦显微镜进行高分辨率成像的数据采集。而对于以细菌的运动方式特别是游泳运动为对象开展的研究工作,则要求相机具有较高的时间分辨率。一般的60帧/s的分辨率难以满足要求,有条件的话建议配备高速相机进行数据采集。总之,显微镜和相机的性能越好,拍摄的图像质量越高,采集到的信息就越丰富,后续的图像处理相对就更容易。
硬件方面还包括对应于不同研究对象而设计的特定容器。以研究生物被膜为例,常用的一个观察生物被膜演变的容器为流体室(Flow cell)。其通常设计为一个长40 mm宽4 mm深1 mm的长方体凹槽,其顶部由尺寸合适的盖玻片通过硅胶密封,凹槽两端各有一个通孔通过橡皮管分别与注射器和废液收集器相连。培养液通过注射泵从流体室一端流入,在另一端流出进入废液收集器,以保证实验期间流体室内的养分充足。观察时,将流体室倒置,观察细菌在玻璃表面上形成生物被膜的过程[51]。另外也可以制备微流控器件,利用微流控来进行细菌的行为研究。
软件方面主要是可以对采集的图像数据进行图像识别和统计分析的软件。其中,图像处理和统计分析中所用算法的有效性和快速性直接决定着软件的性能。鉴于细菌的多样性和研究问题的特异性,目前还没有一款软件(不论是商用软件还是用户自己开发的软件)可以做到通用。大部分商用软件,比如MetaMorph和其他的显微镜图像处理软件以及免费软件Image J等,都具备初级的图像识别和统计分析的功能,能够进行简单的细菌形貌和运动快慢的测量。另外,也有用Matlab或IDL等计算机语言编写的图像处理程序包来满足基本的比如观测物体形状和大小的需求。进一步的深入研究则需用户自己开发有针对性的程序包。
2 细菌显微追踪技术在生物被膜研究中的应用细菌显微追踪技术是近些年发展起来的一项新技术,现已逐渐应用到微生物学、细胞生物学和生理病理学等学科的研究工作中,成为现代生物学微观研究的重要工具。它可以实时记录细菌的形态、运动状态、生理反应等信息,且对样品无损伤,获得的图像清晰度高,便于得到更为准确的微观定量信息。传统的生物被膜观测方法得到的是对被膜中群体细胞平均后的信息,而细菌显微追踪技术可同时追踪大量单个细菌细胞的行为演变,从而可以在单细胞水平上研究细菌的运动方式和生物被膜的形成过程,因此受到研究人员的青睐,并使生物被膜的研究有所突破。
2.1 利用显微追踪技术研究单细胞水平上细菌的运动方式随着现代荧光标记和显微镜技术的发展,研究者们逐渐将显微追踪技术应用到探索细菌的代谢、表型和运动等方面,通过了解单细胞的运动行为来研究多细胞组织的异质性以及生物被膜的形成[52]。细菌的运动行为与其运动器官如鞭毛、菌毛以及分泌物有直接的关系,其运动按照介导方式大致可分为由鞭毛介导的游泳运动(Swimming)、由菌毛介导的蹭行(Twitching)以及鞭毛和菌毛共同介导的群集运动(Swarming)等。其中游泳和蹭行主要是细菌的个体行为,而群集是细菌的群体行为,属于典型的协同运动。
2.1.1 细菌游泳(Swimming)细菌游泳指细菌通过鞭毛的旋转在溶液中运动的现象,高速游动的细菌速率可达几十微米每秒。
多鞭毛细菌由于鞭毛的旋转方式不同,其游泳模式通常有两种方式,即前进(Run)和翻滚(Tumble)。Howard C. Berg教授课题组发现大肠杆菌Escherichia coli的周身鞭毛逆时针旋转可以聚集成束推动细菌快速向前游动(即Run,图 3A),而顺时针旋转则使鞭毛解束,脱离的鞭毛旋转产生不同方向的推进力,使得菌体在原地翻转(即Tumble),导致大肠杆菌的趋向运动[53]。之后该研究组利用荧光标记细菌和鞭毛,结合显微镜技术,实现了对细菌运动状态的实时记录,发现菌体上的不同鞭毛可在不同时间各自改变方向,其中一根鞭毛的方向改变甚至都可以导致原地翻转产生[54]。Macnab利用暗视野显微镜技术观察细菌鞭毛的运动状况,发现鞭毛是以前进和翻转交替进行的方式游动[55]。单个大肠杆菌细胞在近玻璃表面是按照圆形轨迹游泳。而在半固体琼脂基质上,细胞分化成细长的,鞭毛过表达的表型,在表面上协同迁移,称为群集运动(Swarming)。基于此,研究人员设计了一种用聚二甲基硅氧烷(PDMS)制作的透明狭窄的微通道,用于观察单个大肠杆菌细胞在琼脂基底上的移动,发现微通道中的细胞优先沿着右壁游泳[56]。

|
| 图 3 单细胞水平上的细菌运动研究实例. (A)近固体表面大肠杆菌的游泳运动呈圆形轨迹[56];(B) 铜绿假单胞菌的(ⅰ) “直立行走”和(ⅱ) “匍匐前进”行为[29];(C)铜绿假单胞菌匍匐前进中可发生(ⅰ)弹弓似的旋转运动和(ⅱ)平移运动[30];(D)霍乱弧菌的“漫游”、“转圈”运动和初始粘附[65];(E)大肠杆菌(ⅰ, ⅱ)群集运动边缘处的非对称流动和(ⅲ)形成的菌落图案[85] Figure 3 Examples of research on bacteria motility at the single cell level. (A) Cells swim in circular trajectories near a solid planar surface[56]. (B) Twitching motility observed in Pseudomonas aeruginosa: (ⅰ) walking and (ⅱ) crawling[29]. (C) Two modes of motion of Pseudomonas aeruginosa: (ⅰ) rotational motion; (ⅱ) translational motion[30]. (D) Near-surface motion and initial attachment of Vibrio cholerae[65]. (E) Swarming patterns of Escherichia coli[85]. |
| |
与大肠杆菌等多鞭毛细菌不同,铜绿假单胞菌和新月柄杆菌Caulobacter crescentus等单鞭毛细菌的游泳模式通常被描述为向前-反向-轻弹(Forward-reverse-flick),其运动方式遵循不同的物理机理,采用一种正反甩尾方式在液体中游泳[57],通过鞭毛按顺时针或逆时针交替旋转而向前和向后移动,若无外加因素,后退的菌体会沿前进的相同路径退回,但布朗运动使单鞭毛细菌的泳动轨迹呈“之”字形[58]。布朗大学的研究人员发现新月柄杆菌的游动依靠细胞体的螺旋运动,鞭毛只是推力的一部分[59]。他们搭建了一个特定的3D可追踪型显微镜平台,能够通过载物台的移动锁定目标细菌,确保细菌位于显微镜正中,使其得以实时、近距离追踪细菌细胞的运动,并利用阻力理论(Resistive force theory)模型,证实了细菌躯体的螺旋运动导致其向前比向后的游动速度快。Howard C. Berg教授课题组的最新研究发现新月柄杆菌在向后泳动时鞭毛马达产生更大的扭矩,这可能有助于菌体在分裂时游动细胞快速从母体细胞脱离。在存在边界的情况下,鞭毛细菌会贴近壁游泳,此时前进和后退速度明显不同,细菌运动轨迹更接近圆形[60]。研究结果表明,细菌运动明显受到螺距角(Pitch angle)的影响,细菌向前运动相对于向后运动更稳定。向前运动时,细胞平行于边界移动并将保持此状态。然而,向后运动时,其运动状态取决于细菌是否正在接近或离开边界[58]。这一现象在大肠杆菌中也有报道,在固体边界附近,大肠杆菌做顺时针圆周运动[61],轨迹的曲率半径随细菌体的长度增加而增加[62]。
2.1.2 蹭行运动(Twitching)蹭行运动指细菌利用Ⅳ型菌毛(TFP)在固体表面上爬行,在假单胞菌、奈瑟菌属Neisseria中十分常见。它不需要鞭毛的参与,一般沿着细菌长轴的方向,速率最大不超过0.1 μm/s。Bradley于1979年提出蹭行运动是通过细菌表面的菌毛系统完成的,菌毛通过伸长,与表面结合再收缩的方式牵引细菌运动[63]。
铜绿假单胞菌的运动为典型的蹭行运动,其使用Ⅳ型菌毛作为线性致动器,用于表面勘探,以实现定向爬行。Gerard C. L. Wong教授课题组使用细菌显微追踪技术研究铜绿假单胞菌在玻璃表面上的蹭行运动,发现其缺失鞭毛的突变株ΔfliM (排除鞭毛对于蹭行运动的影响)具有两种TFP驱动的表面运动方式,即高定向持久性的“匍匐前进” (Crawling)和快速低定向的“直立行走” (Walking) (图 3B)。匍匐前进有较好的定向性,其运动速率相较于典型的随机运动,既有快速模式(超扩散,Super-diffusive),也有慢速模式(次扩散,Sub-diffusive)[29]。而直立行走的方向随机,其运动速率一般是快速模式。一般认为,直立行走对于细菌搜索近邻环境更为有效,而长程的类趋化行为则使用匍匐前进的运动方式更为有效。在蹭行中,细菌需要不断地释放和收缩TFP,实验中发现释放动作会导致细菌有快速的“弹弓” (Slingshot)运动,细菌通过过度转向有效地转动细胞体(图 3C),而且这种“弹弓”运动跟基底的表面特性有关[30, 64]。另外研究还发现细菌的站立状态更有利于细菌的表面脱离,细菌先通过菌毛的收缩从匍匐状态转变为站立状态,再通过鞭毛的转动实现表面的脱离[29]。这种鞭毛和菌毛在近表面的相互作用对于霍乱弧菌Vibrio cholerae的表面定殖有着重要的影响。霍乱弧菌用它们的鞭毛和甘露糖敏感血凝素(MSHA) Ⅳ型菌毛协同作用以实现运动状态的切换和表面粘附过程。研究人员观察到霍乱弧菌的两种运动行为:“漫游” (Roaming),其特点是蜿蜒的轨迹;“转圈” (Orbiting),其特点是重复的高曲率轨道,只有后者这种模式才可以使细胞不可逆粘附并形成微菌落,而MSHA四型菌毛在其中起着关键作用[31, 65] (图 3D)。
与铜绿假单胞菌的表面运动不同,粘细菌Myxococcus xanthus的运动系统非常具有特色,由两个独立的遗传系统调控:一是A运动系统(Adventurous,单独性运动),主要负责从群体细胞分离的单个细胞的运动;二是S运动系统(Social,社会性运动),主要负责细胞群体的运动[66]。两种运动系统在不同的固体介质表面展现出不同的选择优势,A运动主要在较干燥和较硬的表面(如1.5%琼脂),S运动主要在较湿润和较软的表面(如0.3%琼脂)[67]。S运动需要TFP、胞外多糖和O-抗原[68]的参与,当一个细胞靠近群体中的其他细胞时,其TFP抓住附近细胞的特定胞外糖类,这种接触又可以引发TFP的收缩,从而引发多个细胞粘连而进行定向移动[69]。当单个细胞的突变株A-S+在固体介质表面时,它表现为不运动,但当附近有其他相同类型的细胞存在时,却能呈现运动趋势,表明S运动需要细胞之间的近邻相互作用,细胞与细胞之间通过紧密联系进行集体运动[70]。粘细菌的这种依赖TFP的S运动,通常被称为滑行运动(Gliding),类似于致病菌如奈瑟氏菌、假单胞菌等的蹭行运动[71-73]。而A运动是从细胞后部分泌的黏液推动细胞前进,细胞滑行过后留下痕迹。当一个细胞碰撞到另一个细胞的侧边时,其黏液的推动会引起细胞躯体弯曲,使该细胞与另一细胞平行,聚集成细胞并行排列的团簇,这种聚集是短暂的,二者之间未粘附,很快会互相滑过[74-76]。当细胞缺失S运动时,单个细胞仍然可以独立地运动[66]。而被拴系(Tethering)的粘细菌会出现缓慢垂直地抖动(Jiggling),使其向水平方向转变,为后续的爬行做准备[77]。
2.1.3 群集运动(Swarming)群集(Swarming)是指运动细菌以群体方式协调性地依靠鞭毛和TFP在培养基表面运动,属于典型的协同运动。
群集与生物被膜[78]、子实体的形成[68, 79-80]、病原体的侵入和微生物的扩散及共生等过程有着非常密切的联系。如果运动的表面是一个宿主,那么病原菌会通过群集运动形成生物被膜,达到感染的目的。由于受到周围环境的影响,群集细菌的生理指标常会发生一些周期性的变化,主要表现为:一是群集细菌会在形态、代谢、某些蛋白表达等方面出现显著分化(如菌体拉长、鞭毛过表达等);二是群集是集体行为,单个细菌是无法在相应培养基上做这种扩散运动的;三是群集需要表面活性剂的参与,因而可以通过控制胞外多糖的合成分泌来影响表面活性剂的产量进而调控细菌的群集运动[81]。群集运动既不是一个饥饿反应,也不是一个严格的生长发育阶段,而是细菌对外界环境的刺激所做出的一种激烈且可逆的较为复杂的适应过程[82]。群集运动一般通过鞭毛的旋转、slime的分泌和收缩TFP向前推进[71]。由鞭毛介导的自驱动系统,如在枯草芽孢杆菌Bacillus subtilis和大肠杆菌中,是基于化学物质梯度或营养物水平导致的远程细胞间相互作用来运动,即趋化性(Chemotaxis)。但是像粘细菌这种基于TFP运动的细菌没有这种远程传递系统来引导集体运动,它们依赖于相邻细胞间的接触信号和社会性运动的信号来运动(类趋化运动)。
利用显微追踪技术可以获得群集运动中单个细胞的运动行为信息。如Howard C. Berg教授课题组利用暗场和相差显微镜技术追踪大肠杆菌的群集运动,将氧化镁粒子沉积于可形成群集运动的大肠杆菌菌落上,发现氧化镁粒子层下的细菌仍自由移动,未受到扰动,氧化镁粒子向空白琼脂处的扩散速率比细菌的群集运动速率小很多,而这种差异在细菌群集边缘处更加明显[83]。细菌的这种在两个紧密接触的界面中快速移动的能力有助于其侵染过程。该课题组随后又结合荧光染料,使群集过程中细菌的鞭毛可视化,观察了群集运动中单个细胞的运动轨迹和运动方式,发现当细菌处于群集运动的边缘处静止时,鞭毛向外伸展;当细菌反转的时候,鞭毛的手性改变;当细菌侧向移动时,会被相邻细菌挤到一侧;而当细菌向前移动时,与游泳运动一样,鞭毛绑定成束[84]。做群集运动的大肠杆菌菌落边缘会呈现手性不对称的流动,表现为沿菌落边缘的顺时针流动,这种现象是由于鞭毛粘住底部基质而逆时针旋转,驱使细菌周围的流体顺时针转动所致[85] (图 3E)。也有研究人员利用活细胞实时成像技术获得了不同浓度琼脂上枯草芽孢杆菌周期性群集运动和生长的定量信息,发现其中的细菌可在生长状态和群集状态之间进行周期性的切换[86]。这些发现拓展了对群集运动动力学的理解,并有望将其应用于细菌驱使的微流体装置中。
粘细菌的群集运动是通过粘液的分泌和TFP的收缩和伸展实现的,包含依赖粘液的A运动和依赖TFP的S运动,在群集运动过程中会出现特定的细胞排列[68],以获得充足的营养物质和氧气。当环境中缺乏营养物质时,细胞聚集形成子实体,部分细胞分化形成粘孢子;当营养物质充足时,粘细菌生长活跃形成群集菌落。而粘细菌对抗饥饿环境形成多细胞子实体结构的能力衍生于群集运动边缘出现的多层的山丘状(Mound)结构[87],这种现象在黄色粘球菌和多种粘细菌中均发生。粘细菌的群集运动使大量菌体聚集,产生大量抗生素和消化酶,利于其捕食和获得食物。Wu等通过将实验数据与模拟结果相结合,定量分析群集运动过程中S运动和A运动各自的贡献,构建了以细胞为基础的模型来研究粘细菌群集过程中的社会性相互作用[71]。通过分析延时拍摄的细菌群集运动边缘的视频,发现滑行方向的周期性反转对群集很重要,这种反转可定向排列细胞,形成细胞平行排列的聚集体[87]。
抑制或阻止致病菌的群集运动可以用来影响其生物被膜的形成。比如一氧化氮(NO)可以阻止细菌群集行为,从而诱导生物被膜分散[88]。SbrR (PA2895) 是铜绿假单胞菌进行呼吸道感染时所必需的基因,其表达会抑制其同源胞质功能因子SbrL (PA2896) 的活性。细菌缺乏SbrR因子后在群体运动中表现出严重缺陷,导致生物被膜更易形成,Dove等发现可以表征sigma因子/抗sigma因子系统,相互调节铜绿假单胞菌表面的群集行为,来调节其群集运动性和生物被膜形成[89]。
2.2 利用显微追踪技术在单细胞水平上研究生物被膜形成过程的调控从浮游状态到细菌生物被膜,细菌经历了从低密度到高密度、从无组织状态到有组织状态的过程,在此过程中,细菌会分泌大量的胞外物质包括多糖和蛋白质来完成生物被膜的搭建。在细菌完成了表面附着后,细菌通过多种信号调控途径引导细胞个体之间的协同运动,包括群体感应系统、双因子调控系统[90]、sigma因子、环二核苷酸信号系统、mRNA结合蛋白-Spna、RNA酶以及毒素-非毒素系统等[91],其中有几种基础的细菌信号调控途径分别是群体感应(Quorum sensing, QS)、环二核苷酸信号系统(c-di-GMP)和多糖调控系统,其在细菌的生物被膜形成中发挥重要的作用。
2.2.1 群体感应系统(Quorum sensing)细菌的群体感应是细菌根据细胞密度变化进行基因表达调控的一种生理行为,具有群体感应的细菌能产生并释放成为自体诱导物的信号分子,当其积累到一定阈值时可与调控蛋白结合从而启动细菌中特定基因的表达。细菌的群体感应系统对生物被膜的形成和生物被膜结构的稳定起着关键作用[23, 92-93]。Davies等在1998年就发现细胞信号在常见细菌分化过程中是必需的,群体感应对生物被膜的完整性有重要影响,抑制群体感应信号可以抑制生物被膜的产生[22]。它对革兰氏阴性菌生物被膜的调节主要是通过酰基高丝氨酸内酯类信号分子(Acyl-homoserine lactone,Acyl-HSL)来实现的。其中铜绿假单胞菌的群体感应调控过程如图 4A所示,4个自诱导分子合成酶合成相应的自诱导分子,即3-氧十二烷酰高丝氨酸内酯(3-oxo-C12-HSL)、N-丁酰基高丝氨酸内酯(C4-HSL)、2-庚基-3-羟基-4-喹诺酮(PQS)和2-(2-羟苯基)噻唑-4-甲醛(IQS),当这些自诱导分子达到阈值时,转录激活因子激活合成相应受体,二者结合,直接或间接调控基因表达[94]。铜绿假单胞菌使用Acyl-HSL等信号通过群体感应调控基因表达,影响多糖、鼠李糖脂和其他毒力因子的产生及表面运动性,进而影响生物被膜的组装和结构[95]。其中C4-HSL和N-3-羰基十二酰基高丝氨酸内酯(3OC12-HSL)是微菌落分化发育所需的,生物被膜中C4-HSL的表达量要远多于液体培养状态下的量[96]。相比铜绿假单胞菌野生株的高度结构化的生物被膜,不产生Acyl-HSL的群体感应系统缺陷株则不能形成生物被膜。

|
| 图 4 生物被膜形成过程的调控研究实例. (A)铜绿假单胞菌的群体感应调控系统[94];(B)金黄色葡萄球菌受群体感应系统调控在特殊地形表面的存在状态,红色表示不存在QS的细胞群,黄色表示正受QS调控的细胞群[105];(C) c-di-GMP的结构式及代谢通路[107];(D)铜绿假单胞菌受多糖调控的(ⅰ)表面到访频率分布和(ⅱ)细菌分布[28];(E)多糖Psl对于生物被膜结构的影响(上为Psl过表达菌株WFPA801,下为野生型PAO1)[121] Figure 4 Examples of biofilm regulation. (A) Quorum sensing circuits in Pseudomonas aeruginosa[94]. (B) Effect of surface morphology and flow on quorum sensing of Staphylococcus aureus in microfluidic channels. Red, QS-off cells. Yellow, QS-on cells[105]. (C) Structure and metabolism of c-di-GMP[107]. (D) In Pseudomonas aeruginosa: (ⅰ) polysaccharide-dependent visit frequency map, and (ⅱ) distribution of cells on the surface[28]. (E) Effect of Psl on biofilm architecture: top, Psl overproducer WFPA801; bottom, wt PAO1[121]. |
| |
除了生物被膜的结构,群体感应也对细菌的运动行为有着重要的影响。以粘细菌为例[97],其子实体的发育过程包含以下阶段:1) 营养生长的细胞对外界环境信号作出反应,进入发育阶段;2) 细胞聚集或相互交流,聚集最先出现在菌体密度较大的地方,形成环形区域,内部菌体平行排列;3) 细胞表面产生介导细胞粘连的分子;4) 随着子实体生长,细胞进行有序重排或成串排列;5) 形成特定形状的子实体;6) 营养细胞分化成粘孢子。在该过程中,细菌的运动行为不论是从运动速度或者细胞排列方式等都有显著的变化。而运动行为的变化及其相关基因表达是受到多种信号分子的高度调控的[75, 98-99]。粘细菌通常通过多种胞外多肽蛋白分子来感知群体密度[100],目前在粘细菌中发现的有5类:Asg、Bsg、Csg、Dsg、Esg[101-102],其中A信号和C信号及其传导途径在子实体发育及产孢过程中起着非常重要的作用,它是诱导细胞的波纹运动、聚集行为、生孢相关基因表达的信号。不同浓度的C信号对应不同的发育阶段[103],而当体内过表达C信号时,又会诱导发育的各个阶段提前发生[104]。
群体感应信号分子是细菌用来与其他细胞通讯以应对环境条件变化的有效工具,而这也为控制生物被膜提供了一个新方向。通过研究群体感应信号分子和周围环境条件的关系,期待可以通过控制环境因素来干扰或切断群体感应信号分子的传递进而有效减缓生物被膜的形成。Kim等利用金黄色葡萄球菌Staphylococcus aureus和霍乱弧菌作为研究对象,研究了不同流速不同基质表面的生物被膜生长状态,发现高流速可抑制QS,流速的压力迫使细菌形成生物被膜来传递自体诱导物,而且离流速入口距离的远近不同,QS反应也有明显差异,而细菌活动的介质表面的凹陷裂缝处QS可正常运行(图 4B)。该工作揭示了QS在流体系统中时间和空间上的不均一性,为防治肠道疾病、治理管道堵塞等提供了新的思路和策略[105]。
2.2.2 c-di-GMP调控系统双-(3-5)-环二聚鸟苷一磷酸(c-di-GMP)是在生物被膜生命周期中起重要作用的动态细胞内信号分子,是细菌中广泛使用的第二信使,由2个分子GTP经二鸟苷酸环化酶(DGC)催化缩合,形成环状二核苷酸。c-di-GMP通过与相应的受体蛋白结合并激活受体蛋白而发挥作用,最终由磷酸二酯酶(PDE)降解[106-107] (图 4C)。它的作用非常广泛,参与调节细菌的多种生理功能,如生物被膜形成、表面群集运动等。细胞内c-di-GMP的浓度对于细菌适应环境变化、调节生命活动是非常重要的,因此c-di-GMP浓度的调节和控制对细菌具有重要意义。c-di-GMP发挥高效信号作用需要其对应的代谢蛋白、受体、目标物距离较近,此时其合成和降解均会比较少;而当DGC/PDE远离效应器/目标分子时,DGC会催化合成更多的c-di-GMP;当c-di-GMP与效应蛋白距离较远时,其信号通路可能会被抑制[106]。
常规测量c-di-GMP通常采用化学提取法,但该方法获得的只是整个生物被膜中c-di-GMP的平均浓度,无法获知单细胞水平上生物被膜内c-di-GMP的分布以及单个细胞内的分布情况。研究人员开发了一种实时成像的定量方法,以绿色荧光蛋白(GFP)的表达量反应c-di-GMP浓度,以青色荧光蛋白(CFP)作为生物量指示剂,精确给出了c-di-GMP在生物被膜中的分布情况。数据表明c-di-GMP在生物被膜形成早期的小菌落中分布较均匀,而在成熟生物被膜中多分布于菌落外缘,表明这种第二信使随时间和菌落变化是动力学可变的,也实时反映了生物被膜异质性的程度[108]。Christen等通过显微镜和基于荧光共振能量转移(FRET)的传感器监测了单个细胞中c-di-GMP的浓度,发现c-di-GMP分布随细胞分裂的不同阶段而不同,且在形成的子代中也呈不对称分布[109]。c-di-GMP的这种空间和时间上的不对称分布可以调控胞外细胞器的生物合成和功能,进而影响细菌的表面粘附和生物被膜形成。c-di-GMP通过影响大量的调控因子和路径,从而在生物被膜形成过程中的各个阶段发挥作用[110]。其中对细菌运动性的调控是一个重要方式。如FleN是铜绿假单胞菌的一种重要的鞭毛调节因子,fleN基因突变的菌株会丧失运动性,且可产生多根鞭毛,表现为杂乱的“翻滚” (Tumbling)行为。FleN通过与c-di-GMP结合,调控铜绿假单胞菌的单鞭毛合成,进而改变其运动方式[111]。铜绿假单胞菌中的FleQ是最早确定的能与c-di-GMP结合的转录因子,FleQ与c-di-GMP结合可以活化鞭毛合成蛋白的表达并抑制胞外多糖合成相关基因的转录,以此调控胞外多糖的产生和生物被膜的形成。Matsuyama等通过解析FleQ与c-di-GMP的结合位点和作用机制,发现在溶液中c-di-GMP可以驱使FleQ低聚体的重组,并且这种作用不会影响其对EPS相关基因表达的抑制和活化功能[112]。
c-di-GMP同时也是介导霍乱弧菌的运动性和生物被膜基质形成的关键信号分子,促使细菌从浮游态向生物被膜的转变。通过调节c-di-GMP的表达,可以影响霍乱弧菌生物被膜的形成过程,进而阻止其在人体内定殖及感染过程。霍乱弧菌的转录抑制试验表明高浓度的c-di-GMP促进msh (MSHA菌毛的操纵子编码)、vps (Vibrio polysaccharide)基因簇等其他生物被膜相关基因的转录,抑制鞭毛基因的转录[65, 113]。而且,c-di-GMP合成酶和磷酸二酯酶可以调控c-di-GMP的浓度和vps基因表达,最终影响生物被膜的形成[114]。
2.2.3 多糖调控系统多糖是构成生物被膜骨架的主要成分,因而多糖调控系统在细菌生物被膜的形成过程中扮演着举足轻重的角色。铜绿假单胞菌的胞外多糖主要包括Psl、Pel和alginate多糖,而不同的成分在细菌生物被膜形成过程中发挥不同的作用[19, 21, 115-117]。其中Psl多糖由一个重复的五糖单元多聚而成,五糖单元由D-葡萄糖、D-甘露糖和L-鼠李糖组成,它充当了2个二鸟甘酸环化酶siaD和sadC的刺激信号,可以增加二者的产量,进而升高细胞内c-di-GMP的浓度,影响铜绿假单胞菌生物被膜的形成[118-120]。研究人员通过将多糖在不同阶段的分布轨迹可视化,发现在附着期间,Psl以螺旋模式锚定在细胞表面上,促进细胞-细胞之间的相互作用和基质的装配;而在生物被膜成熟期间,Psl从细菌表面的化学解离破坏了Psl基质以及生物被膜结构。Psl积累在呈3-D结构的小菌落外围,导致在微菌落中心形成无Psl基质的“自由腔” (Matrix-free area);在生物被膜解离阶段,解离的浮游细胞出现在该基质自由腔中,死细胞和胞外DNA (eDNA)也集中在无Psl基质区域,而细胞死亡和自溶的控制基因引起基质腔和小菌落破散[21]。
Psl多糖通常作为一种“粘合剂”,主要用于增强细胞在表面的粘附。Zhao等利用细菌显微追踪技术,研究了在生物被膜形成的初期阶段铜绿假单胞菌在玻璃表面的运动模式,发现了Psl多糖的一种新功能,即可以作为细菌集聚信号引导细菌的表面运动。研究发现,当细菌在表面蹭行时,其分泌的Psl多糖会在表面留下痕迹。由于细菌菌毛与多糖的相互作用,这些痕迹会影响后来细菌的运动行为,使得这些Psl浓度高的区域吸引其他细菌的到访,而其他细菌的到访又会使该区域累积更多的Psl进而吸引更多的细菌到访,由此形成一个正反馈,即Psl聚集导致更多的细胞聚集,一旦聚集了足够多的细菌(约50个左右),这些细菌的行为就会发生改变,不再单个行动,而是形成微菌落结构(Microcolonies),从而成为触发生物被膜形成的原始基础菌落(图 4D)[28]。中国科学院微生物研究所马旅雁团队也发现TFP介导的细菌迁移可以导致Psl纤维状基质的形成,该Psl纤维状基质在生物被膜的中间位置(营养匮乏区)呈现辐射状,暗示有促进细菌向营养物质迁移的作用(图 4E)[121]。Psl作为维持生物被膜结构的骨架成分之一,在细菌粘附过程中起关键作用,若过表达则会影响生物被膜的整体结构,使之呈蘑菇状,表面粗糙度高,而野生型菌株形成的生物被膜相对平坦,覆盖面积大[19, 121]。
这些研究结果表明如果能阻止或干扰Psl多糖在表面的分布,也许就能在某种程度上抑制生物被膜的形成。有意思的是,在2015年,中国科学院微生物研究所马旅雁课题组和山东大学的谷立川课题组解析了Psl合成途径中重要的糖苷水解酶PslG的晶体结构,通过结构分析及同源蛋白比对发现PslG具有糖苷内切酶的特征,PslG可以通过水解Psl抑制铜绿假单胞菌生物被膜的形成,甚至可以水解已经成熟的生物被膜。研究发现,纳摩尔浓度的PslG可在半小时内将生物被膜的物质量降低90%以上,显示了其瓦解生物被膜的高效性。经PslG处理过的生物被膜对抗生素的抗性下降了4–8倍,并且对巨噬细胞更为敏感[122]。除PslG外,还有其他可以抑制生物被膜的物质,比如在辛酸存在的条件下,低浓度的鼠李糖脂有望用于抑制或者破坏生物被膜[123]。
2.2.4 其他机制调控细菌生物被膜形成生物被膜的形成过程是一个细菌跟外界环境相互作用并通过自身的生理调控来适应外界环境的过程。除了上面提到的QS系统、c-di-GMP系统和多糖系统外,生物被膜的形成也受许多其他因素(如环境因素中空间胁迫或抗生素胁迫)的直接影响。
Howard A. Stone教授课题组利用微流控技术,研究了在特定空间内霍乱弧菌生物被膜的生长模式和结构[48, 124],发现表面粘附导致的压迫,使霍乱弧菌的生物被膜从2D分枝状结构向致密且排列有序的3D聚集体结构转变,该过程由基因rbmA控制。Cynthia Winchurch教授课题组利用高分辨率、相差显微镜延时拍摄和计算机视觉算法,研究了铜绿假单胞菌在半固体表面的运动行为,发现在生物被膜形成前期,会有结合紧密的细胞群(处于同一高度的细菌)在半固体表面迁移,产生沟壑,形成复杂的运动轨迹网络,以方便后面的细胞迁移。其中,eDNA有利于整个沟垄中细胞保持连贯排列,避免细胞碰撞,确保群体的高效迁移[32]。
在有抗生素的环境下,细菌的群体行为也会发生适应性的改变。研究显示在羧苄青霉素存在的情况下,铜绿假单胞菌通过TFP-TFP相互作用可以促进细胞-细胞相互作用,限制单个细胞运动,减慢群体扩展,进而帮助群体规避有毒物质[125]。SOS应答系统(SOS response system)是细菌DNA损伤诱导反应系统,普遍存在于大肠杆菌、霍乱弧菌等重要病原菌中,具有遗传毒性的环丙沙星可以诱导大肠杆菌的SOS反应,将杆状大肠杆菌变为含多染色体的细丝状物,诱导染色体间等位基因重组,然后繁殖有抗性的无丝状后代,母体长丝死亡,从而增强大肠杆菌对环丙沙星的抗性[126]。而抗生素在渗透进生物被膜的过程中,由于生物被膜自身的特性,抗生素的浓度随进入生物被膜的深度而减弱。因而研究抗生素浓度梯度对细菌的影响,对理解生物被膜的抗药性十分重要。Robert Austin教授课题组利用微流控技术创建了具有不同抗生素浓度的异质空间环境,观察了大肠杆菌在不同毒性梯度中的结构变化,并通过全基因组测序,揭示了抗药性是细菌对抗生素胁迫产生的生物反应的进化[127]。
3 前景展望细菌显微追踪技术是以成像技术为基础的新型微生物研究手段。近几年成像技术在快速性、灵敏性以及三维成像方面都取得了很好的进展,为显微追踪技术的发展提供了很好的支持。2010年Saar等开发了以受激拉曼散射(SRS)为基础,以核磁共振成像(MRI)为辅助的新型显微成像技术,此成像技术快速灵敏,能够捕获血细胞挤进毛细血管时的“视频”图像,现已广泛应用于捕获器官、肿瘤以及其他大型结构的静态图像[128]。Erturk等开发出一种研究神经组织再生结构的新方法,不仅能检查完整组织中的单个神经细胞,还能描绘出它的三维图像,并利用荧光染料追踪其在脊髓中的路径[129]。Su等利用红光和蓝光偏移波束建立了全息信息,配合先进的软件,实现了显微镜下对快速移动物体的追踪,获得了其精确的三维运动轨迹[37]。未来,整合这些先进的成像技术,发展新型的微生物显微追踪技术,将会在微生物学领域,尤其是在由多种微生物组成的复杂微生物群落的研究领域,发挥重要的作用。
自然界中的微生物群落是由不同种微生物共同组成的。不同种微生物之间的相互作用对于微生物群落的演变以及个体微生物的进化都具有重要意义,因而对不同种微生物之间相互作用的研究是理解微生物群落的关键。例如在口腔环境中,口腔生物被膜先由核梭菌Fusobacterium mucleatum产生,为随后定殖其中的牙龈卟啉单胞菌Porphymonas gingivalis提供了空间上的支持,而后者可以激活口腔中的蛋白酶,前者依靠这些蛋白酶吸收营养物质,彼此获益[130]。Benomar等研究了普通脱硫弧菌Desulfovibrio vulgaris Hildenborough和丙酮丁醇梭菌Clostridium acetobutylicum共生体系中二者之间紧密的相互作用,发现营养物质的胁迫会导致共生细菌之间的相互作用,包括蛋白质等细胞分子的交换,特定基因表达量的差异等[131]。目前作为研究热点的肠道微生物群组更是包含了数量众多的微生物种类。它们在人体能量代谢、营养物质吸收、胃肠道功能及免疫等方面发挥重要作用,一旦肠胃微生物的内稳态被打破,就会诱发多种人类疾病[132-136]。研究者在肠道菌群中建立了共生基因组模型,认为宿主基因组和微生物组是一个整体,接受自然选择,宿主与其肠道微生物的组成和功能是一个长期的协同进化过程,拓展了共生体如何塑造宿主进化的观念[132]。共生微生物间通常会形成胞间纳米管来互相传递营养物质和信号等,而对于病原微生物,其遗传性或非遗传性的抗药性也都可以通过纳米管的传递从相邻细胞获得[133-134]。微生物之间也会相互竞争,而当细菌之间的竞争损害了菌群的合作网络时,菌群稳定性会提升且宿主能够从细菌之间的竞争中受益[134]。因此研究这些不同种微生物之间的关系以及微生物和宿主细胞的关系对于理解肠道微生物群组的生态是至关重要的。近年来以高通量测序为基础的肠道宏基因组学、功能基因组学、代谢组学和蛋白质组学的不断发展,极大地促进了肠道微生物研究的发展。但当前在单细胞水平上进行肠道微生物之间以及微生物和宿主细胞之间相互作用的研究相对较少,尤其是信号传导方式、不同种微生物如何影响寄主基因表达、如何协同维持体内微生物和寄主的内稳态等问题背后的机制并不清楚。而细菌显微追踪技术可以充分发挥其在单细胞水平上进行微生物行为研究的技术优势,同时结合基因组学、代谢组学以及其他生物学研究工具,可以深入了解肠道微生物种群之间以及种群和寄主之间的关系,这也是未来十年肠道微生物菌群分析方法研究的重要方向[137]。
4 总结细菌显微追踪技术是在成像技术上结合图像识别和统计分析的算法而发展起来的一项新技术。具有高时空分辨率,操作方便,可以对细菌的形貌、运动状态、动态生理反应等信息进行实时的原位观测等优势,尤其可以在单细胞水平上对细菌的运动进行精确高保真的统计分析,为研究细菌的群体行为比如生物被膜提供了良好的技术平台。但是,目前的细菌显微追踪技术还主要局限于对二维图像数据的采集和分析,获得的也只是表观上的信息,对成分、三维结构等信息鉴别能力有限,而且受设备硬件的限制,无法探索在活体生物环境下(in vivo)的微生物群体行为。因此,细菌显微追踪技术的未来方向应该是向三维空间的追踪和图像分析技术发展,并开发可以进行活体生物环境下的观测手段。通过与基因组学、代谢组学以及其他生物学研究工具的紧密结合,细菌显微追踪技术不仅在生物被膜领域也将在微生物行为研究的其他领域有着广阔的应用前景。
| [1] | Costerton JW, Stewart PS, Greenberg EP. Bacterial biofilms: a common cause of persistent infections. Science, 1999, 284(5418): 1318–1322. DOI: 10.1126/science.284.5418.1318 |
| [2] | O'toole G, Kaplan HB, Kolter R. Biofilm formation as microbial development. Annu Rev Microbiol, 2000, 54(1): 49–79. DOI: 10.1146/annurev.micro.54.1.49 |
| [3] | Stoodley P, Sauer K, Davies DG, et al. Biofilms as complex differentiated communities. Annu Rev Microbiol, 2002, 56(3): 187–209. |
| [4] | Mann EE, Wozniak DJ. Pseudomonas biofilm matrix composition and niche biology. FEMS Microbiol Rev, 2012, 36(4): 893–916. DOI: 10.1111/j.1574-6976.2011.00322.x |
| [5] | Krzyściak W, Jurczak A, Kościelniak D, et al. The virulence of Streptococcus mutans and the ability to form biofilms. Eur J Clin Microbiol Infect Dis, 2014, 33(4): 499–515. DOI: 10.1007/s10096-013-1993-7 |
| [6] | Tan SYE, Chew SC, Tan SYY, et al. Emerging frontiers in detection and control of bacterial biofilms. Curr Opin Biotechnol, 2014, 26: 1–6. |
| [7] | Donlan RM. Biofilms: microbial life on surfaces. Emerging Infect Dis, 2002, 8(9): 881–890. DOI: 10.3201/eid0809.020063 |
| [8] | Costerton JW, Geesey GG, Cheng KJ. How bacteria stick. Sci Am, 1978, 238(1): 86–95. DOI: 10.1038/scientificamerican0178-86 |
| [9] | Zobell CE. The effect of solid surfaces upon bacterial activity. J Bacteriol, 1943, 46(1): 39–56. |
| [10] | Characklis WG. Attached microbial growths-Ⅱ. Frictional resistance due to microbial slimes. Water Res, 1973, 7(9): 1249–1258. DOI: 10.1016/0043-1354(73)90002-X |
| [11] | Jones HC, Roth IL, Sanders Ⅲ WM. Electron microscopic study of a slime layer. J Bacteriol, 1969, 99(1): 316–325. |
| [12] | Donlan RM. New approaches for the characterization of prosthetic joint biofilms. Clin Orthop Relat Res, 2005, 437: 12–19. |
| [13] | Costerton JW, Cheng KJ, Geesey GG, et al. Bacterial biofilms in nature and disease. Annu Rev Microbiol, 1987, 41(1): 435–464. DOI: 10.1146/annurev.mi.41.100187.002251 |
| [14] | Monroe D. Looking for chinks in the armor of bacterial biofilms. PLoS Biol, 2007, 5(11): e307. DOI: 10.1371/journal.pbio.0050307 |
| [15] | Costerton JW, Lewandowski Z, Caldwell DE, et al. Microbial biofilms. Annu Rev Microbiol, 1995, 49(1): 711–745. DOI: 10.1146/annurev.mi.49.100195.003431 |
| [16] | Donlan RM, Costerton JW. Biofilms: survival mechanisms of clinically relevant microorganisms. Clin Microbiol Rev, 2002, 15(15): 167–193. |
| [17] | O'toole GA, Kolter R. Flagellar and twitching motility are necessary for Pseudomonas aeruginosa biofilm development. Mol Microbiol, 1998, 30(2): 295–304. DOI: 10.1046/j.1365-2958.1998.01062.x |
| [18] | Klausen M, Aaes-J rgensen A, Molin S, et al. Involvement of bacterial migration in the development of complex multicellular structures in Pseudomonas aeruginosa biofilms. Mol Microbiol, 2003, 50(1): 61–68. DOI: 10.1046/j.1365-2958.2003.03677.x |
| [19] | Ma LY, Jackson KD, Landry RM, et al. Analysis of Pseudomonas aeruginosa conditional Psl variants reveals roles for the Psl polysaccharide in adhesion and maintaining biofilm structure postattachment. J Bacteriol, 2006, 188(23): 8213–8221. DOI: 10.1128/JB.01202-06 |
| [20] | Flemming HC, Neu TR, Wozniak DJ. The EPS matrix: the " house of biofilm cells". J Bacteriol, 2007, 189(22): 7945–7947. DOI: 10.1128/JB.00858-07 |
| [21] | Ma LY, Conover M, Lu HP, et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathog, 2009, 5(3): e1000354. DOI: 10.1371/journal.ppat.1000354 |
| [22] | Davies DG, Parsek MR, Pearson JP, et al. The involvement of cell-to-cell signals in the development of a bacterial biofilm. Science, 1998, 280(5361): 295–298. DOI: 10.1126/science.280.5361.295 |
| [23] | Miller MB, Bassler BL. Quorum sensing in bacteria. Annu Rev Microbiol, 2001, 55(1): 165–199. DOI: 10.1146/annurev.micro.55.1.165 |
| [24] | Pawley J. Handbook of Biological Confocal Microscopy. US: Springer, 2006. |
| [25] | Betzig E, Patterson GH, Sougrat R, et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science, 2006, 313(5793): 1642–1645. DOI: 10.1126/science.1127344 |
| [26] | Rust MJ, Bates M, Zhuang XW. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods, 2006, 3(10): 793–796. DOI: 10.1038/nmeth929 |
| [27] | Berk V, Fong JCN, Dempsey GT, et al. Molecular architecture and assembly principles of Vibrio cholerae biofilms. Science, 2012, 337(6091): 236–239. DOI: 10.1126/science.1222981 |
| [28] | Zhao K, Tseng BS, Beckerman B, et al. Psl trails guide exploration and microcolony formation in Pseudomonas aeruginosa biofilms. Nature, 2013, 497(7449): 388–391. DOI: 10.1038/nature12155 |
| [29] | Conrad JC, Gibiansky ML, Jin F, et al. Flagella and pili-mediated near-surface single-cell motility mechanisms in P. aeruginosa. Biophys J, 2011, 100(7): 1608–1616. DOI: 10.1016/j.bpj.2011.02.020 |
| [30] | Jin F, Conrad JC, Gibiansky ML, et al. Bacteria use type-Ⅳ pili to slingshot on surfaces. Proc Natl Acad Sci USA, 2011, 108(31): 12617–12622. DOI: 10.1073/pnas.1105073108 |
| [31] | Utada AS, Bennett RR, Fong JCN, et al. Vibrio cholerae use pili and flagella synergistically to effect motility switching and conditional surface attachment. Nat Commun, 2014, 5: 4913. DOI: 10.1038/ncomms5913 |
| [32] | Gloag ES, Turnbull L, Huang A, et al. Self-organization of bacterial biofilms is facilitated by extracellular DNA. Proc Natl Acad Sci USA, 2013, 110(28): 11541–11546. DOI: 10.1073/pnas.1218898110 |
| [33] | Troian SM. Introduction to microfluidics. Phys Today, 2006, 59(12): 58. |
| [34] | Boehm DA, Gottlieb PA, Hua SZ. On-chip microfluidic biosensor for bacterial detection and identification. Sens Actuators B, 2007, 126(2): 508–514. DOI: 10.1016/j.snb.2007.03.043 |
| [35] | Gu SQ, Zhang YX, Zhu Y, et al. Multifunctional picoliter droplet manipulation platform and its application in single cell analysis. Anal Chem, 2011, 83(19): 7570–7576. DOI: 10.1021/ac201678g |
| [36] | Long ZC, Nugent E, Javer A, et al. Microfluidic chemostat for measuring single cell dynamics in bacteria. Lab Chip, 2013, 13(5): 947–954. DOI: 10.1039/c2lc41196b |
| [37] | Su TW, Xue L, Ozcan A. High-throughput lensfree 3D tracking of human sperms reveals rare statistics of helical trajectories. Proc Natl Acad Sci USA, 2012, 109(40): 16018–16022. DOI: 10.1073/pnas.1212506109 |
| [38] | Sugimoto S, Okuda KI, Miyakawa R, et al. Imaging of bacterial multicellular behaviour in biofilms in liquid by atmospheric scanning electron microscopy. Sci Rep, 2016, 6: 25889. DOI: 10.1038/srep25889 |
| [39] | Sgier L, Freimann R, Zupanic A, et al. Flow cytometry combined with visne for the analysis of microbial biofilms and detection of microplastics. Nat Commun, 2016, 7: 11587. DOI: 10.1038/ncomms11587 |
| [40] | Atci E, Babauta JT, Sultana ST, et al. Microbiosensor for the detection of acetate in electrode-respiring biofilms. Biosens Bioelectron, 2016, 81: 517–523. DOI: 10.1016/j.bios.2016.03.027 |
| [41] | Crocker JC, Grier DG. Methods of digital video microscopy for colloidal studies. J Colloid Interface Sci, 1996, 179(1): 298–310. DOI: 10.1006/jcis.1996.0217 |
| [42] | Mason TG, Weitz DA. Optical measurements of frequency-dependent linear viscoelastic moduli of complex fluids. Phys Rev Lett, 1995, 74(7): 1250–1253. DOI: 10.1103/PhysRevLett.74.1250 |
| [43] | Mason TG, Ganesan K, van Zanten JH, et al. Particle tracking microrheology of complex fluids. Phys Rev Lett, 1997, 79(17): 3282–3285. DOI: 10.1103/PhysRevLett.79.3282 |
| [44] | Zhao K, Mason TG. Frustrated rotator crystals and glasses of brownian pentagons. Phys Rev Lett, 2009, 103(20): 208302. DOI: 10.1103/PhysRevLett.103.208302 |
| [45] | Zhao K, Bruinsma R, Mason TG. Entropic crystal-crystal transitions of brownian squares. Proc Natl Acad Sci USA, 2011, 108(7): 2684–2687. DOI: 10.1073/pnas.1014942108 |
| [46] | Zhao K, Bruinsma R, Mason TG. Local chiral symmetry breaking in triatic liquid crystals. Nat Commun, 2012, 3: 801. DOI: 10.1038/ncomms1803 |
| [47] | Gibiansky ML, Conrad JC, Jin F, et al. Bacteria use type Ⅳ pili to walk upright and detach from surfaces. Science, 2010, 330(6001): 197. DOI: 10.1126/science.1194238 |
| [48] | Yan J, Sharo AG, Stone HA, et al. Vibrio cholerae biofilm growth program and architecture revealed by single-cell live imaging. Proc Natl Acad Sci USA, 2016, 113(36): E5337–E5343. DOI: 10.1073/pnas.1611494113 |
| [49] | Seminara A, Angelini TE, Wilking JN, et al. Osmotic spreading of Bacillus subtilis biofilms driven by an extracellular matrix. Proc Natl Acad Sci USA, 2012, 109(4): 1116–1121. DOI: 10.1073/pnas.1109261108 |
| [50] | Sun MZ, Wartel M, Cascales E, et al. From the cover: motor-driven intracellular transport powers bacterial gliding motility. Proc Natl Acad Sci USA, 2011, 108(18): 7559–7564. DOI: 10.1073/pnas.1101101108 |
| [51] | Nielsen MW, Sternberg C, Molin S, et al. Pseudomonas aeruginosa and Saccharomyces cerevisiae biofilm in flow cells. J Vis Exp, 2011, 47: 2383. |
| [52] | Weaver WM, Tseng P, Kunze A, et al. Advances in high-throughput single-cell microtechnologies. Curr Opin Biotechnol, 2014, 25: 114–123. DOI: 10.1016/j.copbio.2013.09.005 |
| [53] | Berg HC, Turner L. Cells of Escherichia coli swim either end forward. Proc Natl Acad Sci USA, 1995, 92(2): 477–479. DOI: 10.1073/pnas.92.2.477 |
| [54] | Turner L, Ryu WS, Berg HC. Real-time imaging of fluorescent flagellar filaments. J Bacteriol, 2000, 182(10): 2793–2801. DOI: 10.1128/JB.182.10.2793-2801.2000 |
| [55] | Macnab RM. Examination of bacterial flagellation by dark-field microscopy. J Clin Microbiol, 1976, 4(3): 258–265. |
| [56] | Diluzio WR, Turner L, Mayer M, et al. Escherichia coli swim on the right-hand side. Nature, 2005, 435(7046): 1271–1274. DOI: 10.1038/nature03660 |
| [57] | Xie L, Altindal T, Chattopadhyay S, et al. Bacterial flagellum as a propeller and as a rudder for efficient chemotaxis. Proc Natl Acad Sci USA, 2011, 108(6): 2246–2251. DOI: 10.1073/pnas.1011953108 |
| [58] | Goto T, Nakata K, Baba K, et al. A fluid-dynamic interpretation of the asymmetric motion of singly flagellated bacteria swimming close to a boundary. Biophys J, 2005, 89(6): 3771–3779. DOI: 10.1529/biophysj.105.067553 |
| [59] | Liu B, Gulino M, Morse M, et al. Helical motion of the cell body enhances Caulobacter crescentus motility. Proc Natl Acad Sci USA, 2014, 111(31): 11252–11256. DOI: 10.1073/pnas.1407636111 |
| [60] | Lele PP, Roland T, Shrivastava A, et al. The flagellar motor of Caulobacter crescentus generates more torque when a cell swims backwards. Nat Phys, 2016, 12(2): 175–178. |
| [61] | Frymier PD, Ford RM, Berg HC, et al. Three-dimensional tracking of motile bacteria near a solid planar surface. Proc Natl Acad Sci USA, 1995, 92(13): 6195–6199. DOI: 10.1073/pnas.92.13.6195 |
| [62] | Lauga E, Diluzio WR, Whitesides GM, et al. Swimming in circles: motion of bacteria near solid boundaries. Biophys J, 2006, 90(2): 400–412. DOI: 10.1529/biophysj.105.069401 |
| [63] | Bradley DE. A function of Pseudomonas aeruginosa PAO polar pili: twitching motility. Can J Microbiol, 1980, 26(2): 146–154. DOI: 10.1139/m80-022 |
| [64] | Zhang RR, Ni L, Jin ZY, et al. Bacteria slingshot more on soft surfaces. Nat Commun, 2014, 5: 5541. DOI: 10.1038/ncomms6541 |
| [65] | Teschler JK, Zamorano-sánchez D, Utada AS, et al. Living in the matrix: assembly and control of Vibrio cholerae biofilms. Nat Rev Microbiol, 2015, 13(5): 255–268. DOI: 10.1038/nrmicro3433 |
| [66] | Hodgkin J, Kaiser D. Genetics of gliding motility in Myxococcus xanthus (myxobacterales): two gene systems control movement. Mol Genet Genomics, 1979, 171(2): 177–191. DOI: 10.1007/BF00270004 |
| [67] | Shi W, Zusman DR. The two motility systems of Myxococcus xanthus show different selective advantages on various surfaces. Proc Natl Acad Sci USA, 1993, 90(8): 3378–3382. DOI: 10.1073/pnas.90.8.3378 |
| [68] | Pelling AE, Li YN, Cross SE, et al. Self-organized and highly ordered domain structures within swarms of Myxococcus xanthus. Cytoskeleton, 2006, 63(3): 141–148. DOI: 10.1002/(ISSN)1097-0169 |
| [69] | Li YN, Sun H, Ma XY, et al. Extracellular polysaccharides mediate pilus retraction during social motility of Myxococcus xanthus. Proc Natl Acad Sci USA, 2003, 100(9): 5443–5448. DOI: 10.1073/pnas.0836639100 |
| [70] | Mauriello EMF, Mignot T, Yang ZM, et al. Gliding motility revisited: how do the myxobacteria move without flagella?. Microbiol Mol Biol Rev, 2010, 74(2): 229–249. DOI: 10.1128/MMBR.00043-09 |
| [71] | Wu YL, Jiang Y, Kaiser D, et al. Social interactions in myxobacterial swarming. PLoS Comput Biol, 2007, 3(12): e253. DOI: 10.1371/journal.pcbi.0030253 |
| [72] | Mattick JS. Type Ⅳ pili and twitching motility. Annu Rev Microbiol, 2002, 56(1): 289–314. DOI: 10.1146/annurev.micro.56.012302.160938 |
| [73] | Shi WY, Sun H. Type Ⅳ pilus-dependent motility and its possible role in bacterial pathogenesis. Infect Immun, 2002, 70(1): 1–4. DOI: 10.1128/IAI.70.1.1-4.2002 |
| [74] | Yu R, Kaiser D. Gliding motility and polarized slime secretion. Mol Microbiol, 2007, 63(2): 454–467. DOI: 10.1111/j.1365-2958.2006.05536.x |
| [75] | Kaiser D. Coupling cell movement to multicellular development in myxobacteria. Nat Rev Microbiol, 2003, 1(1): 45–54. DOI: 10.1038/nrmicro733 |
| [76] | Wu YL, Jiang Y, Kaiser AD, et al. Self-organization in bacterial swarming: lessons from myxobacteria. Phys Biol, 2011, 8(5): 055003. DOI: 10.1088/1478-3975/8/5/055003 |
| [77] | Sun H, Zusman DR, Shi WY. Type Ⅳ pilus of Myxococcus xanthus is a motility apparatus controlled by the frz chemosensory system. Curr Biol, 2000, 10(18): 1143–1146. DOI: 10.1016/S0960-9822(00)00705-3 |
| [78] | Verstraeten N, Braeken K, Debkumari B, et al. Living on a surface: swarming and biofilm formation. Trends Microbiol, 2008, 16(10): 496–506. DOI: 10.1016/j.tim.2008.07.004 |
| [79] | Kaiser D. Bacterial swarming: a re-examination of cell-movement patterns. Curr Biol, 2007, 17(14): R561–R570. DOI: 10.1016/j.cub.2007.04.050 |
| [80] | Thutupalli S, Sun MZ, Bunyak F, et al. Directional reversals enable Myxococcus xanthus cells to produce collective one-dimensional streams during fruiting-body formation. J R Soc Interface, 2015, 12(109): 20150049. DOI: 10.1098/rsif.2015.0049 |
| [81] | Wang SW, Yu S, Zhang ZY, et al. Coordination of swarming motility, biosurfactant synthesis, and biofilm matrix exopolysaccharide production in Pseudomonas aeruginosa. Appl Environ Microbiol, 2014, 80(21): 6724–6732. DOI: 10.1128/AEM.01237-14 |
| [82] | Yeung ATY, Torfs ECW, Jamshidi F, et al. Swarming of Pseudomonas aeruginosa is controlled by a broad spectrum of transcriptional regulators, including MetR. J Bacteriol, 2009, 191(18): 5592–5602. DOI: 10.1128/JB.00157-09 |
| [83] | Zhang RJ, Turner L, Berg HC. The upper surface of an Escherichia coli swarm is stationary. Proc Natl Acad Sci USA, 2010, 107(1): 288–290. DOI: 10.1073/pnas.0912804107 |
| [84] | Turner L, Zhang RJ, Darnton NC, et al. Visualization of flagella during bacterial swarming. J Bacteriol, 2010, 192(13): 3259–3267. DOI: 10.1128/JB.00083-10 |
| [85] | Wu YL, Hosu BG, Berg HC. Microbubbles reveal chiral fluid flows in bacterial swarms. Proc Natl Acad Sci USA, 2011, 108(10): 4147–4151. DOI: 10.1073/pnas.1016693108 |
| [86] | Yoon S, Kim K, Kim JK. Live-cell imaging of swarming bacteria in a fluidic biofilm formed on a soft agar gel substrate. J Visualization, 2013, 16(2): 123–131. DOI: 10.1007/s12650-013-0159-0 |
| [87] | Wu YL, Kaiser AD, Jiang Y, et al. Periodic reversal of direction allows myxobacteria to swarm. Proc Natl Acad Sci USA, 2009, 106(4): 1222–1227. DOI: 10.1073/pnas.0811662106 |
| [88] | Pasmore M, Todd P, Smith S, et al. Effects of ultrafiltration membrane surface properties on Pseudomonas aeruginosa biofilm initiation for the purpose of reducing biofouling. J Membrane Sci, 2001, 194(1): 15–32. DOI: 10.1016/S0376-7388(01)00468-9 |
| [89] | McGuffie BA, Vallet-Gely I, Dove SL. Sigma factor and anti-sigma factor that control swarming motility and biofilm formation in Pseudomonas aeruginosa. J Bacteriol, 2015, 198(5): 755–765. |
| [90] | Rasamiravaka T, Labtani Q, Duez P, et al. The formation of biofilms by Pseudomonas aeruginosa: a review of the natural and synthetic compounds interfering with control mechanisms. BioMed Res Int, 2015, 2015: 759348. |
| [91] | Martínez LC, Vadyvaloo V. Mechanisms of post-transcriptional gene regulation in bacterial biofilms. Front Cell Infect Microbiol, 2014, 4: 38. |
| [92] | Ng WL, Bassler BL. Bacterial quorum-sensing network architectures. Annu Rev Genet, 2009, 43(1): 197–222. DOI: 10.1146/annurev-genet-102108-134304 |
| [93] | Chen X, Schauder S, Potier N, et al. Structural identification of a bacterial quorum-sensing signal containing boron. Nature, 2002, 415(6871): 545–549. DOI: 10.1038/415545a |
| [94] | Papenfort K, Bassler BL. Quorum sensing signal-response systems in gram-negative bacteria. Nat Rev Microbiol, 2016, 14(9): 576–588. DOI: 10.1038/nrmicro.2016.89 |
| [95] | Shrout JD, Tolker-Nielsen T, Givskov M, et al. The contribution of cell-cell signaling and motility to bacterial biofilm formation. MRS Bull, 2011, 36(5): 367–373. DOI: 10.1557/mrs.2011.67 |
| [96] | Singh PK, Schaefer AL, Parsek MR, et al. Quorum-sensing signals indicate that cystic fibrosis lungs are infected with bacterial biofilms. Nature, 2000, 407(6805): 762–764. DOI: 10.1038/35037627 |
| [97] | Zhang Y, Ducret A, Shaevitz J, et al. From individual cell motility to collective behaviors: insights from a prokaryote, Myxococcus xanthus. FEMS Microbiol Rev, 2012, 36(1): 149–164. DOI: 10.1111/j.1574-6976.2011.00307.x |
| [98] | S gaard-Andersen L, Overgaard M, Lobedanz S, et al. Coupling gene expression and multicellular morphogenesis during fruiting body formation in Myxococcus xanthus. Mol Microbiol, 2003, 48(1): 1–8. DOI: 10.1046/j.1365-2958.2003.03399.x |
| [99] | Zusman DR, Scott AE, Yang ZM, et al. Chemosensory pathways, motility and development in Myxococcus xanthus. Nat Rev Microbiol, 2007, 5(11): 862–872. DOI: 10.1038/nrmicro1770 |
| [100] | Lazazzera BA. Quorum sensing and starvation: signals for entry into stationary phase. Curr Opin Microbiol, 2000, 3(2): 177–182. DOI: 10.1016/S1369-5274(00)00072-2 |
| [101] | Downard J, Ramaswamy SV, Kil KS. Identification of esg, a genetic locus involved in cell-cell signaling during Myxococcus xanthus development. J Bacteriol, 1993, 175(24): 7762–7770. DOI: 10.1128/jb.175.24.7762-7770.1993 |
| [102] | Hagen DC, Bretscher AP, Kaiser D. Synergism between morphogenetic mutants of Myxococcus xanthus. Dev Biol, 1978, 64(2): 284–296. DOI: 10.1016/0012-1606(78)90079-9 |
| [103] | Li S, Lee BU, Shimkets LJ. csgA expression entrains Myxococcus xanthus development. Gene Dev, 1992, 6(3): 401–410. DOI: 10.1101/gad.6.3.401 |
| [104] | Kruse T, Lobedanz S, Berthelsen NMS, et al. C-signal: a cell surface-associated morphogen that induces and co-ordinates multicellular fruiting body morphogenesis and sporulation in Myxococcus xanthus. Mol Microbiol, 2001, 40(1): 156–168. DOI: 10.1046/j.1365-2958.2001.02365.x |
| [105] | Kim MK, Ingremeau F, Zhao AS, et al. Local and global consequences of flow on bacterial quorum sensing. Nat Microbiol, 2016, 1: 15005. DOI: 10.1038/nmicrobiol.2015.5 |
| [106] | R?mling U, Galperin MY, Gomelsky M. Cyclic di-GMP: the first 25 years of a universal bacterial second messenger. Microbiol Mol Biol Rev, 2013, 77(1): 1–52. DOI: 10.1128/MMBR.00043-12 |
| [107] | Mills E, Pultz IS, Kulasekara HD, et al. The bacterial second messenger c-di-GMP: mechanisms of signalling. Cell Microbiol, 2011, 13(8): 1122–1129. DOI: 10.1111/cmi.2011.13.issue-8 |
| [108] | Nair HA, Periasamy S, Yang L, et al. Real time, spatial, and temporal mapping of the distribution of c-di-GMP during biofilm development. J Biol Chem, 2017, 292(2): 477–487. DOI: 10.1074/jbc.M116.746743 |
| [109] | Christen M, Kulasekara HD, Christen B, et al. Asymmetrical distribution of the second messenger c-di-GMP upon bacterial cell division. Science, 2010, 328(5983): 1295–1297. DOI: 10.1126/science.1188658 |
| [110] | Ha DG, O'Toole GA. C-di-GMP and its effects on biofilm formation and dispersion: a Pseudomonas aeruginosa review. Microbiol Spectr, 2015, 3(2): 1–15. |
| [111] | Dasgupta N, Arora SK, Ramphal R. fleN, a gene that regulates flagellar number in Pseudomonas aeruginosa. J Bacteriol, 2000, 182(2): 357–364. DOI: 10.1128/JB.182.2.357-364.2000 |
| [112] | Matsuyama BY, Krasteva PV, Baraquet C, et al. Mechanistic insights into c-di-GMP-dependent control of the biofilm regulator FleQ from Pseudomonas aeruginosa. Proc Natl Acad Sci USA, 2016, 113(2): E209–E218. DOI: 10.1073/pnas.1523148113 |
| [113] | Krasteva PV, Fong JCN, Shikuma NJ, et al. Vibrio cholerae VpsT regulates matrix production and motility by directly sensing cyclic di-GMP. Science, 2010, 327(5967): 866–868. DOI: 10.1126/science.1181185 |
| [114] | Tischler AD, Camilli A. Cyclic diguanylate (c-di-GMP) regulates Vibrio cholerae biofilm formation. Mol Microbiol, 2004, 53(3): 857–869. DOI: 10.1111/j.1365-2958.2004.04155.x |
| [115] | Yang L, Hu YF, Liu Y, et al. Distinct roles of extracellular polymeric substances in Pseudomonas aeruginosa biofilm development. Environ Microbiol, 2011, 13(7): 1705–1717. DOI: 10.1111/j.1462-2920.2011.02503.x |
| [116] | Colvin KM, Gordon VD, Murakami K, et al. The Pel polysaccharide can serve a structural and protective role in the biofilm matrix of Pseudomonas aeruginosa. PLoS Pathog, 2011, 7(1): e1001264. DOI: 10.1371/journal.ppat.1001264 |
| [117] | Colvin KM, Irie Y, Tart CS, et al. The Pel and Psl polysaccharides provide Pseudomonas aeruginosa structural redundancy within the biofilm matrix. Environ Microbiol, 2012, 14(8): 1913–1928. DOI: 10.1111/j.1462-2920.2011.02657.x |
| [118] | Franklin MJ, Nivens DE, Weadge JT, et al. Biosynthesis of the Pseudomonas aeruginosa extracellular polysaccharides, alginate, Pel, and Psl. Front Microbiol, 2011, 2: 167. |
| [119] | Irie Y, Borlee BR, O'connor JR, et al. Self-produced exopolysaccharide is a signal that stimulates biofilm formation in Pseudomonas aeruginosa. Proc Natl Acad Sci USA, 2012, 109(50): 20632–20636. DOI: 10.1073/pnas.1217993109 |
| [120] | Ghafoor A, Hay ID, Rehm BHA. Role of exopolysaccharides in Pseudomonas aeruginosa biofilm formation and architecture. Appl Environ Microbiol, 2011, 77(15): 5238–5246. DOI: 10.1128/AEM.00637-11 |
| [121] | Wang SW, Parsek MR, Wozniak DJ, et al. A spider web strategy of type Ⅳ pili-mediated migration to build a fibre-like Psl polysaccharide matrix in Pseudomonas aeruginosa biofilms. Environ Microbiol, 2013, 15(8): 2238–2253. DOI: 10.1111/emi.2013.15.issue-8 |
| [122] | Yu S, Su TT, Wu HJ, et al. PslG, a self-produced glycosyl hydrolase, triggers biofilm disassembly by disrupting exopolysaccharide matrix. Cell Res, 2015, 25(12): 1352–1367. DOI: 10.1038/cr.2015.129 |
| [123] | De Rienzo MA, Stevenson PS, Marchant R, et al. Pseudomonas aeruginosa biofilm disruption using microbial surfactants. J Appl Microbiol, 2016, 120(4): 868–876. DOI: 10.1111/jam.13049 |
| [124] | Drescher K, Dunkel J, Nadell CD, et al. Architectural transitions in Vibrio cholerae biofilms at single-cell resolution. Proc Natl Acad Sci USA, 2016, 113(14): E2066–E2072. DOI: 10.1073/pnas.1601702113 |
| [125] | Anyan ME, Amiri A, Harvey CW, et al. Type Ⅳ pili interactions promote intercellular association and moderate swarming of Pseudomonas aeruginosa. Proc Natl Acad Sci USA, 2014, 111(50): 18013–18018. DOI: 10.1073/pnas.1414661111 |
| [126] | Bos J, Zhang QC, Vyawahare S, et al. Emergence of antibiotic resistance from multinucleated bacterial filaments. Proc Natl Acad Sci USA, 2015, 112(1): 178–183. DOI: 10.1073/pnas.1420702111 |
| [127] | Zhang QC, Lambert G, Liao D, et al. Acceleration of emergence of bacterial antibiotic resistance in connected microenvironments. Science, 2011, 333(6050): 1764–1767. DOI: 10.1126/science.1208747 |
| [128] | Saar BG, Freudiger CW, Reichman J, et al. Video-rate molecular imaging in vivo with stimulated Raman scattering. Science, 2010, 330(6009): 1368–1370. DOI: 10.1126/science.1197236 |
| [129] | Ertürk A, Mauch CP, Hellal F, et al. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nat Med, 2011, 18(1): 166–171. DOI: 10.1038/nm.2600 |
| [130] | Darenfed H, Grenier D, Mayrand D. Acquisition of plasmin activity by Fusobacterium nucleatum subsp. nucleatum and potential contribution to tissue destruction during periodontitis. Infect Immun, 1999, 67(12): 6439–6444. |
| [131] | Benomar S, Ranava D, Cárdenas ML, et al. Nutritional stress induces exchange of cell material and energetic coupling between bacterial species. Nat Commun, 2015, 6: 6283. DOI: 10.1038/ncomms7283 |
| [132] | Shapira M. Gut microbiotas and host evolution: scaling up symbiosis. Trends Ecol Evol, 2016, 31(7): 539–549. DOI: 10.1016/j.tree.2016.03.006 |
| [133] | Pande S, Shitut S, Freund L, et al. Metabolic cross-feeding via intercellular nanotubes among bacteria. Nat Commun, 2015, 6: 6238. DOI: 10.1038/ncomms7238 |
| [134] | Dubey GP, Ben-Yehuda S, et al. Intercellular nanotubes mediate bacterial communication. Cell, 2011, 144(4): 590–600. DOI: 10.1016/j.cell.2011.01.015 |
| [135] | Chatzidaki-Livanis M, Geva-Zatorsky N, Comstock LE. Bacteroides fragilis type Ⅵ secretion systems use novel effector and immunity proteins to antagonize human gut bacteroidales species. Proc Natl Acad Sci USA, 2016, 113(13): 3627–3632. DOI: 10.1073/pnas.1522510113 |
| [136] | Coyte KZ, Schluter J, Foster KR. The ecology of the microbiome: networks, competition, and stability. Science, 2015, 350(5): 663–666. |
| [137] | Xu J, Ma B, Su XQ, et al. Emerging trends for microbiome analysis: from single-cell functional imaging to microbiome big data. Engineering, 2017, 3(1): 66–70. DOI: 10.1016/J.ENG.2017.01.020 |
 2017, Vol. 33
2017, Vol. 33