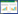中国科学院微生物研究所、中国微生物学会主办
文章信息
- 甘田生, 龚湘君
- Tiansheng Gan, Xiangjun Gong
- 生物被膜的物理特性及其表征
- Characterization of the physical properties of biofilms
- 生物工程学报, 2017, 33(9): 1390-1398
- Chinese Journal of Biotechnology, 2017, 33(9): 1390-1398
- 10.13345/j.cjb.170062
-
文章历史
- Received: February 22, 2017
- Accepted: July 27, 2017
生物被膜是由吸附于固体表面或气液界面的微生物相互粘附在一起,并嵌入其所分泌的胞外多聚物中而形成的固着微生物群落[1]。只要条件适宜,绝大多数细菌都可在各种表面形成生物被膜。细菌生物被膜几乎涉及到我们人类生产和生活的各个方面。生物被膜的形成和发展过程中涉及到很多的物理学过程。因此,从物理学的视角对生物被膜进行研究,有助于我们对生物被膜的形成、发展以及组织结构有更加完整的认识和理解,为开发控制生物被膜的策略提供新的思路[2]。从生物被膜的形成过程来看,首先发生的是细菌在表面的粘附。在细菌附着到表面的过程中,涉及到两个极为重要的物理性质,一是细菌在近界面附近的运动,二是细菌与表面之间的相互作用力。细菌的粘附决定了细菌生物被膜的形成以及后续的发展。当生物被膜初步形成并生长成熟,对其形态结构的表征以及机械性能的研究有助于我们从整体上把握生物被膜的形貌和机械稳定性。本文将从细菌粘附过程、生物被膜的形态结构以及生物被膜的机械性能等三个方面对生物被膜进行论述,同时介绍相关的表征技术。
1 细菌的粘附及其表征细菌粘附到表面是生物被膜形成的第一步,也是最为关键的一步。细菌和表面间存在着多种相互作用,其中,细菌在非生物表面的粘附主要通过非特异性相互作用,如疏水相互作用;而在活体或者衰亡的组织表面的粘附则主要通过特异性分子间的结合作用,如凝集素、配体、粘附素等[3]。细菌的粘附过程可以分为两步:“停靠” (Docking)和“锁定”(Locking)[4]。“停靠”过程主要是浮游细菌刚刚粘附至表面上,处于一个可逆的状态,即细菌既可脱附也可继续留在表面生长。这一过程可再细分为两步。首先,浮游细菌被流体冲刷或者带鞭毛的能动型细菌游走至界面附近几十纳米处;接下来,细菌需要克服细菌表面与可附着表面之间的排斥作用力,一般是静电作用力,才可附着到表面。“锁定”则是可逆附着的细菌通过分泌胞外多聚物将其自身牢牢地固着在表面上。之后,细菌开始增长繁殖,逐渐形成生物被膜。
1.1 细菌的近界面运动行为从细菌粘附的过程可以得知,细菌在界面附近的运动行为对细菌的粘附起到非常关键的作用。有研究表明,细菌自身的运动行为会对生物被膜的形成以及其形态结构有显著影响[5-6]。因此,研究近界面附近细菌的运动行为对我们了解细菌的粘附以及生物被膜的形成具有非常重要的意义。早在1972年,Henrichsen就利用了显微照相机系统地观察了40种细菌在表面上的运动行为,并归纳出了6种运动模式:游动(Swimming)、涌动(Swarming)、滑翔(Gliding)、蹭行(Twitching)、滑行(Sliding)和突进(Darting)[7]。随着现代荧光标记和显微镜技术的发展,科学家们对细菌的运动行为进行了更为细致的研究。细菌采取的运动形式和细菌自身的运动器官如鞭毛、菌毛等有关,例如,游动主要是由于鞭毛的旋转[8],涌动依靠鞭毛和胞外分泌物[9],而蹭行则来自于菌毛的释放和收缩[10]。传统的光学显微技术可获知细菌在二维聚焦平面上的运动行为,然而其丢失了纵向上不在聚焦面的行为信息。数字全息显微技术(Digital holographic microscopy,DHM)是正在兴起的三维成像技术,可以实时获取物体的三维形貌以及空间位置等信息[11]。DHM具有对测试样品无损伤、无需染色、高通量和定量化等优点。利用此工具,我们课题组研究了大肠杆菌在不同性质的聚合物表面的粘附过程[12],结果发现细菌尾部和表面碰撞是细菌在疏水性聚合物表面上粘附的主要诱因。
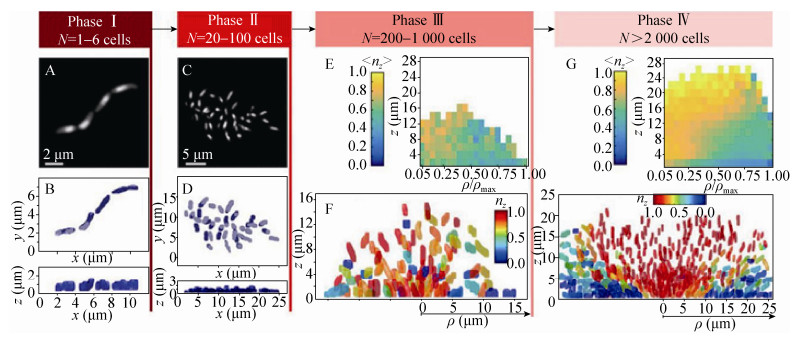
1.2 细菌与表面相互作用力的表征当细菌靠近表面至50 nm左右时,细菌能否吸附到表面取决于细菌和表面之间的相互作用力。这些相互作用力与细菌和表面之间的距离有关,如图 1所示。当距离大于50 nm时,范德华力起主要作用;而当距离在10–20 nm之间时,范德华力和静电作用力同时作用;在距离进一步减少至1.5 nm时,除了范德华力和静电作用力,一些特异性相互作用也可能存在[13]。为研究这些相互作用如何影响着细菌的粘附过程,研究者们发展了许多技术方法去测量细菌或者相关的生物分子和表面之间的相互作用力,例如,流室实验(Flow-chamber experiments)、表面力仪(Surface force apparatus)、生物膜力学探针(Biomembrane force probe)、光镊和磁镊(Optical and magnetic tweezers)、原子力显微镜(Atomic force microscopy,AFM)[14]。AFM具有纳米级的分辨率和较宽的力测量范围(5 pN–100 nN),因此得到了非常广泛的应用。它不仅可用于测量单个受体-配体结合的相互作用(约60–80 pN),也能够测量整个细菌和表面之间的相互作用(大于1 nN)[15]。
|
| 图 1 细菌与表面之间的作用力与距离之间的关系 Figure 1 Interaction forces vs. distance between bacteria and surface. |
| |
AFM利用一根对力非常敏感的微悬臂去探测针尖与表面之间的相互作用力,进而获得样品表面的纳米级形貌[16]。将单个细菌固定在微悬臂上制备成细菌探针,即可用于测量细菌与各种表面以及细菌与细菌之间的相互作用力,这就是所谓的单细胞力谱(Single-cell force spectroscopy,SCFS) (图 2)[17]。SCFS可以获得细菌粘附过程中非特异性和特异性相互作用力的直接、定量的信息。Herman等研究了表皮葡萄球菌Staphylococcus epidermindis表面的粘附素SdrG和血浆中的纤维蛋白原(Fibrinogen)之间的特异性相互作用[18]。他们发现,单个SdrG-Fibrinogen之间结合作用力非常强,约为2 nN。表皮葡萄球菌在植入生物材料表面形成生物被膜的能力与这种极强的特异结合作用力高度有关,极强的结合作用有利于生物被膜抵抗来自其生长环境的剪切力。Alsteens等将表达了白色念珠菌粘附蛋白Als5p的酵母细胞固定到无针尖探针上,然后利用SCFS测量了该蛋白与疏水表面之间的非特异性相互作用[19]。由于蛋白的疏水重复单元和疏水表面间的结合,他们测量得到较大的作用力(1.25 nN)和较长的断裂长度(400 nm)。
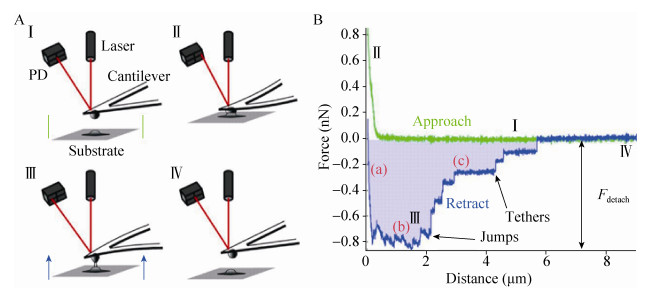
细菌粘附至表面后,开始分泌和积累胞外多聚物。附着的细菌开始增殖,浮游细菌也会继续粘附到表面,最后形成的生物被膜具有非常复杂的三维结构。在大多数生物被膜中,胞外多聚物的质量占到生物被膜干质量的90%以上。胞外多聚物维持着生物被膜的机械稳定性,能够将细菌固定于生物被膜邻近的区域中,便于细菌之间发生交流,形成协同的“细菌社会”[20]。人们对生物被膜的认识有赖于各种成像表征技术的发展。激光共聚焦显微镜(Confocal laser scanning microscopy,CLSM)是研究生物被膜结构最为常用的工具。利用特定的荧光分子作标记,CLSM可以在生物被膜的生长环境中对其形态结构实现原位和无损的观察研究。Bridier等利用高通量的CLSM研究了60种条件致病菌形成的生物被膜的形态结构[21]。他们发现不同生物被膜之间的结构多样性,例如绿脓杆菌Pseudomonas aeruginosa生物被膜的蘑菇状结构,沙门氏菌Salmonella enteric生物被膜中存在的孔洞结构。高分辨率CLSM的出现使得追踪细菌个体在生物被膜中空间分布及形态成为可能。Stewart等利用高分辨率CLSM研究了表皮葡萄球菌Staphylococcus epidermis生物被膜在不同浓度的氯化钠溶液中的结构变化[22]。结果表明,高盐浓度会降低细菌在生物被膜中的密度。Drescher等利用高分辨率CLSM对霍乱弧菌Vibrio cholerae生物被膜中所有的细菌进行成像[23]。他们通过分析细菌在生物被膜中的空间位置、大小和取向来研究生物被膜在生长过程中的结构转变,结果表明生物被膜的生长可以分为4个阶段(图 3)。在第一阶段,少数细菌粘附在表面形成高度有序的一维线状结构;第二阶段,吸附在表面上的细菌形成二维的菌落,而细菌在菌落中是无规排布的;第三阶段,形成具有较低细菌密度(约为200–1 000个细菌)的三维菌落;第四阶段是形成细菌密度很高的菌落(大于2 000个细菌)。
除了CLSM外,扫描电子显微镜(Scanning electron microscope,SEM)、透射电子显微镜(Transmission electron microscope,TEM)以及原子力显微镜(Atomic force microscope,AFM)也在生物被膜形态结构的研究中具有广泛的应用。相比于CLSM,SEM、TEM和AFM具有更高的空间分辨率,可看到生物被膜中更为精细的结构。然而,利用SEM表征生物被膜样品时,样品需要经过固定、染色、干燥和喷涂导电涂层等制备过程,这些预处理过程可能都会改变生物被膜本来的结构。近年来出现的低温场发射扫描电子显微镜(cryoFESEM)、可变压扫描电子显微镜(VPSEM)和环境扫描电子显微镜(ESEM)可以在一定程度上保持生物被膜本来的结构状态,因而在生物被膜的高分辨率成像中得到广泛的应用[24-27]。TEM可用于观察生物被膜中的胞外多聚物。通过利用特定的染色剂对胞外多聚物进行染色,进而可以研究胞外多聚物的性质以及细菌和这些多聚物之间的结合方式[28-30]。
AFM是空间分辨率可以达到纳米级的成像工具,可以在空气以及溶液环境下对样品进行成像,在生物样品的研究中具有非常重要的应用。AFM可以在溶液状态下直接对生物被膜进行原位成像,而不需要对其进行特别的处理,这样可以最大程度保持生物被膜的结构完整性[31-33]。然而,AFM也存在一些局限性,例如成像范围较小。而且,在溶液中成像时,AFM探针容易损伤软的、凝胶状的生物被膜。因此,有时候需要先将生物被膜干燥后,在空气中对其进行成像进而获得高分辨率的形貌图。然而干燥过程也会改变生物被膜的形态结构,因此,在这种情况下得到的结果与真实情况有一定的偏离[34-35]。
3 生物被膜的机械性能及其表征生物被膜在生长过程中会受到很多环境压力,如海洋环境中的水压、输水管道的流体剪切力以及静脉导管中的血液流动等[36]。生物被膜必须具有一定的机械稳定性,才能在受到这些流体剪切力后不会像液体一样流动。同时由于细菌需要生长繁殖,生物被膜也不会完全像固体一样。研究表明,生物被膜可视为一种粘弹性的聚合物材料,即具有流体粘滞性和固体弹性的综合性质[37]。研究生物被膜的机械性能是一个非常重要的科学和经济问题。生物被膜的物理机械性能决定着生物被膜的形态和机械稳定性,影响到生物被膜中的物质传输以及生物被膜的物理清除过程。
为了研究生物被膜的机械性能,研究者们除了使用平板剪切流变仪和平板压缩实验等常用方法外[38-39],还采用了很多新颖的技术方法,例如,流动池方法(Flow cell method)[40-41]、微悬臂法(Micro-cantilever method)[42-43]、流体动力学测定(Fluid dynamic gauging)[44]、磁镊(Magnetic tweezers)[45]以及原子力显微镜(AFM)[31, 34, 46-48]等。利用这些技术方法可以测量得到生物被膜的力-位移或应力-应变曲线、弹性模量、粘度、松弛时间、储能和损耗模量、内聚能和粘附力等力学参数。
Shaw等利用平板流变仪研究了44种不同的生物被膜,包括在流变仪中原位培养的以及从自然界中收集回来的生物被膜[49]。尽管不同生物被膜的剪切模量和粘度差别很大,但是有趣的是,它们具有同样的弹性松弛时间,约为18 min (图 4)。松弛时间是生物被膜受到机械应力产生的可逆形变转化为永久形变(即生物被膜遭到破坏)需要的时间。这一松弛时间(18 min)和生物被膜生长过程中细菌的倍增时间具有同一量级,可能对于生物被膜的生存来说,这一时间尺度在生物被膜抵抗外应力时具有非常重要的意义。Lieleg等研究了生物被膜经过化学试剂处理后机械性能的变化[50]。他们发现,生物被膜中细菌的活性和生物被膜的机械稳定性没有必然的联系,单纯杀死细菌并不能将生物被膜从表面上清除。因此,一些可以降低生物被膜机械稳定性的抗菌剂也许会有更好的效果。
AFM除了可以对生物被膜进行高分辨率的成像外,在测量生物被膜机械性能方面也具有非常广泛的应用。AFM可以测量生物被膜的内聚能[46]、弹性模量[34, 47]和粘附力[31, 48]等性能。磁力调制原子力显微镜(Magnetic force modulation AFM,MF-AFM)的发展使得AFM可用于研究样品的机械性能对扰动频率的依赖性。MF-AFM利用一根磁性探针以一定的力压进样品表面一定的深度,然后通过一个外加磁场使得探针在设定的频率范围内振动,将探针振动的振幅和相位角测量出来后可用于计算样品在不同频率下的机械性能。我们课题组利用MF-AFM研究了大肠杆菌生物被膜在其生长过程中机械性能的变化,测量了距离生物被膜表面不同深度下的有效刚度(Effective stiffness)[51]。随着培养时间的增长,生物被膜的结构变得越来越均匀。测量得到的有效刚度随频率的变化符合幂律规则,说明生物被膜的一些流变行为和软玻璃态材料有着相似之处。
4 总结在本文中,我们讨论了细菌粘附、生物被膜组织结构和机械性能等方面的一些研究进展。浮游细菌粘附到表面是触发生物被膜形成和发展的关键过程,对这一过程进行深入的研究分析是十分必要的。我们课题组搭建了三维数字全息显微镜(DHM)观察细菌在界面附近的运动行为以及细菌与表面的碰撞过程,研究在生物被膜形成初期细菌的粘附机理。DHM实时的三维成像技术将会使其成为生物被膜研究中的重要工具。生物被膜可看作是一种非常复杂的粘弹性材料,研究生物被膜的结构和机械稳定性对于了解它如何能在各种恶劣环境中生存和发展是十分重要的。为此,我们课题组搭建了磁力调制原子力显微镜(MF-AFM)对生物被膜的机械性能的研究。MF-AFM兼具高分辨率成像和微流变表征的功能,在生物被膜结构以及机械性能的研究中具有非常广阔的应用前景。
| [1] | Donlan RM, Costerton JW. Biofilms: survival mechanisms of clinically relevant microorganisms. Clin Microbiol Rev, 2002, 15(2): 167–193. DOI: 10.1128/CMR.15.2.167-193.2002 |
| [2] | Mazza MG. The physics of biofilms-an introduction. J Phys D Appl Phys, 2016, 49(20): 203001. DOI: 10.1088/0022-3727/49/20/203001 |
| [3] | Carpentier B, Cerf O. Biofilms and their consequences, with particular reference to hygiene in the food industry. J Appl Bacteriol, 1993, 75(6): 499–511. DOI: 10.1111/j.1365-2672.1993.tb01587.x |
| [4] | Dunne WM Jr. Bacterial adhesion: seen any good biofilms lately?. Clin Microbiol Rev, 2002, 15(2): 155–166. DOI: 10.1128/CMR.15.2.155-166.2002 |
| [5] | Wood TK, Barrios AFG, Herzberg M, et al. Motility influences biofilm architecture in Escherichia coli. Appl Microbiol Biotechnol, 2006, 72(2): 361–367. DOI: 10.1007/s00253-005-0263-8 |
| [6] | Lemon KP, Higgins DE, Kolter R. Flagellar motility is critical for Listeria monocytogenes biofilm formation. J Bacteriol, 2007, 189(12): 4418–4424. DOI: 10.1128/JB.01967-06 |
| [7] | Henrichsen J. Bacterial surface translocation: a survey and a classification. Bacteriol Rev, 1972, 36(4): 478–503. |
| [8] | Scharf BE, Fahrner KA, Turner L, et al. Control of direction of flagellar rotation in bacterial chemotaxis. Proc Natl Acad Sci USA, 1998, 95(1): 201–206. DOI: 10.1073/pnas.95.1.201 |
| [9] | Kearns DB. A field guide to bacterial swarming motility. Nat Rev Microbiol, 2010, 8(9): 634–644. DOI: 10.1038/nrmicro2405 |
| [10] | Skerker JM, Berg HC. Direct observation of extension and retraction of type Ⅳ pili. Proc Natl Acad Sci USA, 2001, 98(12): 6901–6904. DOI: 10.1073/pnas.121171698 |
| [11] | Yu X, Hong J, Liu CG, et al. Review of digital holographic microscopy for three-dimensional profiling and tracking. Opt Eng, 2014, 53(11): 112306. DOI: 10.1117/1.OE.53.11.112306 |
| [12] | Qi M, Gong XJ, Wu B, et al. Landing dynamics of swimming bacteria on a polymeric surface: effect of surface properties. Langmuir, 2017, 33(14): 3525–3533. DOI: 10.1021/acs.langmuir.7b00439 |
| [13] | Preedy E, Perni S, Nipiĉ D, et al. Surface roughness mediated adhesion forces between borosilicate glass and gram-positive bacteria. Langmuir, 2014, 30(31): 9466–9476. DOI: 10.1021/la501711t |
| [14] | Dufrêne YF. Sticky microbes: forces in microbial cell adhesion. Trends Microbiol, 2015, 23(6): 376–382. DOI: 10.1016/j.tim.2015.01.011 |
| [15] | Müller DJ, Helenius J, Alsteens D, et al. Force probing surfaces of living cells to molecular resolution. Nat Chem Biol, 2009, 5(6): 383–390. DOI: 10.1038/nchembio.181 |
| [16] | Binnig G, Quate CF, Gerber C. Atomic force microscope. Phys Rev Lett, 1986, 56(9): 930–933. DOI: 10.1103/PhysRevLett.56.930 |
| [17] | Helenius J, Heisenberg CP, Gaub HE, et al. Single-cell force spectroscopy. J Cell Sci, 2008, 121(11): 1785–1791. DOI: 10.1242/jcs.030999 |
| [18] | Herman P, El-Kirat-Chatel S, Beaussart A, et al. The binding force of the staphylococcal adhesin SdrG is remarkably strong. Mol Microbiol, 2014, 93(2): 356–368. DOI: 10.1111/mmi.2014.93.issue-2 |
| [19] | Alsteens D, Beaussart A, Derclaye S, et al. Single-cell force spectroscopy of Als-mediated fungal adhesion. Anal Methods, 2013, 5(15): 3657–3662. DOI: 10.1039/c3ay40473k |
| [20] | Flemming HC, Wingender J. The biofilm matrix. Nat Rev Microbiol, 2010, 8(9): 623–633. |
| [21] | Bridier A, Dubois-Brissonnet F, Boubetra A, et al. The biofilm architecture of sixty opportunistic pathogens deciphered using a high throughput CLSM method. J Microbiol Methods, 2010, 82(1): 64–70. DOI: 10.1016/j.mimet.2010.04.006 |
| [22] | Stewart EJ, Satorius AE, Younger JG, et al. Role of environmental and antibiotic stress on Staphylococcus epidermidis biofilm microstructure. Langmuir, 2013, 29(23): 7017–7024. DOI: 10.1021/la401322k |
| [23] | Drescher K, Dunkel J, Nadell CD, et al. Architectural transitions in Vibrio cholerae biofilms at single-cell resolution. Proc Natl Acad Sci USA, 2016, 113(14): E2066–E2072. DOI: 10.1073/pnas.1601702113 |
| [24] | Kachlany SC, Levery SB, Kim JS, et al. Structure and carbohydrate analysis of the exopolysaccharide capsule of Pseudomonas putida G7. Environ Microbiol, 2001, 3(12): 774–784. DOI: 10.1046/j.1462-2920.2001.00248.x |
| [25] | Priester JH, Horst AM, van de Werfhorst LC, et al. Enhanced visualization of microbial biofilms by staining and environmental scanning electron microscopy. J Microbiol Methods, 2007, 68(3): 577–587. DOI: 10.1016/j.mimet.2006.10.018 |
| [26] | Alhede M, Qvortrup K, Liebrechts R, et al. Combination of microscopic techniques reveals a comprehensive visual impression of biofilm structure and composition. FEMS Immunol Med Microbiol, 2012, 65(2): 335–342. DOI: 10.1111/j.1574-695X.2012.00956.x |
| [27] | Weber K, Delben J, Bromage TG, et al. Comparison of SEM and VPSEM imaging techniques with respect to Streptococcus mutans biofilm topography. FEMS Microbiol Lett, 2014, 350(2): 175–179. DOI: 10.1111/fml.2014.350.issue-2 |
| [28] | Vitkov L, Hermann A, Krautgartner WD, et al. Chlorhexidine-induced ultrastructural alterations in oral biofilm. Microsc Res Techiniq, 2005, 68(2): 85–89. DOI: 10.1002/(ISSN)1097-0029 |
| [29] | Hu XB, Xu K, Wang Z, et al. Characteristics of biofilm attaching to carriers in moving bed biofilm reactor used to treat vitamin C wastewater. Scanning, 2013, 35(5): 283–291. DOI: 10.1002/sca.v35.5 |
| [30] | Perras AK, Wanner G, Klingl A, et al. Grappling archaea: ultrastructural analyses of an uncultivated, cold-loving archaeon, and its biofilm. Front Microbiol, 2014, 5: 397. |
| [31] | Volle CB, Ferguson MA, Aidala KE, et al. Spring constants and adhesive properties of native bacterial biofilm cells measured by atomic force microscopy. Colloids Surf B Biointerfaces, 2008, 67(1): 32–40. DOI: 10.1016/j.colsurfb.2008.07.021 |
| [32] | Volle CB, Ferguson MA, Aidala KE, et al. Quantitative changes in the elasticity and adhesive properties of Escherichia coli ZK1056 prey cells during predation by Bdellovibrio bacteriovorus 109J. Langmuir, 2008, 24(15): 8102–8110. DOI: 10.1021/la8009354 |
| [33] | Qin ZQ, Zhang JD, Hu YF, et al. Organic compounds inhibiting S. epidermidis adhesion and biofilm formation. Ultramicroscopy, 2009, 109(8): 881–888. DOI: 10.1016/j.ultramic.2009.03.040 |
| [34] | Abe Y, Polyakov P, Skali-Lami S, et al. Elasticity and physico-chemical properties during drinking water biofilm formation. Biofouling, 2011, 27(7): 739–750. DOI: 10.1080/08927014.2011.601300 |
| [35] | Techaruvichit P, Takahashi H, Kuda T, et al. Adaptation of Campylobacter jejuni to biocides used in the food industry affects biofilm structure, adhesion strength, and cross-resistance to clinical antimicrobial compounds. Biofouling, 2016, 32(7): 827–839. DOI: 10.1080/08927014.2016.1198476 |
| [36] | Peterson BW, He Y, Ren YJ, et al. Viscoelasticity of biofilms and their recalcitrance to mechanical and chemical challenges. FEMS Microbiol Rev, 2015, 39(2): 234–245. DOI: 10.1093/femsre/fuu008 |
| [37] | Wilking JN, Angelini TE, Seminara A, et al. Biofilms as complex fluids. MRS Bulletin, 2011, 36(5): 385–391. DOI: 10.1557/mrs.2011.71 |
| [38] | Pavlovsky L, Younger JG, Solomon MJ. In situ rheology of Staphylococcus epidermidis bacterial biofilms. Soft Matter, 2013, 9(1): 122–131. DOI: 10.1039/C2SM27005F |
| [39] | Waters MS, Kundu S, Lin NJ, et al. Microstructure and mechanical properties of in situ Streptococcus mutans biofilms. ACS Appl Mater Interfaces, 2014, 6(1): 327–332. DOI: 10.1021/am404344h |
| [40] | Stoodley P, Lewandowski Z, Boyle JD, et al. Structural deformation of bacterial biofilms caused by short-term fluctuations in fluid shear: an in situ investigation of biofilm rheology. Biotechnol Bioeng, 1999, 65(1): 83–92. DOI: 10.1002/(ISSN)1097-0290 |
| [41] | Mathias JD, Stoodley P. Applying the digital image correlation method to estimate the mechanical properties of bacterial biofilms subjected to a wall shear stress. Biofouling, 2009, 25(8): 695–703. DOI: 10.1080/08927010903104984 |
| [42] | Poppele EH, Hozalski RM. Micro-cantilever method for measuring the tensile strength of biofilms and microbial flocs. J Microbiol Methods, 2003, 55(3): 607–615. DOI: 10.1016/S0167-7012(03)00198-2 |
| [43] | Aggarwal S, Hozalski RM. Determination of biofilm mechanical properties from tensile tests performed using a micro-cantilever method. Biofouling, 2010, 26(4): 479–486. DOI: 10.1080/08927011003793080 |
| [44] | Möhle RB, Langemann T, Haesner M, et al. Structure and shear strength of microbial biofilms as determined with confocal laser scanning microscopy and fluid dynamic gauging using a novel rotating disc biofilm reactor. Biotechnol Bioeng, 2007, 98(4): 747–755. DOI: 10.1002/(ISSN)1097-0290 |
| [45] | Galy O, Latour-Lambert P, Zrelli K, et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophys J, 2012, 103(6): 1400–1408. DOI: 10.1016/j.bpj.2012.07.001 |
| [46] | Ahimou F, Semmens MJ, Novak PJ, et al. Biofilm cohesiveness measurement using a novel atomic force microscopy methodology. Appl Environ Microbiol, 2007, 73(9): 2897–2904. DOI: 10.1128/AEM.02388-06 |
| [47] | Safari A, Habimana O, Allen A, et al. The significance of calcium ions on Pseudomonas fluorescens biofilms——a structural and mechanical study. Biofouling, 2014, 30(7): 859–869. DOI: 10.1080/08927014.2014.938648 |
| [48] | Lau PCY, Dutcher JR, Beveridge TJ, et al. Absolute quantitation of bacterial biofilm adhesion and viscoelasticity by microbead force spectroscopy. Biophys J, 2009, 96(7): 2935–2948. DOI: 10.1016/j.bpj.2008.12.3943 |
| [49] | Shaw T, Winston M, Rupp CJ, et al. Commonality of elastic relaxation times in biofilms. Phys Rev Lett, 2004, 93(9): 098102. DOI: 10.1103/PhysRevLett.93.098102 |
| [50] | Lieleg O, Caldara M, Baumgäertel R, et al. Mechanical robustness of Pseudomonas aeruginosa biofilms. Soft Matter, 2011, 7(7): 3307–3314. DOI: 10.1039/c0sm01467b |
| [51] | Gan TS, Gong XJ, Schönherr H, et al. Microrheology of growing Escherichia coli biofilms investigated by using magnetic force modulation atomic force microscopy. Biointerphases, 2016, 11(4): 041005. DOI: 10.1116/1.4968809 |
 2017, Vol. 33
2017, Vol. 33