中国科学院微生物研究所、中国微生物学会主办
文章信息
- 齐兴柱, 汪军, 刘磊
- Qi Xingzhu, Wang Jun, Liu Lei
- Foc4 2个谷胱甘肽S-转移酶基因的克隆、鉴定及其在外源氧化胁迫下的表达
- Cloning and characterization of two glutathione S-transferases cDNAs in Foc4 and expression under exogenous oxidative stress
- 生物工程学报, 2017, 33(6): 995-1005
- Chinese Journal of Biotechnology, 2017, 33(6): 995-1005
- 10.13345/j.cjb.160474
-
文章历史
- Received: December 11, 2016
- Accepted: March 20, 2017
2 海南大学 海洋学院,海南 海口 570228;
3 中国热带农业科学院 环境与植物保护研究所,海南 海口 571101
2 College of Ocean, Hainan University, Haikou 570228, Hainan, China;
3 Institute of Environment and Plant Protection, Chinese Academy of Tropical Agricultural Sciences, Haikou 571101, Hainan, China
尖孢镰刀菌古巴专化型4号小种(Fusarium oxysporum f. sp. cubense race 4,Foc4) 是香蕉枯萎病的病原菌,自2001年首次在海南省三亚凤凰镇发现以来,对海南省乃至我国香蕉种植业造成了重大影响。谷胱甘肽S-转移酶(Glutathione S-transferases,GSTs;EC 2.5.1.18) 是一种多功能酶,属于一个超级蛋白家族,广泛存在于动物、植物、真菌和细菌细胞中[1]。典型的GSTs是由两个亚基组成的可溶性蛋白,通过催化毒素与还原性谷胱甘肽(Glutathione,GSH)结合,或者被动地与各种内源性或外源性异型生物质,包括致癌物、治疗药物、氧化应激产物(如活性氧等)结合,参与细胞中的代谢、诱导胁迫、信号转导等过程,然后通过转运蛋白将连有谷胱甘肽的代谢物质转运入液泡,从而达到解毒和保护细胞免受多种外界环境因素的毒害[2-8]。
近年来,许多研究证据表明,当动植物受病菌侵染或寄生虫侵害时,往往会引起寄主细胞内活性氧的升高,而GSTs能够调控细胞内氧化还原水平,从而协助保护生物体免受损害[9-12]。由于GSTs对处于逆境胁迫环境中的细胞具有重要的解毒作用,人们对各种动植物的GSTs基因进行了广泛研究。目前许多参与植物抗逆/抗病响应的GSTs被分离鉴定,比如水稻的OsGST2显著响应稻瘟病菌Magnaporthe grisea的诱导[13];在向日葵中,干旱、盐、草酸、核盘菌及其代谢物均能诱导向日葵HaGSTU1基因的表达量出现显著变化[14];在番茄中,多个GSTs基因家族成员响应SA和盐胁迫[15]等。在动物中,也有报道西氏贝蛔虫Baylisascaris schroederi在侵入大熊猫肠道过程中,GSTs参与清除宿主免疫系统触发的活性氧物质[16];在白菜黑斑病菌Alternaria brassicicola中,异硫氰酸盐(ITC)诱导的GSTs活性分析表明,在白菜黑斑病菌清除寄主有毒代谢物及活性氧物质并成功入侵寄主的过程中,5个分别属于GSTFuA、GTT1、Ure2pB、GSTO和MAPEG亚家族的GSTs都发挥了必不可少的作用,比如属于GSTFuA亚家族的GSTs表现出了GSH转移酶活性,而属于GTT1亚家族的GSTs则表现出了过氧化物酶的活性[17]。
前期的研究表明,Foc4在侵染香蕉苗时,在入侵位点会遭遇寄主细胞的活性氧迸发现象[18],同时,本课题组通过对转录组测序数据分析发现,Foc4中GSTs基因多达27个,同时也发现,Foc4在有香蕉苗诱导时,细胞内多个GSTs的表达上调。而目前国内外文献均未见有关Foc4 GSTs功能研究的报道。为了进一步探究GSTs在Foc4入侵香蕉苗的过程中是否参与解除寄主细胞产生的强氧化胁迫环境,本研究克隆了Foc4中2个表达上调最显著的GSTs基因完整的cDNA编码区序列并利用生物信息学方法对其进行了归类和命名,分析了2个GSTs基因在外源氧化胁迫条件下的表达特性,为探究它们在Foc4入侵香蕉苗过程中的功能提供数据。
1 材料与方法 1.1 试验用菌株及培养条件Foc4的野生型B2菌株从海南省乐东县发病(香蕉枯萎病)的田间巴西蕉根部采集,由本实验室分离鉴定并保存。真菌菌株的培养采用葡萄糖-马铃薯培养基(固体PDA、液体PDB)。为了提取H2O2处理不同时间的菌体总RNA,将在PDB液体培养基中培养6 d的Foc4 B2菌株在超净工作台中用6层擦镜纸过滤收集滤液(即孢子液),然后按1:50的比例将孢子液接种到新的PDB培养基中,28 ℃、120 r/min振荡培养12 h,然后加入H2O2至终浓度10 μmol/mL,继续振荡培养,并分别于1、5、12和24 h收集菌体,液氮研磨,提取总RNA。克隆用大肠杆菌菌株DH5α和原核表达用菌株BL21(DE3) 均购自北京天根生化科技有限公司。
1.2 试剂和引物植物总RNA提取试剂盒,PCR Mix反应混合物均购自北京天根生化科技有限公司。反转录试剂盒采用TaKaRa公司的RNA LA PCRTM Kit。荧光定量PCR试剂盒采用2×one-step SYBR-Real-time RT-PCR Kit (北京百泰克公司产品)。PCR引物(表 1)由北京三博远志生物技术有限公司合成。GSTs酶活性检测采用北京EBT公司的谷胱甘肽S-转移酶活性检测试剂盒。
| Primer name | Primer sequence (5'-3') | Temperature (℃) | Amplified length (bp) |
| 5'-Gst1cds | ATGTCCTCAGACTCAGTCGTTTA | 55.9 | 606 |
| 3'-Gst1cds | CTCGGATTTGATGTACTCAGC | 54.9 | |
| 5'-Gst2cds | GACTTATTCGCTCTTCATCGG | 56.9 | 689 |
| 3'-Gst2cds | ACTCCTTGGTCAAGTCCTTGAG | 57.6 | |
| 5'-Gst1 | CCCAATCCTTCGTTTCCTC | 56.8 | 255 |
| 3'-Gst1 | ATCGCAAAGTCGGTGTAGC | 56.0 | |
| 5'-Gst2 | TACATCGCTGAGCAATACCC | 56.1 | 396 |
| 3'-Gst2 | CGTCTCCTTGATACCGTCCT | 56.5 | |
| 5'-Actin | CCGTGACATCAAGGAGAAGC | 57.8 | 431 |
| 3'-Actin | GGAAAGTGGACAGGGAAGCA | 59.8 | |
| 5'-Gst1exp | TACGGATCCATGTCCTCAGACTCAGTC | 66.9 | 625 |
| 3'-Gst1exp | CACGTCGACACTCGGATTTGATGTACT | 67.7 | |
| 5' Gst2exp | CGTGGATCCATGACTTATTCGCTCTTC | 68.4 | 709 |
| 3'-Gst2exp | TCAGTCGACACTCCTTGGTCAAGTCCT | 68.6 |
利用H2O2处理后的野生型B2菌株转录组信息获得了2个表达上调明显的GSTs基因的cDNA信息,其编码的假想蛋白GenBank登录号分别为EMT69530.1和EMT62696.1。利用其cDNA序列信息在其起始密码子和终止密码子附近设计引物(表 1),并以10 μmol/mL H2O2诱导5 h的总RNA反转录的cDNA为模板,采用普通PCR方法扩增、克隆后获得了Foc4的2个GSTs基因编码区序列。分别将2个GSTs基因编码区序列预测的蛋白质序列在NCBI中进行Blast,获得其他相关物种的GSTs基因的蛋白序列,参考Morel和Edgar等[19]以及Calmes等[17]的文献也获得一部分相关物种的GSTs基因的蛋白序列。
所应用的生物信息学分析软件包括:NCBI blast在线软件http://blast.ncbi.nlm.nih.gov/Blast.cgi;NCBI结构域预测在线软件CDD http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.Cgi。ClustalX软件对Foc4的2个GSTs蛋白序列及所获得的同源蛋白序列进行多重序列比对,以鉴定它们之间的同源性;MEGA7.0软件[20]对所获得的序列构建了邻位相接的系统发育树,并用Bootstrap方法(1 000 replicates;seed=64 238) 对该树进行分析。
1.4 RT-PCR分析基因表达按北京天根公司的植物总RNA提取试剂盒说明书提取处理后菌株的总RNA,然后利用2×one-step SYBR-Real-time RT-PCR Kit (Bioteke Corp.产品),以3 μg总RNA作为反转录模板,RT-PCR引物为:5'-Gst1、3'-Gst1,5'-Gst2、3'-Gst2、5'-Actin、3-Actin (表 1)。PCR反应体系的总体积为25 μL,其中含模板(总RNA) 1.5 μL,2×one-step SYBR mix 12.5 μL,引物1.3 μL (0.2 μmol/L),RT-PCR MIX 1.25 μL,RNase-free H2O 7.15 μL。PCR反应在BIO-RAD公司的Mini OpticonTM Rreal-Time PCR System上进行。PCR热循环条件如下:94 ℃变性30 s;退火温度设梯度温度48-50 ℃,并退火15 s;72 ℃延伸15 s;然后读取荧光值,重复40个循环;最后从45 ℃到90 ℃,每隔0.2 ℃读取溶解曲线荧光值,每读数一次,停留2 s。运行完毕后,从Opticon Monitor 3.1软件读取C(t)值,以β-actin基因作为内参对照,利用2-ΔC(t)法计算各基因的相对表达水平。
1.5 Fogst1和Fogst2基因的原核表达以含有Fogst1和Fogst2基因编码序列的T载体为模板,含有BamHⅠ和SalⅠ的引物5'-Gst1exp、3'-Gst1exp、5'-Gst2exp、3'-Gst2exp进行PCR扩增并克隆到PM18-T载体,再将质粒pMT18-T-Fogst1和pMT18-T-Fogst2用BamHⅠ和SalⅠ双酶切,回收目的片段连接至用同样酶切的表达载体pET28a,得到重组表达质粒pET28a-Fogst1和pET28a-Fogst2,转入大肠杆菌表达菌株BL21(DE3) 中。菌株BL21(DE3) pET28a-Fogst1和菌株BL21(DE3) pET28a-Fogst2在LB培养基中于37 ℃培养过夜,再以1:100的比例转接到新鲜LB培养基中,在37 ℃培养至A600为0.4-0.6,加入IPTG至终浓度分别为0、0.2、1和2 mmol/L,37 ℃诱导目的蛋白表达约3.5 h。取10 mL菌液离心收集菌体,去上清后加1 mL PBS缓冲液,菌体重新悬浮后超声破碎菌体并离心,取16 μL上清及4μL 5×蛋白上样缓冲液混匀变性后进行SDS-PAGE,考马斯亮蓝染色后观察结果。同时以转入空白质粒的大肠杆菌BL21(DE3) pET28a作对照。
1.6 Fogst1和Fogst2的原核表达产物酶活性分析测定原理为GSTs催化GSH与CDNB结合,其结合产物的吸收峰波长为340 nm;通过测定340 nm波长处吸光度上升速率计算GSTs的活性。将H2O2处理好的菌丝进行冰浴超声波破碎后,经离心,取上清作为待测的粗酶液置于冰上。测定方法如下:空白管:取1支石英比色皿,加入100 μL试剂一,900 μL试剂二和100 μL试剂三,迅速混匀后于340 nm测定吸光度变化,记录10 s和310 s吸光度为A1和A2;测定管:取1支石英比色皿,加入100 μL待测粗酶液,900 μL试剂二和100 μL试剂三,迅速混匀后于340 nm测定吸光度变化,记录10 s和310 s吸光度为A3和A4。活性单位定义为在常温下,每毫升液体每分钟催化1 nmol/L CDNB与GSH结合为1个酶活单位。GSTs酶活性计算公式如下:

|
其中,ε:产物摩尔消光系数,9.6×103L/(mol·cm);d:比色皿光径,1 cm;106:1 mol=1×106μmol;V反总:反应体系总体积,1 100 μL=0.001 1 L;V样:加入反应体系中粗酶液体积,100 μL=0.1 mL;t:反应时间,5 min。
2 结果与分析 2.1 Foc4 2个GSTs酶基因的克隆鉴定前期的研究工作中发现,Foc4在入侵位点会遭遇寄主细胞因活性氧迸发而产生的强氧化胁迫环境。为探讨Foc4遭遇外源氧化胁迫时的分子应对机制,采用H2O2模拟外源强氧化胁迫环境,处理Foc4野生型B2菌株,并对其进行转录组测序,结果发现2个编号分别为c6215.graph_c0和c9910.graph_c0的基因相对于未用H2O2处理Foc4野生型B2菌株的表达显著上调。利用其cDNA序列信息在其起始密码子和终止密码子附近设计引物(表 1),采用TaKaRa公司的反转录试剂盒(RNA LA PCRTM Kit)对Foc4野生型B2菌株的总RNA进行反转录后PCR扩增,获得了c6215.graph_c0和c9910.graph_c0的cDNA序列,其cDNA编码序列电泳结果如图 1所示。将PCR扩增获得的2个cDNA片段克隆后送深圳华大基因研究院测序,测序结果与现有的c6215.graph_c0和c9910.graph_c0的cDNA序列比对表明成功获得了c6215.graph_c0和c9910.graph_c0的cDNA的编码序列。将2个基因编码区预测的蛋白质序列输入到NCBI blast在线软件,进行blast查询比对和结构域查询,初步确定2个基因均为谷胱甘肽S转移酶基因。因此,分别将c6215.graph_c0命名为Fogst1,c9910.graph_c0命名为Fogst2。

|
| 图 1 两个谷胱甘肽S-转移酶基因Fogst1和Fogst2 cDNA编码序列的PCR扩增 Figure 1 PCR amplification of cDNA coding sequence of two glutathione S-transferases (Fogst1 and Fogst2). |
| |
通过Blast在线查询和参考Mélanie Morel[19]和Calmes Benoit[17]的文献获得部分真菌的GSTs基因的蛋白序列。利用ClustalX软件对Foc4野生型B2菌株的2个GSTs酶蛋白序列及同源蛋白序列进行多重序列比对,以鉴定它们之间的同源性,并利用MEGA7.0软件[21]对所获得的多重序列比对结果构建了邻位相接的系统发育树(NJ-tree),如图 2所示。系统发育分析结果表明:Fogst1被归类为σ型(Sigma-class) GSTs酶,Fogst2被归类为目前功能未知而且尚未命名的一类亚型(GST_N family unknown subfamily)。

|
| 图 2 Foc4 2个谷胱甘肽S-转移酶蛋白及其同源蛋白的分子进化树(NJ),Bootstrap (1 000 replicates; seed=64 238) (2个谷胱甘肽S-转移酶Fogst1和Fogst2分别用●标记。系统分析所用的同源蛋白序列均来自NCBI) Figure 2 The Phylogenetic tree (NJ) of the two GSTs of Foc4 and their homologous, Bootstrap (1 000 replicates; seed=64 238). The two GSTs (Fogst1 and Fogst2) were marked by ● respectively. The homologous protein sequences used in the phylogenetic analysis were derived from NCBI. Accession number of each sequence is as follows: Saccharomyces cerevisiae (NP_014170, NP_012304, NP_013977, NP_013002); Kluyveromyces lactis (XP_453933, XP_451011, XP_455627); Eremothecium gossypii (NP_982752); Schizosaccharomyces pombe (NP_588298, NP_588517, NP_588171); Neurospora crassa (XP_960040, XP_963068, XP_011394507, XP_956141); Chaetomium thermophilum var. (XP_006696933); Stachybotrys chartarum IBT (KEY65772); Metarhizium robertsii (XP_007819855); Marssonina brunnea f. sp. (XP_007288281); Arabidopsis thaliana (NP_199312, NP_001031671, NP_199315); Magnaporthe oryzae (XP_003714355); Talaromyces marneffei (XP_002143473); Aspergillus parasiticus (KJK65656); Talaromyces verruculosus (KUL91727); Aspergillus kawachii (GAA85136); Pseudogymnoascus sp. (KFX90885); Pochonia chlamydosporia (OAQ62447, OAQ61443); Fusarium oxysporum f. sp. pisi (EXA32327); Microdochium bolleyi (KXJ85708); Fusarium avenaceum (KIL87172); Nectria haematococca (XP_003048601); Fusarium sp. FIESC (CEG04586); Metarhizium brunneum (XP_014539941); Acremonium chrysogenum (KFH42026); Purpureocillium lilacinum (OAQ83436); Beauveria bassiana (KGQ09649, XP_008596612); Cordyceps confragosa (OAA70173); Colletotrichum orbiculare (ENH82170). |
| |
为了验证Fogst1和Fogst2基因编码蛋白的功能,构建了Fogst1和Fogst2的原核表达载体。将Fogst1和Fogst2完整的编码区用BamHⅠ和SalⅠ分别从pMT18-T-Fogst1和pMT18-T-Fogst2载体切下,连入经同样酶切的表达载体pET28a,分别得到pET28a-Fogst1和pET28a-Fogst2。分别将pET28a-Fogst1和pET28a-Fogst2转入大肠杆菌表达菌株BL21(DE3),不同浓度IPTG诱导3.5 h收集菌体,同时以转入pET28a空质粒的菌体作对照。SDS-PAGE检测显示,图 3A的1、2、3和4号泳道分别为2、1、0.2和0 mmol/LIPTG诱导后的BL21(DE3) pET28a-Fogst1总蛋白电泳图,5号泳道为BL21(DE3) pET28a的总蛋白电泳图;图 3B中,1、2、3、5号泳道分别为2、1、0.2和0 mmol/L IPTG诱导后的BL21(DE3) pET28a-Fogst2总蛋白电泳图,4号泳道为BL21(DE3) pET28a的总蛋白电泳图。从图 3可以看出,经IPTG诱导的BL21(DE3) pET28a-Fogst1菌株在分子量约为28 kDa处有明显的蛋白表达条带,与预期的Fogst1分子量基本一致,而且在0.2、1和2 mmol/L IPTG诱导下均有大量表达(图 3A)。经IPTG诱导的BL21(DE3) pET28a-Fogst2菌株在分子量约为30 kDa处有明显的蛋白表达条带,与预期的Fogst2分子量基本一致,而且也是在0.2、1和2 mmol/L的IPTG诱导下均有较大量的表达。菌体超声破碎离心后,仅以上清液点样电泳,即可见清晰表达条带,说明2个重组蛋白主要以可溶形式存在于细胞中。GST可以催化CDNB与GSH反应,其产物在340 nm处有特征吸收,可用分光光度法检测。基于该原理,以北京EBT公司的谷胱甘肽S转移酶活性检测试剂盒分别对经2 mmol/L IPTG诱导的BL21(DE3) pET28a-Fogst1和BL21(DE3) pET28a-Fogst2菌体破碎后的上清液进行了GSTs活性检测。结果2种菌体上清中,均检测到了明显的GSTs活性(表 2),证明Fogst1和Fogst2编码的蛋白质属于Foc4的GSTs家族。
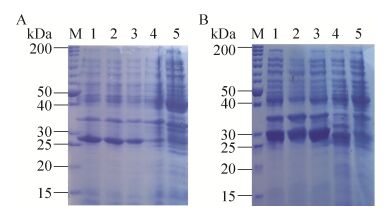
|
| 图 3 SDS-PAGE分析重组Fogst1 (A)和Fogst2 (B)蛋白在大肠杆菌BL21(DE3) 中的表达 Figure 3 SDS-PAGE analysis of the expression of recombinant protein Fogst1 (A) and Fogst2 (B) in Escherichia coli (BL21(DE3)). |
| |
| Sample | Activity of GSTs (nmol/(min·mL)) |
| Supernatant of BL21(DE3) pET28a | 0.69 |
| Supernatant of BL21(DE3) pET28a-Fogst1 | 10.35 |
| Supernatant of BL21(DE3) pET28a-Fogst2 | 13.57 |
前期工作中的外源氧化胁迫条件细下的转录组测序表明,Fogst1、Fogst2两个基因转录本相对于未处理样品大幅增加,因此我们推测这两个GSTs基因在此过程中必定发挥了作用,为了进一步验证转录组测序数据中关于这两个GSTs基因的数据是否准确,利用终浓度为10 μmol/mL的外源H2O2模拟这种强氧化胁迫环境,采用半定量RT-PCR技术分析了两个GSTs基因在H2O2处理1、5、12、24 h的相对表达量(图 4)。结果表明:1) 无论是Fogst1还是Fogst2,在前5 h的表达均远高于12 h和24 h的表达,说明在外源H2O2存在的情况下,为参与清除外源H2O2,Fogst1和Fogst2基因表达均迅速上调;2) 在12-24 h的时间段,表达量已经逐渐恢复到对照样品的水平,说明培养液中外源H2O2因病原菌的清除及自身的降解已经差不多消失完全,对Foc4的菌丝生长的影响已经很轻微了。
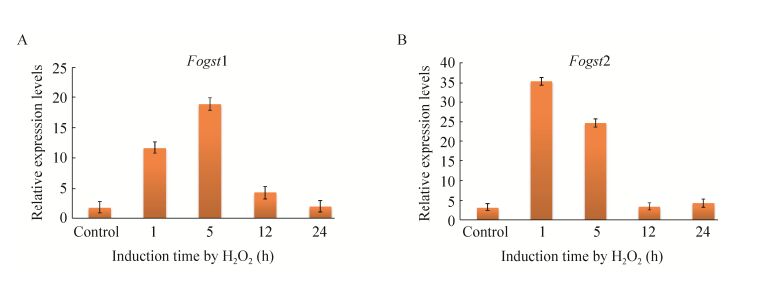
|
| 图 4 Foc4的2个谷胱甘肽S-转移酶基因Fogst1和Fogst2在外源H2O2诱导条件下的相对表达状况 Figure 4 The relative expression levels of two GSTs (Fogst1 and Fogst2) of Foc4 under exogenous oxidative stress. |
| |
植物对病原菌入侵的最快防卫反应之一就是活性氧的急促释放,称为氧化爆发(Oxidative burst),这种现象在植物抵抗病原菌入侵过程中具有重要作用。前期的研究工作中我们发现Foc4在入侵香蕉苗后引起了香蕉苗根部活性氧迸发[14]。为了应对植物寄主所产生的强氧化胁迫环境并成功入侵寄主,病原菌必须通过自身基因表达调控,细胞代谢改变等一系列变化来应对这种强氧化胁迫环境。为此,以H2O2模拟外源强氧化胁迫环境处理Foc4野生型B2菌株并对其进行转录组测序,测序结果发现Fogst1和Fogst2表达显著上调。因此对这2个GSTs基因进行了克隆鉴定和表达分析验证。
GSTs同工酶是一个超级蛋白家族,在其他物种中的研究也相对较多,对GSTs酶的分类也比较成熟,但真菌中GSTs酶的研究相对较晚。虽然在许多真菌中都有发现GSTs基因,但对真菌中GST的分类研究相对还很少,根据等电点的不同、底物特异性、对抑制剂的敏感性、免疫学特性和氨基酸序列相似性的不同,GSTs被分为许多亚型。如哺乳动物中可溶性GSTs有8种,分别为α、μ、π、σ、θ、ζ、κ和ω型[17],植物中的可溶性GSTs有θ、ζ、φ、τ、λ型5种,昆虫中的GST被划分为3类,分别为σ、δ和ε型[22-23]。在真菌中GST的研究还相对较少,且已经鉴定的真菌GSTs有些与已有的亚型相同,有些与已有的亚型不相同。比如,Morel和Ngadin等[24]利用已有的基因组数据,通过同源比对及进化分析等方法将19种子囊菌、8种担子菌以及2种接合菌的GSTs超家族蛋白分为Omega、GTT1、GTT2、Ure2p、MAK16、EFBγ和GTE七大类,其中粟酒裂殖酵母Schizosaccharomyces pombe中仅发现7个GSTs,而在褐腐菌Postia placenta中发现46个分属于所有7类。而Morel和Edgar等[19]又认为,真菌胞浆GSTs酶有8大类,分别为GTT 1、GTT 2、URE 2p、MAK 16、EFb1、GSTFuA、GSTO和GHR。传统上,两个蛋白质的氨基酸序列一致性超过40%即认为它们属于同一种类型的蛋白质,当同工酶的氨基酸序列一致性低于20%即认为它们不属于同一种类型的蛋白质[25]。然而,如果以这些基本序列作为唯一的标准,仍有许多“非规范的”GSTs酶在多种真菌中被发现,因而增加了GSTs酶分类的复杂性。比如,有几个通常被归为GSTs酶(EF1Bg,MAK16) 的蛋白家族,是基于其在结构上与GSTs酶的相似性,而不是以具有谷胱甘肽依赖的活性而被归类为GSTs酶。而我们根据氨基酸基本序列采用同源比对及进化分析鉴定的Foc4的2个GSTs酶中,Fogst1属于σ亚型,Fogst2不属于以上分类中的任何一种亚型。但根据2个GSTs基因原核表达产物的活性检测表明Fogst1和Fogst2均具有GSTs酶活性说明两者均为Foc4的GSTs酶。同时也说明真菌GSTs酶亚型分类的复杂性,目前的真菌GSTs酶亚型分类并不完善。
为了解Fogst1和Fogst2是否参与Foc4与香蕉苗相互作用的过程,本研究运用RT-PCR技术分析了利用外源H2O2模拟香蕉苗根部所处的活性氧迸发环境来诱导Fogst1和Fogst2基因的表达情况,结果表明Fogst1和Fogst2可能参与了Foc4清除外源H2O2的过程。
4 结论根据以上研究结果,得出如下结论:1) 从Foc4中克隆得到2个GSTs基因Fogst1和Fogst2, 说明2个基因在Foc4中真实存在,其中,Fogst1的编码序列长609 bp,编码202个氨基酸残基,Fogst2的编码序列长693 bp,编码230个氨基酸残基。2) Fogst1属于GSTs酶超蛋白质家族中的σ亚型。3) Fogst1和Fogst2原核表达及活性检测表明两者均具有GSTs酶活性,说明两者均为Foc4中的GSTs酶。4) 在外源氧化胁迫存在的条件下,Fogst1和Fogst2的表达均有上调,进一步推测Fogst1和Fogst2均可能参与了消除Foc4入侵香蕉苗时香蕉苗根部因活性氧迸发产生的强氧化胁迫环境。
| [1] | Ren HL, Xu DD, Gopalakrishnan S, et al. Gene cloning of a sigma class glutathione S-transferase from abalone (Haliotis diversicolor) and expression analysis upon bacterial challenge. Dev Comp Immunol, 2009, 33(9): 980–990. DOI: 10.1016/j.dci.2009.04.003 |
| [2] | Conn S, Curtin C, Bézier A, et al. Purification, molecular cloning, and characterization of glutathione S-transferases (GSTs) from pigmented Vitis vinifera L. cell suspension cultures as putative anthocyanin transport proteins.. J Exp Bot, 2008, 59(13): 3621–3634. DOI: 10.1093/jxb/ern217 |
| [3] | Olsvik PA, Nordtug T, Altin D, et al. Transcriptional effects on glutathione S-transferases in first feeding Atlantic cod (Gadus morhua) larvae exposed to crude oil. Chemosphere, 2010, 79(9): 905–913. DOI: 10.1016/j.chemosphere.2010.03.026 |
| [4] | Salinas AE, Wong MG. Glutathione S-transferases -a review. Curr Med Chem, 1999, 6(4): 279–309. |
| [5] | Torres-Rivera A, Landa A. Glutathione transferases from parasites: a biochemical view. Acta Trop, 2008, 105(2): 99–112. DOI: 10.1016/j.actatropica.2007.08.005 |
| [6] | Ding YC, Hawkes N, Meredith J, et al. Characterization of the promoters of Epsilon glutathione transferases in the mosquito Anopheles gambiae and their response to oxidative stress. Biochem J, 2005, 387(3): 879–888. DOI: 10.1042/BJ20041850 |
| [7] | Jowsey IR, Thomson AM, Flanagan JU, et al. Mammalian class Sigma glutathione S-transferases: catalytic properties and tissue-specific expression of human and rat GSH-dependent prostaglandin D-2 synthases. Biochem J, 2001, 359(3): 507–516. DOI: 10.1042/bj3590507 |
| [8] | Cho SG, Lee YH, Park HS, et al. Glutathione S-transferase mu modulates the stress-activated signals by suppressing apoptosis signal-regulating kinase 1. J Biol Chem, 2001, 276(16): 12749–12755. DOI: 10.1074/jbc.M005561200 |
| [9] | Guo M, Chen Y, Du Y, et al. The bZIP Transcription Factor MoAP1 Mediates the Oxidative Stress Response and Is Critical for Pathogenicity of the Rice Blast Fungus Magnaporthe oryzae. PLoS Pathog, 2011, 7(2): e1001302. DOI: 10.1371/journal.ppat.1001302 |
| [10] | Guo M, Guo W, Chen Y, et al. The basic leucine zipper transcription factor Moatf1 mediates oxidative stress responses and is necessary for full virulence of the rice blast fungus Magnaporthe oryzae. Mol Plant Microbe Interact, 2010, 23(8): 1053–1068. DOI: 10.1094/MPMI-23-8-1053 |
| [11] | Hayes JD, Pulford DJ. The glutathione S-transferase supergene family: regulation of GST and the contribution of the isoenzymes to cancer chemoprotection and drug resistance. Crit Rev Biochem Mol Biol, 1995, 30(6): 445–600. DOI: 10.3109/10409239509083491 |
| [12] | Fujita M, Hossain MZ. Molecular cloning of cDNAs for three tau-type glutathione S-transferases in pumpkin (Cucurbita maxima) and their expression properties. Physiol Plant, 2003, 117(1): 85–92. DOI: 10.1034/j.1399-3054.2003.1170111.x |
| [13] | Agrawal GK, Jwa NS, Rakwal R. A pathogen-induced novel rice (Oryza sativa L.) gene encodes a putative protein homologous to type Ⅱ glutathione S-transferases. Plant Sci, 2002, 163(6): 1153–1160. DOI: 10.1016/S0168-9452(02)00331-X |
| [14] |
Ma LG, Meng QL, Zhang YH, et al. Clone and function of a glutathione-S-transferase gene from sunflower (Helianthus annuus).
Chin J Oil Crop Sci, 2015, 37(5): 635–643.
(in Chinese). 马立功, 孟庆林, 张匀华, 等. 向日葵谷胱甘肽-S-转移酶基因的克隆及抗病功能研究. 油料作物学报, 2015, 37(5): 635-643. DOI:10.7505/j.issn.1007-9084.2015.05.007 |
| [15] | Csiszár J, Horváth E, Váry Z, et al. Glutathione transferase supergene family in tomato: salt stress-regulated expression of representative genes from distinct GST classes in plants primed with salicylic acid. Plant Physiol Biochem, 2014, 78: 15–26. DOI: 10.1016/j.plaphy.2014.02.010 |
| [16] | Xie Y, Zhou X, Chen L, et al. Cloning and characterization of a novel sigma-like glutathione S-transferase from the giant panda parasitic nematode, Baylisascaris schroederi. Parasit Vectors, 2015, 8: 44. DOI: 10.1186/s13071-014-0629-9 |
| [17] | Calmes B, Morel-Rouhier M, Bataillé-Simoneau N, et al. Characterization of glutathione transferases involved in the pathogenicity of Alternaria brassicicola. BMC Microbiol, 2015, 15: 123. DOI: 10.1186/s12866-015-0462-0 |
| [18] | Qi XZ, Guo LJ, Yang LY, et al. Foatf1, a bZIP transcription factor of Fusarium oxysporum f. sp. cubense, is involved in pathogenesis by regulating the oxidative stress responses of Cavendish banana (Musa spp.). Physiol Mol Plant Pathol, 2013, 84: 76–85. DOI: 10.1016/j.pmpp.2013.07.007 |
| [19] | Morel M, Meux E, Mathieu Y, et al. Xenomic networks variability and adaptation traits in wood decaying fungi. Microb Biotechnol, 2013, 6(3): 248–263. DOI: 10.1111/1751-7915.12015 |
| [20] | Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol, 1987, 4(4): 406–425. |
| [21] | Sheehan D, Meade G, Foley VM, et al. Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily. Biochem J, 2001, 360(1): 1–16. DOI: 10.1042/bj3600001 |
| [22] | Edwards R, Dixon DP. Plant glutathione transferases. Methods Enzymol, 2005, 401: 169–186. DOI: 10.1016/S0076-6879(05)01011-6 |
| [23] | Agianian B, Tucker PA, Schouten A, et al. Structure of a Drosophila sigma class glutathione S-transferase reveals a novel active site topography suited for lipid peroxidation products. J Mol Biol, 2003, 326(1): 151–165. DOI: 10.1016/S0022-2836(02)01327-X |
| [24] | Morel M, Ngadin AA, Droux M, et al. The fungal glutathione S-transferase system. Evidence of new classes in the wood-degrading basidiomycete Phanerochaete chrysosporium. Cell Mol Life Sci, 2009, 66(23): 3711–3725. DOI: 10.1007/s00018-009-0104-5 |
| [25] | Hayes JD, Flanagan JU, Jowsey IR. Glutathione transferases. Annu Rev Pharmacol Toxicol, 2005, 45(1): 51–88. DOI: 10.1146/annurev.pharmtox.45.120403.095857 |
 2017, Vol. 33
2017, Vol. 33




