中国科学院微生物研究所、中国微生物学会主办
文章信息
- 唐鸿志, 王伟伟, 张莉鸽, 黄玲, 陆歆毓, 许平
- Tang Hongzhi, Wang Weiwei, Zhang Lige, Huang Ling, Lu Xinyu, Xu Ping
- 合成生物学在环境修复中的应用
- Application of synthetic biology in environmental remediation
- 生物工程学报, 2017, 33(3): 506-515
- Chin J Biotech, 2017, 33(3): 506-515
- 10.13345/j.cjb.160417
-
文章历史
- Received: November 1, 2016
- Accepted: December 15, 2016
环境保护与污染防治,是实现国家经济和环境可持续发展战略的重要前提。环境废弃物中所含的难降解有机污染物严重污染大气、水和土壤。目前,在环境各介质中检测到抗生素、内分泌干扰物和阻燃剂等新兴难降解有机污染物,这些污染物对生物和人体具有潜在的危害,并且难以降解。高效降解环境中有害污染物的微生物制品研究,能够为环境保护与污染防治提供技术支撑。利用我国丰富的微生物与基因资源,运用合成生物学的手段,有目的地重新设计和改造现有降解菌株,构建新的能够降解某一特定污染物或同时降解多个污染物的菌株。针对复合型污染物,如污染水体,进行微生物系统的理性设计和组装,合理构建具有抗逆特性的针对环境难降解有机污染物的高效降解菌群,为环境修复提供理论与技术保障。
1 难降解有机污染物现状难降解有机污染物是指在自然环境条件下持久稳定存在的、几乎不能被生物降解的一类有害物质,其中多数为人工合成的化学物质。难降解有机污染物具有持久性、生物富集性、长距离迁移能力及生物有害性等特点,其中新兴污染物是近年来才被意识到或者尚未意识到其危害的物质。新兴污染物有可能是新物质,也可能是近年来才发现其危害或具有潜在危害的物质,其数目种类繁多,性质多样,几乎无处不在。常见的分类包括:药品 (主要以抗生素为主)、洗护用品、增塑剂、阻燃剂以及杀虫剂等等。
1.1 抗生素污染抗生素应用广泛,是一类常见的新兴污染物。近些年,不断有研究报道抗生素在环境中的残留及转归现状,抗生素的污染及其产生的一系列生态效应和对人群健康的影响开始引起人们的广泛关注。医用抗生素和兽用抗生素的滥用情况日益加重,抗生素被机体摄入吸收后,绝大部分以原形通过粪便和尿液排出体外,对土壤和水体等造成污染。研究表明,在我国广东北江一带,大环内酯类抗生素污染最为严重,其含量范围为11.7−114.6 ng/L,表层水中磺胺甲恶唑含量达14.7 ng/L,阿奇霉素达到25.0 ng/L,在沉积物中氧氟沙星达到5.4 ng/g,四环素的含量也达到3.3 ng/g[1]。这一数据显示,抗生素的污染已经成为环境中比较严重的污染问题。以四环素类抗生素为例,它是新兴污染物Pharmaceutical and personal care products (PPCPs) 中的一种,在畜禽以及水产养殖中应用十分广泛。在自然条件下,四环素类抗生素的生态毒性较强,土壤和水中残留的四环素类抗生素和其代谢产物会逐渐破坏土壤以及水的生态系统,影响动物、植物和微生物的生长活动。多项研究表明,四环素类抗生素可能通过抑制叶绿体合成酶的活性进而对植物生长产生抑制作用,影响植物发芽率和根的生长,同时其对藻类抑制作用尤为明显,进而破坏完整的生物链[2]。张树清等对我国规模化养殖场畜禽粪便的主要成分进行抽样分析,结果表明,在畜禽粪便中可以检测到高含量的四环素类抗生素残留,猪粪中土霉素平均质量浓度为9.09 mg/kg,最高可达134.75 mg/kg;四环素平均质量浓度为5.22 mg/kg,最高可达78.57 mg/kg;金霉素平均质量浓度为3.57 mg/kg,最高可达121.78 mg/kg[3]。Hamscher等也在畜禽粪便中检测到高含量的四环素和金霉素,质量浓度分别为4.0、0.1 mg/kg,土壤环境中四环素和金霉素的残留质量浓度分别达到86.2−198.7、4.6−7.3 μg/kg[4]。Aust等研究者也发现加拿大土壤环境中四环素的质量浓度可达到52 μg/kg[5]。综上,四环素类抗生素在环境中的残留十分严重,对环境造成了潜在威胁。目前抗生素的种类多达几千种,常用的有数百种之多。与此同时,新型抗生素层出不穷,不断推陈出新,对治理抗生素污染提出了更高的要求。
1.2 内分泌干扰物污染内分泌干扰物是一类典型的新兴污染物,又称环境激素。欧盟将其定义为一类能够对正常生物体或其后代内分泌功能产生不利影响的外源物质[6]。这类污染物主要是外源性干扰内分泌系统,通过直接摄入、间接积累等途径,虽不直接作为有毒物质给人体和动物造成危害,但是在这类污染物质长期刺激作用下,会对动物和人体造成严重的生殖能力下降、幼体死亡、行为异常、生殖器障碍、甚至种类灭绝等危害[7-8]。内分泌干扰物种类广泛,包括70%−80%的农药 (滴滴涕及其分解产物、六氯苯、六六六、艾氏剂、狄氏剂)、阻燃剂 (六溴环十二烷、多溴联苯醚等)、类固醇雌激素 (17α-乙炔基雌二醇、17β-雌二醇、己烷雌酚等) 以及一些有机化合物 (烷基酚 (AP)、烷基酚聚氧乙烯醚 (APE)、双酚A、邻苯二甲酸酯 (PAE)、多氯联苯类 (PCB) 等)[7]。据报道,有机氯农药在禁用20年后,六六六 (HCHs)、硫丹、七氯、狄氏剂在鱼肉中均有不同程度的检出,其中六六六的残留水平最高达到了0.33 mg/kg[9]。类固醇类雌激素在环境介质中被广泛检出,鸡蛋中检出雌二醇、雌酮、雌三醇、睾酮[10];动物组织中检出己烯雌酚浓度达到0.2 ng/g[11];尿液中检出4-壬基酚、双酚、雌二醇和雌酮等[12]。
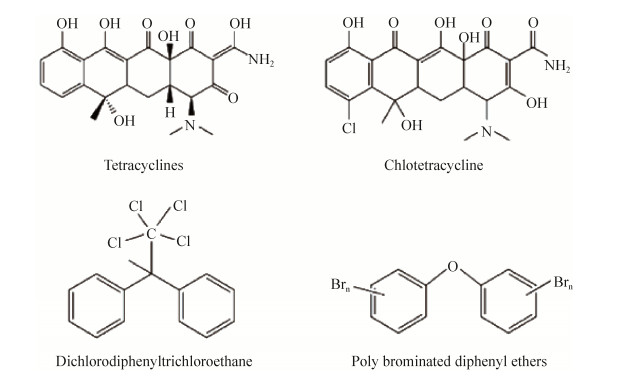
几种常见新兴污染物质四环素、金霉素、滴滴涕和多溴联苯醚的结构如图 1所示。大多数新兴污染物结构非常复杂,化学性质稳定,传统的物理化学方法降解效率不高,并会导致二次污染。微生物降解环境污染物具有环保、高效和价格低廉等优点,被认为是最具潜力的降解方法。据报道,丙二酸柠檬酸杆菌Citrobacter amalonaticus在35 ℃、pH 5.5的条件下,72 h内降解86.1%的四环素[13]。菌株SW-5、LJ245和LJ302在适宜条件下都能对四环素达到90%的降解效率[14]。假单胞菌可以完全降解滴滴涕到4-氯苯甲酸[15]。但是随着科学技术的发展,新兴污染物种类快速增加,目前面临的主要问题有,现有降解菌不可能对所有新兴污染物质具有降解作用;很多新兴污染物质目前还不能被任何微生物降解;使用已有降解菌株降解效率低;在极端环境下降解菌难以实现降解作用等。合成生物学的发展,为解决这些问题提供了新的思路与技术。
2 合成生物学生物修复构想现代化学工业的发展导致新的化合物不断出现,特别是一些人工合成的自然界原本不存在的化合物,自然存在的微生物尚未依靠自然进化获得降解它们的能力,许多菌株往往仅具备代谢通路中的一种或几种催化元件,因此不能将污染物彻底矿化。通过研究细菌中代谢通路的信息,不断发现代谢通路中与代谢产物相关的重要元件,如降解元件、转运元件、趋化元件和抗逆元件等,使我们可以利用合成生物学的技术,有选择性地开发和利用这些重要的生物元件,将不同来源微生物的元件进行设计和组合并构建具备全新代谢网络的工程菌,从而实现环境新兴污染物的彻底矿化,以达到环境修复的目的。
2.1 生物修复关键基因元器件挖掘 2.1.1 脱卤酶元件脱卤酶元件在含卤污染物矿化过程中起到脱卤作用。许多新兴污染物含有卤素原子,去除卤素原子既可以降低该底物生物降解的难度,又可以避免有毒中间产物的产生。
如有机氯农药 (Organochlorine pesticides,OCPs),在五氯酚降解菌鞘氨醇单胞菌Sphingomonaschlorophenolium ATCC39723[16-17]的基础上,研究者成功导入细胞色素P-450cam突变基因,使降解菌具备将六氯苯酚转化为五氯苯酚的能力,进一步通过pcpB编码的单加氧酶反应生成四氯对苯二酚,后经过pcp基因逐步脱氯,实现六氯苯酚的完全矿化[18]。Nagata等发现,少动鞘氨醇单胞菌Sphingomonas paucimobilis UT26的水解脱卤酶LinB是一种具有广泛底物特异性的卤代烷脱卤酶,作用于以六六六代谢中间产物四氯环己二烷为代表的底物,使其碳氯键水解断裂,生成烷基-酶中间产物,最终实现对底物的降解[19]。除此以外,从杀菌剂百菌清降解菌株假单胞菌Pseudomonas sp. CTN-3中分离出能在厌氧和有氧条件下,不需要辅因子的存在即可以进行水解脱氯反应的水解脱氯酶基因chd,为含氯新兴污染物的完全降解提供了研究基础[20]。
又如溴代联苯醚 (PBDEs),作为一类在环境中广泛存在的污染物,其降解途径和降解基因一直处于探索阶段,有关基因编码和表达的报道甚少,更多研究集中在其母环化合物联苯醚 (PCBs) 上。随着对PCBs降解相关的关键调控基因bph和etb[21-22]的深入研究,研究者发现其对PBDEs的降解也发挥着重要作用。将bph和etb基因导入PCBs降解菌-红球菌Rhodococcus erythropolis中,发现其能够使多种多溴联苯醚同系物发生脱溴转化,etb基因重组菌可以降解多种高溴代联苯醚,而bph重组菌则倾向于低溴代联苯醚如一溴、二溴、三溴代联苯醚的脱溴转化[23]。
2.1.2 氧化酶元件氧化酶在芳香烃类有机污染物降解途径中发挥重要的功能,可以将苯环羟基化或切割化。苯双加氧酶将苯氧化成顺苯二氢二醇,在维氏假单胞菌Pseudomonas veronii1YdBTEX2和1YB2中存在此类苯双加氧酶相关基因ipb[24]。甲苯单加氧酶可以将甲苯逐步氧化形成苯甲酸,在恶臭假单胞菌Pseudomonas putida mt-2的大质粒pWW0上包含降解甲苯和间二甲苯的单加氧酶编码基因簇uwcmabn[25],甲苯双加氧酶则是将甲苯分解成顺甲苯二氢二醇,在菌株Pseudomonas putidaF1中存在苯甲酸双加氧酶编码基因todC1C2BA[26]。萘双加氧酶同属一种复合酶,在Pseudomonas putida NCIN9816-4中萘双加氧酶编码基因为nahAa、nahAb、nahAc、nahAd[27]。联苯双加氧酶由还原酶、氧化还原蛋白和铁硫蛋白组成,组成基因包括4个,在菌株Pseudomonas sp. LB400中包含编码基因bphAEFG,可以催化联苯进行双加氧反应[28]。二苯并呋喃双加氧酶可催化二苯并呋喃进行侧面双加氧,再利用下游基因逐步矿化二苯并呋喃,其中地杆菌属Terrabacter sp. DBF63包含二苯并呋喃双加氧酶编码基因dbfA1A2,是催化二苯并呋喃降解的第一步关键基因[29]。
2.1.3 转运蛋白元件目前,从已有研究报道菌株谷氨酸棒状杆菌Corynebacterium glutamicum中发现,4-羟基苯甲酸和原儿茶酸的转运蛋白 (PcaK/NCg1031)、香草酸的转运蛋白 (VanK/NCg2302) 以及苯甲酸的转运相关蛋白 (BenK/NCg2325,BenE/NCg2326),这些转运蛋白在生物修复过程中起着十分重要的作用[30]。龙胆酸转运蛋白GenK在Corynebacteriumglutamicum ATCC13032代谢龙胆酸过程中发挥关键作用[31]。
2.1.4 趋化蛋白元件趋化蛋白元件的发掘能够给污染物降解工程菌株构建提供一定的参考作用。芳香烃化合物感应的甲基趋化受体蛋白的相关研究发现丛毛单胞菌Comamonas testosteroni CNB-1中MCP2201蛋白可以感应TCA循环中产生的芳香族化合物。另外,发现甲基趋化受体MCP2901可以与芳香族化合物结合,通过柠檬酸循环触发趋化反应,还可以启动其下游趋化信号的传导[32-33]。在海洋微生物食碱菌属Alcanivorax dieselolei中发现趋化蛋白CheW2,能够被长链烷烃诱导并传递信号给下游途径,进而激活下游的趋化信号[34]。
2.2 抗逆基因元器件挖掘在过去的几年中,我国微生物学者对环境中有机污染物的微生物降解机制进行了大量研究,阐明了尼古丁、联苯等环境有毒有害物质的微生物降解途径及资源化利用的机制[35-36]。但是在实际应用中往往出现高温、高盐、过酸、过碱或者高渗的恶劣环境,对微生物的生存及代谢具有抑制作用。因此,从微生物自然抗逆功能基因出发,利用离子泵、相似相容性溶质和全局性调控因子等在极端微生物中存在的抗逆机制要素,运用合成生物学手段,构建适配底盘微生物的高效抗逆元件,有目的地重新设计和改造现有降解菌株,对提高微生物在环境压力中的耐受性及其在高效处理环境污染物方面的应用具有重大意义。
2.2.1 离子泵离子泵是一类能够驱使特定的离子逆电化学梯度穿过质膜的蛋白质。细胞内离子泵主要包括钠钾泵和质子泵。若能利用合成生物学方法将这些膜离子泵相关的基因整合到底盘微生物中,对于环境污染修复,尤其是在利用嗜盐、嗜酸和嗜碱微生物抗逆基因构建降解环境污染物的工程菌株方面具有广阔的应用前景。最典型的一类钠离子泵是Na+/H+逆向转运蛋白,它可以偶联氢质子输入系统,泵出细胞内的钠离子,从而维持细胞内的离子浓度平衡。Yang等克隆了盐芽孢杆菌Halobacillus dabanensisD-8T中染色体DNA上编码Na+/H+转运蛋白的nhaH基因,并在大肠杆菌KNabc中进行异源表达,使得菌体能够在0.2 mol/L NaCl或者5 mmol/L LiCl中生存[37]。K+转运系统中的基本元件多为单向转运蛋白,在外界盐浓度较高时,通过在胞内积累K+实现菌体内外渗透压平衡。嗜盐单胞菌Halomonas elongata中的Trk系统和嗜盐古菌中的Kdp系统,是两种主要的K+转运系统[38-39]。Greie等的研究指出,嗜盐古菌中的kdpFABC基因簇可在环境K+浓度变化时由下游基因cat3诱导表达,调节K+转运,使得细胞内外环境达到平衡状态[39]。
2.2.2 相似相容性溶质分泌相似相容性溶质如甜菜碱、氨基酸、糖、多元醇等可以在高渗条件下积累到较高浓度,提高细胞内水活度,在低渗条件下分解或者被排出到周围环境中,使细胞内外渗透压达到平衡,维持细胞正常的代谢活动。Yu等在大肠杆菌中异源表达来自于沙冬青属Ammopiptanthus nanus的甜菜碱醛脱氢酶基因anBADH,使得大肠杆菌在700 mmol/L NaCl和55 ℃下表现出了良好的耐盐及耐热性能[40]。玫瑰盐水球菌Salinicoccus roseus和西班牙盐水球菌Salinicoccushispanicus中存在的脯氨酸是主要的渗透平衡作用调节因子[41]。在大肠杆菌Escherichia coli和鼠伤寒沙门氏菌Salmonella typhimuriu中也存在着由蛋白PutP、ProP和ProU构成的既相互独立又可协同作用的脯氨酸转运系统[42]。除此之外,分别扩增枯草芽孢杆菌Bacillus subtilis中的proA和proB基因,构建融合基因proBA,在大肠杆菌JM83中进行表达,能显著提高菌体内脯氨酸的积累,增强了大肠杆菌对高渗环境的耐受性能[43]。
2.2.3 全局性调控因子能够增强微生物抗逆性能的全局性调控因子主要有sRNA以及热激蛋白 (HSP) 等。sRNA是存在于原核生物中的一种能够通过与mRNA结合进而影响其稳定性或者翻译过程,并最终调控靶基因表达的非编码RNA。热激蛋白是一类能够在高温下与胞内变性的蛋白质结合,修复折叠错误的蛋白,并维持其空间构象和功能的分子伴侣。Negrete等报道,在pH 6.0的弱酸性条件下,将gadY基因在大肠杆菌K-12 1655中进行组成型表达,可以激活胞内的赖氨酸脱羧酶系统 (LDS) 及gadW等耐酸基因的表达,使得菌体生长过程中产生的乙酸含量从8 g/L降低至4 g/L,减轻了对大肠杆菌生长的抑制作用,促进了菌体的生长[44]。孙翔英等以启动子T7和gapA基因作为调控元件,组装并构建了诱导型耐热元器件T7-T.te-HSP20和组成型耐热元器件gapA-T.te-HSP20,有效提高了大肠杆菌的耐热性能和有机溶剂耐受性[45]。Fiocco等在乳杆菌属Lactobacillus plantarum WCFS1中过表达hsp18.5、hsp18.55以及hsp19.3这3种热激蛋白的基因,降低了37 ℃或者40 ℃高温以及12 ℃低温环境对菌体生长速率的影响,同时提高了菌体对丁醇和乙醇等有机溶剂的耐受性[46]。除此之外,传统热激基因如htpG、hsp90、groESL、dnaKJ等也是微生物在抵抗环境变化中发挥重要功能的基因元件[47-49]。
3 展望为实现环境中难降解有机污染物的有效降解,应用合成生物学技术,开发新兴污染物质降解菌株,已成为环境微生物领域研究前沿。不同于传统的从环境中筛选能够代谢特定污染物质的天然微生物为重点的环境生物技术,以合成生物学技术为基础的生物学技术研发重点是在细胞中插入天然或者非天然功能元器件、构建自然界中原不存在的合成代谢途径,形成全新的定向降解的生物细胞工厂。为此研究者可通过合成生物学技术的应用,定向改造微生物代谢路径,使不具降解性能的菌株具有降解某种污染物的能力,提高已报道降解菌株降解效率,增强新兴污染物降解菌株环境适应性能,使其能够在高盐、酸碱、高渗透压条件下保持降解活性,将有利于新兴污染物的环境修复。
研究者也可通过恒化器培养的方法,利用微生物的适应性进化,经过人为施加选择压力,使菌株定向进化,进而筛选得到适应性菌群中的优良菌株。在多种微生物参与的反应器中,研究者可以利用合成生物学技术,人工构建微生物群落生态关系,建立不同微生物间群体感应,使它们能够进行细胞间交流并通过相关基因表达调控来协调群体行为,以使微生物能够在更宽泛的生长环境中长期健康生存并维持平稳的代谢状态,从而在环境修复领域发挥不可替代的作用。
| [1] |
Jiang HY, Zhang MD, Zhou RJ, et al. Occurrence and potential sources of antibiotics in Beijiang River, Southern China.
Asian J Ecotoxicol, 2015, 10(5): 132–140.
(in Chinese). 蒋昊余, 张孟迪, 周仁钧, 等. 北江流域抗生素污染水平和来源初探. 生态毒理学报, 2015, 10(5): 132-140. |
| [2] |
Zhang H, Luo Y, Zhou QX. Research advancement of eco-toxicity of tetracycline antibiotics.
J Agro-Environ Sci, 2007, 27(2): 407–413.
(in Chinese). 张浩, 罗义, 周启星. 四环素类抗生素生态毒性研究进展. 农业环境科学学报, 2007, 27(2): 407-413. |
| [3] |
Zhang SQ, Zhang FD, Liu XM, et al. Determination and analysis on main harmful composition in excrement of scale livestock and poultry feedlots.
Plant Nutr Fert Sci, 2005, 11(6): 822–829.
(in Chinese). 张树清, 张夫道, 刘秀梅, 等. 规模化养殖畜禽粪主要有害成分测定分析研究. 植物营养与肥料学报, 2005, 11(6): 822-829. |
| [4] | Hamscher G, Sczesny S, H per H, et al. Determination of persistent tetracycline residues in soil fertilized with liquid manure by high-performance liquid chromatography with electrospray ionization tandem mass spectrometry. Anal Chem, 2002, 74(7): 1509–1518. DOI: 10.1021/ac015588m |
| [5] | Aust MO, Godlinskl F, Travls GR, et al. Distribution of sulfamethazine, chlortetracycline and tylosin in manure and soil of Canadian feedlots after subtherapeutic use in cattle. Environ Pollut, 2008, 156(3): 1243–1251. DOI: 10.1016/j.envpol.2008.03.011 |
| [6] |
Lv XM. Research progress of endocrine disrupting chemicals (EDCs) and pharmaceuticals and personal care products (PPCPs).
Environ Monitor China, 2012, 28(4): 118–123.
(in Chinese). 吕小明. 典型新兴环境污染物的研究进展. 中国环境监测, 2012, 28(4): 118-123. |
| [7] |
Wu DS. Endocrine disrupting chemicals and health of human being.
J Environ Health, 2001, 18(4): 195–197.
(in Chinese). 吴德生. 内分泌干扰物与人类健康. 环境与健康杂志, 2001, 18(4): 195-197. |
| [8] |
Li J, Si JL. Introduction of environmental endocrine disruptors.
J Environ Health, 2002, 19(1): 83–84.
(in Chinese). 李杰, 司纪亮. 环境内分泌干扰物质简介. 环境与健康杂志, 2002, 19(1): 83-84. |
| [9] |
Tang YJ, Liu Q, Zeng CS, et al. Detection and evalution of organochlorine pesticides in fishes of Meijiang River.
J Jiaying Univ: Nat Sci, 2015, 33(8): 76–81.
(in Chinese). 唐以杰, 刘青, 曾翠珊, 等. 梅江鱼类有机氯农药含量检测和评价. 嘉应学院学报:自然科学, 2015, 33(8): 76-81. |
| [10] |
Wu QL, Shen Q. Determination of four kinds of anabolic steroids residues in the egg.
J Anhui Agric Sci, 2005, 33(5): 852, 878.
(in Chinese). 吴启陆, 沈清. 高效液相色谱同时测定鸡蛋中4种激素残留量的方法. 安徽农业科学, 2005, 33(5): 852, 878. |
| [11] |
Wan MM, Chen ZL, Liu YH, et al. Determination of diethylstilbestrol in animal tissues by HPLC/ECD.
Chin J Vet Drug, 2004, 38(7): 4–6.
(in Chinese). 万美梅, 陈杖榴, 刘雅红, 等. 动物组织中己烯雌酚残留的高效液相色谱/电化学检测方法研究. 中国兽药杂志, 2004, 38(7): 4-6. |
| [12] | Mao LS, Sun CJ, Zhang H, et al. Determination of environmental estrogens in human urine by high performance liquid chromatography after fluorescent derivatization with p-nitrobenzoyl chloride. Anal Chim Acta, 2004, 522(2): 241–246. DOI: 10.1016/j.aca.2004.04.071 |
| [13] |
Ma ZQ, Ma YL, Xie L, et al. Experimental study on microbial degradation of tetracycline residues in antibiotic waste.
Environ Sci Technol, 2012, 35(1): 46–49, 62.
(in Chinese). 马志强, 马玉龙, 谢丽, 等. 微生物法降解药渣中残留四环素的试验研究. 环境科学与技术, 2012, 35(1): 46-49, 62. |
| [14] |
Zhang HY. Study of degradation characteristics of microorganism strains and main degradation products for chlortetracycline[D]. Beijing: Beijing Institute of Technology, 2015 (in Chinese). 张惠艳. 金霉素降解菌株特性及代谢产物研究[D]. 北京: 北京理工大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10007-1015030556.htm |
| [15] | Kamanavalli CM, Ninnekar HZ. Biodegradation of DDT by a Pseudomonas species. Curr Microbiol, 2004, 48(1): 10–13. DOI: 10.1007/s00284-003-4053-1 |
| [16] | Cai M, Xun LY. Organization and regulation of pentachlorophenol-degrading genes in Sphingobium chlorophenolicum ATCC 39723. J Bacteriol, 2002, 184(17): 4672–4680. DOI: 10.1128/JB.184.17.4672-4680.2002 |
| [17] | Dai MH, Copley SD. Genome shuffling improves degradation of the anthropogenic pesticide pentachlorophenol by Sphingobium chlorophenolicum ATCC 39723. Appl Environ Microbiol, 2004, 70(4): 2391–2397. DOI: 10.1128/AEM.70.4.2391-2397.2004 |
| [18] | Yan DZ, Liu H, Zhou NY. Conversion of Sphingobium chlorophenolicum ATCC 39723 to a hexachlorobenzene degrader by metabolic engineering. Appl Environ Microbiol, 2006, 72(3): 2283–2286. DOI: 10.1128/AEM.72.3.2283-2286.2006 |
| [19] | Nagata Y, Prokop Z, Sato Y, et al. Degradation of β-hexachlorocyclohexane by haloalkane dehalogenase LinB from Sphingomonas paucimobilis UT26. Appl Environ Microbiol, 2005, 71(4): 2183–2185. DOI: 10.1128/AEM.71.4.2183-2185.2005 |
| [20] | Wang GL, Li R, Li SP, et al. A novel hydrolytic dehalogenase for the chlorinated aromatic compound chlorothalonil. J Bacteriol, 2010, 192(11): 2737–2745. DOI: 10.1128/JB.01547-09 |
| [21] | Iwasaki T, Miyauchi K, Masai E, et al. Multiple-subunit genes of the aromatic-ring-hydroxylating dioxygenase play an active role in biphenyl and polychlorinated biphenyl degradation in Rhodococcus sp. strain RHA1. Appl Environ Microbiol, 2006, 72(8): 5396–5402. DOI: 10.1128/AEM.00298-06 |
| [22] | Ohtsubo Y, Nagata Y, Kimbara K, et al. Expression of the bph genes involved in biphenyl/PCB degradation in Pseudomonas sp. KKS102 induced by the biphenyl degradation intermediate, 2-hydroxy-6-oxo-6-phenylhexa-2, 4-dienoic acid. Gene, 2000, 256(1/2): 223–228. |
| [23] | Robrock KR. Aerobic and anaerobic microbial degradation of polybrominated diphenyl ethers (PBDEs)[D]. Berkeley: University of California, 2008. |
| [24] | de Lima-Morales D, Chaves-Moreno D, Wos-Oxley ML, et al. Degradation of benzene by Pseudomonas veronii 1YdBTEX2 and 1YB2 is catalyzed by enzymes encoded in distinct catabolism gene clusters. Appl Environ Microbiol, 2016, 82(1): 167–173. DOI: 10.1128/AEM.03026-15 |
| [25] | Kim J, Pérez-Pantoja D, Silva-Rocha R, et al. High-resolution analysis of the m-xylene/toluene biodegradation subtranscriptome of Pseudomonas putida mt-2. Environ Microbiol, 2016, 18(10): 3327–3341. DOI: 10.1111/1462-2920.13054 |
| [26] | Zylstra GJ, Gibson DT. Toluene degradation by Pseudomonas putida F1. Nucleotide sequence of the todC1C2BADE genes and their expression in Escherichia coli. J Biol Chem, 1989, 264(25): 14940–14946. |
| [27] | Suen WC, Gibson DT. Isolation and preliminary characterization of the subunits of the terminal component of naphthalene dioxygenase from Pseudomonas putida NCIB 9816-4. J Bacteriol, 1993, 175(18): 5877–5881. DOI: 10.1128/jb.175.18.5877-5881.1993 |
| [28] | Erickson BD, Mondello FJ. Nucleotide sequencing and transcriptional mapping of the genes encoding biphenyl dioxygenase, a multicomponent polychlorinated-biphenyl-degrading enzyme in Pseudomonas strain LB400. J Bacteriol, 1992, 174(9): 2903–2912. DOI: 10.1128/jb.174.9.2903-2912.1992 |
| [29] | Nojiri H, Kamakura M, Urata M, et al. Dioxin catabolic genes are dispersed on the Terrabacter sp. DBF63 genome. Biochem Biophys Res Commun, 2002, 296(2): 233–240. DOI: 10.1016/S0006-291X(02)00873-2 |
| [30] | Chaudhry MT, Huang Y, Shen XH, et al. Genome-wide investigation of aromatic acid transporters in Corynebacterium glutamicum. Microbiology, 2007, 153(3): 857–865. DOI: 10.1099/mic.0.2006/002501-0 |
| [31] | Xu Y, Wang SH, Chao HJ, et al. Biochemical and molecular characterization of the gentisate transporter GenK in Corynebacterium glutamicum. PLoS ONE, 2012, 7(7): e38701. DOI: 10.1371/journal.pone.0038701 |
| [32] | Ni B, Huang Z, Fan Z, et al. Comamonas testosteroni uses a chemoreceptor for tricarboxylic acid cycle intermediates to trigger chemotactic responses towards aromatic compounds. Mol Microbiol, 2013, 90(4): 813–823. DOI: 10.1111/mmi.12400 |
| [33] | Huang Z, Ni B, Jiang CY, et al. Direct sensing and signal transduction during bacterial chemotaxis toward aromatic compounds in Comamonas testosteroni. Mol Microbiol, 2016, 101(2): 224–237. DOI: 10.1111/mmi.2016.101.issue-2 |
| [34] | Wang WP, Shao ZZ. The long-chain alkane metabolism network of Alcanivorax dieselolei. Nat Commun, 2014, 5: 5755. DOI: 10.1038/ncomms6755 |
| [35] | Tang HZ, Wang LJ, Wang WW, et al. Systematic unraveling of the unsolved pathway of nicotine degradation in Pseudomonas. PLoS Genet, 2013, 9(10): e1003923. DOI: 10.1371/journal.pgen.1003923 |
| [36] | Li QG, Wang XY, Yin GB, et al. New metabolites in dibenzofuran cometabolic degradation by a biphenyl-cultivated Pseudomonas putida strain B6-2. Environ Sci Technol, 2009, 43(22): 8635–8642. DOI: 10.1021/es901991d |
| [37] | Yang LF, Jiang JQ, Zhao BS, et al. A Na+/H+ antiporter gene of the moderately halophilic bacterium Halobacillus dabanensis D-8T: cloning and molecular characterization. FEMS Microbiol Lett, 2006, 255(1): 89–95. DOI: 10.1111/fml.2006.255.issue-1 |
| [38] | Kraegeloh A, Amendt B, Kunte HJ. Potassium transport in a halophilic member of the Bacteria domain: identification and characterization of the K+ uptake systems TrkH and TrkI from Halomonas elongata DSM 2581T. J Bacteriol, 2005, 187(3): 1036–1043. DOI: 10.1128/JB.187.3.1036-1043.2005 |
| [39] | Strahl H, Greie JC. The extremely halophilic archaeon Halobacterium salinarum R1 responds to potassium limitation by expression of the K+-transporting KdpFABC P-type ATPase and by a decrease in intracellular K+. Extremophiles, 2008, 12(6): 741–752. DOI: 10.1007/s00792-008-0177-3 |
| [40] | Yu HQ, Wang YG, Yong TM, et al. Heterologous expression of betaine aldehyde dehydrogenase gene from Ammopiptanthus nanus confers high salt and heat tolerance to Escherichia coli. Gene, 2014, 549(1): 77–84. DOI: 10.1016/j.gene.2014.07.049 |
| [41] | Ventosa A, Nieto JJ, Oren A. Biology of moderately halophilic aerobic bacteria. Microb Mol Biol Rev, 1998, 62(2): 504–544. |
| [42] | Csonka LN. Physiological and genetic responses of bacteria to osmotic stress. Microbiol Rev, 1989, 53(1): 121–147. |
| [43] |
Liu RJ, Chen T, Cao JW. Construction of fused osmoregulation proBA gene from a salt-tolerant mutant of Bacillus subtilis and its influence on the osmotolerance of Escherichia coli.
Acta Microbiol Sin, 2005, 45(1): 23–26.
(in Chinese). 刘瑞杰, 陈婷, 曹军卫. 渗透压调节基因proBA的融合表达对大肠杆菌耐高渗胁迫能力的影响. 微生物学报, 2005, 45(1): 23-26. |
| [44] | Negrete A, Shiloach J. Constitutive expression of the sRNA GadY decreases acetate production and improves E. coli growth. Microb Cell Fact, 2015, 14: 148. DOI: 10.1186/s12934-015-0334-1 |
| [45] |
Sun XY, Liu YQ, Sun H, et al. Construction of heat resistance devices for Escherichia coli and their application.
CIESC J, 2014, 65(8): 3128–3135.
(in Chinese). 孙翔英, 刘月琴, 孙欢, 等. 大肠杆菌耐热元器件的构建及其应用. 化工学报, 2014, 65(8): 3128-3135. |
| [46] | Fiocco D, Capozzi V, Goffin P, et al. Improved adaptation to heat, cold, and solvent tolerance in Lactobacillus plantarum. Appl Microbiol Biotechnol, 2007, 77(4): 909–915. DOI: 10.1007/s00253-007-1228-x |
| [47] | Garcie C, Tronnet S, Garénaux A, et al. The bacterial stress-responsive Hsp90 chaperone (HtpG) is required for the production of the genotoxin colibactin and the Siderophore Yersiniabactin in Escherichia coli. J Infect Dis, 2016, 214(6): 916–924. DOI: 10.1093/infdis/jiw294 |
| [48] | Fan DJ, Liu CP, Liu LS, et al. Large-scale gene expression profiling reveals physiological response to deletion of chaperone dnaKJ in Escherichia coli. Microbiol Res, 2016, 186-187: 27–36. DOI: 10.1016/j.micres.2016.03.001 |
| [49] | Luan GD, Dong HJ, Zhang TR, et al. Engineering cellular robustness of microbes by introducing the GroESL chaperonins from extremophilic bacteria. J Biotechnol, 2014, 178: 38–40. DOI: 10.1016/j.jbiotec.2014.03.010 |
 2017, Vol. 33
2017, Vol. 33