中国科学院微生物研究所、中国微生物学会主办
文章信息
- 季翔宇, 赵会伟, 娄春波
- Xiangyu Ji, Huiwei Zhao, Chunbo Lou
- 人工基因元件的生理学研究进展
- Advances in synthetic physiology of artificial genetic parts
- 生物工程学报, 2017, 33(3): 393-403
- Chin J Biotech, 2017, 33(3): 393-403
- 10.13345/j.cjb.170008
-
文章历史
- Received: January 9, 2017
- Accepted: February 24, 2017
2 中国科学院大学,北京 100049
2 University of Chinese Academy of Sciences, Beijing 100049, China
合成生物学旨在用工程学原理指导人工生命系统设计和实现预想的功能,或经由这种“创造去学习”的方式辅助我们深化对生命系统运作方式的理解。在合成生物学兴起的十几年中,人工基因元件设计和组装完成了许多重要突破:人工基因元件设计方面,大量工作积累了一批精细刻画的优质元件,为复杂回路的设计提供了可靠的工具箱。在此基础上近期研究者已可根据目标真值表自动设计多输入-输出的复杂布尔逻辑电路并生成DNA序列,在大肠杆菌Escherichia coli中实现了良好的预测性[1];在组装技术研究方面,Gibson assembly[2]、Golden gate[3]及酵母同源重组技术[4]代表的DNA组装技术可实现不同尺度的多片段组装,使人工系统的快速构建成为可能。
尽管在人工基因元件的设计和组装中取得了巨大突破,合成生物学至今仍未解决的一个最主要瓶颈问题是人工基因元件“放入”底盘生物体后,对细胞生理产生不可预知的干涉性影响。反过来,这些干涉性影响导致理论上“模块化”的生物元件的功能变得不可预知。依照工程学原理,人造生物系统在理想状态下应可以被拆解为功能上相互独立的模块,模块间的拼装不会导致模块功能的改变[5],使其构建系统可以像电子系统一样被扩展和尺度放大 (Scale-up)[6]。鉴于此,合成生物学领域的大量工作都注重正交性元件的开发 (Part-mining)[7-11],致力于元件之间相互作用的最小化。然而近年来的工作表明,基因回路并不能严格地和宿主细胞隔离,而是与细胞的生理状态耦合形成一个整体[12],这就使我们陷入虽然拥有功能和行为经过精细刻画的元件,但仍然不能避免针对每个回路进行点对点 (Ad hoc) 优化[13]的窘境,元件一旦脱离了刻画时的细胞生理状态使用,其行为就有可能偏离预期,使得“由下而上” (Bottom-up) 的通路构建陷入巨大工作量的“design-build-test-learn”循环[14]。
近期已有工作开始关注细胞生理系统对人工基因元件的影响并提出特定元件的设计原则。本文将对合成生物学人工生命系统与底盘细胞生理系统的相互作用方式及其不利影响进行梳理,对表征和消除这种相互作用的技术方法、设计原则进行综述,并提出合成生理学的概念,为今后合成生物学构建的人工系统与宿主或底盘细胞生理系统相互作用的研究提供框架和方向。
1 合成生物学元件与底盘细胞生理系统的相互作用方式合成生物学元件与底盘细胞的相互作用是紧密联系、互为因果的,本章节根据已有的实验证据将其归纳为3点:1) 基因元件过表达引发细胞生长压力;2) 基因元件特异调控会导致细胞毒性;3) 细胞体内特殊生理机制会影响元件的内禀参数。下文分别加以论述。
1.1 基因元件过表达引发细胞生长压力细胞利用有限的资源完成营养物质摄取、能量代谢、DNA复制和细胞分裂等诸多生理过程,为了优化自身的生长,细胞需要根据环境平衡分配这些资源。由合成生物学原理设计并人为构建的基因元件或系统则打破了这种平衡,引发细胞生长压力 (Burden)。
首先,人工构建的基因元件在DNA复制和蛋白表达过程中都占用了底盘细胞的资源。过多的内源[15]或外源蛋白[16]过量表达都会造成细胞生长放缓。机制研究表明相比于能量和物质消耗,这一现象更主要来源于底盘细胞RNA聚合酶和核糖体的占用[17-18]。过量表达蛋白导致细胞生长停滞,还会引起ppGpp等途径激活细胞应激反应[16],造成细胞生理状态的全局性切换,这也是下文提到的细胞体内特殊生理机制会影响元件内禀参数的原因之一。
细胞生长减缓对人工基因元件的利用造成诸多不良影响。除削弱元件预测性外,生长压力带来的负向筛选作用会减弱人工基因回路的遗传稳定性。尽管底盘细胞DNA的基础突变率极低,如果突变体相对元件宿主具有生长优势便会很快占据群落的主体,使元件在群体层次上失效。例如,You等设计的控制细胞群体大小的元件在传代培养3-6 d后就由于产生的逃脱调控的突变体具有生长优势,大量增殖而失效[19]。
1.2 基因元件特异调控会导致细胞毒性基因元件除了跟预期目标靶位点相互作用以外,还可能对未预料的特定基因发生特异调控作用,并对底盘细胞生理功能产生影响,导致细胞毒性 (Toxicity)。虽然这种基于调控产生毒性的表现往往也是细胞生长缓慢乃至死亡,但由于这些元件的表达量不足以造成严重的细胞资源的占用,我们将其与生物元件过表达引发细胞生长压力区别开来,称之为细胞毒性。
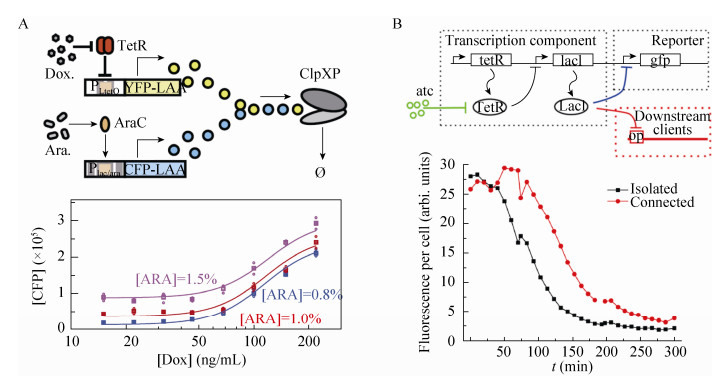
为了构造更为复杂的人工生命系统,获取能够同时使用而不相互干涉的正交元件库是必需的[20]。获取正交元件的思路之一是运用生物信息学手段从不同物种挖掘属于同一家族的元件,但由于这些元件和底盘细胞没有经过漫长的共同进化,它们之间可能存在意料之外的相互作用[6]。如图 1A所示,Stanton等对tetR家族的阻遏蛋白进行了挖掘,发现的一些元件在其表达量足够对回路输出产生调控作用时,细胞的生长也开始受到影响[10]。考虑到基因组序列上存在许多不活跃的调控蛋白结合位点[21],这些阻遏蛋白的细胞毒性很可能来源于转录因子与宿主DNA序列特异性结合而导致的相互作用。另外,即使单个阻遏蛋白表现的细胞毒性较小,用多个阻遏蛋白构建回路时其细胞毒性可能积累对细胞生长造成严重影响[1]。产生细胞毒性的机制不局限于调控蛋白-DNA序列互作层面,基于蛋白-蛋白互作设计的元件也可能造成细胞毒性。Rhodius等从ECF σ因子、抗σ因子 (Extracytoplasmic function σs,anti σs) 中挖掘转录调控元件时, 发现部分σ因子、抗σ因子在较高表达量时,由于与内源RNA聚合酶竞争结合由α2ββ′ω亚基组成的核心酶 (Core enzyme),导致内源基因异常表达产生细胞毒性[9]。
不只是一些新挖掘的元件,许多已经在多种宿主中广泛使用的优质元件其细胞毒性也限制了其作用的发挥和推广。CRISPRi-dCas9 (CRISPR interference-dCas9) 调控系统被认为是优秀的可编程的调控元件,目前已被广泛用于基因回路构建和代谢工程调控[11, 22],然而dCas9随其表达量升高、抑制效果增强会导致细胞生长迟滞的现象在多篇文章中都有所报道[23-24],如图 1B所示。一些研究者认为该毒性的来源是CRISPR具有的脱靶效应[25]。CRISPRi-dCas9的毒性导致其在实际使用时,需要耗费使用者大量精力平衡转录调控开关的正面影响与其对细胞生长的负面影响。
合成生物学的元件设计最终要针对下游应用问题的解决而调整,需要宿主细胞的健康快速生长,因此元件的细胞毒性是其应用的瓶颈问题之一。
1.3 细胞体内特殊生理机制会影响元件内禀参数元件的内禀参数是指用于数学描述元件行为的参数,如启动子的启动强度,阻遏蛋白的解离常数、希尔系数等。由于这些参数都是在特定条件下由实验测定或拟合得到的,变更培养条件或更换元件宿主从而改变细胞的生理条件会影响元件的内禀参数。影响元件内禀参数的细胞体内特殊生理机制主要包括:1) 细胞资源竞争导致内禀参数耦合;2) 追溯效力 (Retroactivity);3) 细胞生长状态影响元件表达相关参数,本节将对这些机制分别展开介绍。
合成生物学元件和宿主细胞共同利用细胞内的酶等有限资源,资源竞争会导致设计上互不干涉的构建产生功能上的耦合。如图 2A所示,Cookson等在实验中发现带有LAA蛋白降解标签的黄色荧光蛋白、青色荧光蛋白在同一Escherichia coli细胞中表达时,提高其中一个荧光蛋白的表达量会导致另一荧光蛋白荧光值提高。这是由于两者共用E. coli内源的ClpXP降解机器,蛋白过表达造成降解机器过载,使得其内禀参数蛋白降解速率产生耦合。这一机制被称为排队效应 (Queueing-up effect)[26]。与此类似,RNA对核糖体的竞争也会导致翻译层次的耦合。Tabor等的实验结果显示含有类似RBS序列的sRNA (Small RNA) 大量表达会结合细胞内的核糖体,导致细胞内的翻译效率下降,表现为同时表达的GFP表达量降低。
追溯效力 (Retroactivity) 是指信号通路下游的系统给上游系统带来信号反馈,从而影响上游系统功能的效应[5]。以合成生物学系统中常用的,阻遏蛋白结合操作子序列介导的信号传导过程为例:上游模块表达阻遏蛋白作为输出信号分子,由于这个阻遏蛋白特异识别多个下游模块的操作子序列,并结合到操作子DNA上完成转录调控,因此阻遏蛋白的浓度就作为下游模块输入信号影响其输出信号。如果阻遏蛋白在上游模块中还结合了其他模块的操作子DNA序列,会导致作为下游模块输入信号的转录因子有效浓度下降,因此额外操作子的存在将影响上下游模块组装的可预测性。如图 2B所示,Jayanthi等利用一个简易系统说明了回溯活性的影响[27]。在构建上游模块时,他们将融合了降解标签LVA的绿色荧光蛋白置于lacⅠ阻遏蛋白抑制的PLac启动子表达下,lacⅠ则由诱导剂atc (无水四环素) 诱导表达。当添加1个相当于其他模块且含有lacO操作子序列的质粒时,待测试模块的行为发生了变化:加入atc抑制绿色荧光蛋白表达和去除atc解开绿色荧光蛋白表达抑制时,该实验组相对于没有额外lacO操作子的对照组都有一定程度的延迟,这就是由于其他模块与上游待测模块竞争了lacⅠ所导致的结果。
在细胞体内,基因元件的相关参数会随着细胞生长状态改变而改变。细胞内基因的实际拷贝数、RNA、蛋白丰度都受生长速率影响[28];细胞内外的信号则会对基因表达产生全局性的调控[29]。Zhang等在实验中发现,在E. coli的生长平台期添加IPTG已经无法有效诱导pTAC启动子表达报告基因,正体现了细胞生长状态对元件表达影响的复杂性[30]。
2 人工基因元件-细胞生理系统相互作用的刻画和不利影响的消除方法基于上述人工基因元件与细胞生理系统之间的相互干涉效应,近年来不少研究者开始进行这种相互干涉作用的定量刻画工作并针对性地总结和验证人工合成生物系统的去干涉设计原则,使得合成生理学有了基本的框架和研究范式。本节将总结近期合成生理学范围内的代表性工作,并介绍目前如何通过工程化设计和生物物理模型消除或精准考虑这些因素的影响。
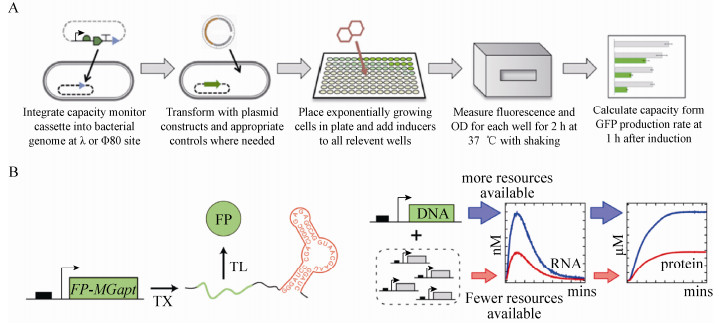
2.1 元件-细胞生理系统相互作用的刻画传统刻画生长压力的方法就是通过观察宿主相对于对照的生长速率变化,然而该方法只有当生长压力足够大时,才能在表型上表现出显著差别。如图 3A所示,Ceroni等开发了一套基于荧光检测的细胞生长压力检测方法,将一个常量表达的绿色荧光蛋白基因用噬菌体整合酶整合到E. coli基因组的特定位点上,考虑到绿色荧光蛋白的表达不受特异性调控,其表达量应该能反映资源分配对细胞全局表达的影响。该工作中作者用单位细胞产生的荧光表征或监测细胞资源的容量 (Capacity),测试不同质粒拷贝数、启动子强度、RBS强度组合下的人工基因元件对细胞资源的占用情况,得出相比于DNA复制、转录过程,其翻译过程中人工基因元件对核糖体的占用是导致宿主生长压力的主要来源[31]。该方法为优化元件表达策略提供了开创性的定量和评价方法。除此之外,也有工作还关注利用无细胞系统 (Cell-free system) 定量研究特定构建对宿主资源的占用 (图 3B)[32]。无细胞系统的优势在于实验简便,反应体系小,便于高通量化,但由于只保留了转录翻译系统,系统的循环能力较弱,诸如NTP水解以及酶降解的因素会导致蛋白合成的过早终止,使元件在系统中的行为与体内 (in vivo) 的行为之间存在相当大的差别。
对元件-细胞生理系统的相互作用进行刻画,一方面为研究者在设计基因回路时比较不同设计方案提供了实验技术支持;另一方面该类工作能找出相互作用中的关键因素,有助于发现更多具有普适性的回路设计原则。
2.2 元件-细胞生理系统相互作用的消除针对基因元件-细胞生理系统相互作用导致其功能预测性削弱,研究者可以通过某些工程化设计方案消除这些影响;或通过强大的生物物理模型精准考虑这些因素的影响,将其包含在元件行为的预测模型中,以提高对元件组装后行为预测的准确性。
关于减小人工基因元件对宿主细胞造成的生长压力,最直接的方法是在完成同样功能的设计中选择消耗资源较少的一种。例如Arkin实验室利用fim整合酶系统构建了一系列记忆存储装置,当装置接受诱导剂的输入信号时重组酶的表达可以使特定的DNA序列反向,并利用这段DNA序列的方向储存接收输入信号的“记忆”[33-34]。选择执行记忆储存功能的装置时,这种利用重组酶系统的设计相比利用转录因子调控实现的扳式开关 (Toggle switch)[35]很可能是一个更好的选择,因为后者储存信息依靠的是特定转录因子在细胞内的高浓度,需要不断地占用细胞的资源表达转录因子维持这一状态。
解决基因元件特异调控导致的细胞毒性,则可从3个方面入手:1) 在开发合成生物学元件时,注意测试元件的细胞毒性,舍弃具有毒性的元件,提升合成生物学工具箱的质量。关于这一点,前文提到的Voigt实验室开发tetR家族的阻遏蛋白工具箱的工作提供了很好的范例[10];2) 通过实验深入挖掘元件毒性的产生机理,据此在回路设计中进行规避。如Kimelman等对无法在E. coli中得到克隆的DNA序列进行分析,挖掘其中具有毒性 (或表达产物具有毒性) 的DNA序列,其中很重要的一部分是具有DnaA box的序列。在E. coli中DnaA蛋白结合到DnaA box上启动DNA复制。该工作之后通过实验证明多拷贝的DnaA box与DnaA相互作用会导致细胞毒性。这一类的研究提供的信息能够有效指导我们在设计中选择可靠的元件;3) 表达具有潜在毒性的元件时,应考虑采用诱导激活的表达方式,并采用诸如sRNA抑制等策略减小其泄露表达量[20]。
对于细胞体内特殊生理机制会影响元件内禀参数的问题,一个可行的解决办法是将人工基因元件与细胞生理系统解耦合。利用与宿主细胞正交的外源表达系统可以将元件的表达从宿主细胞中隔离开来。这一方面应用较为广泛的是T7转录系统,T7家族RNA聚合酶来源于噬菌体,识别的启动子序列特征与E. coli等宿主细胞内源启动子有相当大的差别,保证了正交性,常被用于合成生物学元件的表达。如Kushwaha等使用T7 RNA聚合酶-启动子构建了一套基于反馈调控的表达系统,在E. coli、Pseudomonas putida、Bacillus subtilis中都实现了元件的稳定表达[36]。另一个例子是Cameron等利用烟草病毒来源的Lon蛋白酶在E. coli中控制人工基因元件的降解,避免E. coli内源的降解系统过载,如图 4所示[37]。追溯效力导致的影响则可以通过绝缘子元件的设计将上下游信号通路隔离而解决。Mishra等在酿酒酵母Saccharomyces cerevisiae中的转录因子浓度作为输入-输出的信号通路上下游间添加了基于磷酸化快反应的绝缘装置,削弱了由追溯效力导致的信号传导延滞[38]。

|
| 图 4 利用与宿主细胞正交的外源系统可以将元件的表达和降解从宿主细胞中隔离开来 (改编自文献[38]和文献[39]) Figure 4 The expression and degradation of genetic parts can be decoupled from hosts by using orthogonal heterologous systems. Fig. A adapted from Kushwaha, M. et al., Nat Commun.[38]. Fig. B adapted from Cameron, D.E. and J.J. Collins, Nat Biotechnol.[39] |
| |
另一种思路是通过强大的生物物理模型精准考虑元件-细胞生理系统相互作用的影响,将其包含在元件行为的模型中提高预测的准确性。在这方面的成功范例是Salis实验室关于RBS calculator的一系列工作。RBS calculator基于热力学原理考虑mRNA二级结构、mRNA-rRNA相互作用计算不同转录本的翻译起始效率,输入不同宿主的16S rRNA序列可以有效实现跨宿主的计算[39-40]。
3 结论与展望过去十几年间,合成生物学元件工具箱以及组装方法的开发经历了“从零到一”的飞跃,然而元件-宿主细胞的相互作用却阻碍了人工设计的生命系统的复杂度进一步提升。近期许多研究者开始关注细胞生理系统对人工基因元件的影响以及相关元件设计原则等合成生物学领域基本工程科学问题。这些研究对未来合成生理学的方向很有启发作用。未来合成生理学的研究应在以下几点展开:1) 注重刻画元件在不同培养条件下功能和行为,并对实验结果表述和定量方式进行标准化,便于数据的整合和分享;2) 开发基于转录组、蛋白质组等多层次高通量技术,降低获取合成生物学构建表型数据的资金和时间成本,并作针对性地数据挖掘,为理解元件-宿主相互作用的产生机制提供数据支持;3) 根据需要开发粗粒化和精细的全细胞模型,用于描述资源分配等细胞生理规律,增强人工合成生物系统的预测性;4) 研发元件-宿主隔离技术和策略,总结消除两者相互作用影响的设计原则,例如前文提到的利用正交外源表达系统将元件表达从宿主细胞中隔离的策略。合成生理学的深入研究将使我们真正实现人工生命系统设计的精准化,促进合成生物学成果在应用领域的高效转化。
| [1] | Nielsen AAK, Der BS, Shin J, et al. Genetic circuit design automation. Science, 2016, 352(6281): aac7341. DOI: 10.1126/science.aac7341 |
| [2] | Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods, 2009, 6(5): 343–345. DOI: 10.1038/nmeth.1318 |
| [3] | Engler C, Kandzia R, Marillonnet S. A one pot, one step, precision cloning method with high throughput capability. PLoS ONE, 2008, 3(11): e3647. DOI: 10.1371/journal.pone.0003647 |
| [4] | Gibson DG, Benders GA, Axelrod KC, et al. One-step assembly in yeast of 25 overlapping DNA fragments to form a complete synthetic Mycoplasma genitalium genome. Proc Natl Acad Sci USA, 2008, 105(51): 20404–20409. DOI: 10.1073/pnas.0811011106 |
| [5] | Del Vecchio D, Ninfa AJ, Sontag ED. Modular cell biology: retroactivity and insulation. Mol Syst Biol, 2008, 4(1): 161. |
| [6] | Arkin AP, Fletcher DA. Fast, cheap and somewhat in control. Genome Biol, 2006, 7(8): 114. DOI: 10.1186/gb-2006-7-8-114 |
| [7] | Green AA, Silver PA, Collins JJ, et al. Toehold switches: de-novo-designed regulators of gene expression. Cell, 2014, 159(4): 925–939. DOI: 10.1016/j.cell.2014.10.002 |
| [8] | Temme K, Hill R, Segall-Shapiro TH, et al. Modular control of multiple pathways using engineered orthogonal T7 polymerases. Nucleic Acids Res, 2012, 40(17): 8773–8781. DOI: 10.1093/nar/gks597 |
| [9] | Rhodius VA, Segall-Shapiro TH, Sharon BD, et al. Design of orthogonal genetic switches based on a crosstalk map of σs, anti-σs, and promoters. Mol Syst Biol, 2013, 9(1): 702. |
| [10] | Stanton BC, Nielsen AAK, Tamsir A, et al. Genomic mining of prokaryotic repressors for orthogonal logic gates. Nat Chem Biol, 2014, 10(2): 99–105. |
| [11] | Cress BF, Jones JA, Kim DC, et al. Rapid generation of CRISPR/dCas9-regulated, orthogonally repressible hybrid T7-lac promoters for modular, tuneable control of metabolic pathway fluxes in Escherichia coli. Nucleic Acids Res, 2016, 44(9): 4472–4485. DOI: 10.1093/nar/gkw231 |
| [12] | Klumpp S, Hwa T. Bacterial growth: global effects on gene expression, growth feedback and proteome partition. Curr Opin Biotechnol, 2014, 28: 96–102. |
| [13] | Endy D. Foundations for engineering biology. Nature, 2005, 438(7067): 449–453. DOI: 10.1038/nature04342 |
| [14] | Cheng AA, Lu TK. Synthetic biology: an emerging engineering discipline. Annu Rev Biomed Eng, 2012, 14(1): 155–178. DOI: 10.1146/annurev-bioeng-071811-150118 |
| [15] | Dong H, Nilsson L, Kurland CG. Gratuitous overexpression of genes in Escherichia coli leads to growth inhibition and ribosome destruction. J Bacteriol, 1995, 177(6): 1497–1504. DOI: 10.1128/jb.177.6.1497-1504.1995 |
| [16] | Shachrai I, Zaslaver A, Alon U, et al. Cost of unneeded proteins in E. coli is reduced after several generations in exponential growth. Mol Cell, 2010, 38(5): 758–767. DOI: 10.1016/j.molcel.2010.04.015 |
| [17] | Gyorgy A, Jiménez JI, Yazbek J, et al. Isocost lines describe the cellular economy of genetic circuits. Biophys J, 2015, 109(3): 639–646. DOI: 10.1016/j.bpj.2015.06.034 |
| [18] | Scott M, Gunderson CW, Mateescu EM, et al. Interdependence of cell growth and gene expression: origins and consequences. Science, 2010, 330(6007): 1099–1102. DOI: 10.1126/science.1192588 |
| [19] | You LC, Cox RS Ⅲ, Weiss R, et al. Programmed population control by cell-cell communication and regulated killing. Nature, 2004, 428(6985): 868–871. DOI: 10.1038/nature02491 |
| [20] | Nielsen AA, Segall-Shapiro TH, Voigt CA. Advances in genetic circuit design: novel biochemistries, deep part mining, and precision gene expression. Curr Opin Chem Biol, 2013, 17(6): 878–892. DOI: 10.1016/j.cbpa.2013.10.003 |
| [21] | Burger A, Walczak AM, Wolynes PG. Abduction and asylum in the lives of transcription factors. Proc Natl Acad Sci USA, 2010, 107(9): 4016–4021. DOI: 10.1073/pnas.0915138107 |
| [22] | Lv L, Ren YL, Chen JC, et al. Application of CRISPRi for prokaryotic metabolic engineering involving multiple genes, a case study: controllable P (3HB-co-4HB) biosynthesis. Metab Eng, 2015, 29: 160–168. DOI: 10.1016/j.ymben.2015.03.013 |
| [23] | Nielsen AA, Voigt CA. Multi-input CRISPR/Cas genetic circuits that interface host regulatory networks. Mol Syst Biol, 2014, 10(11): 763. DOI: 10.15252/msb.20145735 |
| [24] | Lee YJ, Hoynes-O'Connor A, Leong MC, et al. Programmable control of bacterial gene expression with the combined CRISPR and antisense RNA system. Nucleic Acids Res, 2016, 44(5): 2462–2473. DOI: 10.1093/nar/gkw056 |
| [25] | Wang HF, Russa ML, Qi LS. CRISPR/Cas9 in genome editing and beyond. Annu Rev Biochem, 2016, 85(1): 227–264. DOI: 10.1146/annurev-biochem-060815-014607 |
| [26] | Cookson NA, Mather WH, Danino T, et al. Queueing up for enzymatic processing: correlated signaling through coupled degradation. Mol Syst Biol, 2011, 7: 561. |
| [27] | Jayanthi S, Nilgiriwala KS, Del Vecchio D. Retroactivity controls the temporal dynamics of gene transcription. ACS Synth Biol, 2013, 2(8): 431–441. DOI: 10.1021/sb300098w |
| [28] | Klumpp S, Zhang ZG, Hwa T. Growth rate-dependent global effects on gene expression in bacteria. Cell, 2009, 139(7): 1366–1375. DOI: 10.1016/j.cell.2009.12.001 |
| [29] | Chuang SE, Daniels DL, Blattner FR. Global regulation of gene expression in Escherichia coli. J Bacteriol, 1993, 175(7): 2026–2036. DOI: 10.1128/jb.175.7.2026-2036.1993 |
| [30] | Zhang HM, Chen SB, Shi HD, et al. Measurements of gene expression at steady state improve the predictability of part assembly. ACS Synth Biol, 2016, 5(3): 269–273. DOI: 10.1021/acssynbio.5b00156 |
| [31] | Ceroni F, Algar R, Stan GB, et al. Quantifying cellular capacity identifies gene expression designs with reduced burden. Nat Methods, 2015, 12(5): 415–418. DOI: 10.1038/nmeth.3339 |
| [32] | Siegal-Gaskins D, Tuza ZA, Kim J, et al. Gene circuit performance characterization and resource usage in a cell-free "breadboard". ACS Synth Biol, 2014, 3(6): 416–425. DOI: 10.1021/sb400203p |
| [33] | Ham TS, Lee SK, Keasling JD, et al. A tightly regulated inducible expression system utilizing the fim inversion recombination switch. Biotechnol Bioeng, 2006, 94(1): 1–4. DOI: 10.1002/(ISSN)1097-0290 |
| [34] | Ham TS, Lee SK, Keasling JD, et al. Design and construction of a double inversion recombination switch for heritable sequential genetic memory. PLoS ONE, 2008, 3(7): e2815. DOI: 10.1371/journal.pone.0002815 |
| [35] | Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli. Nature, 2000, 403(6767): 339–342. DOI: 10.1038/35002131 |
| [36] | Kushwaha M, Salis HM. A portable expression resource for engineering cross-species genetic circuits and pathways. Nat Commun, 2015, 6: 7832. DOI: 10.1038/ncomms8832 |
| [37] | Cameron DE, Collins JJ. Tunable protein degradation in bacteria. Nat Biotechnol, 2014, 32(12): 1276–1281. DOI: 10.1038/nbt.3053 |
| [38] | Mishra D, Rivera PM, Lin A, et al. A load driver device for engineering modularity in biological networks. Nat Biotechnol, 2014, 32(12): 1268–1275. DOI: 10.1038/nbt.3044 |
| [39] | Salis HM, Mirsky EA, Voigt CA. Automated design of synthetic ribosome binding sites to control protein expression. Nat Biotechnol, 2009, 27(10): 946–950. DOI: 10.1038/nbt.1568 |
| [40] | Farasat I, Kushwaha M, Collens J, et al. Efficient search, mapping, and optimization of multi-protein genetic systems in diverse bacteria. Mol Syst Biol, 2014, 10(6): 731. DOI: 10.15252/msb.20134955 |
 2017, Vol. 33
2017, Vol. 33