中国科学院微生物研究所、中国微生物学会主办
文章信息
- 马怡茗, 柯欣, 李晓霞, 舒伟学, 杨文翰, 刘亚勇, 颜霞, 贾良辉, 颜华
- Ma Yiming, Ke Xin, Li Xiaoxia, Shu Weixue, Yang Wenhan, Liu Yayong, Yan Xia, Jia Lianghui, Yan Hua
- 角蛋白酶基因gm2886在密旋链霉菌ACT12中的表达及鉴定
- Expression and characterization of a keratinase encoding gene gm2886 in Streptomyces pactum ACT12 strain
- 生物工程学报, 2017, 33(12): 1968-1978
- Chinese Journal of Biotechnology, 2017, 33(12): 1968-1978
- 10.13345/j.cjb.170081
-
文章历史
- Received: March 1, 2017
- Accepted: April 11, 2017
2 中国科学院微生物研究所,北京 100101
2 Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
角蛋白是一类在自然界中广泛存在的不可溶蛋白,是表皮、羽毛、羊毛、鱼鳞、头发、蹄以及指甲的主要组成部分。由于它紧密交联的氢键、疏水作用以及二硫键的存在,角蛋白相当稳定且难降解,也不易被大部分商品化的蛋白酶如木瓜蛋白酶、胶原酶、胃蛋白酶和胰蛋白酶水解[1-3]。以禽类加工中产生的废弃物羽毛(90%为角蛋白,富含丝氨酸、脯氨酸、赖氨酸、色氨酸、胱氨酸)为例,全世界每年产生约8 000 000 t羽毛废弃物,这些羽毛废弃物常常被掩埋或焚烧,导致严重的环境污染问题,其资源化利用益发显得重要。
角蛋白酶(Keratinase)是一大类可以降解羽毛等不可溶底物和多种蛋白的蛋白水解酶,主要是丝氨酸蛋白酶或者金属蛋白酶类。由于可以特异性分解角蛋白,所以在饲料工业、食品工业、皮革加工、洗涤剂行业、医药工业及环境治理等方面具有广阔的应用前景[4-6],越来越引起研究者的关注。目前在细菌、真菌和放线菌[7]中均已经发现了可以产生角蛋白酶从而降解角蛋白的菌株,其中又以芽胞杆菌属的细菌居多。放线菌可以产生丰富的次级代谢产物,同时,放线菌的生活史有基质菌丝和气生菌丝阶段,这样的特点非常有助于侵入羽毛等硬质角蛋白,从而有助于角蛋白的完全降解,所以角蛋白降解放线菌及其角蛋白酶的研究得到了关注。
我们从家禽养殖场的羽毛堆积废弃物中分离到1株能高效快速降解羽毛的放线菌Fea-10,经16S rDNA测序初步鉴定为微白黄链霉菌Streptomyces albidoflavus。查阅文献发现Verneuil等曾报道微白黄链霉菌K1-02具有很强的角蛋白酶活性,他们纯化得到了角蛋白酶SAKase,并对其N端的31个氨基酸进行了测序[8],但该蛋白的完整编码基因一直未见报道。我们对Fea-10进行全基因组测序,利用BioEdit软件进行本地搜索,发现了极可能编码角蛋白酶的基因gm2886,拟通过异源表达鉴定其表达产物并对表达出的蛋白进行功能研究。
以往角蛋白酶基因的异源表达研究中应用较多的表达宿主有大肠杆菌、枯草芽胞杆菌和毕赤酵母,链霉菌表达系统则较少有人关注。链霉菌GC含量高达70%以上,革兰氏染色阳性,与革兰氏阴性的大肠杆菌显著不同。因此,链霉菌来源的基因更易于在链霉菌宿主中表达。此外,链霉菌有着丰富的外分泌系统,外源基因分泌性表达,可以简化表达产物的分离和纯化过程。因此我们克隆了gm2886,构建了链霉菌表达载体,实现了gm2886的表达纯化,得到重组蛋白GM2886-His6,并对其酶学性质进行了研究。
1 材料 1.1 菌株、质粒、培养基和抗生素菌株:微白黄链霉菌Streptomyces albidoflavus Fea-10、密旋链霉菌Streptomyces pactum ACT12、大肠杆菌Escherichia coli Top10和大肠杆菌Escherichia coli S17-1 (均由本实验室保藏)。质粒:pMD18-T (TaKaRa公司)、pET28a、pIJ8641 (含有红霉素抗性基因启动子PermE)、pSET152 (本实验室保存)。培养基:LB培养基、高氏一号培养基、改良2CMY培养基、2%脱脂牛奶培养基和TSBY培养基。抗生素:Ampicillin (终浓度100 μg/mL),Kanamycin (终浓度50 μg/mL),Apramycin (在LB培养基中终浓度为50 μg/mL,在高氏1号及改良2CMY终浓度为10 μg/mL),萘啶酮酸(终浓度为25 μg/mL)。
1.2 工具酶和引物工具酶:本实验使用的限制性内切酶、T4 DNA连接酶以及Taq酶均购自TaKaRa公司,Pfu高保真酶购自Thermo公司;PCR引物的合成与测序均在生工生物工程(上海)股份有限公司完成。
2 方法 2.1 DNA操作常规的分子生物学操作参照《分子克隆指南》[9],链霉菌基因组的提取参照《链霉菌操作手册》[10]。
2.2 角蛋白酶基因的获取及序列分析根据文献,结合微白黄链霉菌Fea-10的全基因组,利用软件BioEdit进行local blast;序列比对在NCBI上进行,信号肽在http://www.cbs.dtu.dk/services/SignalP/网站上进行预测,蛋白大小及等电点在http://web.expasy.org/compute_pi/网站上进行分析。
2.3 gm2866基因的克隆以及链霉菌表达载体的构建设计引物F1 (带有EcoRⅠ,序列见表 1,下同)和R1 (带有XhoⅠ),以微白黄链霉菌Streptomyces albidoflavus Fea-10基因组DNA为模版,使用Pfu酶PCR扩增获得gm2886的完整基因,使用EcoRⅠ和XhoⅠ双酶切后连入pET28a构建载体pET28a-gm2886 (提供His Tag),测序验证。使用EcoRⅠ和BamHⅠ双酶切pIJ8641获得PermE即片段1。使用引物F2 (带有BamHⅠ和RBS)和R2 (带有XbaⅠ和His Tag),以pET28a-gm2886为模板,PCR扩增,BamHⅠ和XbaⅠ双酶切PCR产物获得片段2。pSET152用EcoRⅠ和XbaⅠ双酶切回收后,和片段1、2共孵育连接,获得带有His6融合标签的链霉菌组成型表达载体pSET152::Perm::gm2886,测序验证。
| Primer name | Primer sequence (5′–3′) | Size (bp) |
| F1 (underline EcoRⅠ) | GTGAATTCGTGAACCACCGACGCATACC | 28 |
| R1 (underline XhoⅠ) | GTCTCGAGTCAGCCGATCTCGACGCC | 26 |
| F2 (underline BamHⅠ) | GTGGATCCAGGAGGACGTGACGTGAACCACCGACGC | 36 |
| R2 (underline XbaⅠ) | GTTCTAGATCAGTGGTGGTGGTGGTGGTG | 29 |
| Underlined letters indicate restriction enzyme digestion sites, Boldface letters indicate RBS in F2 and His tag in R2. | ||
将测序正确的pSET152::Perm::gm2886转入大肠杆菌S17-1,通过大肠杆菌-链霉菌属间接合转移的方法将表达载体转入密旋链霉菌ACT12,筛选阳性接合子。接合子在TSBY培养基发酵96 h,离心除去菌体,将发酵液使用50%的硫酸铵4 ℃沉淀过夜,4 ℃、10 000 r/min离心20 min收集沉淀,沉淀溶解于20 mmol/L Tris-HCl (pH 7.9),0.45 μm的滤膜过滤后,使用Ni柱进行纯化。纯化后的蛋白使用Nanodrop进行浓度测定,并4 ℃透析过夜。
2.5 酶活测定方法使用酪蛋白(Sigma公司)进行重组角蛋白酶酶活的测定,参照Li等[11]和Liu等[12]的方法,并进行了改进。将蛋白样品使用20 mmol/L Tris-HCl (pH 7.9)稀释至终浓度0.012 mg/mL后取1 mL,40 ℃水浴预热2 min,加入1 mL同样预热过的2%酪蛋白溶液,精确反应10 min后立刻加入2 mL 0.4 mol/L TCA终止反应,并继续在40 ℃保温20 min,待残余蛋白沉淀完全后离心取上清。上清液使用0.22 μm的滤膜过滤后取1 mL,加入5 mL 0.4 mol/L碳酸钠以及适当浓度(0.5 mL)的福林试剂,摇匀,40 ℃保温发色20 min,测定OD660。定义每释放出1 μg酪氨酸,酶活升高1个单位。
2.6 温度和pH对酶活影响的测定以酪蛋白为底物,反应温度为30-80 ℃分别测定重组蛋白的酶活,测得重组蛋白的最适反应温度;重组蛋白的温度稳定性分析则通过在最适pH下,30-80 ℃度不同温度处理30 min后,测定酶活;酶最适pH通过测定最适温度下在pH 4.0-12.0的不同缓冲液中的酶活(pH 3.0-6.0,50 mmol/L磷酸二氢钠-柠檬酸钠缓冲液;pH 7.0-8.0,50 mmol/L Tris-HCl缓冲液;pH 9.0-12.0,50 mmol/L甘氨酸-氢氧化钠缓冲液)。每种反应均进行3次重复取平均值。
2.7 金属离子和化学试剂对酶活影响测定以酪蛋白为底物,测定金属离子和化学试剂对纯化的重组蛋白GM2886-His6酶活的影响。分别在4 mL的反应体系中加入5 mmol不同的金属离子(Ca2+、Mg2+、K+)或者不同的化学试剂(5 mmol EDTA、5 mmol PMSF、1% DMSO、1% SDS、1%异丙醇、1% DTT以及1% β-巯基乙醇),按上述酪蛋白作为底物的试验方法进行酶活测定。以未加入任何金属离子和化学试剂时测定的酶活作为对照。每种反应测定3次重复,取平均值。
2.8 不同底物对酶活影响的测定分别以可溶的酪蛋白(Casein)、偶氮酪蛋白(Azo-casein)、牛血清白蛋白(BSA)、血红蛋白(Hemoglobin)和不可溶的天青角蛋白(Azure keratin)、羽毛粉(Feather)为底物测定重组蛋白对不同底物的酶活[13-15]。可溶性底物首先用20 mmol/L Tris-HCl (pH 7.9)配置成2%的溶液。可溶性底物测定酶活的反应总体系为2 mL,体系中含有100 μL纯化的重组蛋白(0.12 mg/mL),1 mL 2%可溶底物溶液,900 μL pH 10.0的50 mmol/L甘氨酸-氢氧化钠缓冲液,50 ℃反应20 min,加入2 mL 0.4 mol/L TCA终止反应,继续置于40 ℃水浴锅保温20 min。离心取上清1 mL,加入1 mL 3%茚三酮溶液和1 mL醋酸缓冲液(pH 4.5),摇匀,置于100 ℃水浴锅10 min,加入3 mL的60%乙醇,摇匀测定OD570。定义每释放出1 μg酪氨酸,酶活升高1个单位。其中偶氮酪蛋白在加入TCA后直接测定上清的OD440。定义OD每升高0.01,酶活增加1个单位。不可溶底物酶活测定的反应总体系为4 mL,其中含有200 μL纯化的GM2886-His6 (0.12 mg/mL),20 mg不可溶底物,10 mmol/L DTT,补50 mmol/L甘氨酸-氢氧化钠缓冲液至总体积为4 mL。37 ℃、180 r/min振摇1 h,天青角蛋白直接测定OD595,羽毛测定OD280。定义OD每升高0.01,酶活增加1个单位。
3 结果与分析 3.1 gm2886编码多肽的氨基酸序列分析及与已知链霉菌角蛋白酶同源性比较利用BioEdit软件在Fea-10基因组内进行本地搜索,遴选可能的角蛋白酶基因,得到候选基因gm2886。gm2886基因全长1 083 bp,GC含量较高,达71%。编码360个氨基酸的多肽,其中包括34个氨基酸的信号肽,138个氨基酸的N-末端前肽以及一个188个氨基酸的成熟蛋白酶结构域。其成熟酶序列与NCBI上已知的角蛋白酶成熟酶序列同源性对比结果如图 1所示。它和灰色链霉菌外泌角蛋白酶SGPD[16]的序列相似度最高,为83%。

|
| 图 1 GM2886与几种已知的链霉菌来源角蛋白酶的成熟酶序列同源性分析 Figure 1 Sequence alignment of mature protein sequence of GM2886 and several other known keratinases. |
| |
重组质粒pSET152::Perm::gm2886转化S17-1并通过接合转移转入密旋链霉菌ACT12后,使用阿普霉素筛选出阳性接合子,并用2%脱脂牛奶平板对接合子进行复筛,以野生型密旋链霉菌ACT12作为对照,出现降解圈即为有蛋白水解活性。结果如图 2所示。

|
| 图 2 密旋链霉菌ACT12/pSET152::Perm::gm2886牛奶平板复筛 Figure 2 Secondary screening of Streptomyces pactum ACT12/pSET152::Perm::gm2886 using milk-agar plate. 1: wild type ACT12; 2: ACT12/pSET152::Perm::gm2886. |
| |
挑取阳性接合子,使用TSBY培养基发酵96 h后,离心取发酵液上清加入等体积的饱和硫酸铵溶液至50%,4 ℃过夜,10 000 r/min、4 ℃离心收集沉淀,将其溶解于20 mmol/L Tris-HCl (pH 7.9)即为粗蛋白液。粗蛋白液过滤后使用镍柱亲和纯化。如图 3所示,发酵液上清中可以纯化到一个大小约为36 kDa的蛋白,说明该蛋白是外泌表达蛋白。

|
| 图 3 纯化到的重组蛋白的SDS-PAGE分析 Figure 3 SDS-PAGE analysis of purified GM2886-His6. 1: premixed protein marker; 2: purified GM2886-His6. |
| |
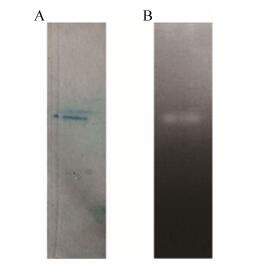
使用酪蛋白为底物进行非变性蛋白电泳,在重组蛋白对应位置可以出现透明条带(图 4),表明该纯化到的蛋白具有蛋白水解活性。
|
| 图 4 以酪蛋白为底物的非变性蛋白电泳分析 Figure 4 Zymogram analysis of proteases excreted by S. pactum ACT12/pSET152::Perm::gm2886. (A) GM2886-His6 stained with Coomassie G-250. (B) GM2886-His6 with casein. |
| |
对纯化到的重组蛋白产量进行测定,得到重组蛋白GM2886-His6在密旋链霉菌ACT12中的异源表达产量为1.108 mg/L。以酪蛋白为底物测得重组蛋白的最大酶活为5.36 kU/mg。
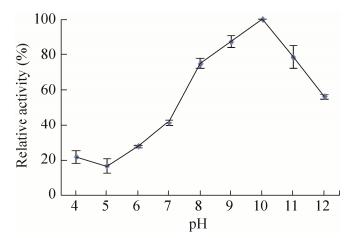
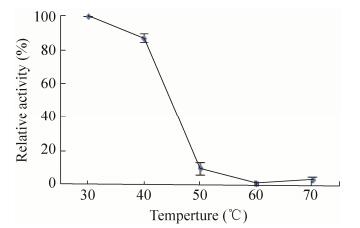
3.4 重组蛋白GM2886-His6最适pH、最适温度及温度稳定性分析以酪蛋白为底物分别测定重组蛋白在不同反应温度下的酶活,结果如图 5所示,在30-70 ℃均有酶活,且在50 ℃时酶活最高,所以50 ℃是重组蛋白GM2886-His6的最适反应温度。分别以酪蛋白为底物测定重组蛋白在不同pH下的酶活,结果如图 6所示,在pH 4.0-11.0时均有酶活,在pH 10.0时酶活最大,所以pH 10.0为重组蛋白GM2886-His6的最适pH。将重组蛋白分别在30-80 ℃下保温30 min后测定以酪蛋白为底物时的酶活,结果如图 7,在30-40 ℃处理下仍有酶活。

|
| 图 5 反应温度对重组角蛋白酶酶活的影响 Figure 5 Effect of temperature on recombinant keratinases. |
| |
|
| 图 6 pH对重组角蛋白酶酶活的影响 Figure 6 Effect of pH on recombinant keratinases. |
| |
|
| 图 7 重组角蛋白酶的温度稳定性 Figure 7 Temperature stability of recombinant keratinases. |
| |
Ca2+、K+和Mg2+对重组蛋白酶活具有抑制作用。DMSO、SDS对重组蛋白酶活具有较弱的抑制作用,EDTA、异丙醇对重组蛋白酶活没有影响,PMSF明显抑制重组蛋白的酶活,说明该重组蛋白为丝氨酸蛋白酶。β-巯基乙醇和DTT使酶活数据极大提高。结果见表 2。
| Metals | Concentrations (mmol) |
Relative activity (%) |
| Control | 100 | |
| Ca2+ | 5 | 64 |
| Mg2+ | 5 | 32 |
| K+ | 5 | 55 |
| Chemicals | Concentrations | Relative activity (%) |
| PMSF | 5 mmol | 66 |
| EDTA | 5 mmol | 100 |
| DMSO | 1% (V/V) | 93 |
| SDS | 1% (V/V) | 91 |
| Isopropanol | 1% (V/V) | 100 |
| β-Mercaptoethanol | 1% (V/V) | 3 477 |
| DTT | 1% (V/V) | 1 266 |
分别测定纯化的GM2886-His6对不同底物的酶活,结果见表 3。重组蛋白可与大部分可溶底物反应,其中酪蛋白为底物时酶活最高。重组蛋白水解天青角蛋白和羽毛,证明该重组蛋白具有角蛋白酶活性。
| Protein substrate | Specific activity (U) | Relative activity (%) |
| Casein | 36.87±0.080 | 100 |
| BSA | 12.64±0.072 | 34.28 |
| Azo-casein | 16.1±0.174 | 43.67 |
| Hemoglobin | 17.52±0.131 | 47.52 |
| Feather | 11.8±0.464 | 32.00 |
| Azure keratin | 20.5±0.954 | 55.60 |
角蛋白酶由于可以特异性地降解角蛋白,在多个领域具有很大的应用前景,近来被人们广泛关注。目前关于角蛋白酶的研究主要集中于角蛋白酶生产菌的筛选与鉴定[17]、角蛋白酶的纯化与表征[18-19]以及角蛋白酶基因的异源表达[14, 20-21]等方面,其中针对地衣芽胞杆菌Bacillus licheniformis[22-21]和绿脓杆菌Pseudomonas aeruginosa[14-15]来源的角蛋白酶的研究较多,链霉菌来源的角蛋白酶研究较少,主要是弗氏链霉菌Streptomyces fradiae sfp2[11-23]等。角蛋白酶异源表达常用宿主为大肠杆菌E. coli[22-21]、芽胞杆菌Bacillus subtilis等[14-20]以及毕赤酵母Pichia pastoris[15-20],使用链霉菌作为表达宿主的并不常见。
实验室前期分离得到一株短时间内即可降解完整羽毛的微白黄链霉菌菌株Fea-10,在对其进行全基因组测序后通过生物信息学方法筛选到了可能的角蛋白酶编码基因gm2886。我们曾经尝试在大肠杆菌表达系统中表达gm2886,可能由于其序列GC含量比较高、大肠杆菌和链霉菌密码子偏好性以及对角蛋白酶的加工处理机制不同等原因而没能得到重组蛋白,因此我们选取了角蛋白酶酶活较弱的密旋链霉菌ACT12作为异源表达的宿主。另外,鉴于角蛋白酶多属于诱导酶,其自身的启动子需要在羽毛等角蛋白的诱导下起始基因的转录,这样限制了培养基的成分和营养,因此本实验使用组成型的红霉素启动子替代gm2886原始启动子,使其不需要通过羽毛的诱导就能进行高效表达,可以使用营养更为丰富的培养基进行发酵,从而增加菌体生长速度和角蛋白酶产量。经过多方改进,最终我们得到了一个大小约为36 kDa的重组蛋白,活性检测表明其具有蛋白酶活性,在还原剂存在的情况下可以高效地降解水不溶性底物天青角蛋白和羽毛粉,说明gm2886确实为角蛋白酶基因,其成熟酶序列和已报道的角蛋白酶的成熟酶序列具有同源性,最高为83%,有一定的研究价值。
gm2886基因的序列分析表明它具有信号肽、前肽和成熟酶3个结构域。前肽是胞外蛋白酶的一个典型结构,被认为参与帮助成熟酶的正确折叠[25-26]。大多数的研究表明,直接表达酶原编码基因,所产生的具有正常生物学活性的表达产物多为经过切除前肽后的成熟酶,如地衣芽胞杆菌kerA[22]和弗氏链霉菌的sfp2[23-24]。gm2886编码的成熟蛋白酶的理论分子量约为20 kDa (加上组氨酸标签),在ACT12中纯化得到的重组蛋白约为36 kDa,表明重组蛋白的前肽可能并没有被前肽酶切割,但在实验室条件下却仍然表现出蛋白酶活性,这值得我们后续对其进一步研究。
在底物特异性实验中,我们发现重组蛋白GM2886-His6需要在还原剂DTT的存在下才可以水解不可溶底物天青角蛋白和羽毛。一般认为,角蛋白水解主要有两个过程,第一步是在还原剂的参与下裂解二硫键,第二步是肽链的水解[27]。许多微生物如高温放线菌CDF[28]、Thermoactinomyces candidus[29]和Bacillus species[5-30]等,均需在活细胞的存在下才可以完全降解羽毛,表明活细胞自身提供必要的还原剂来高效水解二硫键。本实验底物特异性实验的结果验证了在水解角蛋白时,二硫键的裂解是必要的,且需要有还原剂的存在。
综上所述,本实验通过生物信息学分析获得了角蛋白酶编码基因gm2886,实现了其在密旋链霉菌ACT12中的异源表达,纯化得到了具有羽毛粉降解活性的重组蛋白,并对其酶学性质进行了研究。本研究为解析羽毛高效降解菌Fea-10降解角蛋白的分子机理,从而进一步开发其应用潜力提供了基础;并为同类蛋白的研究提供了借鉴。
| [1] | Kublanov IV, Perevalova AA, Slobodkina GB, et al. Biodiversity of thermophilic prokaryotes with hydrolytic activities in hot springs of Uzon Caldera, Kamchatka (Russia). Appl Environ Microbiol, 2009, 75(1): 286–291. DOI: 10.1128/AEM.00607-08 |
| [2] | Son HJ, Park HC, Kim HS, et al. Nutritional regulation of keratinolytic activity in Bacillus pumilis. Biotechnol Lett, 2008, 30(3): 461–465. DOI: 10.1007/s10529-007-9567-3 |
| [3] | Suzuki Y, Tsujimoto Y, Matsui H, et al. Decomposition of extremely hard-to-degrade animal proteins by thermophilic bacteria. J Biosci Bioeng, 2006, 102(2): 73–81. DOI: 10.1263/jbb.102.73 |
| [4] | Gupta R, Rajput R, Sharma R, et al. Biotechnological applications and prospective market of microbial keratinases. Appl Microbiol Biotechnol, 2013, 97(23): 9931–9940. DOI: 10.1007/s00253-013-5292-0 |
| [5] | Gupta R, Ramnani P. Microbial keratinases and their prospective applications: an overview. Appl Microbiol Biotechnol, 2006, 70(1): 21–33. DOI: 10.1007/s00253-005-0239-8 |
| [6] | Macedo AJ, da Silva WO, Gava R, et al. Novel keratinase from Bacillus subtilis S14 exhibiting remarkable dehairing capabilities. Appl Environ Microbiol, 2005, 71(1): 594–596. DOI: 10.1128/AEM.71.1.594-596.2005 |
| [7] | Verma A, Singh H, Anwar S, et al. Microbial keratinases: industrial enzymes with waste management potential. Crit Rev Biotechnol, 2017, 37(4): 476–491. DOI: 10.1080/07388551.2016.1185388 |
| [8] | Bressollier P, Letourneau F, Urdaci M, et al. Purification and characterization of a keratinolytic serine proteinase from Streptomyces albidoflavus. Appl Environ Microbiol, 1999, 65(6): 2570–2576. |
| [9] | Sambrook J, Russel DW. Molecular Cloning: A Laboratory Manual. New York: Cold Spring Harbour Laboratory Press, 2001. |
| [10] | Bibb MJ, Buttner MJ, Chater KF, et al. Practical Streptomyces Genetics. Colney, Norwich: John Innes Foundation, 2000. |
| [11] | Li JX, Chen DD, Yu ZQ, et al. Improvement of expression level of keratinase Sfp2 from Streptomyces fradiae by site-directed mutagenesis of its N-terminal pro-sequence. Biotechnol Lett, 2013, 35(5): 743–749. DOI: 10.1007/s10529-013-1139-0 |
| [12] | Liu BH, Zhang J, Li B, et al. Expression and characterization of extreme alkaline, oxidation-resistant keratinase from Bacillus licheniformis in recombinant Bacillus subtilis WB600 expression system and its application in wool fiber processing. World J Microbiol Biotechnol, 2013, 29(5): 825–832. DOI: 10.1007/s11274-012-1237-5 |
| [13] | Lin HH, Yin LJ, Jiang ST. Cloning, expression, and purification of Pseudomonas aeruginosa keratinase in Escherichia coli AD494(DE3)pLysS expression system. J Agric Food Chem, 2009, 57(9): 3506–3511. DOI: 10.1021/jf803752j |
| [14] | Lin HH, Yin LJ, Jiang ST. Expression and purification of Pseudomonas aeruginosa keratinase in Bacillus subtilis DB104 expression system. J Agric Food Chem, 2009, 57(17): 7779–7784. DOI: 10.1021/jf901903p |
| [15] | Lin HH, Yin LJ, Jiang ST. Functional expression and characterization of keratinase from Pseudomonas aeruginosa in Pichia pastoris. J Agric Food Chem, 2009, 57(12): 5321–5325. DOI: 10.1021/jf900417t |
| [16] | Sidhu SS, Kalmar GB, Willis LG, et al. Protease evolution in Streptomyces griseus discovery of a novel dimeric enzyme. J Biol Chem, 1995, 270(13): 7594–7600. DOI: 10.1074/jbc.270.13.7594 |
| [17] | Yusuf I, Ahmad SA, Phang LY, et al. Keratinase production and biodegradation of polluted secondary chicken feather wastes by a newly isolated multi heavy metal tolerant bacterium-Alcaligenes sp. AQ05-001. J Environ Manage, 2016, 183: 182–195. DOI: 10.1016/j.jenvman.2016.08.059 |
| [18] | Elhoul MB, Jaouadi NZ, Rekik H, et al. Biochemical and molecular characterization of new keratinoytic protease from Actinomadura viridilutea DZ50. Int J Biol Macromol, 2016, 92: 299–315. DOI: 10.1016/j.ijbiomac.2016.07.009 |
| [19] | Bouacem K, Bouanane-Darenfed A, Jaouadi N Z, et al. Novel serine keratinase from Caldicoprobacter algeriensis exhibiting outstanding hide dehairing abilities. Int J Biol Macromol, 2016, 86: 321–328. DOI: 10.1016/j.ijbiomac.2016.01.074 |
| [20] | Radha S, Gunasekaran P. Purification and characterization of keratinase from recombinant Pichia and Bacillus strains. Prot Exp Purif, 2009, 64(1): 24–31. DOI: 10.1016/j.pep.2008.10.008 |
| [21] | Tiwary E, Gupta R. Extracellular expression of keratinase from Bacillus licheniformis ER-15 in Escherichia coli. J Agric Food Chem, 2010, 58(14): 8380–8385. DOI: 10.1021/jf100803g |
| [22] | Hu H, He J, Yu B, et al. Expression of a keratinase (kerA) gene from Bacillus licheniformis in Escherichia coli and characterization of the recombinant enzymes. Biotechnol Lett, 2013, 35(2): 239–244. DOI: 10.1007/s10529-012-1064-7 |
| [23] | Li J, Shi PJ, Han XY, et al. Functional expression of the keratinolytic serine protease gene sfp2 from Streptomyces fradiae var. k11 in Pichia pastoris. Prot Exp Purif, 2007, 54(1): 79–86. DOI: 10.1016/j.pep.2007.02.012 |
| [24] |
Li J, Shi PJ, Zhang WZ, et al. Gene cloning and expression of serine protease SFP2 from Streptomyces fradiae var. k11.
Chin J Biotech, 2005, 21(5): 782–788.
(in Chinese). 李江, 石鹏君, 张王照, 等. 弗氏链霉菌丝氨酸蛋白酶基因的克隆及表达. 生物工程学报, 2005, 21(5): 782-788. |
| [25] | Baardsnes J, Sidhu S, MacLeod A, et al. Streptomyces griseus protease B: secretion correlates with the length of the propeptide. J Bacteriol, 1998, 180(12): 3241–3244. |
| [26] | Baker D, Silen JL, Agard DA. Protease pro region required for folding is a potent inhibitor of the mature enzyme. Proteins, 1992, 12(4): 339–344. DOI: 10.1002/(ISSN)1097-0134 |
| [27] | Brandelli A, Daroit DJ, Riffel A. Biochemical features of microbial keratinases and their production and applications. Appl Microbiol Biotechnol, 2010, 85(6): 1735–1750. DOI: 10.1007/s00253-009-2398-5 |
| [28] | Wang LY, Cheng GY, Ren YX, et al. Degradation of intact chicken feathers by Thermoactinomyces sp. CDF and characterization of its keratinolytic protease. Appl Microbiol Biotechnol, 2015, 99(9): 3949–3959. DOI: 10.1007/s00253-014-6207-4 |
| [29] | Ignatova Z, Gousterova A, Spassov G, et al. Isolation and partial characterisation of extracellular keratinase from a wool degrading thermophilic actinomycete strain Thermoactinomyces candidus. Can J Microbiol, 1999, 45(3): 217–222. DOI: 10.1139/w98-230 |
| [30] | Ramnani P, Singh R, Gupta R. Keratinolytic potential of Bacillus licheniformis RG1: structural and biochemical mechanism of feather degradation. Can J Microbiol, 2005, 51(3): 191–196. DOI: 10.1139/w04-123 |
 2017, Vol. 33
2017, Vol. 33




