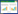中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李恺, 宋雷, 石文昊, 田喜凤
- Li Kai, Song Lei, Shi Wenhao, Tian Xifeng
- 双反相色谱串联平行反应监测靶向定量蛋白质组方法的建立
- Establishment of dual reverse phase chromatography combined parallel reaction monitoring for targeted quantitative proteomics
- 生物工程学报, 2017, 33(11): 1859-1868
- Chinese Journal of Biotechnology, 2017, 33(11): 1859-1868
- 10.13345/j.cjb.170035
-
文章历史
- Received: February 8, 2017
- Accepted: March 28, 2017
2 军事医学科学院放射与辐射医学研究所 国家蛋白质科学中心·北京 北京蛋白质组研究中心 蛋白质组学国家重点实验室,北京 102206;
3 河北省慢性疾病重点实验室 唐山市慢性病临床基础研究重点实验室,河北 唐山 063000
2 State Key Laboratory of Proteomics, Beijing Proteome Research Center, National Center for Protein Sciences (The PHOENIX center, Beijing), Beijing Institute of Radiation Medicine, Beijing 102206, China;
3 Hebei Key Laboratory for Chronic Diseases, Tangshan Key Laboratory for Preclinical and Basic Research on Chronic Diseases, Tangshan 063000, Hebei, China
近年来随着质谱技术的快速发展,蛋白质组学越来越多地应用于各个领域。数据依赖性采集方法(Data dependent acquisition,DDA)是蛋白质组学无标定量的常用技术手段,该方法采用的是基于一级色谱峰的定量方式,定量的深度不足,尤其是对低丰度蛋白定量准确性差。Ding等[1]通过结合双反相色谱正交法,即pH 10的反相色谱降低样本复杂程度[2]后利用pH 3的反相色谱串联高精度质谱仪进行样本鉴定[3],通过缩短洗脱梯度等提升了质谱检测深度,使得质谱检测效率大大提高。
多反应监测(Multiple reaction monitoring,MRM)蛋白靶向定量方法得到广泛关注[4-5],并有取代实验室蛋白定量黄金标准Western blotting方法的势头[6]。该方法在一定时间窗口内对预设的一系列母离子-子离子对进行定量检测[7-8],检测结果在一定动态范围内拥有较高的准确性。随着蛋白质组学定量分析对通量要求的提升,MRM的灵敏度和分辨率成为离子对检测通量的限制因素[9]。此外,基于三重四极杆的MRM方法,在高复杂性样本检测过程中由于分辨率偏低对共洗脱肽段无法进行准确区分,容易造成目标肽段定量结果的假阳性[10-12]。MRM方法中离子对选择[13]、碰撞能量等条件优化过程繁琐,难以满足生物标志物和系统生物学等研究中靶向定量蛋白质组的高通量需求[14-15]。
在大规模靶向定量分析的实际应用中,基于MRM发展而来的平行反应监测(Parallel reaction monitoring,PRM)蛋白靶向定量技术不需筛选碎片离子,利用高分辨的轨道离子阱对所有碎片离子进行检测,降低了干扰离子对结果的影响。PRM方法能够在高通量的检测过程中保证定量的灵敏度和特异性[16-17]。Gallien等利用60 min有效梯度鉴定了酵母全蛋白裂解液中的770条肽段[18];Kim等将PRM技术用于酪氨酸激酶的大规模定量分析,最终在50 min有效梯度内准确定量了83种酪氨酸激酶受体的308条肽段[19]。在翻译后修饰的研究当中Tsuchiya等[20]和Tang等[21]均利用PRM技术进行监测和定量。近期在羊绒分子标志物鉴定[22]和单克隆抗体药物的表征研究[23]中均利用了PRM检测技术。总之,PRM渐渐成为靶向蛋白质组学研究中广泛应用的方法之一。
质谱技术的发展使得仪器灵敏度、分辨率等均有提升,但对于高复杂性样本的鉴定效率无明显提高,Shishkova等通过对比近年已发表文章的质谱数据,发现色谱条件可能是潜在的制约因素[24]。为了提高PRM靶向定量检测的通量和深度、准确性以及重复性,我们建立了一种简单高效的双反相色谱串联PRM靶向蛋白质组定量平台。相对于传统的定量方法,我们主要优化了PRM定量的色谱方法,包括:1)色谱柱的优化:增加色谱柱内径、降低柱长;2)色谱洗脱条件的优化;3)提高液相洗脱流速、缩短洗脱时间。该研究提高了PRM蛋白质组靶向定量的通量和深度,为其更广泛的应用提供了技术基础。
1 材料与方法 1.1 仪器、试剂与材料UltiMate 3000纳升级液相色谱仪和Q Exactive HF质谱仪(美国Thermo Fisher公司)。
质谱级水、乙腈、甲醇(J.T.Baker公司),1.9 μm C18粉末(Dr. Maisch HPLC GmbH),3 μm C18粉末(天津博纳艾杰尔科技有限公司),C18膜(固相萃取片,3M公司),二硫苏糖醇(Dithiothreitol,DTT,纯度≥99.5%,Sigma-Aldrich公司),质谱级甲酸(Formic acid,FA,~98%,SigmaFluka公司),脱氧胆酸钠(Sodium deoxycholate,DOC,国药集团化学试剂有限公司),质谱级胰蛋白酶(Trypsin Gold,Promega公司),0.5-20 μL移液吸头(T-400,Axygen公司)。
1.2 标准液的配制细胞裂解液为50 mmol/L碳酸氢铵,体积分数2%的脱氧胆酸钠,25 mmol/L氯化钠,pH 8.5。精确称取一定量的DTT配制成100 mmol/L的DTT溶液,需现用现配。用水作为溶剂配制10 mmol/L的碳酸氢铵(pH 10)。用水和甲酸配制体积分数为0.1%和1%的FA溶液。利用乙腈和10 mmol/L碳酸氢铵配制乙腈体积分数为6%、9%、12%、15%、18%、21%、25%、30%和35%的反相分离流动相。
1.3 293T细胞样本制备新鲜培养的293T细胞100 μL,加入500 μL细胞裂解液混匀并超声。每50 μL裂解产物加入1 μL的100 mmol/L DTT,95 ℃加热3 min,恢复室温后进行两次酶切(1/50用量,6 h和过夜)。加入150 μL 0.1% FA离心,取上清溶液进行真空干燥。自制C18反相分离柱,以T-400移液吸头为容器,双层直径1 mm的C18膜对吸头封口,填入耐高pH的3 μm粒径C18粉末3 mg,利用0.1% FA对样品脱盐,利用反相分离流动相洗脱对酶切产物预分离,脱盐及预分离的馏分真空干燥后保存在-20 ℃,待用。
1.4 大肠杆菌样本制备制备过程同1.3。
1.5 色谱方法色谱柱使用实验室自制分析柱(规格为1.9 μm C18,120 mm×150 μm,80 mm×150 μm),流动相A为水(含体积分数为0.2%的FA),流动相B为体积分数80%的乙腈(含体积分数为0.2%的FA)。70 min有效梯度:16 min,9%-14% B;36 min,14%-32% B;15 min,32%-40% B;5 min,40%-95% B并持续5 min。35 min有效梯度:22 min,6%-25% B;12 min,25%-45% B;1 min,45%-95% B并持续5 min。样品复溶溶液为体积分数5%甲醇水溶液(含体积分数为0.2%的FA)。
1.6 质谱方法质谱使用电喷雾离子源(Electrospray ionization,ESI源),正离子采集模式,离子传输管温度为320 ℃,喷雾电压2 000 V。不同检测模式下质谱参数详见表 1。
| Data dependent acquisition | Parallel reaction monitoring | |||
| MS1 | MS2 | MS1 | PRM | |
| Resolution | 120 000 | 15 000 | 15 000 | 15 000 |
| AGC target | 3e6 | 2e4 | 5e5 | 1e5 |
| Maximum IT | 80 ms | 40 ms | 80 ms | 40 ms |
| Isolation window | - | 1.6 m/z | - | 1 m/z |
| NCE/stepped | - | 27 | - | 27 |
| Dynamic exclusion | 12 s | - | ||
| Top N | Top 30 | 20 | ||
| Time window | - | 1.5 min | ||
| -Not covered. | ||||
DDA采集数据利用MaxQuant软件进行数据搜库,所用蛋白序列数据库来自NCBI网站,更新于2013年7月1日。参数设定为默认参数,母离子质量精度10 (Parts per million,ppm),子离子质量精度20 ppm。PRM数据使用Proteome Discoverer 1.4软件进行搜库鉴定,母离子质量精度:20 ppm,子离子质量精度:50 (Milli-mass units,mmu)。利用Skyline软件进行二级定量抽提。母离子电荷数目设定为2和3,质量偏差设定为0.05 m/z。抽提时间窗口设定为0.2 min。利用Top 5的碎片离子面积加和代表肽段定量结果。
2 结果与讨论 2.1 色谱柱和洗脱梯度条件测试首先检测了293T全细胞提取物样本,在70 min有效梯度内,8 cm色谱柱检测蛋白数为4 295,肽段数为23 709,与12 cm色谱柱的4 455个蛋白和24 369条肽段相比,基本相当。该结果说明8 cm色谱柱能够对复杂生物样品提供足够的分离能力。同时短色谱柱能够承受更高流速,色谱条件可优化参数范围更广。
质谱检测效率与洗脱梯度时长紧密相关,缩短洗脱梯度减少了检测时长,但加重肽段共洗脱情况,影响质谱检测深度。为了权衡洗脱梯度与检测深度,我们利用预分离的293T样本,分别用35 min和70 min有效洗脱梯度进行深度覆盖检测。结果表明,前者210 min有效检测时长鉴定到6 916种蛋白和52 760条肽段,与后者420 min有效检测时长的7 126种蛋白和56 563条肽段鉴定结果相比,蛋白水平无差别,肽段水平有一定程度降低(图 1A),这说明色谱梯度缩短并未对蛋白鉴定深度造成明显影响,但缩短洗脱梯度后质谱检测效率在蛋白和肽段水平提升明显(图 1B)。综合上述结果,缩短色谱柱长和洗脱梯度后,能够满足预分离样本的分离能力需求,明显提高质谱的检测效率。
|
| 图 1 不同色谱条件下293T预分离样本鉴定结果对比(A)和检测效率对比(B) Figure 1 Evaluation of different chromatographic conditions. (A) Comparison of qualitative results of 293T pre-separation samples with different chromatographic gradient. (B) Comparison of detection efficiencies with different chromatographic gradient. |
| |
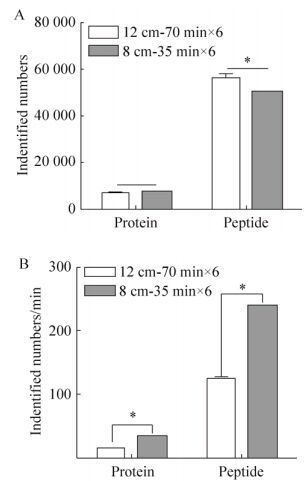
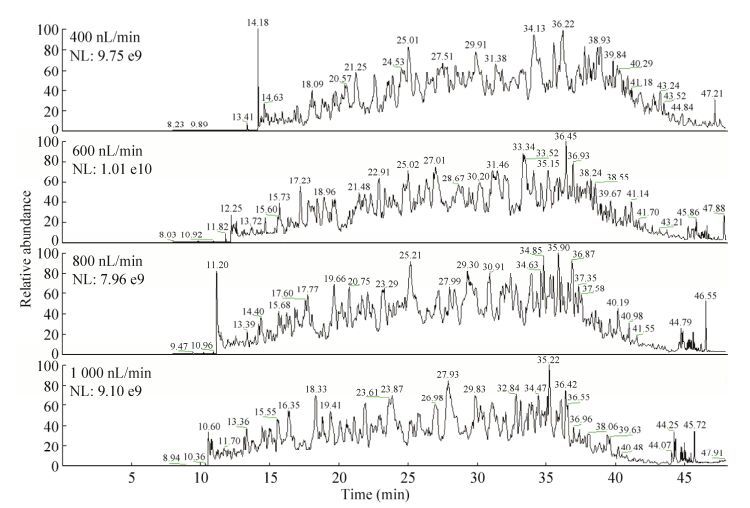
Shishkova等的研究中[24],常用色谱条件为75 μm内径色谱柱,1.7 μm或1.9 μm粒径C18填料,柱长10-50 cm,250-350 nL/min的洗脱流速。为了达到理想分离效果,需使用耐高压设备和色谱柱加热装置等,增加检测平台的搭建难度。本研究通过使用150 μm内径的毛细管一体柱填充1.9 μm粒径的C18粉末,填充长度为8 cm,为保证色谱系统在(低于200 bar)低压力条件下即可达到理想分离效果,我们比较了400、600、800、1 000 nL/min四种流速下293T预分离后的单一组分样本检测结果。四种流速下有效梯度时长无明显变化,但相对于1 000 nL/min的流速条件,其他流速条件下肽段保留时间(Retention time,RT)整体依次延后4.7 min、2.2 min和0.8 min,其中800 nL/min的流速使样本更好地分布在洗脱梯度中央(图 2);4种流速的鉴定结果在蛋白和肽段水平上无明显差别(表 2);鉴定肽段的总离子流(Total ion current,TIC),前两种低流速条件明显提高(图 3A)。为了探究TIC变化的原因,我们对不同流速条件下鉴定肽段进行丰度分布统计。统计结果发现,(e5-e6)和(e9-e10)范围内肽段数目占总数的0.6%左右,不影响整体结果,高流速条件下低丰度肽段(e6-e7)数目明显增多,而对于中高丰度肽段(e7-e8,e8-e9)数目减少(图 3B)。这说明流速的提高对整体定性结果无明显影响,但对低丰度肽段检测的灵敏度有所提高,这是高流速条件的潜在优势之一。同时考虑到高流速会造成系统压力升高,综合分析后选用800 nL/min流速。
|
| 图 2 不同洗脱流速条件时色谱峰分布 Figure 2 Separation of chromatographic peaks with different flow rates. |
| |
| 400 nL/min | 600 nL/min | 800 nL/min | 1 000 nL/min | |
| Number of identified proteins | 3 580/3 764/3 728 | 3 561/3 692/3 765 | 3 589/3 665/3 659 | 3 536/3 624/3 643 |
| Number of identified peptides | 8 859/8 666/8 900 | 8 648/8 989/8 609 | 8 688/8 600/8 326 | 8 495/8 598/8 337 |
|
| 图 3 不同流速条件下TIC对比(A)和肽段丰度分布对比(B) Figure 3 Evaluation of different flow rates. (A) Comparison of total ion current (TIC). (B) Comparison of peptide intensity distribution. |
| |
PRM检测涉及的主要参数中,扫描分辨率、自动增益控制(Automatic gain control,AGC)、目标离子注入时间、扫描周期时间以及目标肽段时间窗口都能够直接影响检测效果。通过调整各项参数,最终确定了相对最优的PRM检测参数(表 1)。
在参数调整过程中发现,当肽段时间窗口放大,肽段检测比例及扫描点数明显减少,而时间窗口大小直接受肽段RT偏移影响。将不同梯度时长的数据进行RT偏移分析,利用偏移时间与相应有效梯度时长之比作为标准化后的偏移率(图 4),结果显示短梯度(35 min有效梯度)相对于长梯度(70 min有效梯度)肽段RT偏移率更低且色谱表现更稳定。证明本研究所优化后的色谱体系不仅能够保证色谱分离能力,提高质谱的检测效率,还能够为PRM靶向定量检测提供一个更加稳定的色谱条件,有利于提高PRM的通量。

|
| 图 4 不同色谱条件下RT偏移情况对比. (A) RT偏移情况(B)标准化的RT偏移 Figure 4 Evaluation of RT shift with different chromatographic conditions. (A) Distribution of RT shift; (B) Normalized RT shift. |
| |
首先,我们对293T预分离样本采用35 min有效梯度进行深度覆盖检测;其次,在深度覆盖鉴定到的6 870蛋白结果中,保留拥有2条以上特异性肽段的蛋白,按照蛋白丰度进行排序,选取丰度最低的1 000种蛋白作为靶向定量的目标蛋白;最后,我们筛选各组分中对应目标蛋白的肽段进行PRM定量分析。肽段筛选条件包括:无修饰,无漏切,肽段长度8-25个氨基酸,图谱评分(Ion score)不低于20,所带电荷数目为2或3等条件。各组分最终分别筛选出337、305、283、335、323和330条目的肽段。最终PRM定量结果中对目的肽段的鉴定率均在93%以上,个别组分高达99%。如果放宽肽段筛选条件,我们在第6个组分中共筛选出了501条目标肽段,其鉴定率为97%。
随后对PRM定量结果进行了评估,包括对低丰度肽段定量的准确性(定义二级碎片离子对应点数大于7为可定量肽段)与稳定性(重复检测的变异系数,Coefficient of variation,CV)。碎片离子对应点数=肽段峰宽(通过Skyline软件抽提获得)/扫描周期时长(通过计算相邻一级谱图时间之差获得)。计算后,扫描周期在1.35 s左右,抽提点数中位数在10个点,CV中位数在10%以内(表 3)。说明优化过后的PRM检测体系能够在35 min有效梯度内对高达400条低丰度肽段进行快速准确定量。
| Fraction | F1 | F2 | F3 | F4 | F5 | F6 | F6-More |
| Number of target peptides | 337 | 305 | 283 | 335 | 323 | 330 | 501 |
| Number of identified peptides | 329 (97%) | 303 (99%) | 278 (98%) | 313 (93%) | 319 (99%) | 327 (99%) | 487 (97%) |
| Number of quantitative peptides (points > 7) | 314 (93%) | 278 (91%) | 260 (92%) | 299 (89%) | 305 (94%) | 315 (95%) | 445 (89%) |
| Median CV | 9% | 9% | 6% | 8% | 5% | 9% |
对150种低丰度目标蛋白(400条肽段)的结果进行分析发现:1)修饰肽段的检测比例42%,远低于无修饰肽段的检测比例95%,挑选肽段应优先挑选非修饰肽段;2)肽段所带电荷数目为2 (占比60%)或3 (占比40%),检测比例无明显差别;3)目标肽段长度在7-15个氨基酸(占比57%)、16-25个氨基酸(占比36%)和26-34个氨基酸(占比7%),三者被检测比例相当,均为92%,氨基酸序列长短对检测效率无明显影响;4)对全部目标肽段的RT分布情况进行观察,发现在洗脱时间内肽段分布均匀;5)在Skyline抽提结果中,Top 5的碎片离子以y离子居多,占碎片离子总数的80%左右,这种结果应该与PRM检测时选用高能碰撞破碎(High-energy collision-induced dissociation,HCD)模式有关,最终出现y离子的偏向性。
为了进一步验证本研究所优化的PRM检测体系的准确性与稳定性,我们进行了倍比稀释定量实验。选取293T双反相预分离后的单一组分样本,按照1:3稀释后用大肠杆菌蛋白作为背景补齐总量。配合实验室先前研究中所创建的肽段线性情况评分数据库筛选出300条目标肽段,覆盖高中低各丰度蛋白。通过比较DDA和PRM的定量结果,发现PRM定量结果中,定量倍数结果以3倍为中心呈正态分布,且77%的肽段倍数在2.5-3.5倍之间;而DDA定量结果中偏离3倍的肽段数量明显偏高(图 5A)。这说明优化后的PRM检测体系在定量上更加准确。

|
| 图 5 倍数稀释实验目标肽段PRM定量结果(A:PRM与DDA定量比例分布情况对比;B:PRM重复定量CV值) Figure 5 PRM targeted peptide quantification results of rate dilution test. (A) Comparison of peptide ratio distribution from PRM and DDA quantification. (B) CV of PRM quantitative repeatability. |
| |
随后对定量肽段的CV值进行了统计。300条肽段中95%的肽段CV稳定在20%以内,且以7%为中心呈正态分布(图 5B)。定量稳定性的确定,需根据具体实验情况进行CV选取,若肽段丰度高可以选取5%,若丰度极低或通量高,选择20%的情况也会存在[25-26]。说明本实验肽段鉴定稳定性在可接受范围。
综上所述,本研究通过双反相高效液相色谱串联PRM靶向定量技术,能够在35 min有效梯度内对150种低丰度目标蛋白(400条肽段)进行快速准确定量,对于不同丰度的蛋白同样能够在单一检测中实现快速准确定量。
3 结论通过质谱参数的优化能够实现PRM检测灵敏度的提升,但高复杂性样本中共洗脱肽段的干扰是质谱靶向定量检测时的主要限制因素,因此PRM技术需要更加高效稳定的色谱系统,这也是该研究的初衷。本研究所创建的双反相高效液相色谱串联PRM靶向定量检测系统,通过降低色谱柱柱长、提高液相洗脱流速、缩短色谱洗脱梯度等方法,提高了色谱系统的稳定性,能够在35 min有效梯度内完成高达400条低丰度肽段的快速准确定量。同时,该系统在相对较低的液相压力条件下也具有理想分离效果,避免了耐高压设备以及色谱柱加热装置等苛刻条件,使检测平台更易搭建。相信更高效、更精准的蛋白质组靶向定量平台的建立,将为进一步推动蛋白质组学从基础研究走向临床转化奠定基础。
| [1] | Ding C, Jiang J, Wei JY, et al. A fast workflow for identification and quantification of proteomes. Mol Cell Proteomics, 2013, 12(8): 2370–2380. DOI: 10.1074/mcp.O112.025023 |
| [2] | Wiczling P, Markuszewski MJ, Kaliszan M, et al. pH/organic solvent double-gradient reversed-phase HPLC. Anal Chem, 2005, 77(2): 449–458. DOI: 10.1021/ac049092r |
| [3] | Delmotte N, Lasaosa M, Tholey A, et al. Two-dimensional reversed-phase × ion-pair reversed-phase HPLC: an alternative approach to high-resolution peptide separation for shotgun proteome analysis. J Proteome Res, 2007, 6(11): 4363–4373. DOI: 10.1021/pr070424t |
| [4] |
Wang JF, Zhao XY, Zhao Y, et al. Influence of overalkylation in enzymatic digestion on the qualitative and quantitative analysis of proteins.
Chin J Chromatogr, 2013, 31(10): 927–933.
(in Chinese). 王继峰, 赵新元, 赵焱, 等. 酶切过程中肽段过烷基化对蛋白质定性和定量分析的影响. 色谱, 2013, 31(10): 927-933. |
| [5] |
Mao XL, Wei JY, Niu M, et al. Establishment of retention time-dependent intelligent selected reaction monitoring mass spectrometric method and its application.
Chin J Chromatogr, 2012, 30(2): 170–177.
(in Chinese). 毛心丽, 卫军营, 牛明, 等. 依赖色谱保留时间的智能化选择反应监测质谱方法的建立及其初步应用. 色谱, 2012, 30(2): 170-177. |
| [6] | Aebersold R, Burlingame AL, Bradshaw RA. Western blots versus selected reaction monitoring assays: time to turn the tables?. Mol Cell Proteomics, 2013, 12(9): 2381–2382. DOI: 10.1074/mcp.E113.031658 |
| [7] | Lange V, Picotti P, Domon B, et al. Selected reaction monitoring for quantitative proteomics: a tutorial. Mol Syst Biol, 2008, 4: 222. |
| [8] | Gallien S, Duriez E, Domon B. Selected reaction monitoring applied to proteomics. J Mass Spectrom, 2011, 46(3): 298–312. DOI: 10.1002/jms.v46.3 |
| [9] | Kiyonami R, Schoen A, Prakash A, et al. Increased selectivity, analytical precision, and throughput in targeted proteomics. Mol Cell Proteomics, 2011, 10(2): M110.002931. DOI: 10.1074/mcp.M110.002931 |
| [10] | Gallien S, Duriez E, Demeure K, et al. Selectivity of LC-MS/MS analysis: implication for proteomics experiments. J Proteomics, 2013, 81: 148–158. DOI: 10.1016/j.jprot.2012.11.005 |
| [11] | Abbatiello SE, Mani DR, Keshishian H, et al. Automated detection of inaccurate and imprecise transitions in peptide quantification by multiple reaction monitoring mass spectrometry. Clin Chem, 2010, 56(2): 291–305. DOI: 10.1373/clinchem.2009.138420 |
| [12] | Sherman J, McKay MJ, Ashman K, et al. How specific is my SRM?: the issue of precursor and product ion redundancy. Proteomics, 2009, 9(5): 1120–1123. DOI: 10.1002/pmic.v9:5 |
| [13] | Karlsson C, Malmström L, Aebersold R, et al. Proteome-wide selected reaction monitoring assays for the human pathogen Streptococcus pyogenes. Nat Commun, 2012, 3: 1301. DOI: 10.1038/ncomms2297 |
| [14] | Picotti P, Bodenmiller B, Mueller LN, et al. Full dynamic range proteome analysis of S. cerevisiae by targeted proteomics. Cell, 2009, 138(4): 795–806. DOI: 10.1016/j.cell.2009.05.051 |
| [15] | Cima I, Schiess R, Wild P, et al. Cancer genetics-guided discovery of serum biomarker signatures for diagnosis and prognosis of prostate cancer. Proc Natl Acad Sci USA, 2011, 108(8): 3342–3347. DOI: 10.1073/pnas.1013699108 |
| [16] | Peterson AC, Russell JD, Bailey DJ, et al. Parallel reaction monitoring for high resolution and high mass accuracy quantitative, targeted proteomics. Mol Cell Proteomics, 2012, 11(11): 1475–1488. DOI: 10.1074/mcp.O112.020131 |
| [17] | Schiffmann C, Hansen R, Baumann S, et al. Comparison of targeted peptide quantification assays for reductive dehalogenases by selective reaction monitoring (SRM) and precursor reaction monitoring (PRM). Anal Bioanal Chem, 2014, 406(1): 283–291. DOI: 10.1007/s00216-013-7451-7 |
| [18] | Gallien S, Duriez E, Crone C, et al. Targeted proteomic quantification on quadrupole-orbitrap mass spectrometer. Mol Cell Proteomics, 2012, 11(12): 1709–1723. DOI: 10.1074/mcp.O112.019802 |
| [19] | Kim HJ, Lin D, Li M, et al. Quantitative profiling of protein tyrosine kinases in human cancer cell lines by multiplexed parallel reaction monitoring assays. Mol Cell Proteomics, 2015, 14(12): 218–224. |
| [20] | Tsuchiya H, Tanaka K, Saeki Y. The parallel reaction monitoring method contributes to an highly sensitive polyubiquitin chain quantification. Biochem Biophys Res Commun, 2013, 436(2): 223–229. DOI: 10.1016/j.bbrc.2013.05.080 |
| [21] | Tang H, Fang HS, Yin E, et al. Multiplexed parallel reaction monitoring targeting histone modifications on the QExactive mass spectrometer. Anal Chem, 2014, 86(11): 5526–5534. DOI: 10.1021/ac500972x |
| [22] |
Yang AF, Li SS, Zhang Y, et al. Developing targeted proteomic technique for identification and quantification of cashmere fibers.
China Science Pap, 2016, 11(6): 663–669.
(in Chinese). 杨阿芳, 李珊珊, 张勇, 等. 基于发展靶向蛋白质组技术的羊绒纤维组成鉴定及含量分析. 中国科技论文, 2016, 11(6): 663-669. |
| [23] |
Cong YT. Method development for the analysis of therapeutic monoclonal antibody and its glycosylation by mass spectrometry[D]. Changchun: Jilin University, 2016 (in Chinese). 丛宇婷. 单克隆抗体药物及其糖基化修饰的质谱分析方法研究[D]. 长春: 吉林大学, 2016. |
| [24] | Shishkova E, Hebert AS, Coon JJ. Now, more than ever, proteomics needs better chromatography. Cell Syst, 2016, 3(4): 321–324. DOI: 10.1016/j.cels.2016.10.007 |
| [25] | Zhou JT, Liu HY, Liu Y, et al. Development and evaluation of a parallel reaction monitoring strategy for large-scale targeted metabolomics quantification. Anal Chem, 2016, 88(8): 4478–4486. DOI: 10.1021/acs.analchem.6b00355 |
| [26] | Bourmaud A, Gallien S, Domon B. Parallel reaction monitoring using quadrupole-orbitrap mass spectrometer: principle and applications. Proteomics, 2016, 16(15/16): 2146–2159. |
 2017, Vol. 33
2017, Vol. 33