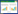中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王远立, 啜国晖, 闫继芳, 石雷, 刘琦
- Wang Yuanli, Chuai Guohui, Yan Jifang, Shi Lei, Liu Qi
- 计算机辅助CRISPR向导RNA设计
- In silico CRISPR-based sgRNA design
- 生物工程学报, 2017, 33(10): 1744-1756
- Chinese Journal of Biotechnology, 2017, 33(10): 1744-1756
- 10.13345/j.cjb.170182
-
文章历史
- Received: May 5, 2017
- Accepted: July 21, 2017
2 合肥工业大学 计算机与信息学院, 安徽 合肥 230009
2 School of Computer and Information, Hefei University of Technology, Hefei 230009, Anhui, China
基于CRISPR系统的基因编辑已经被广泛应用于各种类型的细胞和生物体中[1-5]。该技术主要包括以下类型的基因定点修饰技术:基因敲除(Gene knockout,KO)、基因敲入(Gene knockin,KI)和对基因表达的抑制或激活(CRISPRi/a)[4, 6]。在将CRISPR/Cas9核酸内切酶系统应用到各种基因定点修饰技术的过程中,主要的挑战之一是需要设计高效的sgRNA,并且降低其脱靶风险。合适的外源sgRNA可以通过定位前间区序列邻近基序(Protospacer adjacent motif,PAM)来引导Cas9蛋白到达靶特异性的DNA位点进行切割,其类型由所使用的特定Cas9蛋白质确定(图 1)[5-6]。目前最常用的Cas9是具有PAM NGG的化脓性链球菌(SpyCas9)。同时,根据不同的Cas9物种或变体,研究者先后鉴别出了一些其他种类的PAM序列(图 1)[6-8]。基于CRISPR的基因切割是通过使用针对目标基因敲除的非同源末端连接(Nonhomologous end-joining,NHEJ) DNA修复来引入突变,或是通过使用外源DNA供体模板来触发基因敲入的内生的同源介导双链DNA修复(Homology-directed repair,HDR)来实现的[2]。

|
| 图 1 计算机辅助的sgRNA设计系统可以被大致分为3种类型[69]. (1) 基于序列比对的方法; (2) 基于假设先验的方法; (3) 基于学习的方法.图的上半部分列举了被不同种类Cas9识别的特定类型的PAM Figure 1 In silico sgRNA design based on computational techniques. There are three main in silico sgRNA design systems: (1) alignment-based; (2) hypothesis-driven; (3) learning-based systems. The distinct types of protospacer adjacent motif (PAM) sequences identified for different Cas9 species or variants are listed in the upper part of the figure[69]. |
| |
随着CRISPR技术的迅速发展,研究者积累了大量的基因组编辑数据,若干具有挑战性的计算问题也由此产生。计算机辅助的sgRNA设计已成为基因编辑实验中的关键问题。目前OMIC Tools工具库已经包含多种可用于CRISPR基因编辑的计算工具(http://omictools.com/crispr-cas9-category)[9],但在该领域并没有对现有计算工具的系统总结。因此,本综述将帮助研究者了解和掌握该领域的CRISPR计算工具和计算资源。
2 计算机辅助的CRISPR sgRNA打靶设计CRISPR系统的KO效率主要取决于PAM位置、sgRNA和靶基因位点之间的特异性碱基配对以及随后由NHEJ产生的基因组插入和缺失。此外,DNA序列信息和染色质特征也会对sgRNA的靶向切割产生影响[10-11]。微同源特性与框内移码突变(In-frame mutations)相关,因而可用于预测sgRNA打靶效率。这是因为基于CRISPR的KO通常会产生保留功能性的框内移码突变,从而降低了KO的效率[12]。Bae等率先研究了通过预测微同源特性来提高人体细胞系中CRISPR的KO效率的方法,并通过计算机选择靶位点以减少框内移码突变[12]。在人和小鼠细胞系中,在高效的sgRNA设计中,鸟嘌呤应优先出现在PAM序列最接近的–1和–2位置,这些位置在Cas9初始化时和序列的特性紧密相关[11, 13];而胸腺嘧啶避免存在于靠近PAM的+4/–4位置上[11, 14]。间隔目标下游的DNA的组分也被证明会显著影响sgRNA的效率,而上游的序列核苷酸组分对其没有显著的影响[11, 15]。此外,最近的研究工作同时表明,sgRNA的KO效率和若干表观遗传参数紧密相关[16]。
CRISPR系统同样可以用于下游靶基因的转录抑制(CRISPRi)或激活(CRISPRa)[17]。然而,我们对于序列特征影响CRISPRi/a中sgRNA功效的机制还了解甚少。CRISPRi/a主要靶向基因启动子,其序列特征与编码区不同,这使得sgRNA设计的选定特征与CRISPR KO系统存在差异。与CRISPR/Cas9 KO类似,CRISPRi/a系统更青睐间隔区中占大多数的核苷酸的嘌呤。而二者最重要的差异在于,在用于sgRNA设计的CRISPRi/a系统第3位上没有胞嘧啶富集。此外,与CRISPR/Cas9系统相比,蛋白效应结构域(Protein domain)在CRISPRi/a中是独一无二的,这些结构域在基因定点修饰中起到了关键的作用[11, 18]。到目前为止,只有少量的计算工具可以用于CRISPRi/a系统的sgRNA设计,如CRISPR-ERA[19]和SSC[11],其原因主要是目前CRISPRi/a数据还不足以产生具有鲁棒性的sgRNA设计准则。该领域还需要对可能有助于提高该特定系统中sgRNA功效的特征进行系统的分析。
目前研究者已经开发了用于sgRNA靶向设计的sgRNA设计规则和工具。基于之前对现有资源的总结[6],我们对36个目标sgRNA设计工具进行了全面比较,结果如表 1所示。
| 名称 | 类型 | URL | 是否能脱靶预测 | PAM | 运行平台 | 参考文献 |
| sgRNAcas9 | 序列比对 | http://www.biootools.com/ | 是 | NGG | Perl script/离线/命令行 | [53] |
| Jack Lin's CRISPR/Cas9 gRNA finder | 序列比对 | http://spot.colorado.edu/~slin/cas9.html | 否 | NGG | Web/在线/图形界面 | [54] |
| GT-Scan | 序列比对 | http://gt-scan.braembl.org.au/gt-scan/ | 是 | NGG | Web/在线/图形界面 | [55] |
| CRISPRdirect | 序列比对 | http://crispr.dbcls.jp/ | 是 | NGG, NRG | Web/在线/图形界面 | [56] |
| CRISPR RNA Configurator | 序列比对 | http://dharmacon.gelifesciences.com/ch/gene-editing/crispr-rna-configurator/ | 否 | NGG | Web/在线/图形界面 | |
| CRISPRseek | 序列比对 | http://www.bioconductor.org/ | 是 | NGG, NRG, and extendable | R package/离线/命令行 | [20] |
| CCTop | 序列比对 | http://crispr.cos.uni-heidelberg.de/ | 是 | NGG | Web/在线/图形界面 | [57] |
| Cas-OFFinder | 序列比对 | http://casoffinder.snu.ac.kr/ | 是 | NGG, NAG, NNAGAAW, NNNNGMTT | C++/离线/命令行 | [21] |
| SSFinder | 序列比对 | https://code.google.com/p/ssfinder/ | 否 | NGG | Python script/离线/命令行 | [58] |
| CasFinder | 序列比对 | http://arep.med.harvard.edu/CasFinder/ | 是 | NGG, NAG, NNAGAAW, NNNNGMTT | Python script/离线/命令行 | [59] |
| CRISPR-P | 序列比对 | http://cbi.hzau.edu.cn/crispr/ | 是 | NGG | Web/在线/图形界面 | [26] |
| CRISPRer | 序列比对 | http://jstacs.de/index.php/CRISPRer | 否 | NGG and extendable | Web/在线/图形界面 | |
| CRISPRTarget | 序列比对 | http://bioanalysis.otago.ac.nz/CRISPRTarget | NGG | Web/在线/图形界面 | [60] | |
| CRISPRfinder | 序列比对 | http://crispr.u-psud.fr/Server/ | 否 | NGG and extendable | Web/在线/图形界面 | [61] |
| flyCRISPR | 序列比对 | http://tools.flycrispr.molbio.wisc.edu/targetFinder/ | 是 | NGG | Web/在线/图形界面 | [27] |
| CRISPR gRNA Design tool | 序列比对 | https://www.dna20.com/eCommerce/cas9/input | 否 | NGG, NAG | Web/在线/图形界面 | |
| WGE | 序列比对 | http://www.sanger.ac.uk/htgt/wge/ | 是 | NGG | Web/在线/图形界面 | [62] |
| COD (Cas9 Online Designer) | 序列比对 | http://cas9.wicp.net/ | 是 | NGG, NAG | Web/在线/图形界面 | |
| CRISPOR | 序列比对 | http://crispor.tefor.net/crispor.cgi | 是 | NGG | Web/在线/图形界面 | |
| Protospacer Workbench | 假设先验 | www.protospacer.com | 是 | NGG | Mac OS X/离线/图形界面interface | [25] |
| E-CRISP | 假设先验 | http://www.e-crisp.org/E-CRISP/index.html | 是 | NGG | Web/在线/图形界面 | [24] |
| CRISPR | 假设先验 | http://crispr.mit.edu/ | 是 | NGG | Web/在线/图形界面 | [63] |
| CRISPR-ERA | 假设先验 | http://CRISPR-ERA.stanford.edu | 是 | NGG | Web/在线/图形界面 | [19] |
| CHOPCHOP | 假设先验 | https://chopchop.rc.fas.harvard.edu/ | 是 | NGG | Web/在线/图形界面 | [64] |
| Cas9 design | 假设先验 | http://cas9.cbi.pku.edu.cn/ | 否 | NGG | Web/在线/图形界面 | [65] |
| EuPaGDT | 假设先验 | http://grna.ctegd.uga.edu/index.html | 是 | NGG, NAG | Web/在线/图形界面 | [28] |
| CROP-IT | 假设先验 | http://cheetah.bioch.virginia.edu/AdliLab/CROP-IT/homepage.html | 是 | NGG, NNG | Web/在线/图形界面 | [39] |
| Cas-designer | 假设先验 | http://www.rgenome.net/cas-designer/ | 是 | NGG, NRG, NNAGAAW, NNNNGMTT, NNGRRT | Web/在线/图形界面 | [66] |
| sgRNA Designer (Rule Set 2) | 基于学习 | http://www.broadinstitute.org/rnai/public/analysis-tools/sgrna-design | 否 | NGG | Web/在线/图形界面 | [10] |
| SSC | 基于学习 | http://crispr.dfci.harvard.edu/SSC/ | 否 | NGG | Web/在线/图形界面 | [11] |
| SgRNA Scorer | 基于学习 | http://crispr.med.harvard.edu/sgRNAScorer | 是 | NGG, NAG, NNAGAAW, NNNNGMTT | Web/在线/图形界面 | [16] |
| CRISPR multitargeter | 基于学习 | http://www.multicrispr.net/ | 是 | NGG and extendable | Web/在线/图形界面 | [22] |
| CRISPRscan | 基于学习 | http://www.crisprscan.org/ | 是 | NGG | Web/在线/图形界面 | [52] |
| WU-CRISPR | 基于学习 | http://crispr.wustl.edu/ | 是 | NGG | Web/在线/图形界面 | [23] |
| CRISPR Library Designer (CLD) | 基于学习 | https://github.com/boutroslab/cld | 是 | NGG, NRG, NNAGAAW, NNNNGMTT, NNGRRT | Web/在线/图形界面 | |
| CRISPR-GA | 实验后测序数据分析 | http://crispr-ga.net | Web/在线/图形界面 | [43] | ||
| Microhomology Predictor | 实验后测序数据分析 | http://www.rgenome.net/mich-calculator/ | Web/在线/图形界面 | [12] | ||
| caRpools | 实验后测序数据分析 | http://github.com/boutroslab/caRpools | R package/离线/命令行 | [41] | ||
| MAGeCK | 实验后测序数据分析 | http://liulab.dfci.harvard.edu/Mageck | Python/离线/命令行 | [40] | ||
| CRISPResso | 实验后测序数据分析 | http://crispresso.rocks/ | Web/在线/图形界面 | [44] | ||
| MAGeCK-VISPR | 实验后测序数据分析 | https://bitbucket.org/liulab/mageck-vispr/ | Web/在线/图形界面 | [67] | ||
| CrispRVariants | 实验后测序数据分析 | https://github.com/markrobinsonuzh/CrispRVariants | R package/离线/命令行 | [68] |
我们将这些工具分为3种类型(图 1):1) 基于序列比对的方法(Alignment-based),即sgRNA的选择仅由PAM的位置决定。2) 基于假设先验的方法(Hypothesis-driven),即综合考虑多个特定的因素(例如GC含量、外显子位置等)对sgRNA打靶效率的影响进行打分。3) 基于学习的方法(Learning-based),即利用一个综合考虑不同特征的训练好的模型对sgRNA切割效率预测。我们的基准测试表明,后两种类型的工具比基于序列比对的工具效果更好,因为它们考虑了不同的顺序和染色质特征,而且整合了最新的机器学习模型进行预测。另外,我们建议对于不同的sgRNA设计场景选择不同的计算工具:当用户仅需要基于PAM进行sgRNA设计的时候,建议使用CRISPRseek[20]和Cas-Offinder[21],这两种工具也支持除传统NGG以外的其他PAM类型。此外,对于需要优先选择最优预测性能的高效sgRNA设计的场景,推荐使用基于学习的工具,如sgRNA-designer[10]、CRISPR MultiTargeter[22]、WU-CRISPR[23]、sgRNA Scorer[16]和SSC[11]。其中需要注意的是,sgRNA-designer[10]在其原始版本[15]上使用了基于更新设计规则的logistic回归模型,这在人和小鼠细胞系中表现最好。一些其他的工具,包括E-CRISPR (快速)[24]、CRISPR-ERA (适应于CRISPRi/a)[19]、Protospacer Workbench (适应所有基因组)[25]和CRISPR Library Designer (CLD,适应于CRISPR筛选库设计),也提供了独特且具有吸引力的功能,这使得它们可被应用于不同的sgRNA设计任务。在这些工具中,E-CRISPR具有简单易用的特点,并且能快速识别sgRNA。而Protospacer Workbench拥有友好的图形界面,并且可以适用于除人类和老鼠外的其他基因组。有些sgRNA设计工具是适用于特定物种的,如CRISPR-P适用于植物的基因编辑[26],flyCRISPR适用于果蝇[27],EuPaGDT适用于病原体[28]。因此,在对相应的物种进行基因组编辑实验时,应当首选这些工具。
目前,尚不明确这些sgRNA靶向设计工具在不同细胞类型中的预测结果,因为它们中的大多数设计规则是来源于人和小鼠细胞的。MacPherson等最近设计了CRISPR Software Matchmaker,这是一个基于Excel的程序。它描述了几种sgRNA设计工具的功能,以帮助用户选择合适的工具。然而,目前仍缺乏对于这些工具在不同物种和细胞上的sgRNA预测效果的系统评测。
3 计算机辅助的CRISPR sgRNA脱靶设计对CRISPR系统的研究表明,并不是sgRNA中的每个碱基位点都会匹配目标DNA[6],而这种不匹配或部分匹配常常导致脱靶效应[29]。此外,由结合在特定位点的sgRNA引导的Cas9不一定会导致双链断裂(DSB),除此之外,结合或切割也可能无法产生任何功能性的结果(如框内移码突变)[12]。因此,如何准确和定量地检测真实的功能性切割位点,是基于CRISPR的脱靶分析中的关键问题[30]。目前已经产生了若干种基于实验和测序的技术来检测整个基因组层面sgRNA的脱靶情况。这些技术涵盖了从蛋白质结合位点捕获到DSB捕获[29],而所有这些技术都需要整合全基因组测序和后验数据分析来鉴定其各自的位点。不同的技术会产生不同的sgRNA的脱靶特性。例如,染色质免疫沉淀测序(ChIP-seq)技术主要描述dCas9结合位点,其结果表明,与sgRNA核心或“种子”匹配的sgRNA近端一半的PAM同源就足以启动Cas9结合[6]。然而,实际的裂解还需要在靶位点产生更广泛的碱基配对,以及出现在NHEJ后的插入缺失突变。因此,ChIP-seq技术通常仅捕获dCas9结合位点,而不是真正的DNA切割事件[6, 14, 31-33]。目前,研究者已经借助Digenome-seq[34]和其他DSB捕获技术,例如Guide-seq[35]、IDLV捕获[36]、HTGTS (高通量全基因组转座测序)[37]和BLESS (直接原位断裂标记)[38]等技术,对脱靶效率进行了进一步探究。
计算机辅助的sgRNA脱靶预测也可以通过使用表 1中列出的若干种工具来实现。几乎所有的现有工具都使用简单的序列匹配技术,通过对错配计数来搜索脱靶位点。然而对于可能影响脱靶的序列和染色质特征,目前的研究成果还很少。迄今还没有工具可以准确预测脱靶位点。研究者们最近进行了两项比较研究:一项是将Guide-seq[35]生成的实验性脱靶位点与计算机辅助脱靶预测的MIT CRISPR和E-CRISP进行比较,另一项是将COSMID (一个较高级的CRISPR脱靶位点数据库)与其他几个脱靶预测工具[29]进行比较。两项比较都显示出,对于不同的工具,预测位点和实际位点都有大量差异。产生这些差异的可能原因在于:1) 不同工具之间的碱基错配偏差阈值不同,因此会产生不同的脱靶预测结果。一个可以改善预测结果的方法是通过对不同工具的结果进行整合来获得较为一致的结果。2) 仅基于碱基错配来识别脱靶位点,是无法完整刻画固有的脱靶机制的。例如,像染色质特征等因素也会影响Cas9结合,然而现有工具对其还没有深入的研究。
为了改善脱靶预测性能,需要更好地了解基因和染色质的特征,以及核酸酶DNA结合和切割的机制。最近开发的脱靶预测和鉴定工具(CROP-IT)首次尝试通过整合来自现有Cas9结合和切割数据集的全基因组信息来改善脱靶结合和切割位点预测[39]。然而,与大量的打靶基因组编辑数据训练集相比,脱靶数据集还很少。从模型训练的角度来看,对脱靶预测效果进行改良的另一个关键点在于积累由上述实验和测序技术获得的脱靶基准数据。
4 对CRISPR基因编辑的实验后测序数据分析对CRISPR基因编辑系统的实验后测序数据进行分析也是一个十分重要的课题。它主要涉及CRISPR筛选分析(CRISPR screen analysis)和由NHEJ/HDR产生的基因组插入缺失突变分析。
目前在功能基因组研究中,一个非常重要的问题是鉴定涉及特定生物过程的重要基因。其常规的方法是基于正向遗传学筛选鉴定这些基因,通过高通量的基因定点修饰和基因敲除,将基因功能与特定表型相关联。为此,基于CRISPR的遗传筛选对于研究潜在遗传因素未知的疾病或表型特别有用。通常,基于CRISPR的遗传筛选的目标是使用用于KO或KI的慢病毒CRISPR库,来产生具有多种基因突变的大量细胞群,以鉴定导致特定表现型的基因定点修饰[3]。靶向102至104个基因的常规GeCKO库可以对哺乳动物细胞系进行阴性和阳性选择筛选[11]。最近研究者又开发了两个新的人类和小鼠全基因组库Brunello和Brie。我们将其与传统的库进行了比较[10]。这两个库是通过更新的sgRNA设计规则(规则集2) 设计的,用以最大化目标sgRNA的切割效率,并最小化具有较高切割频率分数的脱靶位点,从而改进了现有的CRISPR筛选库[10]。为了进一步进行CRISPR筛选的数据分析,研究者开发了两种工具,即MAGeCK[40]和caRpools[41](表 1),它们涉及正向/负向的选择。通过对CRISPR筛选后的高通量测序数据应用统计分析,来检测显著选择的基因以及它们的功能。
另外,研究者还开发了用于分析CRISPR实验结果的工具。CRISPR-GA[42]是评估基因组编辑实验质量的计算平台。它基于实验后测序数据,对在预期靶向位点的插入、缺失和同源重组进行了量化分析和表征。CRISP-Resso提供了一套计算工具,用于对目标基因位点易受深度测序影响的基因编辑实验结果进行定性和定量评估[43]。另外还有若干种工具,如Microhomology-Predictor[12],利用实验后测序数据来预测高效sgRNA打靶位点。表 1最后列出了7个基于CRISPR实验后测序数据的基因编辑分析工具。
5 CRISPR计算领域的若干重要开放问题 5.1 对计算机辅助CRISPR sgRNA设计工具的基准分析如上所述,尽管已经有多种可用于高效的sgRNA打靶和脱靶设计的工具,但仍需要通过基准实验数据来比较它们的sgRNA预测效果,需要仔细设计基准数据和测试过程,以确保整个过程是无偏的。另外,目前尚不确定是否可在不同的sgRNA库、细胞类型和生物体上应用一套普适的sgRNA设计规则。我们近期的初步工作表明,基于学习的工具的结果优于基于假设和基于序列比对的sgRNA设计工具[44]。现有的研究工作应用了若干种学习模型(如线性回归、logistic回归、增强回归树、随机森林、支持向量机(SVM)等),并对其在特定数据集中的sgRNA功效预测进行了比较[10],但是仍然不知道它们在不同数据集上的普适预测性能。未来仍然需要通过开发新的计算模型来改善sgRNA预测,以及应用更多的特征选择技术来进行研究,以揭示能增强sgRNA功效的最显著特征。
5.2 sgRNA设计规则的不一致性目前尚不确定是否可在不同的sgRNA库、细胞类型或生物体内重复利用之前研究中发现的序列特征和设计规则。例如在Xu等的研究中,面向HL60和mESC细胞的有效sgRNA设计特征并不相同[11]。他们还认识到,在该研究中可能会遗漏一些细胞特异性序列偏好[11]。而在另一个在bioRxiv上刊登的研究中,研究者在两个不同的数据集上比较了不同的sgRNA KO效率预测模型。其中,sgRNA KO是使用流式细胞术和抗性检验监测的[45]。他们观察到,在流式细胞术数据上,不同模型的预测性能总是比在抗性检验数据上好。这表明在sgRNA KO效率的不同实验测量中,存在大量的批次效应和异质性[45]。尽管在人类CRISPR基因敲除中,微同源特性可用来预测框内移码突变的发生[12],然而当sgRNA效率预测与其他特征相结合时,它被证实是冗余的[10]。我们的研究还表明,微同源特性可能不能作为在多种细胞系中检测框内移码突变的关键指标[46]。总而言之,除了PAM识别和碱基配对之外,其他基因组特征,如核苷酸特性、GC含量和微同源序列模式等,都影响着sgRNA打靶效应。然而,研究者们还没有深入探究染色质特征和sgRNA结构。目前的sgRNA设计规则很可能是不完整的或者是有偏的,仍需要大量数据的整合分析。
5.3 对CRISPR脱靶的高估/低估虽然已经进行了各种研究,但是研究者们仍然难以确定控制sgRNA靶向特异性的规则,特别是控制功能切割位点和突变产生的规则。此外,研究者们仍不清楚除碱基错配之外的其他影响脱靶的因素。目前的证据表明,大部分被ChIP-seq捕获的Cas9脱靶结合事件都是短暂的,功能影响很小。这表明目标结合位点检测技术高估了CRISPR脱靶的效果[47]。与早期技术相比,其他DSB捕获技术可以检测到较少的脱靶事件[48],然而这还需在涵盖了不同sgRNA库和细胞类型的大规模数据集上进一步进行实验证实。目前研究者已经通过各种技术成功地增加了CRISPR特异性,例如在最新的研究中,人们考虑了双切口系统的设计[49],gRNA截短和gRNA延伸[34, 50],采用新型Cas9直系同源物[7],以及对现有Cas9蛋白的改造等[5, 51]。
5.4 个性化的计算机辅助sgRNA设计对于不同细胞类型,不同工具会产生不同的sgRNA KO功效预测结果。而目前所有现有的工具都是针对普适的sgRNA设计而提出的,并没有考虑细胞型异质性。其原因之一是,研究者们还没有深入探究可能影响CRISPR系统的表观遗传和染色质特征,而这些因素是高度细胞类型特异性的。目前,只有很少的计算机辅助打靶/脱靶预测工具对表观遗传学特征进行了全面的探究,尽管如此,它们也未能改善在特定细胞类型的预测性能。来自我们课题组内部评估的一个示例表明,基于学习的sgRNA打靶功效预测工具CRISPRscan[52],在基于人体细胞的基准数据中的表现比其他基于学习的工具更差。对这项工具的后续调研显示,其sgRNA设计规则是来自斑马鱼数据,因此它在人类细胞中的预测效果较差。这表明了细胞类型异质性对功效预测的影响。未来的sgRNA设计应该考虑细胞型异质性,考虑不同细胞类型的表观遗传环境,并为特定的细胞类型输出个性化的sgRNA设计规则,以获得更好的sgRNA设计效果。
6 总结与展望CRISPR/Cas9技术已经迅速发展成为用于功能基因组编辑研究的最新技术。该技术具有诸如易用、高效、特异性和多功能性等巨大优点。计算机辅助的sgRNA设计为CRISPR系统的发展迈出了关键的一步,并推动了生物信息学和计算技术在CRISPR研究中的应用。不过,研究者仍需要继续改进计算机辅助的sgRNA设计,以提高打靶效率,并减少脱靶。需要注意的是:1) 用户应为其特定设计目的选择合适的工具。同时仍需要通过仔细设计的基准来评价其在不同基准数据集中的性能。2) 需要设计高效的模型来整合当前的异质基因组编辑数据,以获得无偏的sgRNA设计规则,并确定与sgRNA打靶和脱靶有关的关键特征。目前所有的工具都是针对普适的sgRNA设计提出的,并未考虑细胞类型的异质性。个性化的sgRNA设计是计算机辅助sgRNA设计的重要发展方向。3) 最后,可利用各种脱靶检测测定和高通量测序技术,对CRISPR系统中的脱靶进行仔细检测,以避免对其低估或高估。总之,计算机辅助的sgRNA设计,将促进更精准的基因组编辑和基因治疗,并减少脱靶。
说明:本论文主要内容来自于笔者英文发表论文[69],中文版本有少量更新,其目的是将原英文论文进行翻译,面向中文的读者群体。该中文翻译版本已经获得了原论文出版机构Elisvier的授权,同意在《生物工程学报》进行发表,特此说明。
| [1] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819–823. DOI: 10.1126/science.1231143 |
| [2] | Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096. DOI: 10.1126/science.1258096 |
| [3] | Hartenian E, Doench JG. Genetic screens and functional genomics using CRISPR/Cas9 technology. FEBS J, 2015, 282(8): 1383–1393. DOI: 10.1111/febs.13248 |
| [4] | Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol, 2014, 32(4): 347–355. DOI: 10.1038/nbt.2842 |
| [5] | Slaymaker IM, Gao LY, Zetsche B, et al. Rationally engineered Cas9 nucleases with improved specificity. Science, 2016, 351(6268): 84–88. DOI: 10.1126/science.aad5227 |
| [6] | Graham DB, Root DE. Resources for the design of CRISPR gene editing experiments. Genome Biol, 2015, 16: 260. DOI: 10.1186/s13059-015-0823-x |
| [7] | Shmakov S, Abudayyeh OO, Makarova KS, et al. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Mol Cell, 2015, 60(3): 385–397. DOI: 10.1016/j.molcel.2015.10.008 |
| [8] | Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759–771. DOI: 10.1016/j.cell.2015.09.038 |
| [9] | Henry VJ, Bandrowski AE, Pepin AS, et al. OMICtools: an informative directory for multi-omic data analysis. Database, 2014, 2014: bau069. DOI: 10.1093/database/bau069 |
| [10] | Doench JG, FusiN, Sullender M, et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat Biotechnol, 2016, 34(2): 184–191. DOI: 10.1038/nbt.3437 |
| [11] | Xu H, Xiao TF, Chen CH, et al. Sequence determinants of improved CRISPR sgRNA design. Genome Res, 2015, 25: 1147–1157. DOI: 10.1101/gr.191452.115 |
| [12] | Bae S, Kweon J, Kim HS, et al. Microhomology-based choice of Cas9 nuclease target sites. Nat Methods, 2014, 11(7): 705–706. DOI: 10.1038/nmeth.3015 |
| [13] | Wang T, Wei JJ, Sabatini DM, et al. Genetic screens in human cells using the CRISPR-Cas9 system. Science, 2014, 343(6166): 80–84. DOI: 10.1126/science.1246981 |
| [14] | Wu XB, Scott DA, Kriz AJ, et al. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat Biotechnol, 2014, 32(7): 670–676. DOI: 10.1038/nbt.2889 |
| [15] | Doench JG, Hartenian E, Graham DB, et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nat Biotechnol, 2014, 32(12): 1262–1267. DOI: 10.1038/nbt.3026 |
| [16] | Chari R, Mali P, Moosburner M, et al. Unraveling CRISPR-Cas9 genome engineering parameters via a library-on-library approach. Nat Methods, 2015, 12(9): 823–826. DOI: 10.1038/nmeth.3473 |
| [17] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173–1183. DOI: 10.1016/j.cell.2013.02.022 |
| [18] | Chen BH, Gilbert LA, Cimini BA, et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell, 2013, 155(7): 1479–1491. DOI: 10.1016/j.cell.2013.12.001 |
| [19] | Liu HL, Wei Z, Dominguez A, et al. CRISPR-ERA: a comprehensive design tool for CRISPR-mediated gene editing, repression and activation. Bioinformatics, 2015, 31(22): 3676–3678. DOI: 10.1093/bioinformatics/btv423 |
| [20] | Zhu LJ, Holmes BR, Aronin N, et al. CRISPRseek: a bioconductor package to identify target-specific guide RNAs for CRISPR-Cas9 genome-editing systems. PLoS ONE, 2014, 9(9): e108424. DOI: 10.1371/journal.pone.0108424 |
| [21] | Bae S, Park J, Kim JS. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics, 2014, 30(10): 1473–1475. DOI: 10.1093/bioinformatics/btu048 |
| [22] | Prykhozhij SV, Rajan V, Gaston D, et al. CRISPR multitargeter: a web tool to find common and unique CRISPR single guide RNA targets in a set of similar sequences. PLoS ONE, 2015, 10(3): e0119372. DOI: 10.1371/journal.pone.0119372 |
| [23] | Wong N, Liu WJ, Wang XW. WU-CRISPR: characteristics of functional guide RNAs for the CRISPR/Cas9 system. Genome Biol, 2015, 16: 218. DOI: 10.1186/s13059-015-0784-0 |
| [24] | Heigwer F, Kerr G, Boutros M. E-CRISP: fast CRISPR target site identification. Nat Methods, 2014, 11(2): 122–123. DOI: 10.1038/nmeth.2812 |
| [25] | MacPherson CR, Scherf A. Flexible guide-RNA design for CRISPR applications using Protospacer Workbench. Nat Biotechnol, 2015, 33(8): 805–806. |
| [26] | Lei Y, Lu L, Liu HY, et al. CRISPR-P: a web tool for synthetic single-guide RNA design of CRISPR-system in plants. Mol Plant, 2014, 7(9): 1494–1496. DOI: 10.1093/mp/ssu044 |
| [27] | Gratz SJ, Ukken FP, Rubinstein CD, et al. Highly specific and efficient CRISPR/Cas9-catalyzed homology-directed repair in Drosophila. Genetics, 2014, 196(4): 961–971. DOI: 10.1534/genetics.113.160713 |
| [28] | Peng D, Tarleton R. EuPaGDT: a web tool tailored to design CRISPR guide RNAs for eukaryotic pathogens. Microb Genom, 2015, 1(4): e000033. |
| [29] | Lee CM, Cradick TJ, Fine EJ, et al. Nuclease target site selection for maximizing on-target activity and minimizing off-target effects in genome editing. Mol Ther, 2016, 24(3): 475–487. DOI: 10.1038/mt.2016.1 |
| [30] | Hendel A, Fine EJ, Bao G, et al. Quantifying on-and off-target genome editing. Trends Biotechnol, 2015, 33(2): 132–140. DOI: 10.1016/j.tibtech.2014.12.001 |
| [31] | Duan JZ, Lu GQ, Xie Z, et al. Genome-wide identification of CRISPR/Cas9 off-targets in human genome. Cell Res, 2014, 24(8): 1009–1012. DOI: 10.1038/cr.2014.87 |
| [32] | O'Geen H, Henry IM, Bhakta MS, et al. A genome-wide analysis of Cas9 binding specificity using ChIP-seq and targeted sequence capture. Nucleic Acids Res, 2015, 43(6): 3389–3404. DOI: 10.1093/nar/gkv137 |
| [33] | Kuscu C, Arslan S, Singh R, et al. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol, 2014, 32(7): 677–683. DOI: 10.1038/nbt.2916 |
| [34] | Kim D, Bae S, Park J, et al. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods, 2015, 12(3): 237–243. DOI: 10.1038/nmeth.3284 |
| [35] | Tsai SQ, Zheng ZL, Nguyen NT, et al. GUIDE-Seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol, 2015, 33(2): 187–197. |
| [36] | Wang XL, Wang YB, Wu XW, et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol, 2015, 33(2): 175–178. DOI: 10.1038/nbt.3127 |
| [37] | Frock RL, Hu JZ, Meyers RM, et al. Genome-wide detection of DNA double-stranded breaks induced by engineered nucleases. Nat Biotechnol, 2015, 33(2): 179–186. |
| [38] | Crosetto N, Mitra A, Silva MJ, et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods, 2013, 10(4): 361–365. DOI: 10.1038/nmeth.2408 |
| [39] | Singh R, Kuscu C, Quinlan A, et al. Cas9-chromatin binding information enables more accurate CRISPR off-target prediction. Nucleic Acids Res, 2015, 43(18): e118. DOI: 10.1093/nar/gkv575 |
| [40] | Li W, Xu H, Xiao TF, et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knockout screens. Genome Biol, 2014, 15(12): 554. DOI: 10.1186/s13059-014-0554-4 |
| [41] | Winter J, Breinig M, Heigwer F, et al. caRpools: an R package for exploratory data analysis and documentation of pooled CRISPR/Cas9 screens. Bioinformatics, 2016, 32(4): 632–634. DOI: 10.1093/bioinformatics/btv617 |
| [42] | Güell M, Yang LH, Church GM. Genome editing assessment using CRISPR Genome Analyzer (CRISPR-GA). Bioinformatics, 2014, 30(20): 2968–2970. DOI: 10.1093/bioinformatics/btu427 |
| [43] | Pinello L, Canver MC, Hoban MD, et al. CRISPResso: sequencing analysis toolbox for CRISPR-Cas9 genome editing. bioRxiv, 2015. DOI: 10.1101/031203 |
| [44] | Yan JF, Chuai GH, Zhou C, et al. Benchmarking CRISPR on-target sgRNA design. Brief Bioinform, 2017. DOI: 10.1093/bib/bbx001 |
| [45] | Fusi N, Smith I, Doench J, et al. In silico predictive modeling of CRISPR/Cas9 guide efficiency. bioRxiv, 2015. DOI: 10.1101/021568 |
| [46] | Chuai GH, Yang FU, Yan JF, et al. Deciphering relationship between microhomology and in-frame mutation occurrence in human CRISPR-based gene knockout. Mol Ther Nucleic Acids, 2016, 5. |
| [47] | Wu XB, Kriz AJ, Sharp PA. Target specificity of the CRISPR-Cas9 system. Quant Biol, 2014, 2(2): 59–70. DOI: 10.1007/s40484-014-0030-x |
| [48] | Kim D, Kim S, Kim S, et al. Genome-wide target specificities of CRISPR-Cas9 nucleases revealed by multiplex Digenome-seq. Genome Res, 2016, 26(3): 406–415. DOI: 10.1101/gr.199588.115 |
| [49] | Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380–1389. DOI: 10.1016/j.cell.2013.08.021 |
| [50] | Arribere JA, Bell RT, Fu BX, et al. Efficient marker-free recovery of custom genetic modifications with CRISPR/Cas9 in Caenorhabditis elegans. Genetics, 2014, 198(3): 837–846. DOI: 10.1534/genetics.114.169730 |
| [51] | Kleinstiver BP, Pattanayak V, Prew MS, et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature, 2016, 529(7587): 490–495. DOI: 10.1038/nature16526 |
| [52] | Moreno-Mateos MA, Vejnar CE, Beaudoin JD, et al. CRISPRscan: designing highly efficient sgRNAs for CRISPR-Cas9 targeting in vivo. Nat Methods, 2015, 12(10): 982–988. DOI: 10.1038/nmeth.3543 |
| [53] | Xie SS, Shen B, Zhang CB, et al. sgRNAcas9: a software package for designing CRISPR sgRNA and evaluating potential off-target cleavage sites. PLoS ONE, 2014, 9(6): e100448. DOI: 10.1371/journal.pone.0100448 |
| [54] | Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823–826. DOI: 10.1126/science.1232033 |
| [55] | O'Brien A, Bailey TL. GT-Scan: identifying unique genomic targets. Bioinformatics, 2014, 30(18): 2673–2675. DOI: 10.1093/bioinformatics/btu354 |
| [56] | Naito Y, Hino K, Bono H, et al. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics, 2015, 31(7): 1120–1123. DOI: 10.1093/bioinformatics/btu743 |
| [57] | Stemmer M, Thumberger T, Del Sol Keyer M, et al. CCTop: an intuitive, flexible and reliable CRISPR/Cas9 target prediction tool. PLoS ONE, 2015, 10(4): e0124633. DOI: 10.1371/journal.pone.0124633 |
| [58] | Upadhyay SK, Sharma S. SSFinder: high throughput CRISPR-Cas target sites prediction tool. Biomed Res Int, 2014, 2014: 742482. |
| [59] | Aach J, Mali P, Church GM. CasFinder: flexible algorithm for identifying specific Cas9 targets in genomes. bioRxiv, 2014. DOI: 10.1101/005074 |
| [60] | Biswas A, Gagnon JN, Brouns SJ, et al. CRISPRTarget: bioinformatic prediction and analysis of crRNA targets. RNA Biol, 2013, 10(5): 817–827. DOI: 10.4161/rna.24046 |
| [61] | Grissa I, Vergnaud G, Pourcel C. CRISPRFinder: a web tool to identify clustered regularly interspaced short palindromic repeats. Nucleic Acids Res, 2007, 35(S2): W52–W57. |
| [62] | Hodgkins A, Farne A, Perera S, et al. WGE: a CRISPR database for genome engineering. Bioinformatics, 2015, 31(18): 3078–3080. DOI: 10.1093/bioinformatics/btv308 |
| [63] | Hsu PD, Scott DA, Weinstein JA, et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol, 2013, 31(9): 827–832. DOI: 10.1038/nbt.2647 |
| [64] | Montague TG, Cruz JM, Gagnon JA, et al. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Res, 2014, 42(W1): W401–W407. DOI: 10.1093/nar/gku410 |
| [65] | Ma M, Ye AY, Zheng WG, et al. A guide RNA sequence design platform for the CRISPR/Cas9 system for model organism genomes. Biomed Res Int, 2013, 2013: 270805. |
| [66] | Park J, Bae S, Kim JS. Cas-Designer: a web-based tool for choice of CRISPR-Cas9 target sites. Bioinformatics, 2015, 31(24): 4014–4016. |
| [67] | Li W, Kster J, Xu H, et al. Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR. Genome Biol, 2015, 16: 281. DOI: 10.1186/s13059-015-0843-6 |
| [68] | Lindsay H, Burger A, Biyong B, et al. CrispRVariants: precisely charting the mutation spectrum in genome engineering experiments. bioRxiv, 2016. DOI: 10.1101/034140 |
| [69] | Chuai GH, Wang QL, Liu Q. In silico meets in vivo: towards computational CRISPR-based sgRNA design. Trends Biotechnol, 2017, 35(1): 12–21. DOI: 10.1016/j.tibtech.2016.06.008 |
 2017, Vol. 33
2017, Vol. 33