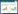中国科学院微生物研究所、中国微生物学会主办
文章信息
- 王路, 刘涛, 陈洋, 孙亚琴, 修志龙
- Lu Wang, Tao Liu, Yang Chen, Yaqin Sun, Zhilong Xiu
- 基于傅里叶变换近红外光谱实时分析 1,3- 丙二醇发酵过程生物量的在线监测方法
- On-line monitoring of biomass in 1,3-propanediol fermentation by Fourier-transformed near-infrared spectra analysis
- 生物工程学报, 2017, ${metaVo.volume}(1): 68-78
- Chin J Biotech, 2017, ${metaVo.volume}(1): 68-78
- 10.13345/j.cjb.160256
-
文章历史
- Received: June 29, 2016
- Accepted: September 26, 2016
2 大连理工大学 生命科学与技术学院,辽宁 大连 116024
2 School of Life Science and Biotechnology, Dalian University of Technology, Dalian 116024, Liaoning, China
发酵技术现已被应用于很多生产领域,关于发酵过程监控与优化方面的研究受到越来越多的关注和探讨[1-3]。相对于很多其他生化工程,发酵过程大多数成分复杂且变化较快[4],因此要对发酵过程实施在线调控,首先要解决的问题就是如何实现对过程重要参数的实时监测[5]。生物量是发酵过程的一个重要参数[6-7],目前采用的细胞干重、分光光度计测量吸光光度值 (OD) 等监测方法比较费时,并且不能实现在线监测,因而造成目前发酵过程控制明显落后于现今工业过程控制的发展水平。
近红外光谱技术拥有无损、快速以及无需样品预处理等优点[8-9],通过结合先进的化学计量学方法,可以实现对工业过程的实时在线监控。目前已经越来越受到人们的关注,被广泛应用于食品、农业、医药以及化工等领域[10-13]。将近红外光谱分析技术应用于生物发酵过程参数的监测也已经有一些报道,但是大都应用离线方法。桂勇利等[14]通过建立近红外定量校正模型对谷氨酸发酵过程中乳酸的浓度进行快速离线检测。Guo等[15]通过近红外光谱技术离线监测乳酸链球菌发酵过程的乳酸链球菌效价、还原糖浓度、细胞浓度和pH这4个关键参数。Cruz等[16]应用近红外光谱技术对废弃食用油发酵过程的胞内物质P (3HB) 等重要参数进行了快速离线检测。
本文针对克雷伯氏杆菌发酵生成1,3-丙二醇过程中的重要参数——生物量,开展在线监测研究,实时采集发酵过程的近红外光谱,对光谱进行敏感谱段选择,通过比较多种光谱预处理方法,对发酵过程生物量应用偏最小二乘算法建立较为准确的在线监测模型。以测量吸光光度值 (OD) 的方法作为检测生物量的离线参考方法,进行实验验证,结果表明本文所建立的模型能够实现对1,3-丙二醇发酵过程中生物量的有效实时在线监测。
1 材料与方法 1.1 主要仪器与1,3-丙二醇发酵过程在线监测实验平台本文使用的仪器和软件主要包括:傅立叶在线近红外分析仪 (型号:TALYS-ASP531,配以波数范围为4 000-12 500 cm-1的浸没式透反射探头,ABB公司),近红外光谱分析与处理软件HorizonMB (ABB公司),5 L发酵罐 (型号:5BG,上海保兴生物设备有限公司),722 s分光光度计 (波长范围:340-1 000 nm,上海精密科学仪器有限公司) 等。通过对仪器的调整,本实验室设计并搭建了基于近红外光谱分析技术的1,3-丙二醇发酵过程在线监测实验平台,如图 1所示。
|
| 图 1 1,3-丙二醇发酵过程在线监测实验平台 Figure 1 Experimental platform for on-line monitoring a 1,3-propanediol fermentation process. |
| |
本实验平台主要用于研究基于近红外光谱分析技术在线监测1,3-丙二醇发酵过程的方法。其中发酵反应在5 L发酵罐内进行,发酵过程中使用生物发酵控制器保持发酵罐内的温度 (使用PT100探头采集温度信息,并使用加热装置和冷水进行调节) 和pH (使用pH计采集酸碱度信息,并使用NaOH进行调节) 稳定于设定值,并控制搅拌桨对发酵液进行稳定的搅拌。通过通入氮气的方式使发酵罐内保持厌氧状态,以满足菌体生长的需要。在发酵反应进行的同时,使用配以浸没式透反射探头的傅里叶在线近红外光谱仪 (探头和光谱仪之间使用光纤连接) 对整个发酵过程进行近红外光谱的在线采集,并可通过计算机进行实时的谱图分析与在线监测工作。
1.2 菌种、培养基组成与实验方法菌种:本文所进行的实验中使用的菌种均为克雷伯氏肺炎菌Klebsiella pneumoniae CGMCC 2028,是大连理工大学生命科学与技术学院课题组筛选诱变、保存于中国微生物菌种保藏中心的菌种。
种子培养基组成如表 1所示。
| Substances | Concentration |
| Glycerol | 20.0 g/L |
| KH2PO4 | 1.3 g/L |
| CaCO3 | 2.0 g/L |
| Ca2+ | 1.0 mL/L |
| Fe2+ | 1.0 mL/L |
| (NH4)2SO4 | 2.0 g/L |
| K2HPO4·7H2O | 4.454 g/L |
| MgSO4·7H2O | 0.2 g/L |
| Trace elements A | 2.0 ml/L |
| Yeast extract powder | 1.0 g/L |
其中微量元素A的组成如表 2所示。
| Substances | Concentration |
| 12M HCl | 0.9 mL/L |
| H3BO3 | 60.0 mg/L |
| ZnCl2 | 70.0 mg/L |
| NiCl2·6H2O | 25.0 mg/L |
| CuCl2·2H2O | 20 mg/L |
| MnCl2·4H2O | 100 mg/L |
| CaCl2·6H2O | 200 mg/L |
| Na2MoO4·2H2O | 35 mg/L |
其中,Fe2+溶液由12 mol/L HCl (4 mL/L) 和FeSO4· 7H2O (5 g/L) 组成,Ca2+溶液由CaCl2 (20 g/L) 组成。
发酵培养基组成如表 3所示。
| Substances | Concentration |
| Glycerol | 60.00 g/L |
| KH2PO4 | 1.36 g/L |
| CaCl2 | 0.29 g/L |
| Citric acid | 0.42 g/L |
| MgCl2·6H2O | 0.26 g/L |
| (NH4)2SO4 | 6.61 g/L |
| Trace elements B | 5.00 mL/L |
| Yeast extract powder | 2.00 g/L |
其中微量元素B的组成如表 4所示。
| Substances | Concentration |
| 12 mol/L HCl | 10.00 mL/L |
| ZnCl2 | 0.68 g/L |
| H3BO3 | 0.06 g/L |
| CoCl2·6H2O | 0.47 g/L |
| CuCl2·2H2O | 0.47 g/L |
| FeCl2·4H2O | 3.97 g/L |
| MnCl2·4H2O | 0.17 g/L |
| Na2MoO4·2H2O | 0.005 g/L |
以上培养基均经过121 ℃、20 min高温灭菌处理。
种子培养方法:按1% (V/V) 的接种量将菌种接种到两瓶100 mL种子培养基内,于37 ℃、150 r/min摇床培养12 h。
发酵实验方法:将培养好的种子接种到装有2 L发酵培养基的5 L发酵罐中,整个发酵过程中通过温度控制设备将发酵液温度保持在 (37±0.5) ℃范围内,并使用5 mol/L的NaOH溶液保持发酵液的pH稳定在 (7±0.5) 范围内。整个发酵过程以0.1 vvm速率通入氮气以保持发酵过程的厌氧状态,并保持250 r/min的搅拌速率,使发酵液保持均匀状态,全程发酵时间13 h。
1.3 发酵过程取样、光谱数据采集与吸光光度值 (OD) 数据采集在发酵过程中每隔1 h对发酵液取样1次,为拓展模型的适用范围并提高建模的准确性,对未接菌时的培养基也进行取样和后续的分析处理。使用测量吸光光度值 (OD) 的方法获取发酵过程生物量的参考数据 (下文称为实测值)。吸光光度值 (OD) 用光密度法以蒸馏水为背景,适当稀释发酵液后在650 nm波长下测定。在取样的同时进行近红外光谱的在线采集。以分辨率16,扫描次数128次 (约50 s),检测器增益237.84,空气背景,吸收光谱的模式在4 000-12 000 cm-1的光谱范围上连续采集近红外光谱3次作为与该样品对应的近红外光谱数据。为对发酵过程进行全面的分析,对取出的样品进行离心处理得到上清液,用水浴锅保持上清液与发酵过程相同的温度 (37 ℃) 并以相同的光谱采集条件进行近红外光谱的离线采集。
2 1,3-丙二醇发酵过程生物量在线动态监测模型建立 2.1 吸光光度值 (OD) 数据分析与处理在上面介绍的实验条件下进行5个批次的发酵实验,得到68个样本用于在线动态监测模型的建立。随机的把样本点分为3份,取其中1份作为验证集,剩余的作为校正集。校正集与验证集的数据特性如表 5所示。从表中可以看到在吸光光度值 (OD) 范围上校正集略大于验证集,在平均数、方差方面都比较相似,根据文献[17]可知这样的数据特性对建立准确可靠的多元校正模型具有重要意义。
| Numbers of samples | Ratio (%) | OD | Mean | Variance | |
| Calibration set | 46 | 67.65 | 0.046 0-8.261 0 | 3.196 5 | 7.262 2 |
| Validation set | 22 | 32.35 | 0.061 2-8.048 3 | 3.148 9 | 6.988 6 |
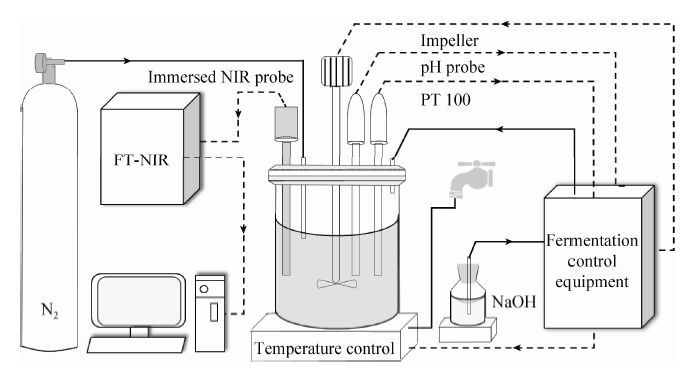
在上文介绍的光谱采集条件下对5个批次的发酵实验进行光谱在线采集,得到原始光谱如图 2所示。
|
| 图 2 1,3-丙二醇发酵过程的原始光谱图 Figure 2 Original spectra of a 1,3-propanediol fermentation process. |
| |
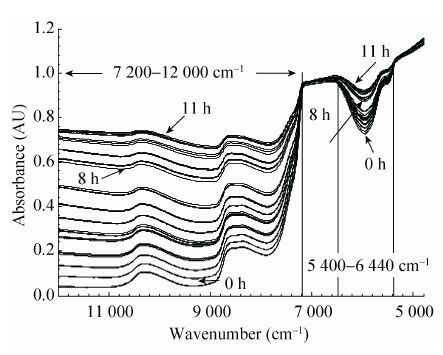
该图为一个批次发酵实验并对含有大量噪声的4 000-4 800 cm-1谱段进行删除处理后所得到的原始光谱图。结合发酵过程信息可以明显看出随着发酵时间的增长,光谱基线不断升高,这与随着发酵时间的增长生物量不断增长 (吸光光度值 (OD) 不断升高) 是相关的。对比图 3发酵过程上清液的光谱图可以看到,排除细胞影响后发酵过程物质浓度变化给光谱带来的影响较小,这说明在线采集光谱的基线变化主要由生物量变化导致 (发酵过程中pH,温度等条件保持恒定,可以排除这些干扰的影响),由底物甘油和主产物1,3-丙二醇等物质吸收带来的影响相对较小。这种现象产生的主要原因是生物量浓度的提高使发酵液对近红外光的散射能力增强,导致光谱的吸光度变高。放大分析光谱在不同波段上的敏感性可以发现,在谱段7 200-12 000 cm-1 (下文称为谱段A) 和谱段5 400-6 440 cm-1 (下文称为谱段B) 上光谱对生物量变化的敏感性更强。
|
| 图 3 发酵过程离心上清液光谱图 Figure 3 The original spectra of supernatant. |
| |
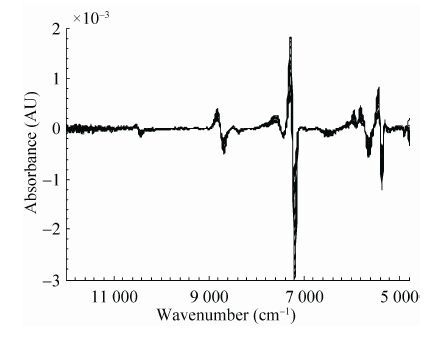
同时A、B这两个谱段也基本涵盖了C-H等基团的吸收区域[18],由于细胞本身也含有很多近红外光谱可以分析的C-H、O-H等基团,会在发酵过程中对光谱的变化产生一定的影响,因此对光谱作以下光谱预处理,以便减少由散射、仪器噪声等带来的干扰。本文主要使用的光谱预处理方法包括多元散射校正 (MSC)[19]、标准正态变量变换 (SNV) [19]、SG平滑[19]及一阶导数和二阶导数处理。经一阶导数和二阶导数处理后的光谱如图 4和图 5所示。从图中可以看到导数处理把光谱的基线拉平,基本上消除了光谱的噪声。

|
| 图 4 经一阶导数处理后的光谱图 Figure 4 The first derivative spectra. |
| |
|
| 图 5 经二阶导数处理后的光谱图 Figure 5 The second derivative spectra. |
| |
在对吸光光度值 (OD) 数据和近红外光谱数据进行上文的分析与处理的基础上应用偏最小二乘算法[20-22]建模。在偏最小二乘算法的建模过程中因子数的选择是一个关键的问题,本文使用了交叉有效性原则对模型的因子数进行选择。此原则主要考察因子的边际贡献率[23],可以减少选取因子数的数量,降低模型过拟合的概率,使模型更具可靠性。1,3-丙二醇发酵过程通常伴有噪声与干扰,如通气带来的气泡与强力搅拌带来的干扰等[24]。因而需要可靠性好的在线动态监测模型来实现对该发酵过程的有效在线监测。
交叉有效性原则主要是通过考察取h个因子数时,Qh2是否大于等于0.097 5这个阈值来决定此因子的取舍,当满足条件时考虑引进此因子,反之则不引进此因子。其公式如式(1) 所示。
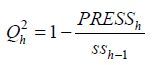
|
(1) |
其中,h为因子数,PRESSh为取h个因子数时,利用除去样本点i的数据集建模后再对该样本点进行预测,并对每个样本点重复上述操作后得到的预测残差平方和,其公式如式(2) 所示。ssh为利用所有样本点,取h个因子数建模时的误差平方和,其公式如式(3) 所示。

|
(2) |
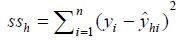
|
(3) |
其中,n为样本数量,h为因子数,yi为实测数据,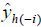
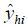
本文使用交叉验证均方根误差 (RMSECV)、校正均方根误差 (RMSEC) 和预测均方根误差 (RMSEP) 来对模型进行评价,其公式如式 (4) 、(5) 和 (6) 所示。
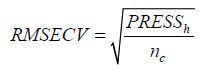
|
(4) |
其中,nc为校正集样本数量,PRESSc为由公式 (2) 计算得到的校正集数据的预测残差平方和。
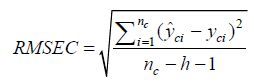
|
(5) |
其中,nc为校正集样本数量,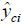

|
(6) |
其中,np为验证集样本数量,
由对近红外光谱的敏感性分析可知,谱段A和谱段B与发酵过程中生物量变化有明显的相关关系,且可以看到相比谱段B,谱段A的光谱吸光度变化范围更大,因而与发酵过程中生物量变化的相关关系更加明显,所以考虑以谱段A为核心谱段来进行谱段选择的建模比较。分别采用了谱段A、谱段A+B、全谱段(4 800-12 000 cm-1)这3种谱段选择方式,并在无预处理,多元散射校正 (MSC),标准正态变量变换 (SNV),SG平滑条件下一阶导数和二阶导数的光谱预处理条件下进行了建模比较。根据不同的谱段选择得到的建模结果分别列在表 6、7、8中。表中使用RMSECV作为评价模型预测偏差的标准 (该值越小说明模型的预测误差越小[25]),使用RMSEP与RMSEC的比值作为评价模型过拟合和欠拟合程度的标准 (该值过大易过拟合,过小易欠拟合,通常认为该值在0.8-1.2范围内时,所建立的模型是可以接受的[25]),使用测定系数 (R2) 作为评价校正集数据实测值与预测值相关性的标准 (该值越接近1说明相关性越好[25])。
| Model | Pretreatment method | RMSECV | RMSEP | RMSEP/RMSEC | Factors | R2 |
| 1 | None | 0.561 5 | 0.449 9 | 0.807 8 | 1 | 0.957 0 |
| 2 | MSC | 0.377 7 | 0.322 0 | 0.896 8 | 3 | 0.982 4 |
| 3 | SNV | 0.363 0 | 0.327 3 | 0.945 4 | 3 | 0.983 6 |
| 4 | The 1st | 0.310 8 | 0.293 8 | 1.086 2 | 4 | 0.990 1 |
| 5 | The 2nd | 0.352 2 | 0.379 6 | 1.199 5 | 4 | 0.986 4 |
| Model | Pretreatment method | RMSECV | RMSEP | RMSEP/RMSEC | Factors | R2 |
| 6 | None | 0.560 8 | 0.449 4 | 0.808 1 | 1 | 0.957 1 |
| 7 | MSC | 0.384 9 | 0.349 7 | 0.955 9 | 2 | 0.981 6 |
| 8 | SNV | 0.369 2 | 0.342 6 | 0.968 1 | 2 | 0.982 7 |
| 9 | The 1st | 0.337 7 | 0.341 5 | 1.039 8 | 2 | 0.985 1 |
| 10 | The 2nd | 0.299 8 | 0.283 6 | 1.048 4 | 4 | 0.990 1 |
分析表 6-8中的数据可以看到,表中所得到的15个模型的参数RMSEP/RMSEC均在0.8-1.2范围内,说明这些模型都没有产生过拟合或欠拟合,但模型1、6、7、8、9、12、13和14通过采用交叉有效性原则选择出的因子数较少,可能会严重影响模型的预测性能。为此,对因子数少于3的模型进行因子数的扩展,以验证是否该原则选择出较少的因子数会明显影响模型预测性能,扩展因子数后的建模结果如表 9、10、11所示。
| Model | Pretreatment method | RMSECV | RMSEP | RMSEP/RMSEC | Factors | R2 |
| 11 | None | 0.288 4 | 0.267 2 | 0.961 9 | 4 | 0.988 4 |
| 12 | MSC | 0.349 0 | 0.328 4 | 0.955 2 | 1 | 0.983 6 |
| 13 | SNV | 0.352 5 | 0.329 8 | 0.949 8 | 1 | 0.983 3 |
| 14 | The 1st | 0.362 4 | 0.383 1 | 1.091 7 | 2 | 0.983 0 |
| 15 | The 2nd | 0.329 3 | 0.324 7 | 1.076 9 | 4 | 0.987 7 |
综合分析表 6-11可以看到,进行因子数扩展后,除对模型1和6稍有一些精度提高外,对其他模型的性能提高均较小,而且对所建立的27个模型进行比较可以看到,未经因子数扩展的模型11所得的交叉验证均方根误差最小且R2和RMSEP/RMSEC均接近1。可知该模型具有较小的预测误差且具有较小的过拟合与欠拟合风险。
| The former model | Model | Pretreatment method | RMSECV | RMSEP | RMSEP/RMSEC | Factors | R2 |
| 1 | 16 | None | 0.540 4 | 0.453 0 | 0.845 0 | 2 | 0.960 4 |
| 17 | 0.385 4 | 0.339 6 | 0.904 3 | 3 | 0.980 7 |
| The former model | Model | Pretreatment method | RMSECV | RMSEP | RMSEP/RMSEC | Factors | R2 |
| 6 | 18 | None | 0.529 3 | 0.451 3 | 0.864 3 | 2 | 0.962 5 |
| 19 | 0.433 7 | 0.374 7 | 0.878 6 | 3 | 0.975 1 | ||
| 7 | 20 | MSC | 0.343 9 | 0.304 5 | 0.931 8 | 3 | 0.985 4 |
| 8 | 21 | SNV | 0.339 1 | 0.311 7 | 0.964 9 | 3 | 0.985 7 |
| 9 | 22 | The 1st | 0.324 3 | 0.291 3 | 0.926 4 | 3 | 0.986 5 |
| The former model | Model | Pretreatment method | RMSECV | RMSEP | RMSEP/RMSEC | Factors | R2 |
| 12 | 23 | MSC | 0.335 2 | 0.312 0 | 0.953 5 | 2 | 0.985 3 |
| 24 | 0.321 2 | 0.299 4 | 1.000 2 | 3 | 0.987 8 | ||
| 13 | 25 | SNV | 0.336 6 | 0.312 5 | 0.951 2 | 2 | 0.985 1 |
| 26 | 0.321 6 | 0.301 8 | 1.002 1 | 3 | 0.987 6 | ||
| 14 | 27 | The 1st | 0.330 1 | 0.291 7 | 0.922 0 | 3 | 0.986 3 |
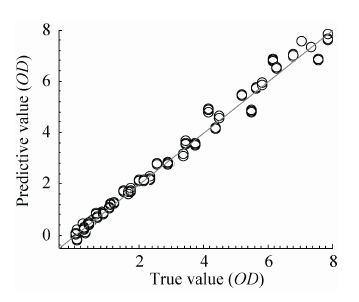
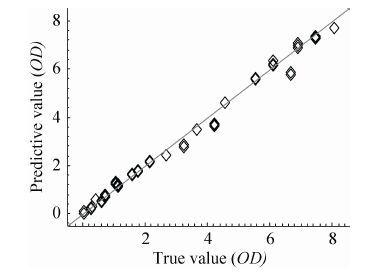
因此,在排除了选择较少因子数导致模型性能严重降低的风险后,本文最终确定模型11为最佳模型。模型11的校正集和验证集的预测值 (此处的预测值为1,3-丙二醇发酵过程生物量在线监测模型给出的结果,后文同) 与实测值 (此处的实测值为使用传统检测方法得到的吸光光度值 (OD),下文同) 比较如图 6和图 7所示。
从图 6和图 7中可以看到,所选择的最佳模型校正集与验证集的预测值与实测值之间具有较小误差,且相关性较好。
|
| 图 6 模型11校正集的预测值与实测值比较 Figure 6 Predictive and true values of calibration set of model 11. |
| |
|
| 图 7 模型11验证集的预测值与实测值比较 Figure 7 Predictive and true values of validation set of model 11. |
| |
选择的最佳模型是利用无光谱预处理的全谱段 (4 800-12 000 cm-1) 近红外光谱得到的。采用全谱段建模说明,在光谱变化不明显的谱段也包含了建模所需要的必要信息。采用含有更多细胞散射信息的原始光谱进行建模说明,细胞散射信息相比于细胞含氢基团的光谱吸收信息,对于准确和可靠的建模具有不可忽视的作用。
4 实验验证通过两个批次的1,3-丙二醇发酵实验 (初始甘油浓度为60 g/L和40 g/L),对本文建立的1,3-丙二醇发酵过程生物量在线监测模型的监测效果和模型的适用性进行验证。在实验中,使用本文建立的模型可以实现对生物量的实时在线监测 (可1 min检测1次数据),相比测量吸光光度值 (OD) 的传统检测方法 (1 h检测1个数据点) 具有明显的实时性优势。从图 8和图 9中可以看到,应用本文建立的模型可以得到与传统方法实测值相比趋势一致且误差较小的实时监测值,并且可以看到本文所建立的模型在培养基物质浓度变化的情况下,也可得到稳定且误差较小的实时监测结果。

|
| 图 8 光谱模型预测值与实测值对比 (甘油60 g/L) Figure 8 Comparison between spectra model prediction and the real measurement (Glycerol 60 g/L). |
| |

|
| 图 9 光谱模型预测值与实测值对比 (甘油40 g/L) Figure 9 Comparison between spectra model prediction and the real measurement (Glycerol 40 g/L). |
| |
模型的在线监测数据及与发酵实验实测数据的比较如表 12所示。对于甘油浓度60 g/L和40 g/L的验证实验,模型的预测值与实测值之间的测定系数 (R2) 分别为0.9976和0.9928,说明预测值与实测值之间具有很好的相关性。预测均方根误差 (RMSEP) 分别为0.3416和0.2743,说明所建立的模型能够较准确的实现对1,3-丙二醇发酵过程生物量的实时在线监测。
| Glycerol 60 g/L | True value | Monitoring value |
| OD | 0.229 3-7.170 2 | 0.529 8-7.675 7 |
| R2 | 0.997 6 | |
| RMSEP | 0.341 6 | |
| Glycerol 40 g/L | True value | Monitoring value |
| OD | 0.338 6-6.941 0 | 0.532 3-6.959 0 |
| R2 | 0.992 8 | |
| RMSEP | 0.274 3 | |
对发酵过程生物量进行在线监测能有效地改善发酵过程的调控优化。本文设计了一个基于近红外光谱分析技术的1,3-丙二醇发酵过程在线监测实验平台,通过对采集的光谱数据进行预处理以及不同谱段的敏感性分析,应用偏最小二乘算法对发酵过程中生物量建立了在线动态监测模型。根据选定的最佳模型,发现细胞散射信息相对于细胞含氢基团的光谱吸收信息,对于准确建模具有重要作用。通过实验测试与离线方法的检测结果对比,验证了本文建立的模型能对该发酵过程生物量进行有效的实时在线监测,相比传统的监测方法具有明显的实时性优势。
| [1] |
Zhuang YP, Chen HZ, Xia JY, et al. Progress in industrial bioprocess engineering in China.
Chin J Biotech, 2015, 31 (6) : 778–796.
(in Chinese). 庄英萍, 陈洪章, 夏建业, 等. 我国工业生物过程 工程研究进展. 生物工程学报, 2015 , 31 (6): 778-796. |
| [2] | Louren?o ND, Lopes JA, Almeida CF, et al. Bioreactor monitoring with spectroscopy and chemometrics: a review. Anal Bioanal Chem, 2012, 404 (4) : 1211–1237. DOI: 10.1007/s00216-012-6073-9 |
| [3] | Landgrebe D, Haake C, H?pfner T, et al. On-line infrared spectroscopy for bioprocess monitoring. Appl Microbiol Biotechnol, 2010, 88 (1) : 11–22. DOI: 10.1007/s00253-010-2743-8 |
| [4] |
Chu XL, Yuan HF, Lu WZ. The on-line near infrared spectroscopy process analytical technique and its applictions.
Mod Sci Instrum, 2004 (2) : 3–21.
(in Chinese). 褚小立, 袁洪福, 陆婉珍. 在线近红外光谱过程 分析技术及其应用. 现代科学仪器, 2004 (2): 3-21. |
| [5] | Arnold SA, Gaensakoo R, Harvey LM, et al. Use of at-line and in-situ near-infrared spectroscopy to monitor biomass in an industrial fed-batch Escherichia coli process. Biotechnol Bioeng, 2002, 80 (4) : 405–413. DOI: 10.1002/(ISSN)1097-0290 |
| [6] | Hall JW, McNeil B, Rollins MJ, et al. Near-infrared spectroscopic determination of acetate, ammonium, biomass, and glycerol in an industrial Escherichia coli fermentation. Appl Spectrosc, 1996, 50 (1) : 102–108. DOI: 10.1366/0003702963906726 |
| [7] | Crowley J, Arnold SA, Wood N, et al. Monitoring a high cell density recombinant Pichia pastoris, fed-batch bioprocess using transmission and reflectance near infrared spectroscopy. Enzyme Microb Technol, 2005, 36(5/6): 621-628. |
| [8] | Cervera AE, Petersen N, Lantz AE, et al. Application of near-infrared spectroscopy for monitoring and control of cell culture and fermentation. Biotechnol Progr, 2009, 25 (6) : 1561–1581. |
| [9] |
Lu WZ. Modern Near Infrared Spectroscopy Analytical Technology. 2nd ed. Beijing: China Petrochemical Press, 2006: 1-11 (in Chinese). 陆婉珍. 现代近红外光谱分析技术. 2 版. 北京: 中国石化出版社, 2006: 1-11. |
| [10] | Huang HB, Yu HY, Xu HR, et al. Near infrared spectroscopy for on/in-line monitoring of quality in foods and beverages: a review. J Food Eng, 2008, 87 (3) : 303–313. DOI: 10.1016/j.jfoodeng.2007.12.022 |
| [11] | Roggo Y, Chalus P, Maurer L, et al. A review of near infrared spectroscopy and chemometrics in pharmaceutical technologies. 2007 : 683 -700(in Chinese). |
| [12] | 褚小立, 陆婉珍. 近五年我国近红外光谱分析技 术研究与应用进展. 光谱学与光谱分析, 2014 , 34 (10): 2595-2605. |
| [13] | Watson DG. Pharmaceutical analysis : a textbook for pharmacy students and pharmaceutical chemists. 2nd ed. London: Elsevier Press, 2005: 128-134. |
| [14] | 桂勇利, 梁静波, 马雷, 等. 基于近红外技术谷氨 酸发酵过程中乳酸浓度预测模型的建立. 食品与 发酵工业, 2014 , 40 (8): 1-6. |
| [15] | Guo WL, Du YP, Zhou YC, et al. At-line monitoring of key parameters of nisin fermentation by near infrared spectroscopy, chemometric modeling and model improvement. World J Microbiol Biotechnol, 2012, 28(3): 993-1002. |
| [16] | Cruz MV, Sarragu?a MC, Freitas F, et al. Online monitoring of P(3HB) produced from used cooking oil with near-infrared spectroscopy. J Biotechnol, 2015, 194 : 1–9. DOI: 10.1016/j.jbiotec.2014.11.022 |
| [17] |
Lu WZ, Yuan HF, Chu XL.
Near Infrared Spectroscopy Instrument. Beijing: Chemical Industry Press, 2010 : 17 -61(in Chinese).
陆婉珍, 袁洪福, 褚小立. 近红外光谱仪器. 北 京: 化学工业出版社, 2010 : 17-61. |
| [18] | Workman J, Weyer L. Practical Guide and Spectral Atlas for Interpretive Near-Infrared Spectroscopy. 2nd ed. New York: CRC Press, 2012: 195-226. |
| [19] | Rinnan ?, Van Den Berg F, Engelsen SB. Review of the most common pre-processing techniques for near-infrared spectra. 2009 : 1201 -1222(in Chinese). |
| [20] | Haaland DM, Thomas EV. Partial least-squares methods for spectral analyses: 2. application to simulated and glass spectral data. Anal Chem, 1988, 60(11): 1202-1208. |
| [21] | Gemperline P. Practical guide to chemometrics. 2nd ed. New York: Crc Press, 2006: 167-216. |
| [22] | Wold S, Sjöström M, Eriksson L. PLS- regression: a basic tool of chemometrics. Chemom Intell Lab Syst, 2001, 58(2): 109-130. |
| [23] |
Wang HW.
Partial least-Squares Regression- Method and Applications. 1999 : 155 -157(in Chinese).
王惠文. 偏最小二乘回归方法及其应用. 北京: 国防工业出版社, 1999 : 155-157. |
| [24] | Menzel K, Zeng AP, Biebl H, et al. Kinetic, dynamic, and pathway studies of glycerol metabolism by Klebsiella pneumoniae, in anaerobic continuous culture: I. (in Chinese). |
| [25] |
Yan YL, Chen B, Zhu DZ, et al.
Near Infrared Spectroscopy-Principles, Technologies and Applications. Beijing: China Light Industry Press, 2013 : 162 -175(in Chinese).
严衍禄, 陈斌, 朱大洲, 等. 近红外光谱分析的原 理、技术与应用. 北京: 中国轻工业出版社, 2013 : 162-175. |
 2017, Vol. ${metaVo.volume}
2017, Vol. ${metaVo.volume}