中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈思, 贾楠, 丁明珠, 元英进
- Chen Si, Jia Nan, Ding Mingzhu, Yuan Yingjin
- 山梨酮脱氢酶模块与酮古龙酸杆菌底盘细胞的适配分析
- Fitness analysis between the L-sorbosone dehydrogenase modules and Ketogulonigenium vulgare chassis
- 生物工程学报, 2016, 32(9): 1224-1232
- Chin J Biotech, 2016, 32(9): 1224-1232
- 10.13345/j.cjb.150523
-
文章历史
- Received: December 10, 2015
- Accepted: January 15, 2016
2 天津化学化工协同创新中心,天津 300072
2 Collaborative Innovation Center of Chemical Science and Engineering (Tianjin), Tianjin 300072, China
维生素C是维生素类药中发展最快、产量最大、用途最广的品种[1]。我国VC二步发酵法由中国科学院微生物研究所和北京制药厂于20世纪70年代初发明。第一步由氧化葡萄糖酸杆菌Gluconobacter oxydans将D-山梨醇氧化为L-山梨糖。第二步由酮古龙酸杆菌Ketogulonigenium vulgare及伴生菌混合培养将L-山梨糖转化为VC的前体2-酮基-L-古龙酸(2-KGA)[2]。K.vulgare作为功能菌完成L-山梨糖到2-KGA的转化,单独生长时十分缓慢且产酸量很低[3],伴生菌负责辅助产酸菌的生长及产酸。其中,山梨糖/山梨酮脱氢酶能将L-山梨糖转化为L-山梨酮,进而转化为维生素C的前体2-KGA。PQQ作为醌酶类辅因子,参与氧化还原反应,调节微生物呼吸链电子传递[4],同时PQQ还作为L-山梨糖/山梨酮脱氢酶的辅因子发挥重要作用。
K.vulgare作为非模式微生物,其遗传背景尚不清晰,基因操作的相关研究有限,糖酸转化的代谢路径不清晰且相关酶系的蛋白结构和催化反应机理不明确,限制了对菌株的工程改造。Cai等[5]利用来源于乳酸菌Lactococcus lactis的叶酸合成基因簇,弥补了K.vulgare体内叶酸代谢路径的缺乏,提高了细胞密度和产酸能力。杜瑾等通过在K.vulgare体内组合表达山梨糖/山梨酮脱氢酶和辅因子PQQ合成路径的模块[6],使混菌发酵的产酸水平提高了20%。但由于基因组信息的缺乏,对关键酶的研究较为局限,该研究中只选用了1个山梨糖脱氢酶基因和1个山梨酮脱氢酶基因。江南大学陈坚课题组通过在氧化葡萄糖酸杆菌内过表达山梨糖脱氢酶和山梨酮脱氢酶,在该菌中实现了由山梨糖到2-KGA的转化过程[7],发酵168 h获得2-KGA 32.4 g/L。此外,本实验室曾借助系统生物学方法对混菌体系进行代谢组学与蛋白组学偶联的解析[8-9],深入挖掘了两菌间相互作用机制。同时,利用混菌培养传代150 d的方式加强混菌的相互作用[10],糖酸转化率提高16%,并借助代谢组[11]与蛋白组[12]成功挖掘了两菌相互强化的分子机制。
本研究中,针对前人较少研究的山梨酮脱氢酶(L-sorbosone dehydrogenase,缩写为SNDH)进行序列比对分析,发现2个山梨酮脱氢酶基因,分别位于基因组(缩写为sndhg)和质粒(缩写为sndhp)上。sndhg和sndhp序列差异很大,同源性只有30.39%。通过在工业应用的产酸菌K.vulgare HKv604中构建两种山梨酮脱氢酶功能模块,比较其对2-KGA产量的影响,同时,结合混菌长期传代加强两菌相互作用的适应性进化策略,让改造后的K.vulgare工程菌株与伴生菌互为进化压力,研究适应性进化实验对2-KGA产量及模块作用的影响。旨在解析两种模块在K.vulgare中的作用机制的同时,增加山梨酮脱氢酶模块与底盘细胞K.vulgare之间的适配,寻找强化K.vulgare产酸路径及削弱缺陷路径的方案。
1 材料与方法 1.1 材料本文所使用的维生素C二步发酵菌株为工业应用菌种酮古龙酸杆菌K.vulgare HKv604与伴生菌内生芽孢杆菌Bacillus endophyticus[13]。构建K.vulgare工程菌株所用的质粒为广宿主低拷贝的pBBR1MCS2[14],由军事科学院微生物研究所张惟材教授惠赠。脱氧核糖核苷酸(dATP、dTTP、dGTP和dCTP)和Taq DNA聚合酶购自北京天根生物技术有限公司。限制性内切酶和连接反应试剂为TaKaRa公司产品,质粒小量抽提试剂盒、琼脂糖凝胶DNA回收试剂盒、基因组提取试剂盒和PCR产物纯化试剂盒为北京天根生物技术有限公司产品。其他化学试剂均为国产分析纯。
1.2 方法 1.2.1 工程菌株构建本实验室先前对菌株K.vulgare Hbe602进行了全基因组测序,包含染色体和两个质粒,结果递交于NCBI库中(CP012908、CP012909、CP012910)。K.vulgare编码2个山梨酮脱氢酶L-sorbosone dehydrogenase,分别位于染色体DNA (简称sndhg)和质粒2 (简称sndhp)上,GI号分别为939480045和939479492,Blast结果显示两个基因间的同源性只有30.39 %。根据基因序列设计引物(表 1),扩增菌株K.vulgare基因组上两个山梨酮脱氢酶基因及PQQ合成基因pqqA,构建组合模块(图 1)。通过电转化法将含有目的基因的质粒导入K.vulgare HKv604中,在含卡那霉素的抗性平板筛选阳性转化子提质粒进行酶切验证。获得了过表达sndhg、sndhp、sndhg-pqqA和sndhp-pqqA的4株K.vulgare工程菌株,分别命名为SyBE_Kv0001116021、SyBE_Kv0001116022、SyBE_Kv00011160210和SyBE_Kv00011160220。
| Prime names | Sequence (5' -3' ) | Restriction sites |
| SNDHGF | CGGggtaccGCCACCAGCATCCAGCAA | KpnⅠ |
| SNDHGR | CCCaagcttATAGGCAGAGGCGTCAAAAC | Hind Ⅲ |
| SNDHPF | CGGggtaccCTGATCCAGTAAGCCACCA | KpnⅠ |
| SNDHPR | CCCaagcttGCCGCATTAACCGTGATAG | Hind Ⅲ |
| pqqAF | GGactagtGCGGAGGAAACCGATCCATT | SpeⅠ |
| pqqAR | ACgagctcTCAGCTATCCGCGCGTGCAT | SacⅠ |
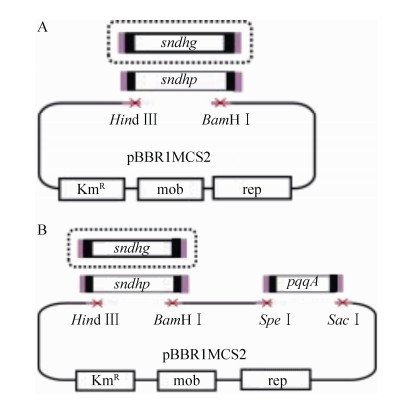
|
| 图 1 工业K.vulgare中L-山梨酮脱氢酶模块(A)及其与辅因子合成基因pqqA组合的功能模块(B)的构建 Figure 1 Construction of the functional modules including (A) genes encoding L-sorbosone dehydrogenase (sndhg, sndhp) and (B) genes encoding L-sorbosone dehydrogenase with PQQ (sndhg-pqqA, sndhp-pqqA) in K.vulgare. |
| |
种子培养基(1 L培养基):玉米浆3 g,牛肉膏3 g,酵母膏3 g,尿素1 g,蛋白胨10 g,KH2PO4 1 g,MgSO4 0.2 g,CaCO3 1 g,定容至0.8 L。配制固体培养基还需添加琼脂20 g。调节pH 6.8,121 ℃灭菌20 min。另称取20 g/L-山梨糖定容至0.2 L单独灭菌,接菌前添加至种子培养基中。
发酵培养基(1 L培养基):玉米浆15 g,尿素12 g,KH2PO4 1 g,MgSO4 0.5 g,CaCO3 2 g,定容至0.8 L,调节pH 6.8-7.0,121 ℃灭菌20 min。另称取80 g山梨糖定容至0.2 L单独灭菌,接菌前添加至发酵培养基中。
1.2.3 酮古龙酸杆菌单菌发酵将活化的菌株用无菌水从平板上吹洗下来,加入50 mL新鲜培养基(含50 μg/mL卡那霉素)内,在30 ℃、250 r/min摇床中培养48 h,转接培养24 h,获得种子液。测定种子液的细胞浓度,计算初始接菌浓度为OD600=0.3所需的种子液体积,加入新鲜培养基中发酵。
1.2.4 底物与产物的检测本研究中底物与产物的测定选用高效液相色谱法(HPLC)。取菌液1 mL,12 000 r/min离心5 min,将上清转移至离心管中,用0.5 mol/L硫酸溶液稀释适当的倍数,水系滤膜过滤后,通过HPLC进行检测。色谱柱选用Bio-Rad HPX-87H,流动相为0.5 mol/L H2SO4,流动相流速为0.6 mL/min,柱温设定为65 ℃,示差检测器检测样品。
1.2.5 混菌传代驯化与发酵改造后的K.vulgare和B.endophyticus通过平板活化和种子液培养获得活性较好的菌种。按初始OD600比为3:1进行种子液混合,接入有50 mL新鲜种子培养液的250 mL摇瓶中进行混菌培养,30 ℃、250 r/min摇床振荡培养24 h。取2 mL混菌培养液转接到50 mL新鲜种子培养基中在相同的条件下进行培养,连续传代50 d取转接后剩余的混菌培养液1 mL,12 000 r/min离心5 min,将上清移至离心管中用于2-KGA的HPLC测定,菌体经1 mL 0.5 mol/L HCl处理用于混菌OD600的测定。对第50天的混菌分纯,将50代K.vulgare与伴生菌B.endophyticus配合混菌发酵,测定产酸量和OD600值。
2 结果与讨论 2.1 山梨酮脱氢酶功能模块对K.vulgare的影响利用传统的分析手段和基因工程手段,研究者们解析了K.vulgare中将山梨糖转化为古龙酸的反应途径及相关酶学特性[15-16],认为山梨酮脱氢酶对参与山梨糖转化为2-KGA的过程中起到重要作用。本文的研究工作通过对比两个山梨酮脱氢酶模块的效果,进一步认识其功能。将工程菌SyBE_Kv0001116021和SyBE_Kv0001116022进行单菌发酵,底物L-山梨糖的浓度为20 g/L。底物消耗和产物2-KGA浓度随时间的变化如图 2所示。在48 h发酵过程中菌株的耗糖速率基本相同,细胞的生长速率也没有太大差别。发酵到36 h后,不再消耗L-山梨糖,2-KGA的产量也趋于稳定。过表达山梨酮脱氢酶基因的菌株产酸量都低于对照菌株,过表达sndhp的菌株生产2-KGA的能力最弱。通过上述数据可知,过表达sndhp并不影响细胞对底物山梨糖的吸收利用和转化,但是山梨糖流向副产物途径的通量较大,导致2-KGA产量明显降低。图 3是24 h测得改造菌株单菌发酵菌液的HPLC结果,底物L-山梨糖的出峰时间为9.50 min,产物峰为7.87 min。观察可知过表达sndhg对L-山梨糖代谢路径没有太大影响。过表达sndhp的菌株发酵液在7.40 min出现了副产物峰,耗糖量并没有降低。本文采用的高效液相色谱87H柱子上离得近的两种物质一般结构比较接近,从图 3可以肯定该副产物与目标产物2-KGA的性质较为接近,这与本研究确定的山梨酮脱氢酶的特异性较差的结论较为一致,而之前的文献报道有提到一种副产物L-艾杜糖酸[17],本研究得到的也有可能是类似物质。结果表明sndhp过表达编码的蛋白酶能很好地利用底物L-山梨糖,但底物有相当一部分被转化为副产物,进一步说明sndhp编码的山梨酮脱氢酶对L-山梨糖转化2-KGA反应专一性较差。
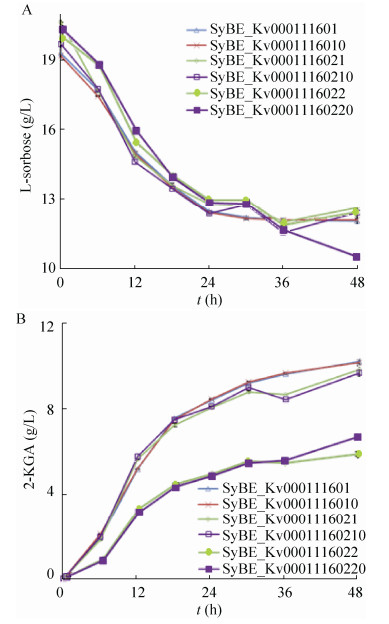
|
| 图 2 山梨酮脱氢酶模块对K.vulgare耗糖(A)与产酸(B)的影响 Figure 2 Influence of sndhg (SyBE_Kv0001116021), sndhp (SyBE_Kv0001116022), sndhg-pqqA (SyBE_ Kv00011160210), sndhp-pqqA (SyBE_Kv00011160220) modules on (A) sorbose consumption and (B) 2-KGA production of K.vulgare. |
| |
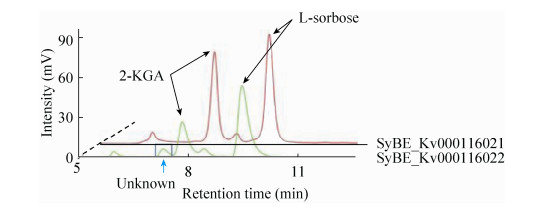
|
| 图 3 改造K.vulgare发酵产物的HPLC检测 Figure 3 HPLC analysis of the engineered strains. |
| |
醌酶与其辅因子PQQ的合成是两个相对独立的过程。大肠杆菌中不含合成PQQ的基因,但有编码PQQ依赖型葡萄糖脱氢酶的基因[18]。耐辐射球菌Deinococcus radiodurans中具有合成PQQ相关基因而不含醌酶合成基因[19]。而K.vulgare中同时具有醌酶和PQQ合成基因,考虑到山梨酮脱氢酶属于醌酶家族,在参与氧化还原反应的过程中需要配合辅因子PQQ进行物质的氧化,单独过表达山梨酮脱氢酶可能会导致细胞体内辅因子不平衡,从而使氧化还原反应受阻。在大肠杆菌、酿酒酵母等模式生物中,针对NADH、NADPH等辅因子的代谢调控已取得显著效果[20-21]。为此,本研究进行了功能模块与辅因子配合表达。
过表达sndhg的菌株SyBE_Kv0001116021,发酵48 h消耗底物0.402 g,2-KGA的产量为9.83 g/L;过表达模块sndhg-pqqA的菌株SyBE_ Kv00011160210消耗底物0.365 g,2-KGA的产量为9.68 g/L。可能是辅因子配合带来的积极影响与辅因子过表达对细胞生长造成的负担相互抵消,导致总体无影响。混菌发酵120 h,原始菌株SyBE_Kv000111601与伴生菌混菌发酵的产酸量为70.5 g/L,含空载体的菌株SyBE_Kv0001116010与伴生菌混菌发酵的产酸量为69.1 g/L,过表达基因sndhg菌株混菌发酵的产酸量为66.3 g/L,过表达sndhg-pqqA的菌株混菌发酵的产酸量为67.6 g/L (图 4)。改造菌株的混菌发酵产酸量较对照菌株低,可能由于代谢负担造成。

|
| 图 4 山梨酮脱氢酶模块及其与辅酶合成模块组合对混菌发酵生产2-KGA的影响 Figure 4 Effects of sndh modules on the 2-KGA production and L-sorbose consumption in the consortium. |
| |
sndhp配合辅因子合成基因pqqA获得的菌株SyBE_Kv00011160220的耗糖速率有了较大提高。过表达sndhp的菌株SyBE_Kv0001116022发酵48 h后消耗底物0.367 g,2-KGA产量为5.75 g/L;过表达sndhp-pqqA的菌株SyBE_Kv00011160220底物消耗为0.477 g,2-KGA的产量为6.55 g/L,提升了13.9%。说明辅因子的加入对sndhp编码的山梨酮脱氢酶参与的氧化还原反应有积极作用。混菌发酵结果,原始菌株混菌发酵的产酸量为70.5 g/L,含有空载体的菌株混菌发酵的产酸量为69.1 g/L,过表达sndhp菌株混菌发酵的产酸量为7.41 g/L,过表达sndhp-pqqA的菌株混菌发酵的产酸量为7.42 g/L。辅因子合成功能模块对sndhp编码的山梨酮脱氢酶催化反应无明显作用。
2.3 混菌配合适应性进化对混菌发酵的影响适应性进化与理性设计改造策略不同,能在大量不同基因出现突变,有效提高功能模块与底盘细胞间的适配性[22]。运用合成生物技术构建工程菌株生产生物燃料、生物化学品等工作中,结合适应性进化能显著提高菌株适应性、底物利用效率和生产速率[23-24]。为此,将改造后的K.vulgare与伴生菌配合进行适应性进化实验,图 5为进化50 d过程中4个工程菌混菌体系2-KGA的变化情况。图中曲线的断点是因为在传代过程中发生了染菌情况,通过对其中的两菌进行了分离纯化后重新混合传代所致。曲线均存在一定的波动,但总体上趋于稳定。适应性进化的机制是生物体随外界环境条件的改变而改变了自身的特性或生活方式,适应于新环境的基因型会留下更多的子代,从而不断进化。以前研究表明,二步混菌生产维生素C的发酵体系中,K.vulgare与其伴生菌内生芽孢杆菌既存在着代谢物质上的交流,营养物质和生存空间上的竞争[9]。伴生菌在生长和产芽孢过程中释放促进产酸菌生长和产酸[25],K.vulgare能够降解培养基中的蛋白为伴生菌提供大量氨基酸,通过促进伴生菌快速生长并进入衰亡期产生芽孢[26]。借助产酸菌与伴生菌的相互作用,通过传代的方式,互为环境压力,使得两菌配合更为默契,从而得到更优化的发酵体系。分离菌株进行混菌发酵,经过传代进化实验菌株SyBE_Kv0001116021的2-KGA的产量提高了15.4%,菌株SyBE_Kv0001116022的2-KGA产量提升179%。菌株SyBE_Kv00011160210在进化前后2-KGA的产量并未发生明显改变,菌株SyBE_Kv00011160220进化后2-KGA产量提高了125% (图 6)。由上述结果可看出适应性进化对菌株的作用效果明显,能很好地削弱副产物路径的作用,提高混菌产酸效率。
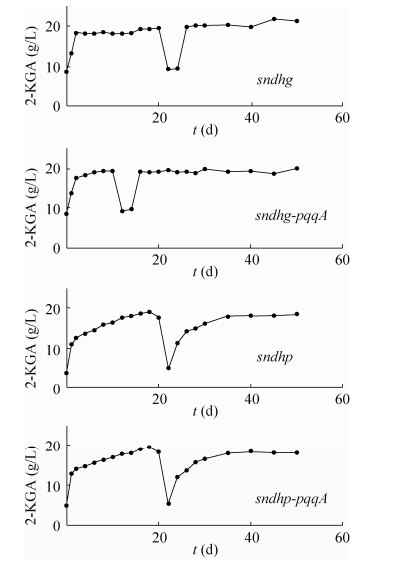
|
| 图 5 混菌传代50 d过程中2-KGA产量 Figure 5 The 2-KGA production of mixed culture in the process of sub-cultivation for 50 d. |
| |

|
| 图 6 传代菌株混菌发酵产酸图 Figure 6 2-KGA production of the sub-cultivated K.vulgare and B.endophyticus consortium. |
| |
本文在工业K.vulgare中构建L-山梨酮脱氢酶基因sndhg、sndhp及其与辅因子组合的sndhg-pqqA和sndhp-pqqA模块,期望通过产酸功能模块与辅因子合成模块的适配性研究,比较其作用差异。研究发现,K.vulgare单独发酵时,配合辅因子模块组合sndhg-pqqA与功能模块单独表达sndhg对比无明显区别,而混菌发酵时sndhg-pqqA过表达使得产酸量获得了少量的提升。sndhp-pqqA工程菌单独发酵时产酸量较sndhp提升13.9%,然而混菌发酵无明显效果。结合适应性进化策略,将上述改造的K.vulgare工程菌株与伴生菌B.endophyticus混合传代培养,两菌间互为动态进化压力。混菌系统在50 d传代完成后,2-KGA的转化率得到了一定的提升,2-KGA的产量分别提高15.4%、179%、0.65%和125%。从另一角度说明该混菌体系中两菌的共同进化作用,使该模块产生副产物的作用削弱。由此可见,混菌适应性进化策略是一种增加功能模块与底盘细胞适配性,进而快速获得优良性状菌种的有效方法。
| [1] | Chauhan AS, Ramteke RS, Eipeson WE. Properties of ascorbic acid and its applications in food processing: a critical appraisal. J Food Sci Technol,1998, 35 (5) : 381 –392. |
| [2] | Yin GL, He JM, Ren SX, et al. Production of vitamin C precursor-2-keto-L-gulonic acid from L-sorbose by a novel bacterial component system of SCB329-SCB933. Ind Microbiol,1997, 27 : 1 –7. |
| [3] | Ma Q, Zhang WW, Zhang L, et al. Proteomic analysis of Ketogulonicigenium vulgare under glutathione reveals high demand for thiamin transport and antioxidant protection. PLoS ONE,2012, 7 (2) : e32156 . DOI: 10.1371/journal.pone.0032156 |
| [4] |
Wang X, Zhang WC. Advances in biosynthesis of pyrroloquinoline quinone.
Lett Biotechnol,2007, 18 (3) : 534 –538.
(in Chinese). 王歆, 张惟材. 吡咯喹啉醌生物合成研究进展. 生物技术通讯, 2007 , 18 (3) : 534-538. |
| [5] | Cai L, Yuan MQ, Li ZJ, et al. Genetic engineering of Ketogulonigenium vulgare for enhanced production of 2-keto-L-gulonic acid. J Biotechnol,2012, 157 (2) : 320 –325. DOI: 10.1016/j.jbiotec.2011.12.004 |
| [6] | Du J, Bai W, Song H, et al. Combinational expression of sorbose/sorbosone dehydrogenases and cofactor pyrroloquinoline quinone increases 2-keto-L-gulonic acid production in Ketogulonigenium vulgare-Bacillus cereus consortium. Metab Eng,2013, 19 : 50 –56. DOI: 10.1016/j.ymben.2013.05.006 |
| [7] | Gao LL, Hu YD, Liu J, et al. Stepwise metabolic engineering of Gluconobacter oxydans WSH-003 for the direct production of 2-keto-L-gulonic acid from D-sorbitol. Metab Eng,2014, 24 : 30 –37. DOI: 10.1016/j.ymben.2014.04.003 |
| [8] | Ma Q, Zhou J, Zhang W, et al. Integrated proteomic and metabolomic analysis of an artificial microbial community for two-step production of vitamin C. PLoS ONE,2011, 6 (10) : e26108 . DOI: 10.1371/journal.pone.0026108 |
| [9] | Du J, Zhou J, Xue J, et al. Metabolomic profiling elucidates community dynamics of the Ketogulonicigenium vulgare-Bacillus megaterium consortium. Metabolomics,2012, 8 (5) : 960 –973. DOI: 10.1007/s11306-011-0392-2 |
| [10] | Zou Y, Hu ML, Lv YJ, et al. Enhancement of 2-keto-gulonic acid yield by serial subcultivation of co-cultures of Bacillus cereus and Ketogulonigenium vulgare. Bioresource Technol,2013, 132 : 370 –373. DOI: 10.1016/j.biortech.2012.10.151 |
| [11] | Ding MZ, Zou Y, Song H, et al. Metabolomic analysis of cooperative adaptation between co-cultured Bacillus cereus and Ketogulonicigenium vulgare. PLoS ONE,2014, 9 (4) : e94889 . DOI: 10.1371/journal.pone.0094889 |
| [12] | Ma Q, Zou Y, Lv YJ, et al. Comparative proteomic analysis of experimental evolution of the Bacillus cereus-Ketogulonicigenium vulgare co-culture. PLoS ONE,2014, 9 (3) : e91789 . DOI: 10.1371/journal.pone.0091789 |
| [13] | Jia N, Du J, Ding MZ, et al. Genome sequence of Bacillus endophyticus and analysis of its companion mechanism in the Ketogulonigenium vulgare-Bacillus strain consortium. PLoS ONE,2015, 10 (8) : e0135104 . DOI: 10.1371/journal.pone.0135104 |
| [14] | Kovach ME, Phillips RW, Elzer PH, et al. pBBR1MCS: a broad-host-range cloning vector. Biotechniques,1994, 16 (5) : 800 . |
| [15] | Asakura A, Hoshino T. Isolation and characterization of a new quinoprotein dehydrogenase, L-sorbose/L-sorbosone dehydrogenase. Biosci Biotech Biochem,1999, 63 (1) : 46 –53. DOI: 10.1271/bbb.63.46 |
| [16] | Miyazaki T, Sugisawa T, Hoshino T. Pyrroloquinoline quinone-dependent dehydrogenases from Ketogulonicigenium vulgare catalyze the direct conversion of L-sorbosone to L-ascorbic acid. Appl Environ Microb,2006, 72 (2) : 1487 –1495. DOI: 10.1128/AEM.72.2.1487-1495.2006 |
| [17] | Zou W, Liu LM, Zhang J, et al. Reconstruction and analysis of a genome-scale metabolic model of the vitamin C producing industrial strain Ketogulonicigenium vulgare WSH-001. J Biotechnol,2012, 161 (1) : 42 –48. DOI: 10.1016/j.jbiotec.2012.05.015 |
| [18] | Blattner FR, Plunkett G, Bloch CA, et al. The complete genome sequence of Escherichia coli K-12. Science,1997, 277 (5331) : 1453 –1462. DOI: 10.1126/science.277.5331.1453 |
| [19] | Shrivastava M, Rajpurohit YS, Misra HS, et al. Survival of phosphate-solubilizing bacteria against DNA damaging agents. Can J Microbiol,2010, 56 (10) : 822 –830. DOI: 10.1139/W10-067 |
| [20] | San KY, Bennett GN, Berrios-Rivera SJ, et al. Metabolic engineering through cofactor manipulation and its effects on metabolic flux redistribution in Escherichia coli. Metab Eng,2002, 4 (2) : 182 –192. DOI: 10.1006/mben.2001.0220 |
| [21] | Heux S, Cachon R, Dequin S. Cofactor engineering in Saccharomyces cerevisiae: expression of a H2O-forming NADH oxidase and impact on redox metabolism. Metab Eng,2006, 8 (4) : 303 –314. DOI: 10.1016/j.ymben.2005.12.003 |
| [22] | Portnoy VA, Bezdan D, Zengler K. Adaptive laboratory evolution--harnessing the power of biology for metabolic engineering. Curr Opin Biotech,2011, 22 (4) : 590 –594. DOI: 10.1016/j.copbio.2011.03.007 |
| [23] | Hong KK, Nielsen J. Adaptively evolved yeast mutants on galactose show trade-offs in carbon utilization on glucose. Metab Eng,2013, 16 : 78 –86. DOI: 10.1016/j.ymben.2013.01.007 |
| [24] | Peng BY, Shen Y, Li XW, et al. Improvement of xylose fermentation in respiratory-deficient xylose-fermenting Saccharomyces cerevisiae. Metab Eng,2012, 14 (1) : 9 –18. DOI: 10.1016/j.ymben.2011.12.001 |
| [25] | Zhang J, Liu J, Shi ZP, et al. Manipulation of B.megaterium growth for efficient 2-KLG production by K.vulgare. Process Biochem,2010, 45 (4) : 602 –606. DOI: 10.1016/j.procbio.2009.11.016 |
| [26] | Zhou J, Ma Q, Yi H, et al. Metabolome profiling reveals metabolic cooperation between Bacillus megaterium and Ketogulonicigenium vulgare during induced swarm motility. Appl Environ Microb,2011, 77 (19) : 7023 –7030. DOI: 10.1128/AEM.05123-11 |
 2016, Vol. 32
2016, Vol. 32




