服务
文章信息
- 陈阿娜, 刘秀霞, 戴晓峰, 詹锦玲, 彭枫, 李璐, 王芬, 李松, 杨艳坤, 白仲虎
- A’na Chen, Xiuxia Liu, Xiaofeng Dai, Jinling Zhan, Feng Peng, Lu Li, Fen Wang, Song Li, Yankun Yang, and Zhonghu Bai
- N 端截短对嗜酸普鲁兰芽孢杆菌普鲁兰酶酶学特性及功能的影响
- Effect of N-terminal truncation of Bacillus acidopullulyticus pullulanase on enzyme properties and functions
- 生物工程学报, 2016, 32(3): 355-364
- Chin J Biotech, 2016, 32(3): 355-364
- 10.13345/j.cjb.150322
-
文章历史
- Received: July 13, 2015
- Accepted: September 8, 2015
2 安徽工程大学 生物与化学工程学院,安徽 芜湖 241000
2 School of Biochemical Engineering, Anhui Polytechnic University, Wuhu 241000, Anhui, China
普鲁兰酶是一类淀粉脱支酶,可高效切割多聚葡萄糖类底物分支部位的α-1,6-糖苷键[1]。该酶常与糖化酶复配使用,应用于淀粉糖化过程中:普鲁兰酶切割淀粉α-1,6-糖苷键分支点,糖化酶切割α-1,4-糖苷键连接的线性低聚糖[2-3]。两者协同作用可有效提高淀粉利用率和糖化得率,并显著缩短糖化时间[4-5]。在特定条件下,普鲁兰酶具有反向合成α-1,6-糖苷键功能,可用于获得具有特殊结构和功能的多糖[6-7],在制药领域具有潜在的应用价值。目前已获得许多微生物编码的普鲁兰酶[8-11],其中来源于嗜酸普鲁兰芽孢杆菌Bacillus acidopullulyticus的普鲁兰酶 (BaPul)酶学性质 (主要是耐热、耐酸性和比酶活) 能较好地满足淀粉糖化过程需求,已实现工业化生产[12]。
BaPul成熟蛋白由921个氨基酸组成,各结构域的连接顺序为CBM41-X45a-X25-X45b-CBM48-GH13。CBM为碳水化合物结合结构域,X是未知功能结构域,GH13是CAZY GH13结构域,其本身由GH13家族 (又称α-淀粉酶家族) 典型的A、B和C三个结构域组成[13-16]。空间结构上,CBM48和GH13结合紧密,它们组成的结构与异淀粉酶相似,可能具有淀粉脱支酶完整的催化功能;处于N端的CBM41、X45和X25与催化功能域连接较为松散,推测这3个结构域可能与大分子底物的结合相关。对其他种属的Ⅰ型普鲁兰酶研究表明,N端结构域的切除并不影响普鲁兰酶对小分子糊精的脱支功能,不影响普鲁兰酶在淀粉糖化过程中的实际应用[17-19]。
因而当前工作中,我们依据BaPul 3D结构 (PDB code 2WAN) 构建不同形式的N端截短突变体,对比考察截短突变体与野生型普鲁兰酶 (WT) 在可溶性表达水平、酶学特性和淀粉糖化过程中应用的差异。为优化/简化BaPul编码基因,解析结构域与功能的关系以及探索酶与底物协同作用机制等方面的研究工作奠定基础。
1 材料与方法 1.1 菌株、质粒和基因大肠杆菌BL21 (DE3) 和质粒pelB-pET28a (+) 分别用于普鲁兰酶及其截短突变体基因重组表达的宿主菌和质粒。pelB-pET28a (+) 的质粒骨架是pET28a (+),pelB信号肽来源于pET22b (+)。构建过程中将pET28a (+) 上Bgl Ⅱ到XhoⅠ之间的片段替换为pET22b (+) 上的等位片段。BaPul全长基因 (GenBank Accession No. Ax203843.1) 及其截短突变体基因由Invitrogen上海公司合成,并连接于pMD18-T载体上。
1.2 重组载体的构建以含有目标基因的pMD18-T载体为模板,通过PCR方法扩增出目标基因。PCR反应条件为:98 ℃预变性2 min;98 ℃变性8 s,65 ℃退火30 s,72 ℃延伸1-2 min,30个循环;72 ℃终延伸2 min。采用限制性内切酶NdeⅠ及 XhoⅠ对扩增产物及pelB-pET28a (+) 表达载体进行双酶切。酶切产物经T4 DNA连接酶连接后,转化BL21 (DE3) 感受态细胞。所用引物及其相关信息见表 1。
| Primer name | Nucleotide sequence (5´-3´)a | Gene |
| WT[F]b | GGGAATTCCATATGGATTCTACTTCGACTAAAGTTATTGTTC | BaPul |
| M1[F] | GGGAATTCCATATGCCATCTGTTTCAAATGCCTATCTTG | BaPul-ΔCMB41 |
| M2[F] | GGGAATTCCATATGGATTCTACTTCGACTAAAGTTATTGTTC | BaPul-ΔX45 |
| M3[F] | GGGAATTCCATATGGATTCTACTTCGACTAAAGTTATTGTTC | BaPul-ΔX25 |
| M4[F] | GGGAATTCCATATGGTAACTGCCGTTCTTGTTGGAGATTTAC | BaPul-ΔCMB41ΔX45 |
| M5[F] | GGGAATTCCATATGCCATCTGTTTCAAATGCCTATCTTG | BaPul-ΔCMB41ΔX25 |
| M6[F] | GGGAATTCCATATGGATTCTACTTCGACTAAAGTTATTGTTC | BaPul-ΔX25ΔX45 |
| [R]c | CCGCTCGAGTTGTTTGAGAATAAGCGTACTTATAG | |
| a Italicized fonts indicate the recognition sites of the corresponding restriction enzymes. b [F] and [R] mean the forward and reverse primers,respectively. c The reverse primers of all variants are the same. | ||
种子培养基采用LB:10 g/L蛋白胨,5 g/L酵母浸出物,和10 g/L NaCl。发酵培养基采用改进的TB:12 g/L蛋白胨,24 g/L酵母浸出物,2.31 g/L KH2PO4,9.85 g/L K2HPO4,和10 g/L甘油,pH 7.0。10 mL培养基装入100 mL三角瓶中灭菌,并在使用前加入卡那霉素至终浓度50 μg/mL。
取-80 ℃保存的菌种按0.1%的接种量接入种子培养基中,置于摇床上37 ℃、230 r/min培养10 h。取种子培养液按1%的接种量接入发酵培养基中,置于摇床上37 ℃、230 r/min培养5.5 h (此时的OD600约为5-6);加入IPTG至终浓度0.1 mmol/L,并下调培养温度至20 ℃,诱导目标蛋白表达,诱导时间18-20 h。
1.4 组分分离取1.0 mL发酵终点的发酵液,4 000 × g、4 ℃离心30 min。收集离心上清并定义该组分为胞外组分。将离心所得菌体用10 mmol/L PBS (pH 7.4) 稀释至OD600 = 4.0-5.0后,用超声破碎方法破碎菌体。超声条件为25%功率,超2 s停2 s,总超声时间6 min。将超声破碎液12 000 × g、4 ℃离心10 min,收集离心上清定义为胞内可溶组分和离心碎片定义为胞内不溶组分[20]。所有组分之和为全细胞蛋白。
1.5 酶活分析普鲁兰酶酶活力测定采用3,5-二硝基水杨酸方法 (简称DNS方法)[21]。取适当稀释的酶液0.1 mL加入到1.9 mL 1%的普鲁兰溶液中,置于60 ℃水浴中温浴10 min,迅速加入3 mL DNS试剂终止酶解反应,并于沸水浴中煮沸 7 min进行显色反应。将上述反应液置于冰水中冷却,并加入20 mL蒸馏水,混匀后于540 nm测定吸光度值。采用pH 5.0的醋酸-醋酸钠配制1%的普鲁兰溶液。1个酶活力单位定义为上述测定条件下,每分钟释放1 μmol还原糖所需的酶量 (还原糖以葡萄糖为标准计)。
1.6 蛋白质纯化及定量目标蛋白C端连接His-tag,纯化采用Ni+柱亲和层析方法,具体操作参见文献[22]。蛋白纯度采用SDS-PAGE分析。蛋白定量采用Bradford方法[23],具体操作参见改良型Bradford蛋白浓度测定试剂盒 (生工生物工程上海股份有限公司)。
1.7 最适pH及酸碱稳定性最适pH测定采用不同pH值的缓冲液配制1%的普鲁兰溶液、重悬和稀释酶液,用于酶活测定。酸碱稳定性测定采用不同pH值的缓冲液重悬和稀释酶液,于4 ℃冰箱放置24 h,在标准条件下测定酶活。以最高酶活为100%,其余pH值下的酶活以相对百分比表示。各pH值所用缓冲液:甘氨酸-盐酸 (pH 3.0,100 mmol/L),柠檬酸-柠檬酸钠 (pH 3.5和5.5,100 mmol/L),醋酸-醋酸钠 (pH 4.0-5.0,100 mmol/L),和Na2HPO4-NaH2PO4(pH 6.0-7.5,100 mmol/L)。
1.8 最适温度及热稳定性最适温度的测定在水浴温度为40-80 ℃条件下测定酶活,其余参数同标准条件。温度稳定性测定时将酶液在60 ℃温浴,每隔一定时间取样测定残余酶活。以未经温浴的酶液酶活作为100%,其余酶活以相对百分比表示。以ln (残余酶活%) 对温浴时间作图,采用线性回归方法求得一级反应速率常数kd。半衰期t1/2=ln2/kd[24]。
1.9 动力学参数用100 mmol/L的醋酸-醋酸钠 (pH 5.0) 配制浓度为0.25-16.0 mg/mL (0.25、0.5、1.0、2.0、4.0、6.0、8.0、10.0、12.0、14.0和16.0) 的普鲁兰溶液,并在标准条件下测定酶活[25]。以反应速率对底物浓度作图,采用非线性回归方法求得Vmax和Km。kcat = Vmax/[E],其中[E]是酶浓度。
1.10 底物特异性及应用用于测定底物特异性的底物浓度为0.25%。以普鲁兰为底物时的酶活计为100%,其余酶活以相对百分比表示。
配制干基浓度为30%的玉米淀粉乳 (按15 L配制),调节pH值至5.5-6.2,加入1 mmol/L氯化钙,加入高温酸性α-淀粉酶 (10-15 U/g干基),在剧烈搅拌下,先加热至72 ℃,保温15 min,再加热至95 ℃,并维持60 min,以达到所需的液化程度(DE值:12-15%),碘反应呈棕红色。液化结束后,迅速将料液用盐酸将pH调至4.2-4.5,并升温至120 ℃,保持10 min,以灭活酶。糖化过程温度下调为60 ℃,加入糖化酶 (120 U/g干基) 和普鲁兰酶 (0.5 U/g干基)。60 ℃保温若干小时后,当用无水酒精检验无糊精存在时,糖化过程结束。将料液pH调至5.0,同时在沸水浴中煮沸10 min,以灭活酶[26]。
2 结果与讨论 2.1 截短突变体的设计依据BaPul的3D结构设计了6种截短突变体,分别是缺失CBM41的变体M1,缺失X45 (包括X45a和X45b) 的变体M2,缺失X25的变体M3,缺失CBM41和X45的变体M4,缺失CBM41和X25的变体M5,和缺失X45和X25的变体M6。野生型及其截短突变体一级结构连接顺序及各结构域的划分见图 1。

|
| 图1 野生型及其截短突变体结构示意图 Figure1 Schematic representation of the WT and the variants. Different color represents different structural domain. Numbers represent the first residues of each domain. The names of the variants are indicated on the left. The predicted molecular sizes are indicated on the right. |
| |
将野生型普鲁兰酶及截短突变体的编码基因亚克隆至pelB-pET28a (+),并转化至BL21 (DE3) 中进行诱导表达。由于缺乏信号肽引导,所有目标蛋白定位于细胞质中,发酵液上清中无可检测酶活。发酵终点时缺失CBM41的M1和M5酶活高于其他重组菌株,分别达到84.6 U/mL和71.0 U/mL,野生型酶活仅为29.1 U/mL,缺失X25的M3酶活与野生型相当。M2、M4和M6发酵液上清和胞内均未检测到酶活,这3个重组菌株的共同特征是切除了X45结构域。SDS-PAGE电泳结果显示,M1和M5的可溶性表达量明显高于M3和WT;缺失X45的M2、M4和M6可溶性蛋白少 (图 2)。X45结构域可能在促进催化结构域正确折叠或维持催化结构域正确构象中发挥作用[17]。后续实验将不再涉及这3个突变体。

|
| 图2 野生型普鲁兰酶及其截短突变体全细胞蛋白的可溶性组分SDS-PAGE Figure2 The total cell protein SDS-PAGE analysis of the WT and the variants. M: protein molecular mass markers; 1: control (vector pET28a(+) without foreign gene); 2: M1; 3: M2; 4: M3; 5: M4; 6: M5; 7: M6; 8: WT. The values are molecular sizes in kilodaltons. |
| |
为研究酶学特性的变化,对野生型及其截短突变体进行了纯化。细胞经超声破碎后,12 000 × g、4 ℃离心10 min,去除细胞碎片及不溶物,离心上清过Ni+柱进行纯化。SDS-PAGE分析结果显示目标蛋白实现电泳纯,可用于酶学性质的测定 (图 3)。纯化后产物的比酶活测定结果表明截短突变体与野生型酶差异较小,因而总酶活比值可近似代表表达量比值。蛋白定量结果表明可溶性蛋白表达量差异较为明显,如M1和M5的表达量分别是野生型的2.7倍和2.1倍 (表 2)。M1和M5酶活提高倍数与表达量提高倍数相当。以上结果表明酶活升高主要是可溶性表达量升高引起的。

|
| 图3 野生型普鲁兰酶及其截短突变体纯化后SDS-PAGE Figure3 SDS-PAGE analysis of the purified WT and variants. M: protein molecular mass markers; 1: M1; 2: M3; 3: M5; 4: WT. The values are molecular sizes in kilodaltons. |
| |
| Variants | Soluble enzyme (μg/mL) | Specific activity (U/mg) |
| M1 (ΔCBM41) | 72.5 ± 2.2 | 1167.3 ± 35.0 |
| M3 (ΔX25) | 31.7 ± 0.7 | 1124.4 ± 24.7 |
| M5 (ΔCBM41ΔX25) | 55.9 ± 1.8 | 1271.1 ± 41.9 |
| WT | 26.6 ± 0.5 | 1096.5 ± 21.9 |
截短突变对最适pH的影响较小,野生型与各突变体最适pH曲线变化趋势几乎相同,M1、M3、M5和野生型的最适pH为5.0。然而,不同突变体的酸碱稳定性有明显差异,尤其是当pH值低于最适pH值时。如在pH 3.0时,M5 (ΔCBM41ΔX25) 的残余酶活不足70%,而其余突变体的残余酶活均高于80%。当pH值偏酸性时,M1变体的稳定性优于野生型 (图 4)。上述结果表明,X25结构域可能对野生型普鲁兰酶在维持酸碱稳定性方面发挥正向促进作用,而CBM41结构域可能不利于维持酸碱稳定性。
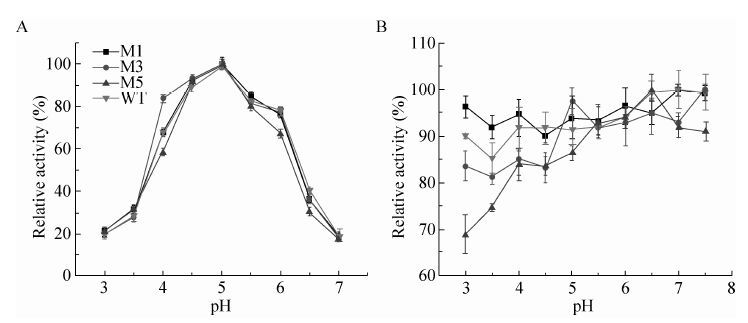
|
| 图4 野生型及其截短突变体的最适pH 及酸碱稳定性 Figure4 The optimal pH and pH stability of the WT and the variants. (A) Optimal pH. (B) pH stability. |
| |
如图 5所示,M1、M3和M5的最适温度 (60 ℃) 与野生型相同。在70 ℃时,M1、M3、M5和WT的相对酶活分别为82%、80%、87%和63%。该结果表明,突变体比野生型具有较好的耐热性。热稳定性测试结果表明切除CBM41变体的半衰期都有所延长,如M1和M5的半衰期分别是野生型的1.3倍和1.2倍。切除X25对热稳定性的影响不大 (表 3)。
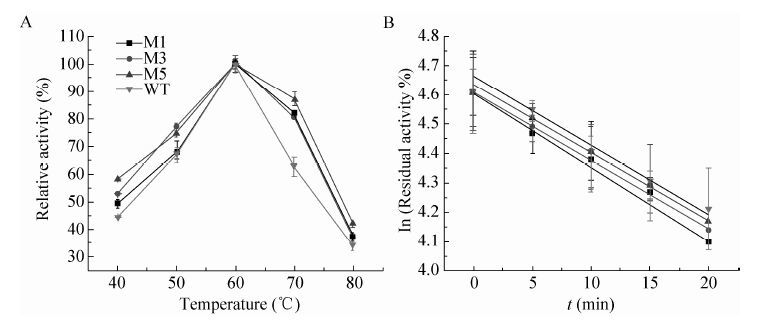
|
| 图5 野生型及其截短突变体的最适温度及热稳定性 Figure5 The optimal temperature and thermostability of the WT and the variants. (A) Optimal temperature. (B) Thermostability. |
| |
| Variants | Half-life (min) |
| M1 (ΔCBM41) | 43.7 |
| M3 (ΔX25) | 31.1 |
| M5 (ΔCBM41ΔX25) | 40.3 |
| Wide-type | 34.4 |
根据已解析的BaPul 3D结构,普鲁兰酶的N端结构域与催化功能域连接较为松散,尤其是CBM41结构域甚至不能以晶体形式呈现[13]。这些柔性结构域可能会对普鲁兰酶整体的热稳定性造成一定的负面影响。前期工作中,我们通过定点突变CBM41结构域中柔性氨基酸,提高了野生型普鲁兰酶的热稳定性[22]。结合本次实验结果我们提出:通过删除或替换蛋白分子结构中的柔性区域,可以有效提高蛋白的热稳定性。
2.6 酶促动力学参数M1和M5变体的Km值分别比野生型提高了2.4倍和3.1倍,M3变体的Km值变化不明显。该结果表明截短的CBM41有助于大分子普鲁兰与酶结合 (Km值越小表示酶对底物的亲和力越大),X25也可能发挥相似的作用。kcat/Km测定结果显示M5催化效率下降明显,M1和M3与野生型相当。结构变化引起功能变化的机制目前尚不清楚,有待于进一步分析。
| Kinetic parameter | M1 | M3 | M5 | WT |
| Km (mg/mL) | 1.42±0.22 | 0.69±0.08 | 1.85±0.10 | 0.60±0.16 |
| kcat/Km (mL/(mg·s)) | 1313.0±55.1 | 1372.5±83.6 | 946.8±35.0 | 1473.29±44.2 |
所有普鲁兰酶的最适底物是普鲁兰多糖;所有普鲁兰酶对高度分支的糖原没有水解活性。所有突变体对小分子糊精的相对活性比野生型略有下降,对分子量较大的可溶性淀粉下降较为明显 (表 5)。上述结果表明,CBM41和未知功能的X25有助于酶与底物结合,尤其是对大分子底物。N端结构域的切除并不影响普鲁兰酶对小分子糊精的脱支功能,但是是否影响在淀粉糖化过程中的实际应用仍需实验检验。
| Substrate | Relative activity (%) | |||
| M1 | M3 | M5 | WT | |
| Pullulan | 100±1.8 | 100±1.0 | 100±0.8 | 100±0.4 |
| Dextrin | 44.7±0.6 | 41.2±1.0 | 38.6±1.2 | 49.4±1.2 |
| Soluble starch | 31.3±0.9 | 36.7±0.3 | 27.4±1.2 | 46.9±0.8 |
| Glycogen | - | - | - | - |
| -: below the detection line. | ||||
糖化终点时,测定各组料液中DE值 (还原性糖以葡萄糖计)。对照组中未添加任何普鲁兰酶变体,糖化终点时DE值为92.1%。当野生型及突变体M1,M3和M5与糖化酶复配使用时,糖化终点时DE值分别为93.6%、94.7%、94.5%和93.1%。添加普鲁兰酶可提高糖化得率,且突变体M1和M3对DE值的贡献高于野生型普鲁兰酶。
3 结论为解析野生型Bacillus acidopullulyticus普鲁兰酶结构域与功能的关系,本文通过蛋白质工程对其N端进行了不同形式的截短。研究过程中主要得出以下结论:缺失CBM41可有效提高突变体的酶活;缺失X25对酶活影响不大;缺失X45的突变体丧失了催化活力,目标蛋白主要以不溶的包涵体形式存在。比酶活测定结果表明截短突变体与野生型酶差异较小,酶活升高主要是可溶性表达量升高引起的。突变体M1、M3和M5的最适pH (5.0) 和最适温度 (60 ℃) 与野生型相同;突变体M1、M3和M5对小分子葡聚多糖底物的水解效率影响较小,上述结果表明切除CBM41和X25结构域的普鲁兰酶变体不影响其在淀粉糖化过程中的应用。
| [1] | Singh RS, Saini GK, Kennedy JF. Continuous hydrolysis of pullulan using covalently immobilized pullulanase in a packed bed reactor. Carbohyd Polym, 2011, 83 (2) : 672–675 (in Chinese). |
| [2] | Nie Y, Yan W, Xu Y, et al. High-level expression of Bacillus naganoensis pullulanase from recombinant Escherichia coli with auto-induction: effect of lac operator. PloS ONE, 2013, 8 (10) : e78416 (in Chinese). |
| [3] | Talekar S, Pandharbale A, Ladole M, et al. Carrier free co-immobilization of alpha amylase,glucoamylase and pullulanase as combined cross-linked enzyme aggregates (combi-CLEAs): a tri-enzyme biocatalyst with one pot starch hydrolytic activity. Bioresour Technol, 2013 : 269–275 (in Chinese). |
| [4] | Roy I, Gupta MN. Hydrolysis of starch by a mixture of glucoamylase and pullulanase entrapped individually in calcium alginate beads. Enzyme Microb Technol, 2004, 34 (1) : 26–32 (in Chinese). |
| [5] | Hii SL, Tan JS, Ling TC, et al. Pullulanase: role in starch hydrolysis and potential industrial applications. Enzyme Res, 2012 : 921362 (in Chinese). |
| [6] | Kitahata S, Tanimoto T, Okada Y, et al. Synthesis of novel heterobranched β-cyclodextrins from α-D-mannosyl-maltotriose and β-cyclodextrin by the reverse action of pullulanase,and isolation and characterization of the products. Biosci Biotechnol Biochem, 2000, 64 (11) : 2406–2411 (in Chinese). |
| [7] | Yim DK, Park YH, Park YH. Production of branched cyclodextrins by reverse reaction of microbial debranching enzymes. Starch-Stärke, 1997, 49 (2) : 75–78 (in Chinese). |
| [8] | Chen WB, Nie Y, Xu Y. Signal peptide-independent secretory expression and characterization of pullulanase from a newly isolated Klebsiella variicola SHN-1 in Escherichia coli. Appl Biochem Biotechnol, 2013, 169 (1) : 41–54 (in Chinese). |
| [9] | Bertoldo C, Armbrecht M, Becker F, et al. Cloning, sequencing and characterization of a heat- and alkali-stable type I pullulanase from Anaerobranca gottschalkii. Appl Environ Microbiol, 2004, 70 (6) : 3407–3416 (in Chinese). |
| [10] | Kang JH, Park KM, Choi KH, et al. Molecular cloning and biochemical characterization of a heat-stable type I pullulanase from Thermotoga neapolitana. Enzyme Microb Technol, 2011, 48 (3) : 260–266 (in Chinese). |
| [11] | Duffner F, Bertoldo C, Andersen JT, et al. A new thermoactive pullulanase from Desulfurococcus mucosus: cloning,sequencing,purification,and characterization of the recombinant enzyme after expression in Bacillus subtilis. J Bacteriol, 2000, 182 (22) : 6331–6338 (in Chinese). |
| [12] | Chen AN, Sun Y, Zhang W, et al. Downsizing a pullulanase to a small molecule with improved soluble expression and secretion efficiency in Escherichia coli. Microb Cell Fact, 2016, 15 (1) : 9.DOI:10.1186/s12934-015-0403-5 (in Chinese). |
| [13] | Turkenburg JP, Brzozowski AM, Svendsen A, et al. Structure of a pullulanase from Bacillus acidopullulyticus. Proteins: Struct Funct Bioinform, 2009, 76 (2) : 516–519 (in Chinese). |
| [14] | Cantarel BL, Coutinho PM, Rancurel C, et al. The carbohydrate-Active EnZymes database (CAZy): an expert resource for Glycogenomics. Nucl Acids Res, 2009, 37 (Suppl. 1) : D233–D238 (in Chinese). |
| [15] | Henrissat B, Davies G. Structural and sequence-based classification of glycoside hydrolases. Curr Opin Struct Biol, 1997, 7 (5) : 637–644 (in Chinese). |
| [16] | Stam MR, Danchin EGJ, Rancurel C, et al. Dividing the large glycoside hydrolase family 13 into subfamilies: towards improved functional annotations of α-amylase-related proteins. Protein Eng Des Sel, 2006, 19 (12) : 555–562 (in Chinese). |
| [17] | Duan XG, Wu J. Enhancing the secretion efficiency and thermostability of a Bacillus deramificans pullulanase mutant (D437H/D503Y) by N-terminal domain truncation. Appl Environ Microbiol, 2015, 81 (6) : 1926–1931 (in Chinese). |
| [18] | England G,Kolkman M,Miller B,et al. Pullulanase variants with increased productivity: US,7968691. 2011-06-28. |
| [19] | Teague MW,Brumm PJ,Allen LN,et al. Pullulanase expression constructs containing α-amylase promoter and leader sequences: US, 6300115. 2001-10-09. |
| [20] | Chen AN, Li YM, Liu XX, et al. Soluble expression of pullulanase from Bacillus acidopullulyticus in Escherichia coli by tightly controlling basal expression. J Ind Microbiol Biotechnol, 2014, 41 (12) : 1803–1810 (in Chinese). |
| [21] | Lappalainen A, Niku-Paavola ML, Suortti T, et al. Purification and characterization of Bacillus acidopullulyticus pullulanase for enzymatic starch modification. Starch-Stärke, 1991, 43 (12) : 477–482 (in Chinese). |
| [22] | Chen AN, Li YM, Nie JQ, et al. Protein engineering of Bacillus acidopullulyticus pullulanase for enhanced thermostability using in silico data driven rational design methods. Enzyme Microb Technol, 2015 : 74–83 (in Chinese). |
| [23] | Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem, 1976 : 248–254 (in Chinese). |
| [24] | Kim HS, Le QAT, Kim YH. Development of thermostable lipase B from Candida antarctica (CalB) through in silico design employing B-factor and RosettaDesign. Enzyme Microb Technol, 2010, 47 (1/2) : 1–5 (in Chinese). |
| [25] | Malle D, Itoh T, Hashimoto W, et al. Overexpression,purification and preliminary X-ray analysis of pullulanase from Bacillus subtilis strain 168. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2006, 62 (Pt 4) : 381–384 (in Chinese). |
| [26] | Svendsen A. Pullulanase variants and methods for preparing such variants with predetermined properties: US,7906306. 2011-03-15. |
 2016, Vol. 32
2016, Vol. 32




