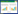服务
文章信息
- 马文瑞, 柳春梅, 杨明峰, 薛飞燕, 陈青, 马兰青, 吕鹤书
- Wenrui Ma, Chunmei Liu, Mingfeng Yang, Feiyan Xue, Qing Chen, Lanqing Ma, Heshu Lü
- 虎杖苯亚甲基丙酮合酶PcPKS2 的表达纯化和晶体生长
- Preparation and crystallization of Polygonum cuspidatum benzalacetone synthase
- 生物工程学报, 2016, 32(2): 250-258
- Chin J Biotech, 2016, 32(2): 250-258
- 10.13345/j.cjb.150234
-
文章历史
- Received: May 20, 2015
- Accepted: October 27, 2015
2. 北京农学院 农业部都市农业 (北方) 重点实验室,北京 102206
2. Key Laboratory of Urban Agriculture (North) of Ministry of Agriculture China, Beijing University of Agriculture, Beijing 102206,China
植物类型Ⅲ聚酮合酶超家族 (PKSs),又称查尔酮合酶 (Chalcone synthase,CHS) 超家族,催化合成多种植物次生代谢产物的分子骨架,主要包括酚类、芪类以及类黄酮化合物等[1, 2]。其中,众多化合物具有其抗肿瘤、心血管保护、抗氧化功能。目前,来源于聚酮化合物的药物每年销售额已超过100亿美元。由于生物合成途径及机制复杂多变,从而使该类化合物具有显著多样的生物学活性。
目前,已经从苔藓、蕨类、裸子和被子植物中分离了14种植物类型Ⅲ PKS基因[3]。CHS是家族中第一个被发现研究较深入的成员,催化来自丙二酰辅酶A (Malonyl-CoA) (延伸底物) 的3个乙酰集团通过连续的缩合反应连接到 4-香豆酰辅酶A (p-Coumaroyl-CoA) (起始底物) 分子上,之后通过克莱森 (Claisen) 型环化反应生成芳香族聚酮化合物柚皮素查尔酮 (Naringenin chalcone),其为类黄酮化合物生物合成的前体[4, 5, 6, 7, 8, 9](图1)。家族中的另一个成员,苯亚甲基丙酮合酶 (Benzalacetone synthase,BAS) 催化4-香豆酰辅酶A与丙二酰辅酶A通过一步脱羧缩合反应生成苯亚甲基丙酮[10, 11, 12, 13],苯亚甲基丙酮是一系列具有重要生物学活性苯丁烷类化合物及其衍生物的前体化合物[14, 15]。目前这些苯丁烷类化合物主要包括大黄中的林氏莲花掌素甙 (Glucoside lindleyin)、生姜中的姜酚 (Gingerol) 和姜黄 (Curcumin),以及覆盆子果实中一种独特的芳香物质覆盆子酮 (Raspberry ketone)。覆盆子酮及其衍生物具有多种生物学活性,如对流感、心肌收缩力衰弱、糖尿病、减肥具有明显功效,可作为植物源农药等[16, 17]。

|
| 图1 CHS和BAS催化反应过程 Fig.1 Enzyme reactions of CHS and BAS. |
蓼科植物虎杖Polygonum cuspidatum (Japanese knotweed,Polygonaceae) 是一种富含聚酮化合物的药用植物,然而虎杖中聚酮化合物的生物合成机制却鲜有了解。最近,从虎杖中分离出第一个苯亚甲基丙酮合酶BAS (PcPKS2) [18]和1个具有CHS和BAS活性的双功能酶 (PcPKS1) [19]。PcPKS1和PcPKS2主要表达部位位于虎杖根茎,该器官是虎杖主要功能次生代谢产物合成的主要部位,提示植物中苯亚甲基丙酮的生物合成可能由BAS和CHS/BAS双功能酶共同承担。
Austin and Noel认为紫花苜蓿Medicago sativa CHS2“门卫”Phe215和Phe265可能调节类型Ⅲ PKS活性位点与辅酶A (CoA) 结合通道之间的空间结构[20] (图2)。掌叶大黄Rheum Palmatum BAS一个显著特点是Phe215 (Ms CHS2顺序) 被Leu取代,这个位置上的取代对于该酶的BAS活性是必需的[11]。RpBAS的Phe265 (Ms CHS2顺序) 与CHS2相比其侧链构象更加靠近Leu215并形成疏水相互作用。两个门卫氨基酸Leu215和Phe 265的构象变化导致了BAS活性腔的入口扩大为CHS2的两倍,影响了其底物专一性[22]。在PcPKS2序列中Phe215和Phe265双双缺失,分别被Leu和Cys取代[18]。PcPKS1是一个同时拥有CHS和BAS活性的双功能酶,且PcPKS1的BAS活性催化效率 (Kcat/Km) 比PcPKS2高70倍[19]。有趣的是,在PcPKS1序列中,Phe215和Phe265保守存在,预示PcPKS1须采用区别于PcPKS2特异的重要氨基酸序列决定BAS活性。

|
| 图2 不同种属CHS、BAS重要氨基酸序列比较 Fig.2 Sequence alignment of crutial amino acids of CHSs and BASs. The sequences of AmCHS2,RpBAS,PcPKS1,PcPKS2 and RiPKS4 are numbered from Ms CHS2. |
蛋白晶体学研究以及基于结构的突变研究表明,PKSs家族不同成员的功能多样性来自于酶催化位点的非常微小的构象变化[17]。在前期研究中已经获得了PcPKS1的晶体[23]。但是,为了能够比较PcPKS2和PcPKS1双功能酶活性差异可能产生的机制,以确定其高效BAS活性的分子机理,需要对PcPKS2以及双功能酶PcPKS1进行三维结构比较。在本研究中,利用原核表达并纯化了PcPKS2,并在此基础上进行了结晶条件的摸索,获得了蛋白晶体,为其结构解析、催化机理研究和该类酶在基因工程中的应用提供了基础。
1 . 材料与方法 1.1 材料pET30a质粒,克隆菌株DH5α,表达菌株BL21 (DE3) pLysS为本实验室保存。质粒提取试剂盒,胶回收试剂盒购自北京全式金生物技术有限公司。PFU DNA聚合酶,T4 DNA连接酶购自TaKaRa公司。限制性内切酶购自NEB Biorad公司。分子量Marker、亲和Ni树脂购自鼎国昌盛公司。分子筛层析柱Superdex 200 10/300 GL和纯化系统Purifier 10购自GE公司。晶体筛选试剂盒购自Hampton Research公司。引物合成和测序由上海生工生物工程有限公司完成,上游NdeⅠ酶切位点引物为:TATACATATGGCGGCTTCAATTGAGGAG,下游SalⅠ酶切位点引物为:TATAGTCGACATGAATGATGG GCACGCTG。
1.2 PcPKS2在大肠杆菌中的表达目标基因经PCR扩增及胶回收后,用NdeⅠ和SalⅠ限制性双酶切,插入经同样酶切后的原核表达载体 pET30(a) (Novagen) 中,从而使PcPKS2以C-端融合有His6标签的重组形式表达。经菌液PCR和酶切鉴定阳性克隆,经过测序公司测序确定插入序列的正确性,pET30-PCPKS2质粒被转化至E. coli BL21 (DE3) pLysS,小试检测可溶性表达。
1.3 PcPKS2大量表达纯化从重组质粒转化BL21 (DE3) pLysS平板上挑取单菌落接入50 mL含卡那霉素和氯霉素的LB培养基中,37 ℃、200 r/min振荡培养过夜;按照1∶100比例将菌液接入1 L LB培养基 (补充卡那霉素至终浓度50 mg/mL,氯霉素终浓度34 mg/mL),200 r/min、37 ℃培养。600 nm光吸收 (A600) 至0.6-0.8,加入诱导剂IPTG至终浓度为0.5 mmol/L。在25 ℃和180 r/min条件下诱导6 h,6 000×g离心20 min收集菌体。
1 L重组大肠杆菌培养物用于纯化,使用 40 mL裂解缓冲液 (20 mmol/L Tris-HCl pH 7.5,300 mmol/L NaCl,20 mmol/L咪唑,1 mmol/L 苯甲基磺酰氟,8 μg/mL亮抑酶肽,4 μg/mL抑酶肽)。细胞破碎采用超声方法,使用400 W功率超声15s 3次 (JY92-IIDN,宁波新芝)。裂解物经高速离心 (10 000×g,12 min),将离心得到的上清与5 mL Ni-Agarose 结合2 h,进一步装柱,与树脂结合的PcPKS2在20 mmol/L Tris-HCl (pH 7.5)、300 mmol/L NaCl、150 mmol/L 咪唑条件下被洗脱。浓缩洗脱下来的目的蛋白至体积约为2.0 mL,加样至Superdex 200 10/300 GL进行凝胶层析。使用缓冲液为20 mmol/L Tris-HCl (pH 7.5)、50 mmol/L NaCl、1 mmol/L EDTA、1 mmol/L DTT。收集主要目标蛋白峰,SDS-PAGE检测纯度。
1.4 蛋白晶体生长条件筛选和优化使用10 kDa截留量的超滤浓缩管,将PcPKS2浓缩至20 mg/mL。使用溶液为 20 mmol/L Tris-HCl (pH 7.5)、150 mmol/L NaCl、1 mmol/L EDTA、1 mmol/L DTT。结晶采用悬滴式气相扩散法。蛋白溶液与沉淀剂溶液1∶1等量混合,置于Linbro 24 孔结晶板,池液500 μL。初次筛选为Hampton Crystal Screen & II的96个结晶条件,经过22 ℃培养24 h后显微镜下观察。单独的PcPKS2或其与催化产物 (苯亚甲基丙酮,终浓度为1 mmol/L) 复合物进行蛋白晶体生长条件的尝试。
1.5 X-射线衍射鉴定蛋白晶体晶体长出后约72 h被快速冷冻,防冻液为30% PEG 8000,5%甘油,利用取样环将晶体快速转至数据收集系统的液氮气流中上样。X-射线衍射利用了FR-E X-射线光源 (日本理学公司,Riguka),收集面探系统为RAXIS4++ (Area Detector Systems Corporation)。收集距离为 160 mm,波长为1.5418 Å。数据初步分析采用了MOSFLM软件 (Collaborative Computational Project,Number 4,1994)[24, 25]。
2 . 结果 2.1 pET30a-PCPKS2重组子的鉴定利用已有PCPKS2基因为模板,PCR扩增得到片段,纯化回收后将其连接在pET30a载体上。菌液PCR鉴定的阳性菌落,提取质粒后用NdeⅠ和SalⅠ进行酶切鉴定。切出片段符合预期1 167 bp (图3),经上海生工生物工程有限公司测序确认插入正确无突变。
|
| 图3 PCPKS2基因PCR扩增及重组质粒双酶切鉴定 Fig.3 PCR amplification of PCPKS2 gene and double enzymatic digestion of the recombinant plasmid. (A) PCR amplification of PCPKS2. M: DNA ladder; 1: PCR product of PCPKS2. (B) Double enzymatic digestion of the recombinant plasmid. M: DNA ladder; 2: digested with NdeⅠand SalⅠ. |
利用大肠杆菌BL21 (DE3) pLysS菌株进行了小量表达以优化表达量和可溶性成分的比例。尝试了利用不同的温度18 ℃、25 ℃和37 ℃下,不同的诱导剂浓度 (0.2 mmol/L、0.5 mmol/L和1.0 mmol/L IPTG) 进行诱导,在不同的诱导时间留样 (2、4、6和16 h) ,进行SDS-PAGE电泳分析确定最佳诱导条件。最终确定了最佳表达条件为25 ℃,0.5 mmol/L IPTG诱导6 h,在这样的条件下,PcPKS2表达量高并大部分呈可溶性。
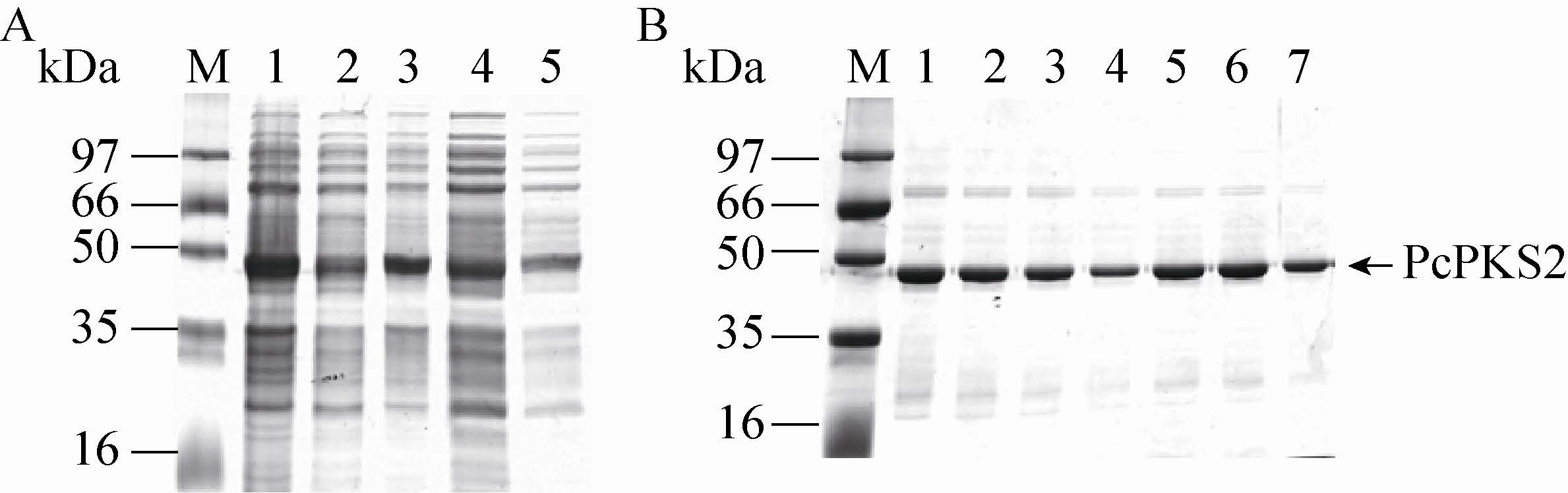
大肠杆菌经裂解后通过5 mL Ni2+金属螯合柱进行纯化,C-端连有His6的PcPKS2与树脂结合,并在150 mmol/L咪唑浓度条件下被洗脱。蛋白样品经SDS-PAGE 分析呈现主要条带,分子量约为43 kDa,与PcPKS2分子量相符,以及几条弱的杂蛋白条带 (图4)。
|
| 图4 SDS-PAGE检测PcPKS2亲和层析纯化结果 (考马斯亮蓝染色) Fig.4 SDS-polyacrylamide gel electrophoresis of recombinant PcPKS2 over the affinity chromatography,stained with Coomassie Blue. (A) PcPKS2 solubility analysis after the cell lysis. M: molecular mass standards; 1: whole cell of E. coli BL21(DE3)pLysS; 2: soluble fraction of the lysate; 3: insoluble fraction of the lysate; 4-5: unbound fraction to the column. (B) Purification over the Ni column. M: molecular mass standards; 1-7: eluted fractions. |
样品进一步进行了分子筛层析纯化,出现单一洗脱峰,其洗脱收集液经进行电泳分析。与亲和层析结果比较表明Superdex 200凝胶层析对几种高分子量杂蛋白的除去效果不明显,但低分子量杂蛋白进一步被去除,使蛋白达到较高的纯度 (图5)。纯化后蛋白被浓缩至20 mg/mL用于蛋白结晶。

|
| 图5 PcPKS2分子筛层析纯化结果 Fig.5 Gel filtration chromatography analysis of PcPKS2. (A) SDS-PAGE of Superdex 200 column elutions of PcPKS2. (B) Gel filtration profile of PcPKS2 through Superdex 200. |
对PcPKS2进行蛋白晶体生长条件的初筛。在初筛约24 h后,在几个结晶条件下可以看到出现晶状沉淀或微晶,其中在Crystal Screen试剂盒的条件15有一些成蔟生长的针状或片状结晶产物 (30% PEG 8 000,100 mmol/L 三水二甲砷酸钠,Hepes pH 6.5,0.2 mol/L (NH4)2SO4) (图6)。初筛时的蛋白晶体个体较小,形状不规则,很多小晶体混杂生长在一起。利用实验室自配的聚乙二醇8 000作为沉淀剂溶液进行实验,同样获得了重复出现的微晶。进一步对该条件进行添加剂条件优化,利用添加剂试剂盒 (Additive Screen conditions,Hampton Research) 进行搜索。其中山梨糖醇明显改善了晶体生长。最终用于结晶的沉淀剂溶液为100 mmol/L Tris (pH 8.0)、17% PEG 8 000、10 mmol/L DTT、3% (W/V) 山梨糖醇。最后经培养约3 d的片状单晶体,利用X-射线衍射鉴定证实为蛋白的单晶。

|
| 图6 PcPKS2晶体图片 Fig.6 Photographs of PcPKS2 crystals. (A) Microcrystals grown in 30% PEG 8 000,100 mmol/L sodium cacodylate trihydrate Na Hepes pH 6.5,0.2 mol/L (NH4)2SO4. (B) Plate-shaped single PcPKS2 crystals grown with the additive of 3% (W/V) sorbitol. |
晶体经衍射鉴定为蛋白晶体,分辨率至最少为3.5 Å (图7)。初步处理显示空间群为P222,晶胞参数为a=82.23,b=83.11,c=119.7 Å。实验中得到的片状晶体因为较薄,还无法用于衍射数据的收集。

|
| 图7 PcPKS2蛋白晶体经X-射线衍射的图片 Fig.7 Typical X-ray diffraction pattern of PcPKS2 crystal. |
虎杖因其富含聚酮化合物是一种被广为应用的药用植物,然而其体内聚酮化合物的生物合成机制却鲜有报道。PcPKS2是第一个被报道的虎杖中发现的BAS。我们针对PcPKS2所具有BAS催化活性的机理展开研究。在原核系统中表达的PcPKS2具有良好的可溶性,使我们能够进行蛋白结晶的研究。
前期工作中,我们在原核生物中超量表达了PcPKS2的全长蛋白。PcPKS2首先被插入到pET30(a) 表达载体BamHⅠ和SalⅠ位点之间,以具有N-端His6标签序列的形式融合表达,然而在后续的结晶实验中,蛋白晶体出现多晶、镶嵌生长的现象,质量差难以优化。在经过序列分析后,后期工作重新构建表达载体,PCPKS2被插入到NdeⅠ和SalⅠ位点之间,从而去除了融合蛋白中N-末端的约50氨基酸表达载体序列,PcPKS2表达为C-端具有His6 标签的融合蛋白。这种改变在后续的结晶实验中被证实有效,因为减少融合蛋白的柔韧性从而增加了PcPKS2蛋白的结构上的均一性,对于蛋白晶体改善是关键的一步。通过尝试不同的沉淀剂和添加剂,最终得到了蛋白的单晶体。
实验中得到的片状单晶体因为较薄,还无法用于衍射数据的收集,因此,今后本研究的工作重点将集中在晶体生长条件的优化上。通过进一步降低晶体生长速度,使晶体内部原子排列更为有序,以及尝试不同的添加剂的组合等优化手段,提高晶体的质量。本研究为其结构功能关系研究以及基于结构的药物设计提供了基础,从而为该类酶在基因工程中的应用奠定基础。
| [1] | Schröder J. The chalcone/stilbene synthase-type family of condensing enzymes//Sankawa U, Ed. Comprehensive Natural Products Chemistry. Vol. 1. Amsterdam: Elsevier, 1999: 749-771. |
| [2] | Shen B. Polyketide biosynthesis beyond the type I, II and III polyketide synthase paradigms. Polyketide synthase paradigms. Curr Opin Chem Biol, 2003, 7(2): 285-295. |
| [3] |
Ma LQ, Shi GL, Ye HC, et al. Plant-specific type III polyketide synthase superfamily: gene structure, function and metabolistes. Chin J Biotech, 2010, 26(11): 1482-1492 (in Chinese). 马兰青, 师光禄, 叶和春, 等. 植物类型III 聚酮 合酶超家族基因结构、功能及代谢产物. 生物工 程学报, 2010, 26(11): 1482-1492. |
| [4] | Reimold U, Kröger M, Kreuzaler F, et al. Coding and 3’ non-coding nucleotide sequence of chalcone synthase mRNA and assignment of amino acid sequence of the enzyme. EMBO J, 1983, 2(10): 1801-1805. |
| [5] | Ferrer JL, Jez JM, Bowman ME, et al. Structure of chalcone synthase and the molecular basis of plant polyketide biosynthesis. Nat Struct Biol, 1999, 6(8): 775-784. |
| [6] | Jez JM, Bowman ME, Noel JP. Structure-guided programming of polyketide chain-length determination in chalcone synthase. Biochemistry, 2001, 40(49): 14829-14838. |
| [7] | Jez JM, Ferrer JL, Bowman ME, et al. Dissection of malonyl-coenzyme a decarboxylation from polyketide formation in the reaction mechanism of a plant polyketide synthase. Biochemistry, 2000, 39(5): 890-902. |
| [8] | Jez JM, Bowman ME, Noel JP. Expanding the biosynthetic repertoire of plant type III polyketide synthases by altering starter molecule specificity. Proc Natl Acad Sci USA, 2002, 99(8): 5319-5324. |
| [9] | Jez JM, Austin MB, Ferrer JL, et al. Structural control of polyketide formation in plant-specific polyketide synthases. Chem Biol, 2000, 7(12): 919-930. |
| [10] | Abe I, Takahashi Y, Morita H, et al. Benzalacetone synthase. A novel polyketide synthase that plays a crucial role in the biosynthesis of phenylbutanones in Rheum palmatum. Eur J Biochem, 2001, 268(11): 3354-3359. |
| [11] | Abe I, Sano Y, Takahashi Y, et al. Site-directed mutagenesis of benzalacetone synthase. The role of the Phe215 in plant type III polyketide synthases. J Biol Chem, 2003, 278(27): 25218-25226. |
| [12] | Abe I, Abe T, Wanibuchi K, et al. Enzymatic formation of quinolone alkaloids by a plant type III polyketide synthase. Org Lett, 2006, 8(26): 6063-6065. |
| [13] | Abe T, Morita H, Noma H, et al. Structure function analysis of benzalacetone synthase from Rheum palmatum. Bioorg Med Chem Lett, 2007, 17(11): 3161-3166. |
| [14] | Borejsza-Wysocki W, Hrazdina G. Biosynthesis of p-hydroxyphenylbutan-2-one in raspberry fruits and tissue cultures. Phytochemistry, 1994, 35(3): 623-628. |
| [15] | Borejsza-Wysocki W, Hrazdina G. Aromatic polyketide synthases (purification, characterization, and antibody development to benzalacetone synthase from raspberry fruits). Plant Physiol, 1996, 110(3): 791-799. |
| [16] | Abe I. Engineering of plant polyketide biosynthesis. Chem Pharm Bull (Tokyo), 2008, 56(11): 1505-1514. |
| [17] | Abe I, Morita H. Structure and function of the chalcone synthase superfamily of plant type III polyketide synthases. Nat Prod Rep, 2010, 27(6): 809-838. |
| [18] | Ma LQ, Pang XB, Shen HY, et al. A novel type III polyketide synthase encoded by a three-intron gene from Polygonum cuspidatum. Planta, 2009, 229(3): 457-469. |
| [19] | Ma LQ, Guo YW, Gao DY, et al. Identification of a Polygonum cuspidatum three-intron gene encoding a type III polyketide synthase producing both naringenin and p-hydroxybenzalacetone. Planta, 2009, 229(5): 1077-1086. |
| [20] | Austin MB, Noel JP. The chalcone synthase superfamily of type III polyketide synthases. Nat Prod Rep, 2003, 20(1): 79-110. |
| [21] |
Lü HS, Liu CM, Lu P, et al. Plant-specific type III polyketide synthases superfamily: crystal structures and functions. Chin J Biotech, 2012, 28(1): 1-14 (in Chinese). 吕鹤书, 柳春梅, 路平, 等. 植物类型III 聚酮合 酶超家族晶体结构与功能. 生物工程学报, 2012, 28(1): 1-14. |
| [22] | Morita H, Shimokawa Y, Tanio M, et al. A structure-based mechanism for benzalacetone synthase from Rheum palmatum. Proc Natl Acad Sci USA, 2010, 107(2): 669-673. |
| [23] | Lü HS, Yang MF, Liu CM, et al. Protein preparation, crystallization and preliminary X-ray analysis of Polygonum cuspidatum bifunctional chalcone synthase/benzalacetone synthase. Acta Crystallogr Sect F Struct Biol Cryst Commun, 2013, 69(Pt 8): 871-875. |
| [24] | Jancarik J, Kim SH. Sparse matrix sampling: a screening method for crystallization of proteins. J Appl Cryst, 1991, 24(4): 409-411. |
| [25] | Collaborative Computational Project, Number 4. The CCP4 suite: programs for protein crystallography. Acta Crystallogr D Biol Crystallogr, 1994, 50(Pt 5): 760-763. |
 2016, Vol. 32
2016, Vol. 32