中国科学院微生物研究所、中国微生物学会主办
文章信息
- 梁敏, 彭婷婷, 石亚伟
- Liang Min, Peng Tingting, Shi Yawei
- 敲除L-periaxin基因的大鼠RSC96细胞系的建立
- Establishment of L-periaxin gene knock-out RSC96 cell line
- 生物工程学报, 2016, 32(12): 1735-1744
- Chin J Biotech, 2016, 32(12): 1735-1744
- 10.13345/j.cjb.160170
-
文章历史
- Received: April 25, 2016
- Accepted: September 6, 2016
Periaxin是施万细胞中特异表达的支架蛋白之一[1],参与髓鞘成熟及稳定[2-3]。Periaxin蛋白在周围神经纤维发育早期即可表达,在髓鞘中的定位是逐渐变化的,在髓鞘形成过程中,其表达在近轴突膜上(靠近轴突),但髓鞘成熟后,它定位在远轴突膜上(靠近基底膜)[4]。Periaxin敲除的小鼠髓鞘纤维可正常发育[5],但是细胞质带(Cajal带)被破坏,并且施万细胞在神经发育过程中的伸长程度受损[6],导致节间距离减小,使神经传导速度降低,影响运动功能。Periaxin的突变也会导致多种神经疾病,包括先天性肌营养不良、神经纤维瘤病和麻风病[7]。至今在Periaxin基因上已发现有18种不同的位点突变可能导致外周脱髓鞘神经疾病CMT4F亚型的发生[8]。Periaxin根据mRNA剪接方式的不同可以编码L-periaxin和S-periaxin两种蛋白亚型,分别含有1 461个氨基酸和147个氨基酸[9-10]。
TALEN (Transcription activator-like effector nucleases)转录激活子样效应因子核酸酶,是继归巢核酸内切酶(Homing endonucleases)、锌指核酸酶(Zinc finger nucleases, ZFNs)技术之后[11],发展起来的又一种对靶向基因进行修饰的新技术。转录激活因子样效应子(Transcription activator-like effectors,TALEs)最初发现于植物病原黄单胞菌中,可以特异性地识别并结合一定的DNA序列[12];ForkⅠ是一种Ⅱs型限制酶,在二聚体的状态下存在切割活性[13]。TALE核酸酶(TALENs)即是由TALEs构成DNA结合结构域与内切核酸酶ForkⅠ构成的切割结构域构成。
TALE蛋白的DNA结合结构域是由数量可变且高度保守的重复单元组成,一般情况下,34个氨基酸组成一个重复单元,每个重复单元序列中第12、13位的氨基酸特异识别DNA链中的A、T、G、C碱基[14]。这样一个能够识别单一碱基的TALE蛋白可作为一个单独的基本单元模块,多个这样的模块按照一定的顺序串连组合,可识别特定的DNA序列。所以DNA序列中的每一个碱基的识别都需要一个对应的基本单元模块,即编码34个氨基酸的TALE重复序列。
2009年,各个重复单元特异识别的DNA碱基被破译。12、13位氨基酸与碱基之间的对应分子密码为:NI对应A,HD对应C,NG对应T,NN对应G/A[11]。根据TALE-DNA分子密码,将识别目的序列上各个碱基的TALE单元依次连接,构成可识别完整碱基序列的TALEs;并与ForkⅠ酶相连接即形成DNA酶TALEN[15]。两个TALEN单体的结合位点间的距离要适当,并且反向相对,使得ForkⅠ的两个单体能在一处聚集并形成具有切割活性的二聚体,从而定点剪切DNA[16]。
RSC96细胞株是原代大鼠施万细胞经长时间培养后自发转化而成的[17-18],为了进一步研究L-periaxin基因在髓鞘形成过程中的作用,我们利用TALEN技术构建了一对针对L-periaxin基因的TALENs载体,获得了敲除L-periaxin基因的RSC96细胞株,为后续研究其在髓鞘形成过程中的作用及腓骨肌萎缩症的防治提供了实验基础。
1 材料与方法 1.1 材料克隆菌株E. coli X10由本实验室保存;大鼠施万细胞RSC96购自武汉博士德生物公司,用含10%无支原体胚胎牛血清的DMEM (含葡萄糖4.5 g/L,含丙酮酸钠0.11 g/L)培养液在37 ℃、5% CO2及饱和湿度的培养箱中培养。
本实验中载体构建使用上海斯丹赛生物技术有限公司TALEN试剂盒中:A、T、G、C四种TALE基本单元模块(即多个能够识别A、T、G、C四个碱基的TALE蛋白),L15: cmv-sp6-NLS-TAL-T-IRES-puro-pA,R11: cmv-sp6-NLS-TAL-T-pA作为左右臂载体。
兔抗L-periaxin及S-periaxin多抗购自上海艾比玛特生物医药有限公司,山羊抗兔抗体购自北京中杉金桥生物技术有限公司。
1.2 TALEN质粒的构建与鉴定按照TALENs设计原则在TALEN序列设计软件中(https://tale-nt.cac.cornell.edu/)确定L-periaxin基因敲除靶位点,选用位于NLS2结构域的一段基因作为靶点,并在其上下游分别设计相应的TALEN左臂识别序列、TALEN右臂识别序列(表 1)。
| Left target (5'-3') | Cleavage site | Right target (5'-3') |
| CCCAAGTTCTCCCGAT | TGCGTCGGGGCCTTA | AAGCCGATGCTGTCAAGGGA |
| GGGTTCAAGAGGGCTA | ACGCAGCCCCGGAAT | TTCGGCTACGACAGTTCCCT |
按照TALEN试剂盒构建方法,通过反复的酶切-连接反应构建可特异识别periaxin基因左、右臂的TALEN重复序列,并将TALEN左、右臂识别序列分别与cmv-sp6-NLS-TAL-T-IRES-puro-pA、cmv-sp6-NLS-TAL-T-pA (质粒图谱见图 1,由上海斯丹赛生物技术有限公司提供)载体重组,分别命名为TALEN-L与TALEN-R。
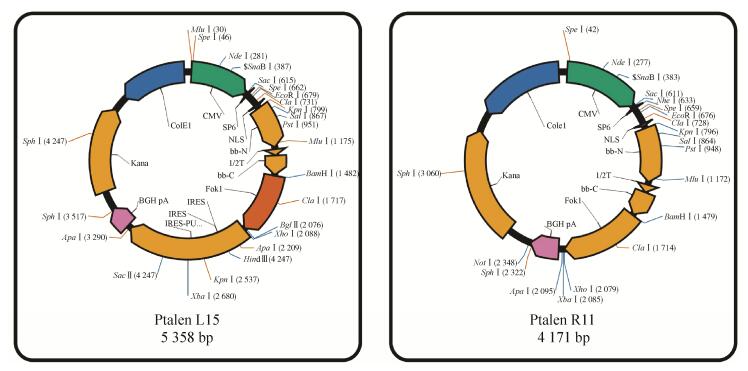
|
| 图 1 TALEN质粒图谱(左图为连接可识别TALEN左臂,即靶位点上游序列的TALEN重复序列的载体;右图为连接可识别TALEN右臂,即靶位点下游序列的TALEN重复序列的载体) Figure 1 The plasmid profile of TALEN. Left: connecting-vector of TALEN-L; Right: connecting-vector of TALEN-R. |
| |
将DMEM (含葡萄糖4.5 g/L,含丙酮酸钠0.11 g/L)培养基中加入10%胎牛血清,向培养RSC96细胞的培养瓶中加入4-5 mL培养基后,置于37 ℃、含5% CO2的饱和湿度培养箱中培养。细胞6 h后可贴壁,1-2 d后可更换细胞培养液,2-3 d后可传代。取生长状态良好的RSC96细胞,于转染前一天接种于培养皿中,当细胞密度达到70%-80%时,将TALEN-L、TALEN-R与pEGFP-N1共转染进入细胞。取2 μg TALEN-L、2 μg TALEN-R、0.5 μg pEGFP-N1与6 μL脂质体混匀后室温孵育20 min,加入细胞中。将转染后的RSC96细胞置于Delta Vision下观察,确定转染效率达到70%后,加入16 μg/mL嘌呤霉素进行药筛。药筛3 d后,得到成功转入TALEN-L和TALEN-R的细胞,用无抗性的培养基再培养数日。
1.4 TALEN表达载体的活性检测及突变率检测根据periaxin基因序列在位于编码NLS结构域的打靶位点上下游各300 bp处设计引物Sense (5'-CCCCTGTCCACTCACTGTGAGATT C-3')和Antisense (5'-GGCTCCTAGCCCAAGG GTTGGCAGG-3'),以药筛后的RSC96细胞全基因组DNA为模板,PCR扩增目的片段。将扩增产物测序。再将PCR产物连接到pMD18-T载体,进行单克隆测序,以单克隆测序结果中含有基因突变的序列数占所有测序序列数比例,确定质粒的突变率。
1.5 单克隆筛选消化经过药筛的细胞接种96孔板,挑选单个细胞继续培养,并将细胞克隆编号。待细胞长至一定数量,将不同编号的细胞分别收取少量细胞进行鉴定,其余细胞继续培养、冻存,用于后续研究。收取的少量细胞分别提取基因组DNA,以上述Sense和Antisense为引物,PCR扩增TALENs作用靶位点处上下游共约650 bp片段,经T7E1酶酶切后,通过琼脂糖凝胶检测,如果出现杂带,则证明该单克隆细胞株为突变型。之后将鉴定为突变的细胞克隆的PCR产物分别与pMD18-T克隆载体连接,每个克隆挑取至少10个单菌落测序。采用同源性分析软件(DNAMAN)进行序列比对,分析确定突变类型。
1.6 Western blotting检测细胞中Periaxin蛋白表达情况收集L-periaxin基因敲除成功的RSC96细胞及对照组RSC96细胞,提取细胞总蛋白,BCA法测蛋白浓度。将待测样品置于聚丙烯酰胺凝胶上,电泳后转移到硝酸纤维素膜(上海生工生物工程股份有限公司)上。经过封闭后,用兔抗L-periaxin及S-periaxin多抗(上海艾比玛特生物医药有限公司) 4 ℃过夜孵育,加入1:1 000稀释的辣根过氧化物酶标记的山羊抗兔IgG (北京中杉金桥生物技术有限公司),室温振荡2 h后洗涤,显色。
1.7 RSC96细胞、L-periaxin缺失的RSC96细胞和体外转染L-periaxin的RSC96细胞的生长曲线的绘制分别取对数生长期的RSC96细胞、L-periaxin缺失的RSC96细胞和体外转染L-periaxin的RSC96细胞接种于96孔板,每孔3 000个细胞,每组9个复孔,分别于接种后的第12、24、36、48、60、72 h加MTT (5 mg/mL),20 μL/孔,37 ℃、5% CO2温箱中继续孵育4 h,弃上清后加DMSO 150 μL/孔,37 ℃振荡10 min,使结晶充分溶解,于490 nm波长处在酶联仪上测定各孔吸光度值,以时间为横轴、吸光度值为纵轴,绘制3组细胞生长情况柱状图。
1.8 流式细胞术检测细胞周期分别将1×106个RSC96细胞、L-periaxin缺失的RSC96细胞和体外转染L-periaxin的RSC96细胞种植于35 mm培养皿,培养过夜之后换无血清培养液培养24 h后收集细胞。用70%冰冷的乙醇4 ℃固定2 h,3 000×g离心5 min,细胞经PBS清洗后重悬于含50 μg/mL的PI (Propidium iodide, 碘化丙啶)和100 μg/mL DNase-free RNase的PBS溶液中。样品在37 ℃避光放置15 min后用流式细胞仪测定DNA含量。
2 结果与分析 2.1 TALEN表达载体鉴定构建TALEN表达载体的设计中TALEN左臂识别序列长度为15 bp,其中每一个碱基的识别需要一个对应的基本单元模块,即编码34个氨基酸的TALEN重复序列(102 bp),所以识别15 bp需要的TALEN重复序列为1 530 bp (102×15 bp),另外L15空载体BamHⅠ和EcoRⅠ酶切位点之间有803 bp,所以一旦构建成功的TALEN-L双酶切后得到的片段大小应为2 333 bp (1 530+803 bp);同理,TALEN右臂识别序列为18 bp,双酶切后得到的片段大小为2 639 bp。将构建的重组质粒TALEN-L与TALEN-R用BamHⅠ和EcoRⅠ进行双酶切鉴定,0.7 %琼脂糖凝胶电泳显示(图 2),TALEN-L得到了约2 333 bp的目的片段和约4 570 bp的载体片段;TALEN-R得到了约2 639 bp的目的片段和约3 386 bp的载体片段,酶切结果揭示TALEN表达载体构建成功。
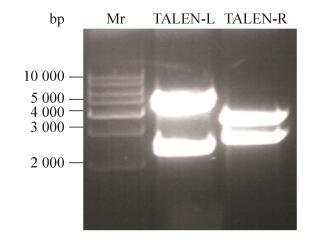
|
| 图 2 TALEN质粒经BamHⅠ和EcoRⅠ酶切后的电泳结果 Figure 2 Enzymatic digestion of TALEN-L and TALEN-R with BamHⅠand EcoRⅠ. |
| |
将TALEN-L、TALEN-R质粒与pEGFP-N1共转染RSC96细胞48 h后,利用Delta Vision拍照,观察转染效率。结果显示,其转染效率达到70% (图 3)。

|
| 图 3 TALEN质粒转染RSC96细胞48 h后转染效果 Figure 3 Effect of transfection RSC96 cell with TALEN-L and TALEN-R after 48 h. |
| |
将转染TALEN-L和TALEN-R质粒的RSC96细胞加入16 μg/mL嘌呤霉素进行药筛3 d,部分细胞死亡,仍有贴壁存活细胞,且状态良好。加入培养基继续培养,数天后,收集转染成功的RSC96细胞提取基因组DNA (图 4A)。PCR扩增TALENs作用靶位点处上下游可见650 bp片段(图 4B),PCR产物测序结果显示,打靶位置处的序列出现套峰,说明打靶成功(图 4C)。将该PCR产物连接pMD18-T载体,挑选13个菌落通过自身引物筛选,目的片段大小仍为650 bp (图 4D)。依据筛选结果,将1-8和10-11号质粒送测序。测序结果显示,2号质粒缺失1 bp;3号质粒缺失7 bp;5号质粒缺失6 bp,表明该质粒有活性,质粒的突变效率为21.6%,可以用于后续实验(表 2)。

|
| 图 4 TALEN质粒活性检测结果(A: TALEN敲除L-periaxin后的RSC96细胞基因组提取结果;B:以基因组为模板对打靶位置进行PCR扩增结果;C:打靶位置PCR产物进行测序结果;D:打靶位置PCR产物连接T载体的筛选结果) Figure 4 Detection of TALEN plasmid activity. (A) Genome extraction from L-periaxin knock-out RSC96 cell. (B) PCR results of L-periaxin gene targeting position. (C) Sequencing results of L-periaxin gene targeting position. (D) Screening results of PCR product connected with T-vector. |
| |
| Left target (5'-3') | Cleavage site | Right target (5'-3') | |
| WT | CCCAAGTTCTCCCGAT | tgcgtcggggcctta | AAGCCGATGCTGTCAAGGGA |
| 2 | CCCAAGTTCTCCCGAT | tgcgtcgggg-ctta | AAGCCGATGCTGTCAAGGGA-1 |
| 3 | CCCAAGTTCTCCCGAT | tgcgtcggg------- | -AGCCGATGCTGTCAAGGGA-7 |
| 5 | CCCAAGTTCTCCCGAT | tgcg--------cctta | AAGCCGATGCTGTCAAGGGA-6 |
| “-”: the lack of base number. | |||
为制备periaxin基因敲除细胞系,利用脂质体转染法将TALENs表达载体转染RSC96细胞,通过挑取单克隆法获得30个细胞克隆。采用T7E1酶切和测序的方法确定细胞克隆的突变类型。
用T7E1酶分别酶切每个克隆的PCR产物,未转染TALENs的细胞作为对照。结果显示,编号为G4的细胞株的PCR产物经T7E1酶切后出现两条带(图 5A),该细胞株为突变体。又依据其PCR产物测序峰图判断(图 5B),其为单拷贝敲除的RSC96细胞株系。之后以同样的方法进行二次敲除,最终得到一株periaxin敲除的RSC96细胞。从测序结果可以看出,所取得的单克隆细胞系中,periaxin基因的一个拷贝出现了23 bp的碱基缺失(图 5B箭头指向缺失碱基位置),另一个拷贝出现了1 bp的碱基缺失,从而导致该缺失位点以后阅读框移码的改变,终止密码子提前出现,后续1 376个氨基酸不能正常表达,不能产生L-periaxin基因编码的蛋白。

|
| 图 5 TALEN敲除L-periaxin的RSC96细胞突变株的鉴定(A:正常RSC96细胞株及编号为G4的TALEN敲除细胞株靶位点处PCR产物的T7E1酶切结果对比;B:正常RSC96细胞株及编号为G4的TALEN敲除细胞株靶位点处测序结果比对) Figure 5 Identification of TALEN knock-out L-periaxin RSC96 cell line. (A) Digestion of the genome of RSC96 and knock-out L-periaxin RSC96 cell (G4) with T7E1. (B) Comparison of the sequence between RSC96 and knock out L-periaxin RSC96 cell (G4) by TALENs. |
| |
L-periaxin和S-periaxin的抗体均为C端抗体,经过Western blotting检测可发现,对照组的RSC96细胞既有L-periaxin的表达,也有S-periaxin的表达;而敲除L-periaxin的RSC96细胞中,由于打靶位置之后的1 376个氨基酸均不能正常表达,L-periaxin C端的抗体检测不到L-periaxin的表达,只有S-periaxin的C端抗体检测到S-periaxin的表达(图 6)。

|
| 图 6 Western blotting检测细胞中Periaxin蛋白表达(A: TALEN打靶位点及L-periaxin和S-periaxin抗体对应的抗原表位区域;B: Western blotting检测敲除L-periaxin的RSC96细胞的Periaxin蛋白表达结果) Figure 6 Detection the expression of Periaxin in RSC96 cell by Western blotting. (A) Knock-out target position of TALEN and antigen epitope regions corresponding to antibodies in L-periaxin or S-periaxin. (B) Detection the periaixn in TALEN knock-out L-periaxin RSC96 by Western blotting. |
| |
通过MTT法检测对照组(WT)、敲除组(periaxin-/-)、体外转染L-periaxin组(periaxin+/+) 3组细胞的生长状况。结果显示(图 7),敲除L-periaxin后细胞生长速度较正常细胞及体外转染L-periaxin基因的RSC96细胞均降低,说明L-periaxin基因可能对细胞生长有一定的促进作用,敲除后影响了细胞的生长。采用SPSS软件处理数据,计量资料以(x±s)表示,组间比较采用独立样本均数t检验,率的比较采用χ2检验,P < 0.05为差异有统计学意义。
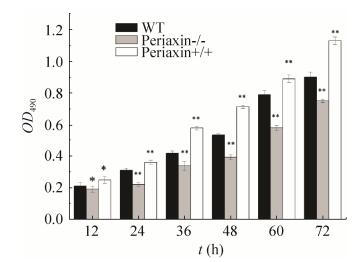
|
| 图 7 MTT法检测细胞活力 Figure 7 Cell viability was determined by the MTT assay. Error bars represent x±s of triplicate experiments.*P < 0.05; **P < 0.01. |
| |
为了进一步验证L-periaxin基因对细胞生长的影响,对照组、敲除组和过表达组3组细胞周期利用流式细胞仪进行检测,结果显示(图 8)敲除组S期比例与对照组相比减少了14.94%,由此可见敲除L-periaxin通过抑制细胞的S期(即DNA合成期),达到抑制细胞增殖,同时也证明了TALEN敲除L-periaxin后,影响了其在施万细胞中的生物学活性。

|
| 图 8 通过流式细胞仪检测periaxin对RSC96细胞增殖与周期的影响 Figure 8 Flow cytometry to evaluate the effect of periaxin on the proliferation and cell cycle progression of RSC96 cell. |
| |
Periaxin基因的突变与缺失可导致腓骨肌萎缩症4F亚型发生,该疾病是一种脱髓鞘型遗传病[19-20]。Periaxin蛋白是外周神经系统中成髓鞘细胞施万细胞中特异且大量表达的蛋白,在髓鞘成熟与维护中发挥重要作用[21]。Periaxin基因突变或缺失,会导致施万细胞不能正常包裹轴突,发生脱髓鞘现象。Gillespie等研究证明缺失了Periaxin的小鼠在6周后坐骨神经变厚,轴突被基底薄片及施万细胞包裹,形成洋葱头样结构[5]。神经纤维的延展速度与节间的生长并不能完全匹配,节间距离减少,轴突直径增加。另外,Periaxin的突变还将影响神经纤维的传导速度,紊乱髓鞘外郎飞氏结的节间长度和直径等[7]。
基因敲除是20世纪80年代发展起来的一门新技术[22]。传统的打靶技术效率低,应用大大受限[23],所以近年来出现了许多新的基因敲除技术,如基因重组、锌指核酸酶等[24]。锌指核酸酶技术能够对靶基因进行定点断裂,显著提高同源重组效率,是一种高效的新型基因打靶技术。已应用于多种动物基因的靶向敲除[22]。但锌指核酸酶技术实验设计复杂,成本高,且特异性不高[25],TALENs可以靶向更长的基因序列[26],并且相对更易构建,能识别任意目标基因序列,不受上下游序列影响,只需要构建特定的转录激活因子。TALENs序列越长,对细胞产生毒副效应的可能性就越小,因为越短的TALENs越有可能结合和改变目标基因位点[22]。TALEN技术不受物种的限制,剪切效率高及脱靶效率低[25],至今已经被成功应用于细胞、斑马鱼、果蝇、大鼠、小鼠及植物上,并且被研究者不断地尝试应用到其他更多的物种中[22]。
本文通过构建TALENs表达载体,高效获得了缺失L-periaxin基因的细胞克隆,同时初步分析了L-periaxin基因的缺失对细胞生长的影响及原因,为进一步研究periaxin在细胞生长及髓鞘形成中的作用奠定了基础,也为后续体外模拟髓鞘化过程的实验提供了必要的实验模型。
| [1] | Gillespie CS, Sherman DL, Blair GE, et al. Periaxin, a novel protein of myelinating Schwann cells with a possible role in axonal ensheathment. Neuron, 1994, 12 (3) : 497–508. DOI: 10.1016/0896-6273(94)90208-9 |
| [2] | Dytrych L, Sherman DL, Gillespie CS, et al. Two PDZ domain proteins encoded by the murine periaxin gene are the result of alternative intron retention and are differentially targeted in Schwann cells. J Biol Chem, 1998, 273 (10) : 5794–5800. DOI: 10.1074/jbc.273.10.5794 |
| [3] | Sherman DL, Fabrizi C, Gillespie CS, et al. Specific disruption of a Schwann cell dystrophin-related protein complex in a demyelinating neuropathy. Neuron, 2001, 30 (3) : 677–687. DOI: 10.1016/S0896-6273(01)00327-0 |
| [4] | Scherer SS, Xu YT, Bannerman PG, et al. Periaxin expression in myelinating Schwann cells: modulation by axon-glial interactions and polarized cocatization during development. Development, 1995, 121 (12) : 4265–4273. |
| [5] | Gillespie CS, Sherman DL, Fleetwood-Walker SM, et al. Peripheral demyelination and neuropathic pain behavior in periaxin-deficient mice. Neuron, 2000, 26 (2) : 523–531. DOI: 10.1016/S0896-6273(00)81184-8 |
| [6] | Court FA, Sherman DL, Pratt T, et al. Restricted growth of Schwann cells lacking Cajal bands slows conduction in myelinated nerves. Nature, 2004, 431 (7005) : 191–195. DOI: 10.1038/nature02841 |
| [7] | Feltri ML, Wrabetz L. Laminins and their receptors in Schwann cells and hereditary neuropathies. J Peripher Nerv Syst, 2005, 10 (2) : 128–143. DOI: 10.1111/jns.2005.10.issue-2 |
| [8] |
Yang Y, Peng TT, Shi YW. Periaxin protein and charcot-marie-tooth disease.
Chin J Biochem Mol Biol, 2015, 31 (2) : 113–120.
(in Chinese). 杨艳, 彭婷婷, 石亚伟. Periaxin蛋白与腓骨肌萎缩症. 中国生物化学与分子生物学报, 2015 , 31 (2): 113-120. |
| [9] | Delague V, Bareil C, Tuffery S, et al. Mapping of a new locus for autosomal recessive demyelinating charcot-marie-tooth disease to 19q13. 1-13.3 in a large consanguineous lebanese family: exclusion of MAG as a candidate gene. Am J Hum Genet, 2000, 67 (1) : 236–243. DOI: 10.1086/302980 |
| [10] | Williams AC, Brophy PJ. The function of the periaxin gene during nerve repair in a model of CMT4F. J Anat, 2002, 200 (4) : 323–330. DOI: 10.1046/j.1469-7580.2002.00038.x |
| [11] | Lloyd A, Plaisier CL, Carroll D, et al. Targeted mutagenesis using zinc-finger nucleases in Arabidopsis. Proc Natl Acad Sci USA, 2005, 102 (6) : 2232–2237. DOI: 10.1073/pnas.0409339102 |
| [12] | Boch J, Scholze H, Schornack S, et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science, 2009, 326 (5959) : 1509–1512. DOI: 10.1126/science.1178811 |
| [13] | Bitinaite J, Wah DA, Aggarwal AK, et al. FokI dimerization is required for DNA cleavage. Proc Natl Acad Sci USA, 1998, 95 (18) : 10570–10575. DOI: 10.1073/pnas.95.18.10570 |
| [14] | Bonas U, Stall RE, Staskawicz B. Genetic and structural characterization of the avirulence gene avrBs3 from Xanthomonas campestris pv. vesicatoria. Mol Gen Genet, 1989, 218 (1) : 127–136. DOI: 10.1007/BF00330575 |
| [15] |
Li YQ, Wang CL, Zhao KJ. Molecular recognition code between pathogenic bacterial TAL-effectors and host target genes: a review.
Chin J Biotech, 2011, 27 (8) : 1132–1141.
(in Chinese). 李岩强, 王春连, 赵开军. 病原菌TAL效应子与寄主靶基因相互识别的分子密码. 生物工程学报, 2011 , 27 (8): 1132-1141. |
| [16] | Li T, Huang S, Jiang WZ, et al. TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and FokI DNA-cleavage domain. Nucleic Acids Res, 2011, 39 (1) : 359–372. DOI: 10.1093/nar/gkq704 |
| [17] | Hai M, Muja N, DeVries GH, et al. Comparative analysis of Schwann cell lines as model systems for myelin gene transcription studies. J Neurosci Res, 2002, 69 (4) : 497–508. DOI: 10.1002/(ISSN)1097-4547 |
| [18] | Badache A, DeVries GH. Neurofibrosarcoma-derived Schwann cells overexpress platelet-derived growth factor (PDGF) receptors and are induced to proliferate by PDGF BB. J Cell Physiol, 1998, 177 (2) : 334–342. DOI: 10.1002/(ISSN)1097-4652 |
| [19] | Fledrich R, Stassart RM, Sereda MW. Murine therapeutic models for charcot-marie-tooth (CMT) disease. Br Med Bull, 2012, 102 (1) : 89–113. DOI: 10.1093/bmb/lds010 |
| [20] | Takashima H, Boerkoel CF, De Jonghe P, et al. Periaxin mutations cause a broad spectrum of demyelinating neuropathies. Ann Neurol, 2002, 51 (6) : 709–715. DOI: 10.1002/ana.10213 |
| [21] | Boerkoel CF, Takashima H, Stankiewicz P, et al. Periaxin mutations cause recessive dejerine-sottas neuropathy. Am J Hum Genet, 2001, 68 (2) : 325–333. DOI: 10.1086/318208 |
| [22] |
Liu XJ, Wang H, Yan F, et al. The development of gene knockout technologies in large and medium animal models.
Prog Physiol Sci, 2015, 46 (1) : 11–16.
(in Chinese). 刘雪静, 王欢, 严放, 等. 大中型动物基因敲除技术的研究进展. 生物科学进展, 2015 , 46 (1): 11-16. |
| [23] |
Li FD, Li Y, Liu H, et al. Production of GHR double-allelic knockout Bama pig by TALENs and handmade cloning.
Hereditas (Beijing), 2014, 36 (9) : 903–911.
(in Chinese). 李飞达, 李勇, 刘欢, 等. 利用TALENs和手工克隆技术高效获得GHR基因敲除巴马猪. 遗传, 2014 , 36 (9): 903-911. |
| [24] |
Xiao A, Hu YY, Wang WY, et al. Progress in zinc finger nuclease engineering for targeted genome modification.
Hereditas (Beijing), 2011, 33 (7) : 665–683.
(in Chinese). 肖安, 胡莹莹, 王唯晔, 等. 人工锌指核酸酶介导的基因组定点修饰技术. 遗传, 2011 , 33 (7): 665-683. DOI:10.3724/SP.J.1005.2011.00665 |
| [25] | Cermak T, Doyle EL, Christian M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res, 2011, 39 (12) : e82. DOI: 10.1093/nar/gkr218 |
| [26] | Mussolino C, Morbitzer R, Lütge F, et al. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity. Nucleic Acids Res, 2011, 39 (21) : 9283–9293. DOI: 10.1093/nar/gkr597 |
 2016, Vol. 32
2016, Vol. 32




